핵산 3차 구조
Nucleic acid tertiary structure핵산 3차 구조는 핵산 [1]폴리머의 3차원 형상이다.RNA와 DNA 분자는 분자 인식에서 촉매 작용까지 다양한 기능을 할 수 있습니다.이러한 기능들은 정확한 3차원 구조를 필요로 한다.이러한 구조는 다양하고 겉보기에는 복잡해 보이지만, 그것들은 분자 구성 요소 역할을 하는 반복적이고 쉽게 인식할 수 있는 3차 구조 모티브로 구성되어 있다.RNA 및 DNA 3차 구조에 대한 가장 일반적인 모티브 중 일부는 아래에 설명되지만, 이 정보는 제한된 수의 해결된 구조에 기초합니다.새로운 RNA와 DNA 분자가 구조적으로 특징지어짐에 따라 더 많은 3차 구조 모티브가 밝혀질 것이다.
나선 구조
이중나선
이중 나선은 생물학적 DNA의 지배적인 3차 구조이며 RNA의 가능한 구조이기도 하다.자연에서는 A-DNA, B-DNA, Z-DNA의 세 가지 DNA 배치가 발견된다고 여겨진다.제임스 D에 의해 기술된 "B" 양식. 왓슨과 프란시스 크릭은 [2]세포에서 우세하다고 여겨진다.제임스 D. 왓슨과 프란시스 크릭은 이 구조를 반지름이 10Ω이고 피치가 34Ω인 이중나선구조로,[3] 10bp마다 축을 완전히 한 바퀴 돈다고 설명했다.이중 나선은 용액에서 10.4-10.5 염기쌍마다 축을 중심으로 한 바퀴 완전히 돈다.이 비틀림 빈도(나선 피치로 알려져 있음)는 각 베이스가 체인의 이웃에 가하는 적층력에 크게 좌우됩니다.이중나선 RNA는 A형 구조와 유사한 배열을 채용하고 있다.
다른 구성도 가능합니다.실제로 현재 사용할 수 있는 것은 F, Q, U, V 및 Y 문자뿐입니다.[4][5]미래에 출현할 가능성이 있는 새로운 DNA 구조를 기술할 수 있습니다.그러나 이러한 형태의 대부분은 인공적으로 생성되었으며 자연적으로 발생하는 생물학적 시스템에서는 관찰되지 않았다.
장구 및 단구 3중주
마이너 그루브 트리플렉스는 유비쿼터스 RNA 구조 모티브입니다.작은 홈과의 상호작용은 종종 리보오스 당의 2'-OH에 의해 매개되기 때문에, 이 RNA 모티브는 DNA 당량과는 매우 다르게 보입니다.마이너 그루브 트리플의 가장 일반적인 예는 A 마이너 모티브 또는 마이너 그루브에 아데노신 염기를 삽입하는 것입니다(위 참조).그러나 다른 핵염기들도 RNA 마이너 그루브와 상호작용하는 것이 관찰되었기 때문에 이 모티프는 아데노신에만 국한되지 않는다.
마이너 홈은 삽입된 베이스를 거의 완벽하게 보완합니다.이를 통해 최적의 판데르발스 접촉, 광범위한 수소 결합 및 소수성 표면 매몰이 가능하며 에너지적으로 매우 유리한 상호작용을 [8]생성할 수 있습니다.단구 3중에는 프리루프 및 헬릭스를 안정적으로 충전할 수 있으므로 I족 인트론,[9] II족 인트론 [10]및 리보솜을 포함한 대형 리보뉴클레오티드 구조의 핵심요소이다.
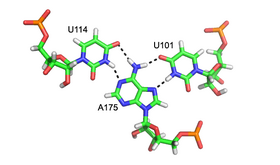
표준 A형 RNA의 주홈은 상당히 좁기 때문에 부홈보다 트리플x 상호작용이 적지만 여러 RNA 구조에서 주홈 트리플x 상호작용을 관찰할 수 있다.이러한 구조는 염기쌍과 Hoogsteen 상호작용의 여러 조합으로 구성됩니다.예를 들어 Watson-Crick형 G-C 쌍으로 이루어진 50S 리보솜에서 관찰된 GGC 트리플플렉스(GGC 아미노(N-2)-N-7, 이미노-카르보닐, 카르보닐-아미노(N-4; 왓슨-크릭))와 유사 G-Ho를 형성한다.깊은 홈 triplexes의 다른 주목할 만한 예들을 포함한다(나는)촉매 중심의 그룹 II개재에서 표시 그림에서 왼쪽[6](ii) 촉매 반응으로 필수적인 3중 나선 구조 관찰의 말단 소체 복원 효소 RNA[7](iii)은 SAM-II riboswitch[13]과(iv)는 요소를로 표현(ENE), 행동하는 RNA안정 요소를 통해톤helriple폴리(A) 꼬리를 가진 [14][15]ix 형성.
B형 DNA의 주요 홈에서 Hoogsteen 또는 반전 Hoogsteen 수소 결합으로부터도 3가닥 DNA가 가능하다.
포플렉스
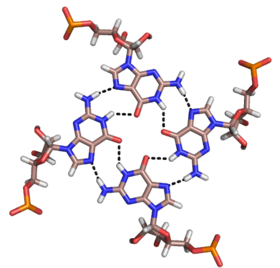
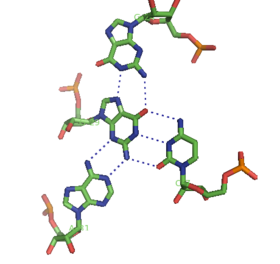
이중나선 및 상기 삼중나선 외에 RNA 및 DNA는 모두 4중나선을 형성할 수 있다.RNA 염기 4중합체에는 다양한 구조가 있다.4개의 연속된 구아닌 잔기는 Hoogsteen 수소 결합에 의해 RNA에서 4중체를 형성하여 "Hoogsteen 고리"를 형성할 수 있다(그림 [11]참조).G-C 및 A-U 쌍은 또한 Watson-Crick 쌍과 마이너 [16]그루브에서 비표준 쌍과의 조합으로 베이스 4중창을 형성할 수 있다.
말라카이트 그린 압타머의 핵심 역시 수소 결합 패턴이 다른 일종의 염기성 4중합체이다([12]그림 참조).포플렉스는 연속적으로 여러 번 반복할 수 있어 매우 안정적인 구조를 만들 수 있습니다.
RNA에서 사중복엽 영역의 독특한 구조는 생물학적 시스템에서 다른 기능을 할 수 있다.두 가지 중요한 기능은 리간드 또는 단백질과의 결합 잠재력과 DNA 또는 RNA의 전체 3차 구조를 안정화시키는 능력이다.강한 구조는 염색체의 텔로미어 및 [17]mRNA의 UTR과 같이 전사 및 복제를 억제하거나 변조할 수 있습니다.베이스 아이덴티티는 리간드 결합에 중요하다.G-쿼텟은 전형적으로 칼륨과 같은 1가 양이온과 결합하는 반면, 다른 염기는 U-U-C-U [16]4중합체에서 하이포산틴과 같은 수많은 다른 리간드와 결합할 수 있다.
이러한 기능과 함께, 리보솜 결합 영역 주변의 mRNA의 G-quadruplex는 [18]박테리아에서 유전자 발현을 조절하는 역할을 할 수 있다.아직 생체 내에서 발견되지 않은 더 흥미로운 구조와 기능이 있을 수 있다.
동축 스태킹

헬리컬 스태킹이라고도 하는 동축 스태킹은 고차 RNA 3차 구조의 주요 결정 요인입니다.동축 스태킹은 두 개의 RNA 이중체가 연속된 나선을 형성할 때 발생하며, 이는 두 개의 나선의 경계면에서 베이스 스태킹에 의해 안정화됩니다.tRNAPhe의 결정 구조에서 [20]동축 스태킹이 확인되었습니다.최근에는 자기 스플라이싱 그룹 I 및 그룹 II 인트론의 다양한 형태를 포함한 많은 리보자임들의 고차 구조에서 동축 적층 현상이 관찰되었다.일반적인 동축 스태킹 모티브에는 키싱 루프 상호작용과 의사 노트가 있습니다.이러한 상호작용의 안정성은 "터너의 규칙"[21]을 적용함으로써 예측할 수 있다.
1994년, 월터와 터너는 짧은 올리고머와 머리핀 스템의 끝에 있는 4개의 뉴클레오티드 돌출부 사이에 나선 나선 나선 계면을 만든 모델 시스템을 사용하여 나선-나선 계면 내에서 가장 가까운 이웃의 적층 상호작용의 자유 에너지 기여도를 결정했다.그들의 실험은 두 개의 나선형 2차 구조 사이의 베이스 스택의 열역학적 기여가 표준 이중 형성의 열역학을 거의 모방한다는 것을 확인했다. (가장 가까운 인접 상호작용은 결과 나선의 열역학적 안정성을 예측한다.)가장 가까운 네이버 상호작용의 상대적인 안정성을 사용하여 기존의 2차 구조를 기반으로 바람직한 동축 스태킹을 예측할 수 있습니다.Walter와 Turner는 RNA 구조의 예측이 [22]평균 67%에서 74% 정확도로 향상되었다는 것을 발견했다.
잘 연구된 대부분의 RNA 3차 구조에는 동축 적층 예가 포함되어 있습니다.대표적인 예로는 tRNA-Phe, 그룹 I 인트론, 그룹 II 인트론 및 리보솜 RNA가 있다.tRNA의 결정 구조는 아미노산 수용체 스템의 T-암과 D-암 및 안티코돈 암의 동축 적층으로부터 발생하는 두 개의 확장된 나선의 존재를 밝혀냈다.tRNA 내의 이러한 상호작용은 항코돈 줄기를 아미노산 줄기에 수직으로 향하게 하여 기능적인 L자형 3차 [20]구조로 이끈다.그룹 I의 인트론에서 P4 및 P6 나선은 생화학적[23] 방법과 결정학적 방법의 조합을 사용하여 동축적으로 쌓이는 것으로 나타났다.P456 결정 구조는 동축 적재가 RNA 나선 패킹을 3차 [24]구조로 안정화시키는 방법을 자세히 보여 주었습니다.Oceanobacillus iheyensis에서 유래한 자기 분할 그룹 II에서 IA 및 IB 줄기는 동축적으로 적층되어 오원 [6]접합의 구성 헬리스의 상대적인 배향에 기여한다.이 방향은 기능성 리보자임의 활성 부위의 적절한 접기를 용이하게 합니다.리보솜에는 70bp [25]길이의 스택세그먼트를 포함한 다수의 동축 스태킹 예가 포함되어 있습니다.
동축 스태킹과 관련된 두 가지 일반적인 모티브는 키싱 루프와 의사 노트입니다.키스 루프 상호작용에서 두 헤어핀의 단일 가닥 루프 영역은 베이스 페어링을 통해 상호작용하며, 복합적이고 동축적으로 쌓인 나선을 형성한다.특히 이 구조는 각 루프의 모든 뉴클레오티드가 염기쌍화 및 적층 상호작용에 참여할 수 있도록 한다.이 모티브는 Lee와 Crothers에 [26]의해 NMR 분석을 통해 시각화 및 연구되었습니다.pseudoknot 모티브는 동일한 RNA 가닥 내에서 업스트림 또는 다운스트림 시퀀스와 함께 헤어핀 루프 베이스쌍의 단일 고립 영역이 있을 때 발생합니다.두 개의 듀플렉스 영역은 종종 서로 겹쳐져 안정적인 동축 적층 복합 나선을 형성합니다.유사 노 모티브의 일례는 매우 안정적인 간염 델타 바이러스 리보자임으로, 골격은 전체적인 이중 유사 [27]노 토폴로지를 나타낸다.
합리적으로 설계된 DNA 구조에서 동축 스택과 유사한 효과가 관찰되었습니다.DNA 종이접기 구조에는 끝이 무딘 이중 나선형이 다수 포함되어 있습니다.이러한 구조는 소수성 적층 [28]상호작용으로 인해 노출된 뭉툭한 끝을 포함하는 가장자리를 따라 서로 달라붙는 것으로 관찰되었다.
기타 모티브
테트라루프수용체 상호작용

테트라루프-수용체 상호작용은 테트라루프 모티브의 루프 뉴클레오티드와 RNA 이중체 내에 위치한 수용체 모티브 사이의 염기쌍화 및 적층 상호작용을 결합하여 RNA 분자의 글로벌 3차 접힘을 안정시키는 3차 접점을 생성한다.테트라룹스 또한 DNA [30]이중체의 가능한 구조이다.
줄기-루프는 크기와 배열이 크게 다를 수 있지만, 4개의 뉴클레오티드의 테트라루프는 매우 흔하며 [31]보통 순서에 따라 세 가지 범주 중 하나에 속합니다.이 3개의 패밀리는 CUYG, UNCG 및 GNRA(오른쪽 그림 참조) 4중 [32]루프입니다.이들 테트라루프족 각각에서 제2 및 제3뉴클레오티드가 RNA 가닥에서 선회를 형성하고 제1 및 제4뉴클레오티드 사이의 염기쌍이 스템루프 구조를 안정화시킨다.일반적으로 테트라루프의 안정성은 루프 내의 염기 구성과 이 "닫히는 염기쌍"[33]의 구성에 따라 결정되었다.테트라루프의 GNRA 계열은 테트라루프 수용체 상호작용에서 가장 일반적으로 관찰된다.또한 UMAC 테트라루프는 유사한 백본 구조를 공유하는 GNRA 루프의 대체 버전인 것으로 알려져 있다. 유사성에도 불구하고 이들은 가능한 [34]장거리 상호작용에서 다르다.

"테트라루프 수용체 모티브"는 2차 RNA 구조의 [36]원위부 스템루프 배열에 대한 테트라루프 내 염기 사이의 수소 결합으로 이루어진 장거리 3차[35] 상호작용이다.수소 결합과 더불어, 적층 상호작용은 이러한 3차 상호작용의 중요한 구성요소이다.예를 들어 GNRA-테트라루프 상호작용에서 테트라루프의 두 번째 뉴클레오티드는 [24]수용체 내의 A플랫폼 모티브(상기 참조)에 직접 쌓인다.테트라루프와 그 수용체의 배열은 종종 공존하는 것이어서 동일한 유형의 3차 접촉이 테트라루프와 그 동족 [37]수용체의 다른 동질 형태와 이루어질 수 있다.
예를 들어 자기 스플라이싱 그룹 I 인트론은 구조 및 기능을 [24][36]위해 테트라루프 수용체 모티브에 의존한다.구체적으로는 수용체 나선 위에 있는 표준 GAA 모티브 스택의 3개의 아데닌 잔기가 수용체와 다중 안정화 수소 결합을 형성한다.GAAA 배열의 첫 번째 아데닌은 수용체 AU 염기와 3중 염기쌍을 형성한다.두 번째 아데닌은 동일한 우리딘과의 수소 결합 및 수용체와의 2'-OH 및 GAA 테트라루프의 구아닌과의 상호작용에 의해 안정화된다.세 번째 아데닌은 삼중 염기쌍을 형성한다.
A 마이너 모티브
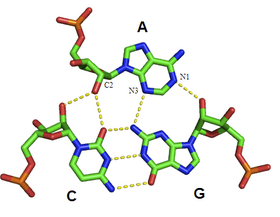
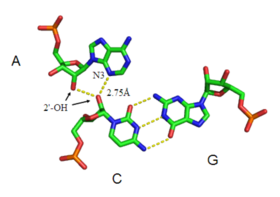
A-마이너 모티브는 유비쿼터스 RNA 3차 구조 모티브입니다.이것은 쌍을 이루지 않은 뉴클레오시드를 RNA 이중체의 작은 홈에 삽입함으로써 형성된다.따라서 그것은 마이너 그루브 트리플의 예시이다.구아노신, 시토신 및 우리딘은 또한 작은 그루브 3중 상호작용을 형성할 수 있지만, 아데닌에 의한 작은 그루브 상호작용은 매우 일반적이다.아데닌의 경우, 삽입 베이스의 N1-C2-N3 가장자리는 이중화 베이스의 2'-OH 중 하나 또는 양쪽과 수소 결합을 형성한다(그림: A-마이너 상호작용 참조).호스트 듀플렉스는 대부분의 경우 G-C 베이스 페어입니다.
A-마이너 모티브는 Watson-Crick 염기쌍의 2'-OH에 상대적인 삽입 베이스의 위치에 따라 유형 0~II의 4가지 [8]클래스로 분리되었다.타입 I 및 II A-minor 모티브에서는 아데닌의 N3가 듀플렉스의 소구내에 깊숙이 삽입되어 있어(그림: A 마이너 상호작용 - 타입 II 상호작용 참조), 염기쌍과의 형상보완성이 양호하다.유형 0과 III와는 달리, 유형 I와 II의 상호작용은 수소 결합 상호작용으로 인해 아데닌에 특이적이다.타입 III의 상호작용에서는 삽입 베이스의 O2'와 N3의 양쪽이 듀플렉스의 마이너홈과 보다 밀접하게 관련되어 있지 않다.유형 0 및 III 모티브는 단일 2'-OH와의 교호작용에 의해 매개되기 때문에 더 약하고 비특이적입니다(그림: A-소수 상호작용 - 유형 0 및 유형 III 교호작용 참조).
A-마이너 모티브는 리보솜에서 가장 일반적인 RNA 구조 모티브 중 하나로, 23S 서브유닛에 [39]대한 tRNA 결합에 기여한다.이들은 대부분 그룹 II [6]인트론의 핵심과 같은 루프와 나선형에서 RNA 이중 상호작용을 안정화시킵니다.
A 마이너의 흥미로운 예는 안티코돈 인식에서의 역할이다.리보솜은 올바른 코돈-안티코돈 쌍과 올바르지 않은 코돈 쌍을 구별해야 합니다.그것은 부분적으로 아데닌 염기를 작은 홈에 삽입함으로써 그렇게 한다.잘못된 코돈-안티코돈 쌍은 왜곡된 나선 형상을 나타내며, 이는 A-마이너 상호작용이 결합을 안정화하는 것을 방지하고 잘못된 tRNA의 [40]해리율을 증가시킨다.
23S 리보솜 RNA의 A 마이너 모티브의 분석은 리보솜 진화와 현대 세균의 큰 [41]서브유닛의 개발을 이끈 사건의 순서와 관련이 있는 구조적 의존성의 계층적 네트워크를 밝혀냈다.
A-마이너 모티브와 그 새로운 서브클래스인 WC/H A-마이너 상호작용은 RNA [15][14]안정화 요소에서 식별된 주요 홈 삼중 나선과 같은 다른 RNA 3차 구조를 강화시키는 것으로 보고되었다.
리보스 지퍼

리보스 지퍼는 2'와 관련된 수소 결합 상호작용에 의해 두 개의 RNA 사슬이 함께 고정되는 RNA 3차 구조 요소이다.리보오스당의 OH는 다른 가닥에 있습니다.2'OH는 수소 결합 공여체이자 수용체 역할을 할 수 있으며, 이를 통해 또 다른 2'[42][43]OH와 함께 분기된 수소 결합을 형성할 수 있다.
많은 형태의 리보스 지퍼가 보고되었지만, 일반적인 유형은 인접한 두 설탕의 2'-OH 그룹 사이의 4개의 수소 결합을 포함합니다.리보스 지퍼는 일반적으로 개별 RNA [44]가닥 간의 상호작용을 안정화하는 배열에서 발생합니다.리보스 지퍼는 시퀀스 특이성이 매우 낮은 스템 루프 상호작용으로 종종 관찰된다.그러나 작고 큰 리보솜 서브유닛에서는 CC/AA 시퀀스의 리보오스 지퍼에 대한 경향이 존재한다. 즉, 첫 번째 사슬의 두 개의 세포신과 두 번째 사슬의 두 개의 아데닌이 쌍을 이룬다.
금속 이온의 역할
기능성 RNA는 종종 플로피 선형 [46]가닥보다는 3차원 모양을 가진 접힌 안정적인 분자입니다.양이온은 RNA 3차 구조의 열역학적 안정화에 필수적이다.RNA를 결합하는 금속 양이온은 1가, 2가 또는 3가일 수 있습니다.칼륨(K+)은 RNA를 결합하는 일반적인 1가 이온이며, RNA를 결합하는 일반적인 2가 이온은 마그네슘(Mg)이다2+.나트륨(Na+), 칼슘(Ca2+), 망간(Mn2+)을 포함한 다른 이온은 RNA를 생체내 및 체외에 결합시키는 것으로 확인되었다.또한 스펠미딘이나 스펠민과 같은 다가의 유기 양이온도 세포에서 발견되며 이것들은 RNA 접힘에 중요한 기여를 한다.코발트 헥사민과 같은 3가 이온이나 테르비움(Tb3+)과 같은 란타니드 이온은 [47][48]RNA에 대한 금속 결합을 연구하는 데 유용한 실험 도구입니다.
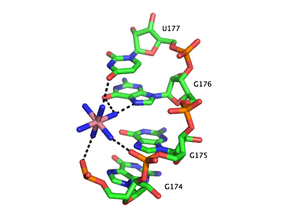
금속 이온은 RNA와 여러 가지 방식으로 상호작용할 수 있다.이온은 RNA backbone에 확산적으로 결합할 수 있으며, 그렇지 않으면 불리한 정전 상호작용을 차폐할 수 있습니다.이 전하 선별은 종종 1가 이온에 의해 충족됩니다.부위 결합 이온은 RNA 3차 구조의 특정 요소를 안정화시킵니다.부위의 결합 상호작용은 물이 금속 결합을 매개하는지 여부에 따라 두 가지 범주로 세분될 수 있다."외구" 상호작용은 금속 이온을 둘러싼 물 분자에 의해 매개된다.예를 들어, 마그네슘 육수화물은 주요 홈에서 구아노신과의 상호작용을 통해 특정 RNA 3차 구조 모티브와 상호작용하고 안정화시킨다.반대로, "내구" 상호작용은 금속 이온에 의해 직접적으로 매개된다.RNA는 종종 여러 단계에서 접히고 이러한 단계는 다른 종류의 양이온에 의해 안정화될 수 있습니다.초기에 RNA는 폴리 음이온성 골격을 중화시키기 위해 1가의 양이온, 2가의 양이온 및 폴리아니온성 아민의 결합을 통해 안정화 된 2차 구조를 형성한다.이 과정의 후반 단계는 RNA 3차 구조의 형성을 포함하며, 이는 칼륨 결합에서 가능한 기여와 함께 마그네슘과 같은 2가 이온의 결합을 통해 거의 안정화된다.
금속 결합 부위는 종종 퓨린의 Hoogsteen 가장자리에 맞춰 RNA 이중체의 깊고 좁은 주요 홈에 국소화됩니다.특히 금속 양이온은 인산염의 촘촘한 패킹으로 인해 고밀도 음전하가 발생하는 골격 비틀림 부위를 안정화시킵니다.결정 구조에서 확인된 RNA 이중체에는 몇 가지 금속 이온 결합 모티브가 있습니다.예를 들어 Tetrahymena thermophila group I 인트론의 P4-P6 도메인에서는 여러 이온결합 부위가 탠덤 G-U 워블 쌍과 탠덤 G-A 미스매치로 구성되며, 탠덤 G-A 미스매치는 2가의 양이온이 O6 및 [49][50][51]N7을 통해 구아노신의 후그스틴 가장자리에 상호작용한다.Tetrahymena군 I 인트론의 또 다른 이온결합 모티브는 A-A플랫폼 모티브이며, 이 모티브에서는 동일한 RNA사슬 내의 연속된 아데노신이 비카논성 의사염기쌍을 [52]형성한다.탠덤 G-U 모티브와 달리 A-A 플랫폼 모티브는 1가 양이온에 우선적으로 결합됩니다.이러한 모티브 중 많은 경우, 1가 또는 2가 양이온이 없는 경우 3차 구조의 유연성 또는 손실을 초래한다.
2가 금속 이온, 특히 마그네슘은 유전자 재조합의 홀리데이 접합 중간체와 같은 DNA 접합의 구조에 중요한 것으로 밝혀졌다.마그네슘 이온은 음으로 대전된 인산염 그룹을 접합부에서 차폐하고 서로 더 가깝게 배치하여 분할된 구성이 [53]아닌 스택된 구성을 가능하게 합니다.마그네슘은 DNA 나노테크놀로지에 사용되는 인공적으로 설계된 구조에서 이러한 결합을 안정화시키는데 필수적입니다. 예를 들어 이중 교차 [54]모티브입니다.
역사
RNA 구조 생물학에서 가장 초기의 연구는 1950년대 초에 DNA에 대한 연구와 거의 일치했다.왓슨과 크릭은 1953년에 발표한 논문에서 리보스의 2'OH 그룹에 의해 몰려드는 반데르발스가 그들이 제안한 모델과 동일한 이중 나선 구조를 RNA가 채택하는 것을 막을 수 있을 것이라고 제안했다 - 현재 우리가 B형 [55]DNA로 알고 있다.이것은 RNA의 3차원 구조에 대한 의문을 불러일으켰다: 이 분자가 어떤 종류의 나선 구조를 형성할 수 있는가? 만약 그렇다면, 어떻게?
1960년대 중반에 단백질 합성에 있어 tRNA의 역할이 집중적으로 연구되었다.1965년 Holley 등은 첫 번째 tRNA 분자를 정제하고 염기서열을 분석했으며, 처음에는 주로 스템 루프 [56]구조를 형성하는 분자의 특정 영역의 능력에 기초한 클로버 잎 구조를 채택할 것을 제안했다.tRNA의 분리는 RNA 구조 생물학에서 첫 번째 큰 횡재임이 입증되었다.1971년 Kim 등은 tRNA에 [57]결합하고 안정화시키는 자연발생 폴리아민인 스펠민을 사용하여 효모 tRNA의 결정을 2-3 Ongström 분해능으로 회절시키는 또 다른 돌파구를 마련했다.
첫 번째 tRNA 구조 이후 상당 기간 동안 RNA 구조 분야는 극적으로 발전하지 않았다.RNA 구조를 연구하는 능력은 RNA 표적을 분리할 수 있는 가능성에 달려 있었다.이것은 여러 해 동안 이 분야에 제한적인 것으로 입증되었는데, 부분적으로는 다른 알려진 표적인 리보솜이 분리 및 결정화하기가 훨씬 더 어려웠기 때문이다.이와 같이 tRNA 구조의 최초 발표 이후 약 20년 동안 소수의 다른 RNA 타깃의 구조만이 해결되었으며, 이들 중 거의 대부분이 전달 RNA [58]패밀리에 속했다.
이 불행한 범위 부족은 핵산 연구의 두 가지 주요 발전, 즉 리보자임의 식별과 시험관내 전사를 통한 생산 능력 때문에 결국 극복될 것이다.Tetrahymena 그룹 I 인트론을 자기촉매 리보자임으로 [59]암시하는 Tom Cech의 발표와 시드니 Altman의 리보핵산가수분해효소 P [60]RNA에 의한 촉매작용에 대한 보고에 이어, [61]1980년대 후반에 해머헤드 리보자임을 포함한 몇 가지 다른 촉매 RNA가 확인되었다.1994년 McKay 등은 2.6 [62]Ongström 분해능에서 DNA 기질에 결합함으로써 리보자임의 자기촉매 활성을 방해한 '해머헤드 RNA-DNA 리보자임-억제제 복합체'의 구조를 발표했다.결정학을 통한 지구구조 결정의 진보에 더해, 1990년대 초에는 RNA 구조생물학의 강력한 기술로 NMR의 구현도 볼 수 있었다.이와 같은 연구는 큰 RNA 분자의 전지구적 접힘을 안정시키는 염기쌍과 염기 적층 상호작용의 보다 정밀한 특성화를 가능하게 했다.
1990년대 중반 RNA 구조생물학의 부활은 핵산 구조 연구 분야에서 진정한 폭발을 일으켰다.해머헤드와4-6 P구조물이 출판된 이후, 이 분야에 많은 공헌이 이루어졌습니다.가장 주목할 만한 예로는 그룹 I과 그룹 II의 [6]침입 구조 및 [38]리보솜이 있다.처음 세 가지 구조는 시험관내 전사를 사용하여 생성되었으며, NMR은 RNA 연구에 있어 두 기술의 필수성에 대한 고환인 네 가지 구조 모두의 부분 성분을 조사하는 역할을 했다.2009년 노벨 화학상은 리보솜에 대한 구조적인 연구로 아다 요나트, 벤카트라만 라마크리슈난, 토마스 스타이츠에게 수여되었으며, 이는 RNA 구조 생물학이 현대 분자 생물학에서 가져 온 중요한 역할을 증명한다.
「 」를 참조해 주세요.
레퍼런스
- ^ IUPAC, 화학 용어집, 제2판('골드북') (1997).온라인 수정판: (2006–) "teriary structure".doi:10.1351/goldbook.T06282
- ^ Richmond TJ, Davey CA (May 2003). "The structure of DNA in the nucleosome core". Nature. 423 (6936): 145–50. Bibcode:2003Natur.423..145R. doi:10.1038/nature01595. PMID 12736678. S2CID 205209705.
- ^ Watson JD, Crick FH (April 1953). "Molecular structure of nucleic acids; a structure for deoxyribose nucleic acid" (PDF). Nature. 171 (4356): 737–8. Bibcode:1953Natur.171..737W. doi:10.1038/171737a0. PMID 13054692. S2CID 4253007.
- ^ Bansal M (2003). "DNA structure: Revisiting the Watson-Crick double helix". Current Science. 85 (11): 1556–1563.
- ^ Ghosh A, Bansal M (2003). "A glossary of DNA structures from A to Z". Acta Crystallogr D. 59 (4): 620–626. doi:10.1107/S0907444903003251. PMID 12657780.
- ^ a b c d e PDB: 3BWP;; PyMOL로 렌더링
- ^ a b PDB: 2K95;; PyMOL로 렌더링
- ^ a b Doherty EA, Batey RT, Masquida B, Doudna JA (April 2001). "A universal mode of helix packing in RNA". Nat. Struct. Biol. 8 (4): 339–43. doi:10.1038/86221. PMID 11276255. S2CID 213577.
- ^ Szewczak AA, Ortoleva-Donnelly L, Ryder SP, Moncoeur E, Strobel SA (December 1998). "A minor groove RNA triple helix within the catalytic core of a group I intron". Nat. Struct. Biol. 5 (12): 1037–42. doi:10.1038/4146. PMID 9846872. S2CID 10908125.
- ^ Boudvillain M, de Lencastre A, Pyle AM (July 2000). "A tertiary interaction that links active-site domains to the 5' splice site of a group II intron". Nature. 406 (6793): 315–8. Bibcode:2000Natur.406..315B. doi:10.1038/35018589. PMID 10917534. S2CID 4336795.
- ^ a b c PDB: 1RAU;; PyMOL로 렌더링
- ^ a b PDB: 1FIT;; PyMOL로 렌더링
- ^ Gilbert SD, Rambo RP, Van Tyne D, Batey RT (February 2008). "Structure of the SAM-II riboswitch bound to S-adenosylmethionine". Nat. Struct. Mol. Biol. 15 (2): 177–82. doi:10.1038/nsmb.1371. PMID 18204466. S2CID 40791601.
- ^ a b Mitton-Fry RM, DeGregorio SJ, Wang J, Steitz TA, Steitz JA (November 2010). "Poly(A) tail recognition by a viral RNA element through assembly of a triple helix". Science. 330 (6008): 1244–7. Bibcode:2010Sci...330.1244M. doi:10.1126/science.1195858. PMC 3074936. PMID 21109672.
- ^ a b Torabi SF, Vaidya AT, Tycowski KT, DeGregorio SJ, Wang J, Shu MD, et al. (January 2021). "RNA stabilization by a poly(A) tail 3'-end binding pocket and other modes of poly(A)-RNA interaction". Science. 371 (6529). doi:10.1126/science.abe6523. ISSN 0036-8075. PMID 33414189. S2CID 231195473.
- ^ a b Batey RT, Gilbert SD, Montange RK (November 2004). "Structure of a natural guanine-responsive riboswitch complexed with the metabolite hypoxanthine". Nature. 432 (7015): 411–5. Bibcode:2004Natur.432..411B. doi:10.1038/nature03037. PMID 15549109. S2CID 2462025.
- ^ Arthanari H, Bolton PH (March 2001). "Functional and dysfunctional roles of quadruplex DNA in cells". Chem. Biol. 8 (3): 221–30. doi:10.1016/S1074-5521(01)00007-2. PMID 11306347.
- ^ Oliver AW, Bogdarina I, Schroeder E, Taylor IA, Kneale GG (August 2000). "Preferential binding of fd gene 5 protein to tetraplex nucleic acid structures". J. Mol. Biol. 301 (3): 575–84. doi:10.1006/jmbi.2000.3991. PMID 10966771.
- ^ PDB: 6tna;; PyMOL을 통해 렌더링됩니다.
- ^ a b Quigley GJ, Rich A (November 1976). "Structural domains of transfer RNA molecules". Science. 194 (4267): 796–806. Bibcode:1976Sci...194..796Q. doi:10.1126/science.790568. PMID 790568.
- ^ "Douglas H. Turner". Turner’s rules. Department of Chemistry, University of Rochester.
- ^ Walter AE, Turner DH, Kim J, Lyttle MH, Müller P, Mathews DH, Zuker M (September 1994). "Coaxial stacking of helixes enhances binding of oligoribonucleotides and improves predictions of RNA folding". Proc. Natl. Acad. Sci. U.S.A. 91 (20): 9218–22. Bibcode:1994PNAS...91.9218W. doi:10.1073/pnas.91.20.9218. PMC 44783. PMID 7524072.
- ^ Murphy FL, Wang YH, Griffith JD, Cech TR (September 1994). "Coaxially stacked RNA helices in the catalytic center of the Tetrahymena ribozyme". Science. 265 (5179): 1709–12. Bibcode:1994Sci...265.1709M. doi:10.1126/science.8085157. PMID 8085157.
- ^ a b c Cate JH, Gooding AR, Podell E, Zhou K, Golden BL, Kundrot CE, Cech TR, Doudna JA (September 1996). "Crystal structure of a group I ribozyme domain: principles of RNA packing". Science. 273 (5282): 1678–85. Bibcode:1996Sci...273.1678C. doi:10.1126/science.273.5282.1678. PMID 8781224. S2CID 38185676.
- ^ Noller HF (September 2005). "RNA structure: reading the ribosome". Science. 309 (5740): 1508–14. Bibcode:2005Sci...309.1508N. doi:10.1126/science.1111771. PMID 16141058. S2CID 16577145.
- ^ Lee AJ, Crothers DM (August 1998). "The solution structure of an RNA loop-loop complex: the ColE1 inverted loop sequence". Structure. 6 (8): 993–1005. doi:10.1016/S0969-2126(98)00101-4. PMID 9739090.
- ^ Ferré-D'Amaré AR, Zhou K, Doudna JA (October 1998). "Crystal structure of a hepatitis delta virus ribozyme". Nature. 395 (6702): 567–74. Bibcode:1998Natur.395..567F. doi:10.1038/26912. PMID 9783582. S2CID 4359811.
- ^ Rothemund PW (March 2006). "Folding DNA to create nanoscale shapes and patterns" (PDF). Nature. 440 (7082): 297–302. Bibcode:2006Natur.440..297R. doi:10.1038/nature04586. PMID 16541064. S2CID 4316391.
- ^ a b c d PDB: 1GID;; PyMOL로 렌더링
- ^ Nakano M, Moody EM, Liang J, Bevilacqua PC (December 2002). "Selection for thermodynamically stable DNA tetraloops using temperature gradient gel electrophoresis reveals four motifs: d(cGNNAg), d(cGNABg),d(cCNNGg), and d(gCNNGc)". Biochemistry. 41 (48): 14281–92. doi:10.1021/bi026479k. PMID 12450393.
- ^ Moore PB (1999). "Structural motifs in RNA". Annu. Rev. Biochem. 68 (1): 287–300. doi:10.1146/annurev.biochem.68.1.287. PMID 10872451.
- ^ Abramovitz DL, Pyle AM (February 1997). "Remarkable morphological variability of a common RNA folding motif: the GNRA tetraloop-receptor interaction". J. Mol. Biol. 266 (3): 493–506. doi:10.1006/jmbi.1996.0810. PMID 9067606.
- ^ Moody EM, Feerrar JC, Bevilacqua PC (June 2004). "Evidence that folding of an RNA tetraloop hairpin is less cooperative than its DNA counterpart". Biochemistry. 43 (25): 7992–8. doi:10.1021/bi049350e. PMID 15209494.
- ^ Zhao Q, Huang HC, Nagaswamy U, Xia Y, Gao X, Fox GE (August 2012). "UNAC tetraloops: to what extent do they mimic GNRA tetraloops?". Biopolymers. 97 (8): 617–628. doi:10.1002/bip.22049. PMID 22605553.
- ^ Williams DH, Gait MJ, Loakes D (2006). Nucleic Acids in Chemistry and Biology. Cambridge, UK: RSC Pub. ISBN 0-85404-654-2.
- ^ a b Jaeger L, Michel F, Westhof E (March 1994). "Involvement of a GNRA tetraloop in long-range RNA tertiary interactions". J. Mol. Biol. 236 (5): 1271–6. doi:10.1016/0022-2836(94)90055-8. PMID 7510342.
- ^ Michel F, Westhof E (December 1990). "Modelling of the three-dimensional architecture of group I catalytic introns based on comparative sequence analysis". J. Mol. Biol. 216 (3): 585–610. doi:10.1016/0022-2836(90)90386-Z. PMID 2258934.
- ^ a b c PDB: 1FFK;; PyMOL로 렌더링
- ^ Nissen P, Ippolito JA, Ban N, Moore PB, Steitz TA (April 2001). "RNA tertiary interactions in the large ribosomal subunit: the A-minor motif". Proc. Natl. Acad. Sci. U.S.A. 98 (9): 4899–903. Bibcode:2001PNAS...98.4899N. doi:10.1073/pnas.081082398. PMC 33135. PMID 11296253.
- ^ Yoshizawa S, Fourmy D, Puglisi JD (September 1999). "Recognition of the codon-anticodon helix by ribosomal RNA". Science. 285 (5434): 1722–5. doi:10.1126/science.285.5434.1722. PMID 10481006.
- ^ Bokov K, Steinberg SV (February 2009). "A hierarchical model for evolution of 23S ribosomal RNA". Nature. 457 (7232): 977–80. Bibcode:2009Natur.457..977B. doi:10.1038/nature07749. PMID 19225518. S2CID 4400869.
- ^ Batey RT, Rambo RP, Doudna JA (August 1999). "Tertiary Motifs in RNA Structure and Folding". Angew. Chem. Int. Ed. Engl. 38 (16): 2326–2343. doi:10.1002/(SICI)1521-3773(19990816)38:16<2326::AID-ANIE2326>3.0.CO;2-3. PMID 10458781.
- ^ Tamura M, Holbrook SR (July 2002). "Sequence and structural conservation in RNA ribose zippers". J. Mol. Biol. 320 (3): 455–74. doi:10.1016/S0022-2836(02)00515-6. PMID 12096903.
- ^ PDB: 3IGI;; PyMOL을 사용하여 렌더링.
- ^ PDB: 1ZZN;; PyMOL로 렌더링
- ^ Celander DW, Cech TR (January 1991). "Visualizing the higher order folding of a catalytic RNA molecule". Science. 251 (4992): 401–7. Bibcode:1991Sci...251..401C. doi:10.1126/science.1989074. PMID 1989074.
- ^ Pyle AM (September 2002). "Metal ions in the structure and function of RNA". J. Biol. Inorg. Chem. 7 (7–8): 679–90. doi:10.1007/s00775-002-0387-6. PMID 12203005. S2CID 42008484.
- ^ Morrow JR, Andolina CM (2012). "Chapter 6. Spectroscopic Investigations of Lanthanide Ion Binding to Nucleic Acids". In Sigel A, Sigel H, Sigel RK (eds.). Interplay between Metal Ions and Nucleic Acids. Metal Ions in Life Sciences. Vol. 10. Springer. pp. 171–197. doi:10.1007/978-94-007-2172-2_6. PMID 22210339.
- ^ Cate JH, Doudna JA (October 1996). "Metal-binding sites in the major groove of a large ribozyme domain". Structure. 4 (10): 1221–9. doi:10.1016/S0969-2126(96)00129-3. PMID 8939748.
- ^ Kieft JS, Tinoco I (May 1997). "Solution structure of a metal-binding site in the major groove of RNA complexed with cobalt (III) hexammine". Structure. 5 (5): 713–21. doi:10.1016/S0969-2126(97)00225-6. PMID 9195889.
- ^ Rüdisser S, Tinoco I (February 2000). "Solution structure of Cobalt(III)hexammine complexed to the GAAA tetraloop, and metal-ion binding to G·A mismatches". J. Mol. Biol. 295 (5): 1211–23. doi:10.1006/jmbi.1999.3421. PMID 10653698.
- ^ Burkhardt C, Zacharias M (October 2001). "Modelling ion binding to AA platform motifs in RNA: a continuum solvent study including conformational adaptation". Nucleic Acids Res. 29 (19): 3910–8. doi:10.1093/nar/29.19.3910. PMC 60250. PMID 11574672.
- ^ Panyutin IG, Biswas I, Hsieh P (April 1995). "A pivotal role for the structure of the Holliday junction in DNA branch migration". The EMBO Journal. 14 (8): 1819–26. doi:10.1002/j.1460-2075.1995.tb07170.x. PMC 398275. PMID 7737132.
- ^ Fu TJ, Seeman NC (April 1993). "DNA double-crossover molecules". Biochemistry. 32 (13): 3211–20. doi:10.1021/bi00064a003. PMID 8461289.
- ^ Watson JD, Crick FH (April 1953). "Molecular structure of nucleic acids; a structure for deoxyribose nucleic acid" (PDF). Nature. 171 (4356): 737–738. Bibcode:1953Natur.171..737W. doi:10.1038/171737a0. PMID 13054692. S2CID 4253007.
- ^ Holley, RW, Apgar, J, Everett, GA, Madison, JT, Marguisse, M, Merrill, SH, Penwick, JR, Zamir (March 1965). "Structure of a ribonucleic acid". Science. 147 (3664): 1462–5. Bibcode:1965Sci...147.1462H. doi:10.1126/science.147.3664.1462. PMID 14263761. S2CID 40989800.
{{cite journal}}: CS1 maint: 여러 이름: 작성자 목록(링크) - ^ Kim SH, Quigley G, Suddath FL, Rich A (April 1971). "High-resolution x-ray diffraction patterns of crystalline transfer RNA that show helical regions". Proc. Natl. Acad. Sci. U.S.A. 68 (4): 841–5. Bibcode:1971PNAS...68..841K. doi:10.1073/pnas.68.4.841. PMC 389056. PMID 5279525.
- ^ Shen LX, Cai Z, Tinoco I (August 1995). "RNA structure at high resolution". FASEB J. 9 (11): 1023–33. doi:10.1096/fasebj.9.11.7544309. PMID 7544309. S2CID 40621440.
- ^ Cech TR, Zaug AJ, Grabowski PJ (December 1981). "In vitro splicing of the ribosomal RNA precursor of Tetrahymena: involvement of a guanosine nucleotide in the excision of the intervening sequence". Cell. 27 (3 Pt 2): 487–96. doi:10.1016/0092-8674(81)90390-1. PMID 6101203. S2CID 17674600.
- ^ Stark BC, Kole R, Bowman EJ, Altman S (August 1978). "Ribonuclease P: an enzyme with an essential RNA component". Proc. Natl. Acad. Sci. U.S.A. 75 (8): 3717–21. Bibcode:1978PNAS...75.3717S. doi:10.1073/pnas.75.8.3717. PMC 392857. PMID 358197.
- ^ Prody GA, Bakos JT, Buzayan JM, Schneider IR, Bruening G (March 1986). "Autolytic Processing of Dimeric Plant Virus Satellite RNA". Science. 231 (4745): 1577–1580. Bibcode:1986Sci...231.1577P. doi:10.1126/science.231.4745.1577. PMID 17833317. S2CID 21563490.
- ^ Pley HW, Flaherty KM, McKay DB (November 1994). "Three-dimensional structure of a hammerhead ribozyme". Nature. 372 (6501): 68–74. Bibcode:1994Natur.372...68P. doi:10.1038/372068a0. PMID 7969422. S2CID 4333072.