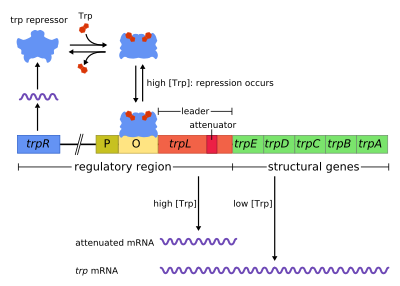
trp 오퍼론
trp operontrp 오퍼론은 트립토판을 생성하는 성분을 코드하는 함께 사용되거나 전사되는 유전자의 그룹인 오퍼론입니다.trp 오퍼론은 많은 박테리아에 존재하지만, 대장균에서 처음 특징지어졌습니다.오퍼론은 환경에 트립토판이 존재할 때 트립토판 합성을 위한 유전자가 발현되지 않도록 조절된다.그것은 유전자 조절에 대해 배우기 위한 중요한 실험 시스템이었고, 유전자 조절을 가르치는데 일반적으로 사용된다.
Trp 오퍼론은 경로의 효소적인 부분을 코드하는 trpE, trpD, trpC, trpB 및 trpA의 다섯 가지 구조 유전자를 포함합니다.그것은 또한 trpR이라고 불리는 억제성 조절 유전자를 포함합니다. trpR은 RNA 중합효소가 조절 단백질을 위해 mRNA를 결합하고 합성하는 프로모터를 가지고 있습니다.trpR에 의해 합성된 단백질은 그 후 전사를 차단하는 작용기에 결합한다.트립토판은 유전자 전사를 효과적으로 차단하는 억제단백질에 결합한다.이 상황에서 억제란 오퍼론에서 유전자를 전사하는 RNA 중합효소의 억제이다.또한 라크 오퍼론과 달리 trp 오퍼론은 단계적 조절을 [1]가능하게 하는 리더펩타이드 및 감쇠기 배열을 포함한다.
그것은 유전자 발현에 대한 억제할 수 있는 음성 조절의 한 예이다.오퍼론의 조절 배열 내에서 오퍼레이터는 트립토판이 존재하는 경우(전사를 방지함) 억제단백질에 결합하고 트립토판이 없는 경우(전사를 허용함) 해방된다.
유전자
Trp Operon은 5개의 구조 유전자를 가지고 있다.그 역할은 다음과 같습니다.
- TrpE(P00895):안트라닐산합성효소는 안트라닐산을 생성한다.
- Trpd (P00904) :TrpE와 협력합니다.
- TrpC(P00909) :포스포리보실란트라네이트 이성질화효소 도메인은 먼저 N-(5-포스포-β-D-리보실)안트라네이트를 1-(2-카르복시페닐아미노)-1-디옥시-D-리불로스5-인산으로 한다.동일한 단백질에서 인돌-3-글리세롤-인산합성효소는 생성물을 (1S,2R)-1-C-(인돌-3-일)글리세롤 3-인산으로 변화시킨다.
- TrpA(P0A877), TrpB(P0A879) : 트립토판합성효소 2개 서브유닛.TrpC 제품과 세린을 조합하여 트립토판을 만듭니다.
억압
오퍼론은 음의 억제 가능한 피드백 메커니즘에 의해 작동한다.trp 오퍼론의 억제제는 구성적으로 낮은 수준에서 발현되는 trpR 유전자에 의해 상류에서 생성된다.합성된 trpR 단량체는 이합체에 결합한다.트립토판이 존재하는 경우 이러한 트립토판 억제기 이합체가 트립토판에 결합되어 억제기 배치가 변경되어 억제기가 연산자에 결합할 수 있게 됩니다.이것은 RNA 중합효소가 오퍼론에 결합하고 전사하는 것을 방지하므로 트립토판은 그 전구체에서 생성되지 않습니다.트립토판이 존재하지 않는 경우, 리프레서는 비액티브한 컨피규레이션에 있어 오퍼레이터 영역을 바인드 할 수 없기 때문에 리프레서에 의해 전사가 금지되지 않습니다.
감쇠
감쇠는 trp 오퍼론에서의 네거티브 피드백의 두 번째 메커니즘입니다.억제 시스템은 세포 내 trp 농도를 목표로 하며, 감쇠는 대전된 tRNA trp[2]농도에 반응한다.따라서, trpR 억제제는 전사의 시작을 변경함으로써 유전자 발현을 감소시키는 반면, 감쇠는 이미 [2]진행 중인 전사의 과정을 변경함으로써 그렇게 한다.TrpR 억제제는 전사를 70배 감소시키지만 감쇠는 10배 감소시켜 약 700배의 누적 [3]억제를 가능하게 한다.감쇠는 RNA 중합효소가 여전히 DNA 염기서열을 전사하는 동안 리보솜이 mRNA를 번역하기 시작한다는 사실에 의해 가능하다.이것에 의해, 변환 프로세스가 오퍼론의 문자 변환에 직접 영향을 줄 수 있습니다.
trp 오퍼론의 전사 유전자의 선두에는 리더 전사체(trpL; P0AD92)[4]라고 불리는 적어도 130개의 뉴클레오티드의 배열이 있다.Lee와 Yanofsky(1977)는 감쇠 효율이 [5]trpL에 내장된 2차 구조의 안정성과 상관관계가 있다는 것을 발견했으며, 터미네이터 구조의 2개 구성 헤어핀은 나중에 Oxender et al.([6]1979)에 의해 설명되었다.이 대본은 1~4로 지정된 4개의 짧은 시퀀스를 포함하며, 각각은 다음 시퀀스를 부분적으로 보완한다.따라서 1-2, 2-3 또는 3-4의 세 가지 2차 구조(헤어핀)가 형성될 수 있다.RNA 중합효소는 리보솜이 부착되기를 기다린 후 시퀀스 1을 지나 전사를 계속하기 때문에 시퀀스 1과 시퀀스 2의 교배는 드물지만, 1-2 헤어핀이 형성된다면 (3-4가 아닌) 2-3 구조의 형성을 방해할 것이다.시퀀스 2-3 사이에 헤어핀 루프가 형성되면 1-2와 3-4 사이에 헤어핀 루프가 형성되는 것을 방지할 수 있다.3-4 구조는 RNA 중합효소를 형성하면 DNA에서 연결이 끊기고 오퍼론의 구조 유전자의 전사가 일어날 수 없다(자세한 설명은 아래 참조).전사 종료를 위한 두 번째 헤어핀의 기능적 중요성은 이 헤어핀의 [5][7][8][9]중심 G+C 쌍을 불안정하게 하는 실험에서 관찰된 감소된 전사 종단 빈도로 설명된다.
리더 전사체의 일부는 리더 펩타이드라고 불리는 14개의 아미노산으로 이루어진 짧은 폴리펩타이드를 코드한다.이 펩타이드에는 인접한 2개의 트립토판 잔기가 포함되어 있는데, 트립토판은 매우 드문 아미노산이기 때문이다(일반 대장균 단백질의 100개 중 1개는 트립토판이다).trpL의 가닥 1은 리더펩타이드의 후행 잔기를 코드하는 영역을 포함한다.Trp, Trp, Arg, Thr, Ser.[2] 이 5개의 코돈에서는 보존이 확인되지만 업스트림 코돈을 변환해도 오퍼론 [2][10][11][12]표현은 변경되지 않습니다.세포 내 트립토판 수치가 낮을 때 리보솜이 이 펩타이드를 번역하려고 시도하면 두 trp 코돈 중 하나에서 멈춥니다.정지하는 동안 리보솜은 물리적으로 전사 시퀀스 1을 차폐하여 1-2 이차 구조의 형성을 막는다.시퀀스 2는 시퀀스 3과 교배하여 2-3 구조를 형성할 수 있으며, 이는 3-4 종단 헤어핀의 형성을 방지하므로 2-3 구조를 안티 종단 헤어핀이라고 한다.2-3 구조의 존재 하에서 RNA 중합효소는 오퍼론을 계속 전사하는 데 자유롭다.상보적 올리고뉴클레오티드를 포함한 돌연변이 분석과 연구는 2-3 구조의 안정성이 오퍼론 발현 [10][13][14][15]수준에 해당함을 보여준다.세포 내 트립토판 수치가 높으면 리보솜은 전체 리더 펩타이드를 중단 없이 번역하고 정지 코돈에서 번역 종료 시에만 멈춥니다.이 시점에서 리보솜은 물리적으로 시퀀스 1과 시퀀스 2를 모두 차폐합니다.따라서 시퀀스 3과 4는 전사를 끝내는 3-4 구조를 형성할 수 있습니다.이 터미네이터 구조는 Trp 탠덤 부근(즉 Trp 또는 Arg 코돈)에서 리보솜이 정지하지 않을 때 형성된다. 리더펩타이드는 번역되지 않거나 풍부한 하전 tRNAtrp로 [2][10]스트랜드 1을 따라 원활하게 번역된다.또한 리보솜은 하류 약 10nts만을 차단하도록 제안되므로 상류 Gly 또는 하류 Thr 중 하나의 리보솜 지연은 종단 헤어핀 [2][10]형성에 영향을 주지 않는 것으로 보인다.결과적으로 trpL 전사가 구성적으로 발현되는 동안 트립토판을 리보솜에 사용할 수 없는 경우에만 오퍼론이 전사됩니다.
이 감쇠 메커니즘은 실험적으로 지원됩니다.첫째, 리더펩타이드 및 리보솜 지연의 번역이 전사 [13]종료를 억제하기 위해 필요하다는 것을 직접 증명한다.또한 변이분석은 항암제 헤어핀의 염기쌍을 불안정하게 하거나 교란시키는 변이분석을 통해 여러 개의 접힘을 증가시킨다.감쇠모델과 일관되게 이 돌연변이는 [10][13]Trp가 부족한 경우에도 감쇠를 완화할 수 없다.한편, 스트랜드 1을 대상으로 한 상보성 올리고뉴클레오티드는 항거미네이터 [10][14]형성을 촉진함으로써 오퍼론 발현을 증가시킨다.또한 히스티딘 오퍼론에서 보상 돌연변이는 감쇠를 [10][15]억제하는 데 있어 가닥 2-3의 짝짓기 능력이 1차 배열보다 더 중요하다는 것을 보여준다.
감쇠 시에는 변환 리보솜이 정지하는 위치에 따라 종단 헤어핀이 [10]형성될지 여부를 결정한다.전사중합효소가 대체구조를 동시에 포착하기 위해서는 구조변조의 시간척도가 [2]전사중합효소와 동등해야 한다.리보솜이 결합하고 합성 직후 리더 트랜스크립트의 변환을 시작하기 위해 trpL 시퀀스 내에 포즈 사이트가 존재합니다.이 부위에 도달하면 RNA 중합효소는 전사를 멈추고 번역이 시작될 때까지 기다리는 것으로 보인다.이 메커니즘에 의해 감쇠의 중요한 요소인 문자 변환과 번역의 동기화가 가능하게 됩니다.
유사한 감쇠기구는 히스티딘, 페닐알라닌 및 트레오닌의 합성을 조절한다.
서브틸리스균의 trp오페론 조절
Trp오페론 유전자는 대장균과 [16]서브틸리스균의 동일한 순서로 배열되어 있다.두 유기체의 trp 오퍼론의 조절은 세포에 존재하는 trp의 양에 따라 달라진다.그러나 B. 서브틸리스에서 트립토판 생합성의 주요 조절은 전사의 [17]억제보다는 감쇠에 의한 것이다.B. 서브틸리스에서 트립토판은 11 서브유닛 트립토판 활성 RNA결합감쇠단백질(TRAP)에 결합하며, TRAP는 trp 리더 [18][19]RNA에 결합하는 능력을 활성화한다.trp 활성 TRAP은 리더 RNA에 결합하면 전사 [17]종료를 초래하는 터미네이터 구조를 형성한다.
레퍼런스
- ^ Klug WS, Cummings MR, Spencer C (2006). Concepts of Genetics (8th ed.). New Jersey: Pearson Education Inc. pp. 394–402. ISBN 978-0-13-191833-7.
- ^ a b c d e f g Yanofsky C (February 1981). "Attenuation in the control of expression of bacterial operons". Nature. 289 (5800): 751–8. Bibcode:1981Natur.289..751Y. doi:10.1038/289751a0. PMID 7007895. S2CID 4364204.
- ^ Lehninger AL, Nelson DL, Cox MM (2008). Principles of Biochemistry (5th ed.). New York, NY: W.H. Freeman and Company. p. 1128. ISBN 978-0-7167-7108-1.
- ^ Bertrand K, Squires C, Yanofsky C (May 1976). "Transcription termination in vivo in the leader region of the tryptophan operon of Escherichia coli". Journal of Molecular Biology. 103 (2): 319–37. doi:10.1016/0022-2836(76)90315-6. PMID 781269.
- ^ a b Lee F, Yanofsky C (October 1977). "Transcription termination at the trp operon attenuators of Escherichia coli and Salmonella typhimurium: RNA secondary structure and regulation of termination". Proceedings of the National Academy of Sciences of the United States of America. 74 (10): 4365–9. Bibcode:1977PNAS...74.4365L. doi:10.1073/pnas.74.10.4365. PMC 431942. PMID 337297.
- ^ Oxender DL, Zurawski G, Yanofsky C (November 1979). "Attenuation in the Escherichia coli tryptophan operon: role of RNA secondary structure involving the tryptophan codon region". Proceedings of the National Academy of Sciences of the United States of America. 76 (11): 5524–8. Bibcode:1979PNAS...76.5524O. doi:10.1073/pnas.76.11.5524. PMC 411681. PMID 118451.
- ^ Stroynowski I, Yanofsky C (July 1982). "Transcript secondary structures regulate transcription termination at the attenuator of S. marcescens tryptophan operon". Nature. 298 (5869): 34–8. Bibcode:1982Natur.298...34S. doi:10.1038/298034a0. PMID 7045685. S2CID 4347442.
- ^ Zurawski G, Yanofsky C (September 1980). "Escherichia coli tryptophan operon leader mutations, which relieve transcription termination, are cis-dominant to trp leader mutations, which increase transcription termination". Journal of Molecular Biology. 142 (1): 123–9. doi:10.1016/0022-2836(80)90210-7. PMID 6159477.
- ^ Stauffer GV, Zurawski G, Yanofsky C (October 1978). "Single base-pair alterations in the Escherichia coli trp operon leader region that relieve transcription termination at the trp attenuator". Proceedings of the National Academy of Sciences of the United States of America. 75 (10): 4833–7. Bibcode:1978PNAS...75.4833S. doi:10.1073/pnas.75.10.4833. PMC 336215. PMID 368800.
- ^ a b c d e f g h Kolter R, Yanofsky C (1982). "Attenuation in amino acid biosynthetic operons". Annual Review of Genetics. 16: 113–34. doi:10.1146/annurev.ge.16.120182.000553. PMID 6186194.
- ^ Lee F, Bertrand K, Bennett G, Yanofsky C (May 1978). "Comparison of the nucleotide sequences of the initial transcribed regions of the tryptophan operons of Escherichia coli and Salmonella typhimurium". Journal of Molecular Biology. 121 (2): 193–217. doi:10.1016/s0022-2836(78)80005-9. PMID 351195.
- ^ Miozzari G, Yanofsky C (November 1978). "Naturally occurring promoter down mutation: nucleotide sequence of the trp promoter/operator/leader region of Shigella dysenteriae 16". Proceedings of the National Academy of Sciences of the United States of America. 75 (11): 5580–4. Bibcode:1978PNAS...75.5580M. doi:10.1073/pnas.75.11.5580. PMC 393010. PMID 364484.
- ^ a b c Zurawski G, Elseviers D, Stauffer GV, Yanofsky C (December 1978). "Translational control of transcription termination at the attenuator of the Escherichia coli tryptophan operon". Proceedings of the National Academy of Sciences of the United States of America. 75 (12): 5988–92. Bibcode:1978PNAS...75.5988Z. doi:10.1073/pnas.75.12.5988. PMC 393102. PMID 366606.
- ^ a b Winkler ME, Mullis K, Barnett J, Stroynowski I, Yanofsky C (April 1982). "Transcription termination at the tryptophan operon attenuator is decreased in vitro by an oligomer complementary to a segment of the leader transcript". Proceedings of the National Academy of Sciences of the United States of America. 79 (7): 2181–5. Bibcode:1982PNAS...79.2181W. doi:10.1073/pnas.79.7.2181. PMC 346154. PMID 6179092.
- ^ a b Johnston HM, Roth JR (February 1981). "DNA sequence changes of mutations altering attenuation control of the histidine operon of Salmonella typhimurium". Journal of Molecular Biology. 145 (4): 735–56. doi:10.1016/0022-2836(81)90312-0. PMID 6167727.
- ^ Merino E, Jensen RA, Yanofsky C (April 2008). "Evolution of bacterial trp operons and their regulation". Current Opinion in Microbiology. 11 (2): 78–86. doi:10.1016/j.mib.2008.02.005. PMC 2387123. PMID 18374625.
- ^ a b Gollnick P, Babitzke P, Antson A, Yanofsky C (2005-11-14). "Complexity in regulation of tryptophan biosynthesis in Bacillus subtilis". Annual Review of Genetics. 39 (1): 47–68. doi:10.1146/annurev.genet.39.073003.093745. PMID 16285852.
- ^ Elliott MB, Gottlieb PA, Gollnick P (January 2001). "The mechanism of RNA binding to TRAP: initiation and cooperative interactions". RNA. 7 (1): 85–93. doi:10.1017/S135583820100173X. PMC 1370072. PMID 11214184.
- ^ Antson AA, Otridge J, Brzozowski AM, Dodson EJ, Dodson GG, Wilson KS, et al. (April 1995). "The structure of trp RNA-binding attenuation protein". Nature. 374 (6524): 693–700. Bibcode:1995Natur.374..693A. doi:10.1038/374693a0. PMID 7715723. S2CID 4340136.
추가 정보
- Morse DE, Mosteller RD, Yanofsky C (1969). "Dynamics of synthesis, translation, and degradation of trp operon messenger RNA in E. coli". Cold Spring Harbor Symposia on Quantitative Biology. 34: 725–40. doi:10.1101/sqb.1969.034.01.082. PMID 4909527.
- Yanofsky C (February 1981). "Attenuation in the control of expression of bacterial operons". Nature. 289 (5800): 751–8. Bibcode:1981Natur.289..751Y. doi:10.1038/289751a0. PMID 7007895. S2CID 4364204.