불완전한 계보 정렬
Incomplete lineage sorting집단 유전학에서 조상의 다형성 유지 또는 종간 다형성이라고도 불리는 불완전한 혈통 [1][2][3]분류는 이전의 분화 [4]사건보다 더 깊어질 때까지 조상의 유전자 복사가 공통의 조상의 복제로 결합하는 데 실패할 때 일어나는 현상을 설명한다.즉, 하나의 유전자가 만들어내는 나무는 개체수나 종 수준의 나무와 달라서 불일치하는 나무를 만들어낸다.종나무의 연속된 노드들 사이에 유지되었던 유전적 다형성의 계통 분류에 의해 야기된 효과는 반형성이라고 불려왔다.그 메커니즘이 무엇이든 간에,[5][6] 그 결과는 생성된 종 수준의 나무가 평가에 사용된 선택된 유전자에 따라 다를 수 있다는 것이다.이는 유전자에 의해 생성된 나무가 개체수나 종 수준의 나무와 동일한 완전한 혈통 분류와는 대조적이다.유전자, 유기체, 샘플링 기술에 따라 다르지만 둘 다 계통 분석의 일반적인 결과입니다.
개념.
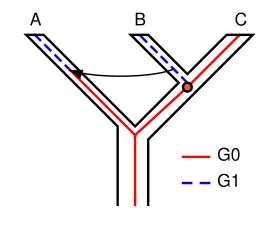
불완전한 혈통 분류의 개념은 계통 발생 기술에 몇 가지 중요한 의미를 갖는다.여러 사양 이벤트에 걸쳐 다형이 지속되면 계보 정렬이 불완전해질 수 있습니다.두 가지 후속 분화 사건이 발생한다고 가정하면, 조상 종이 먼저 A종을 낳고 다음으로 B종과 C종을 발생시킨다고 가정하자.하나의 유전자를 연구할 때, 그것은 여러 가지 버전을 가질 수 있고 다른 특징들이 나타날 수 있다.그림 1의 예에서 유전자 G는 G0과 G1의 두 가지 버전(알레)을 가지고 있다.A, B, C의 조상은 원래 유전자 G의 한 가지 버전인 G0만을 가지고 있었다. 어느 순간 돌연변이가 발생했고 조상 집단은 다형성이 되었고 어떤 개체는 G0과 다른 개체 G1을 가지고 있었다.A종이 갈라졌을 때, B와 C의 조상은 다형성인 반면, G1만 유지되었다.B와 C가 분리되었을 때 B는 G1과 C만 G0으로 유지되었으며, 현재는 G에서도 다형성이 아니다.유전자 G의 나무는 A와 B를 자매로, 종 나무는 B와 C를 자매로 보여준다.만약 이 종들의 계통 발생이 유전자 G에 기초한다면, 그것은 종들 간의 실제 관계를 나타내지 못할 것이다.다시 말해, 가장 가까운 종들이 반드시 가장 가까운 유전자를 물려받지는 않을 것이다.이것은 물론 불완전한 혈통 분류의 단순한 예이며, 실제 연구에서는 보통 더 많은 유전자와 [7][8]종을 포함하는 더 복잡하다.
그림 1에 나타난 특정 종류의 불완전한 혈통 분류는 헤미플라스라고 불리며, 이는 종 나무와 유전자 나무 사이의 불일치가 종 나무의 연속된 노드 간에 유지된 유전적 다형성의 혈통 분류에 의해 발생한다는 것을 의미한다.다른 메커니즘은 같은 명백한 불일치를 야기할 수 있는데, 예를 들어 대립 유전자는 교배종을 통해 종의 경계를 넘어 이동할 수 있고,[9] DNA는 바이러스에 의해 종 간에 전달될 수 있습니다.이것은 그림 2에 나타나 있습니다.여기서 A, B, C의 조상과 B, C의 조상은 G0 버전의 유전자만 가지고 있었다. B와 C의 차이에서 A 돌연변이가 일어나 B는 돌연변이 버전인 G1을 얻었다.잠시 후, 화살표는 G1이 어떤 방법으로 B에서 A로 전달되었음을 나타낸다(예: 교배 또는 수평 유전자 전달).3종의 G의 최종 상태만을 연구하면 그림 1과 같이 A와 B는 B와 C가 아닌 자매로 보이지만 그림 2는 반수식에 의한 것이 아니다.
시사점
불완전한 혈통 분류는 계통학 연구에 중요한 영향을 미친다.계통수 생성 시 이러한 불완전한 계통 분류로 인해 실제 관계와 유사하지 않을 수 있습니다.그러나 교배 또는 수평 유전자 이동에 의한 계통 간 유전자 흐름은 동일한 상충 계통수를 생성할 수 있다.이러한 다양한 과정을 구별하는 것은 어려워 보일 수 있지만, 많은 연구와 다른 통계적 접근법이 이러한 진화적 [10]역학에서 더 나은 통찰력을 얻기 위해 개발되고 있다.불완전한 혈통 분류의 영향을 줄이기 위한 해결책 중 하나는 종이나 개체군 계통 발생을 만들기 위해 여러 유전자를 사용하는 것이다.유전자가 많이 사용될수록 계통 발생은 더욱 신뢰할 수 있게 된다.[8]
이배체 유기체
불완전한 혈통 분류는 보통 성적인 번식과 함께 발생한다. 왜냐하면 그 종은 한 사람 또는 번식 쌍으로 거슬러 올라갈 수 없기 때문이다.유기 부족 인구가 많을 때(즉, 수천 개) 각 유전자는 어느 정도 다양성을 가지며 유전자 나무는 이전에 존재했던 다른 계통으로 구성됩니다.인구가 더 많으면 이러한 조상들의 혈통은 더 오래 지속될 것이다.조상의 개체수가 많고 시기적절하게 분화가 일어나면이 DNA 조각들은 상충되는 관계를 유지합니다.이로 인해 공통 조상 또는 분기점을 [5]결정하는 것이 어려워집니다.
영장류 진화에서
영장류를 연구할 때 침팬지와 보노보는 다른 어떤 분류군보다 서로 관련이 깊고 자매 분류군이다.하지만 보노보 게놈의 1.6%에 있어서, 염기서열은 침팬지보다 인간의 호몰로지와 더 밀접하게 관련되어 있는데, 이것은 아마도 불완전한 혈통 [5]분류의 결과일 것이다.인간과의 23,000개 이상의 DNA 배열에 대한 연구는 약 23%가 침팬지와 [9]인간의 알려진 자매 관계를 지지하지 않는다는 것을 보여주었다.
인류의 진화 속에서
인류의 진화에서 불완전한 혈통 정렬은 선사시대 [11]분화가 일어난 동시에 분류에 실패했을 수 있는 호민 혈통을 도해하는 데 사용됩니다.유전자 검사와 게놈 염기서열 분석의 출현으로, 연구원들은 호민 혈통 사이의 유전적 관계가 신체적 [11]특징에 기초한 그들의 관계에 대한 이전의 이해와 일치하지 않을 수 있다는 것을 발견했다.더욱이, 마지막 공통 조상(LCA)의 분리가 반드시 [12]규격화와 동시에 발생할 필요는 없다.혈통 분류는 고인류학자들이 계통발생학에만 [11]기초한 이전의 분화 모델과는 맞지 않을 수 있는 유전적 관계와 분리를 탐구할 수 있도록 하는 방법이다.
인간 가계도의 불완전한 혈통 분류는 큰 관심 분야이다.고대인류에서 현생인류로의 이행과 다른 유인원의 인간 [13]혈통으로부터의 분리를 고려할 때 많은 미지의 것들이 있다.
유인원과 유인원/인간의 차이
불완전한 혈통 분류는 유전자 간 평균 발산 시간이 종 간 발산 시간과 다를 수 있다는 것을 의미한다.모델에 따르면 인간과 침팬지 게놈의 유전자 간 평균 발산 시간은 인간과 고릴라의 분열 시간보다 더 오래되었다.인간과 침팬지의 공통 조상이 인간과 침팬지, [12]고릴라의 공통 조상에 존재했던 유전물질의 흔적을 남겼다는 뜻이다.그러나 유전자 나무는 [14]종이나 계통수와는 약간 다르다.우리가 인간의, 보노보의 침팬지와 고릴라의 진화 관계로 보이는 계통 발생 나무에서, 이 결과는 보노보와 침팬지의 이산 시간의bonobo-chimpanzee 조상의 일반적인 조상인 humans,[12]에 가까운 곳은 인간과 침팬지들은 일반적인 a을 공유하는지 여부를 나타내는 값에 일을 보여 주nce고릴라로부터 분리된 후 수백만 년 동안 저장되었습니다.이로 인해 불완전한 혈통 정렬 현상이 발생합니다.오늘날 연구자들은 인간과 그들의 상대자들 사이의 진화적 관계를 연구하기 위해 DNA 조각에 의존하고 있으며, 그것이 다른 유형의 [15]인간 게놈으로부터 특이성과 조상 과정에 대한 정보를 제공하기를 희망하고 있다.
바이러스 내

불완전한 혈통 분류는 바이러스 계통역학의 공통적인 특징으로, 한 사람에서 다음 사람으로의 질병 전염으로 대표되는 계통 발생, 즉 집단 수준 트리는 바이러스 전달의 고유한 특징인 개체군 병목 현상으로 인해 유전자 분석에서 생성된 트리와 일치하지 않는 경우가 많다.질병의 사명그림 3은 이러한 현상이 발생하는 방법을 보여 줍니다.이것은 HIV의 범죄 전염과 관련이 있다. 어떤 범죄 사건에서는, 피고와 피해자의 변종으로부터 하나 또는 두 개의 유전자에 대한 계통학적 분석이 전염을 추론하기 위해 사용되었다; 그러나 불완전한 혈통 분류의 공통성은 전염이 그러한 기본적 아나만을 근거로 추정될 수 없다는 것을 의미한다.용리[16]
언어학에서는
Jacques와 List(2019)[17]는 불완전한 혈통 분류 개념을 언어 진화에서 나무와 같지 않은 현상을 설명하기 위해 적용할 수 있음을 보여준다.역사언어학에서 나무 모델의 적용 가능성에 도전하는 모델인 역사적 성문측정법의 지지자인 칼리안과 프랑수아(2019)는 "역사적 성문측정법은 불완전한 혈통 분류를 [18]고려했을 때 가계도 모델에 도전하지 않는다"는 데 동의한다.
「 」를 참조해 주세요.
레퍼런스
- ^ Simpson, Michael G (2010-07-19). Plant Systematics. ISBN 9780080922089.
- ^ Kuritzin, A; Kischka, T; Schmitz, J; Churakov, G (2016). "Incomplete Lineage Sorting and Hybridization Statistics for Large-Scale Retroposon Insertion Data". PLOS Computational Biology. 12 (3): e1004812. Bibcode:2016PLSCB..12E4812K. doi:10.1371/journal.pcbi.1004812. PMC 4788455. PMID 26967525.
- ^ Suh, A; Smeds, L; Ellegren, H (2015). "The Dynamics of Incomplete Lineage Sorting across the Ancient Adaptive Radiation of Neoavian Birds". PLOS Biology. 13 (8): e1002224. doi:10.1371/journal.pbio.1002224. PMC 4540587. PMID 26284513.
- ^ Maddison, Wayne P. (1997-09-01). Wiens, John J. (ed.). "Gene Trees in Species Trees". Systematic Biology. Oxford University Press (OUP). 46 (3): 523–536. doi:10.1093/sysbio/46.3.523. ISSN 1076-836X.
- ^ a b c Rogers, Jeffrey; Gibbs, Richard A. (2014-05-01). "Comparative primate genomics: emerging patterns of genome content and dynamics". Nature Reviews Genetics. 15 (5): 347–359. doi:10.1038/nrg3707. PMC 4113315. PMID 24709753.
- ^ Shen, Xing-Xing; Hittinger, Chris Todd; Rokas, Antonis (2017). "Contentious relationships in phylogenomic studies can be driven by a handful of genes". Nature Ecology & Evolution. 1 (5): 126. doi:10.1038/s41559-017-0126. ISSN 2397-334X. PMC 5560076. PMID 28812701.
- ^ Copetti, Dario; Búrquez, Alberto; Bustamante, Enriquena; Charboneau, Joseph L. M.; Childs, Kevin L.; Eguiarte, Luis E.; Lee, Seunghee; Liu, Tiffany L.; McMahon, Michelle M.; Whiteman, Noah K.; Wing, Rod A.; Wojciechowski, Martin F. & Sanderson, Michael J. (2017-11-07). "Extensive gene tree discordance and hemiplasy shaped the genomes of North American columnar cacti". Proceedings of the National Academy of Sciences. 114 (45): 12003–12008. doi:10.1073/pnas.1706367114. PMC 5692538. PMID 29078296.
- ^ a b Futuyma, Douglas J. (2013-07-15). Evolution (3rd ed.). Sunderland, Massachusetts U.S.A. ISBN 9781605351155. OCLC 824532153.
- ^ a b Avise, John C. & Robinson, Terence J. (2008). "Hemiplasy: A New Term in the Lexicon of Phylogenetics". Systematic Biology. 57 (3): 503–507. doi:10.1080/10635150802164587. PMID 18570042.
- ^ Warnow, Tandy; Bayzid, Md Shamsuzzoha; Mirarab, Siavash (2016-05-01). "Evaluating Summary Methods for Multilocus Species Tree Estimation in the Presence of Incomplete Lineage Sorting". Systematic Biology. 65 (3): 366–380. doi:10.1093/sysbio/syu063. ISSN 1063-5157. PMID 25164915.
- ^ a b c Maddison, Wayne P. (1997-09-01). "Gene Trees in Species Trees". Systematic Biology. 46 (3): 523–536. doi:10.1093/sysbio/46.3.523. ISSN 1076-836X.
- ^ a b c Mailund, Thomas; Munch, Kasper; Schierup, Mikkel Heide (2014-11-23). "Lineage Sorting in Apes". Annual Review of Genetics. 48 (1): 519–535. doi:10.1146/annurev-genet-120213-092532. ISSN 0066-4197. PMID 25251849.
- ^ Nichols, Richard (July 2001). "Gene trees and species trees are not the same". Trends in Ecology & Evolution. 16 (7): 358–364. doi:10.1016/s0169-5347(01)02203-0. ISSN 0169-5347. PMID 11403868.
- ^ "Primate Speciation: A Case Study of African Apes Learn Science at Scitable". www.nature.com. Retrieved 2020-05-30.
- ^ Peyrégne, Stéphane; Boyle, Michael James; Dannemann, Michael; Prüfer, Kay (September 2017). "Detecting ancient positive selection in humans using extended lineage sorting". Genome Research. 27 (9): 1563–1572. doi:10.1101/gr.219493.116. ISSN 1088-9051. PMC 5580715. PMID 28720580.
- ^ Leitner, Thomas (May 2019). "Phylogenetics in HIV transmission: taking within-host diversity into account". Current Opinion in HIV and AIDS. 14 (3): 181–187. doi:10.1097/COH.0000000000000536. ISSN 1746-630X. PMC 6449181. PMID 30920395.
- ^ Jacques, Guillaume; List, Johann-Mattis (2019). "Why we need tree models in linguistic reconstruction (and when we should apply them)". Journal of Historical Linguistics. 9 (1): 128–167. doi:10.1075/jhl.17008.mat. hdl:21.11116/0000-0004-4D2E-4. ISSN 2210-2116. S2CID 52220491.
- ^ Kalyan, Siva; François, Alexandre (2019). "When the waves meet the trees". Journal of Historical Linguistics. 9 (1): 168–177. doi:10.1075/jhl.18019.kal. ISSN 2210-2116. S2CID 198707375.
외부 링크
- Venema, D. (2013-08-01). "Evolution Basics: Incomplete Lineage Sorting and Ancestral Population Sizes". BioLogos. Retrieved 29 June 2018.
- Maddison, Wayne P.; Knowles, L. Lacey; Collins, Tim (2006). "Inferring Phylogeny Despite Incomplete Lineage Sorting". Systematic Biology. 55 (1): 21–30. doi:10.1080/10635150500354928. ISSN 1076-836X. PMID 16507521.
- Joly, Simon; McLenachan, Patricia A.; Lockhart, Peter J. (2009). "A Statistical Approach for Distinguishing Hybridization and Incomplete Lineage Sorting". The American Naturalist. 174 (2): E54–E70. doi:10.1086/600082. PMID 19519219. S2CID 205991354.
- Carstens, Bryan C.; Knowles, L. Lacey; Collins, Tim (2007). "Estimating Species Phylogeny from Gene-Tree Probabilities Despite Incomplete Lineage Sorting: An Example from Melanoplus Grasshoppers". Systematic Biology. 56 (3): 400–411. doi:10.1080/10635150701405560. ISSN 1076-836X. PMID 17520504.
- Scornavacca, C.; Galtier, N. (2017). "Incomplete lineage sorting in mammalian phylogenomics". Systematic Biology. 66 (1): 112–120. doi:10.1093/sysbio/syw082. PMID 28173480.