제한 사이트 관련 DNA 마커
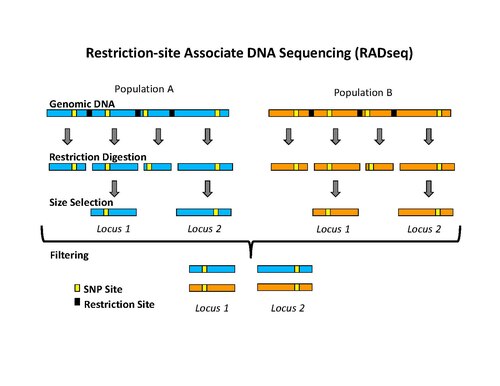
Restriction site associated DNA markers제한 사이트 관련 DNA(RAD) 표지는 연관성 매핑, QTL 매핑, 인구 유전학, 생태 유전학 및 진화 유전학에 유용한 유전자 표지의 일종이다.유전자 매핑에 RAD 마커를 사용하는 것을 RAD 매핑이라고 한다.RAD 마커와 매핑의 중요한 측면은 RAD 태그를 분리하는 과정이다. RAD 태그는 게놈 전체에 걸쳐 제한 효소의 특정 제한 부위의 각 인스턴스 옆에 바로 붙어 있는 DNA 시퀀스다.[1]일단 RAD 태그가 분리되면 주로 단일 뉴클레오티드 폴리모르프리즘(SNP)의 형태로 DNA 시퀀스 다형성을 식별하고 유전자형화하는 데 사용할 수 있다.[1]RAD 태그를 분리하고 분석하여 식별하고 유전형화하는 폴리모형을 RAD 마커라고 한다.null
RAD 태그 격리
각 제한 지점 주변에 측면 DNA 서열을 사용하는 것은 RAD 태그의 중요한 측면이다.[1]게놈에서 RAD 태그의 밀도는 분리 과정에서 사용되는 제한 효소에 따라 달라진다.[2]유전적 다형성 구별을 위해 서로 다른 제한 부위에 의해 발생하는 단편적 길이의 다형성을 사용하는 RFLP나 증폭된 단편적 길이의 다형성(AFP) 등 다른 제한적 사이트 표식기법이 있다.RAD 태그 기법에서 측면 DNA 시퀀스를 사용하는 것을 축소 표현 방법이라고 한다.[3]null
RAD 태그를 분리하기 위한 초기 절차에는 특정 제한 효소로 DNA를 소화하고, 오버행(overhang)에 바이오티닐화 어댑터를 묶고, DNA를 제한 부위 사이의 평균 거리보다 훨씬 작은 조각으로 임의로 깎고, 스트렙타비딘 구슬을 사용하여 바이오티닐화 파편을 분리하는 작업이 포함되었다.[1]이 절차는 초기에 마이크로 어레이 분석을 위해 RAD 태그를 분리하는 데 사용되었다.[1][4][5]최근에는 Illumina 플랫폼에서 고투과 시퀀싱과 함께 사용하도록 RAD 태그 격리 절차가 수정되어 원시 오류 발생률이 크게 감소하고 처리량이 높다는 장점이 있다.[2]새로운 절차에는 특정 제한 효소로 DNA를 소화하는 것(예: SbfI, NsiI,…)이 포함된다. P1이라고 불리는 첫 번째 어댑터를 돌출부에 묶는 것, DNA를 무작위로 깎는 것, 즉 제한 부위 사이의 평균 거리보다 훨씬 작은 조각으로 잘라낸 다음, 깎은 끝을 뭉툭한 끝으로 준비하고 두 번째 어댑터(P)를 묶는 것이 포함된다.2) 및 PCR을 사용하여 두 어댑터가 모두 포함된 조각을 구체적으로 증폭하십시오.중요한 것은 첫 번째 어댑터에 MID(분자 식별자)라고 하는 짧은 DNA 시퀀스 바코드가 들어 있는데, 이 바코드는 함께 풀링되어 동일한 반응으로 배열된 서로 다른 DNA 샘플을 식별하기 위한 표식기로 사용된다.[2][6]RAD 태그를 분석하기 위한 고투과 시퀀싱의 사용은 축소표현 시퀀싱으로 분류될 수 있는데, 여기에는 무엇보다도 RADSeq(RAD-시퀀싱)가 포함된다.[3]null
RAD 마커의 검출 및 유전자형화
RAD 태그가 분리되면 단일 뉴클레오티드 폴리모르프리즘(SNP)과 같은 DNA 시퀀스 다형성을 식별하고 유전자형화하는 데 사용할 수 있다.[1][2]이러한 다형성 부위는 RAD 마커라고 불린다.RAD 태그를 찾는 가장 효율적인 방법은 RAD 태그 시퀀싱, RAD 시퀀싱, RAD-Seq 또는 RADSeq라고 하는 [2][6]고투과 DNA 시퀀싱이다.
고투과 시퀀싱 기술의 개발에 앞서 RAD 태그를 마이크로레이에 혼합해 RAD 마커를 식별했다.[1][4][5]이 접근방식은 미량선의 낮은 민감도로 인해 제한 부위를 교란하고 RAD 태그의 부재로 이어지는 DNA 시퀀스 다형성 또는 RAD 태그 혼성을 교란하는 상당한 DNA 시퀀스 다형성 중 하나만 검출할 수 있다.따라서 미세선으로 달성할 수 있는 유전자 표지자 밀도는 고투과 DNA 시퀀싱으로 가능한 것보다 훨씬 낮다.[7]null
역사
RAD 마커는 처음에는 마이크로레이를 사용하여 구현되었고 나중에 NGS(Next-Generation-Sequencing)에 맞게 조정되었다.[7]2006년경 오리건 대학의 에릭 존슨과 윌리엄 크레스코의 연구소가 공동으로 개발하였다.그들은 D. 멜라노가스터에서 재조합 중단점을 식별하고 세 개의 트리스핀 스틱백에서 QTL을 검출함으로써 RAD 마커의 효용을 확인했다.[1]null
ddRADseq
2012년에는 이중 다이제스트 RADseq(ddr RADseq)라고 하는 수정된 RAD 태그 지정 방법이 제안되었다.[8][9]두 번째 제한 효소를 추가하고, 무작위 피복을 대체하며, 촘촘한 DNA 크기 선택 단계를 통해 저비용 모집단 유전자형을 수행할 수 있다.이것은 선택과 모집단 분화 또는 모집단 적응을 위한 전유전자 스캔에 특히 강력한 도구가 될 수 있다.[9]
원천
- ^ a b c d e f g h Miller MR; Dunham JP; Amores A; Cresko WA; Johnson EA (2007). "Rapid and cost-effective polymorphism identification and genotyping using restriction site associated DNA (RAD) markers". Genome Research. 17 (2): 240–248. doi:10.1101/gr.5681207. PMC 1781356. PMID 17189378.
- ^ a b c d e Baird NA; Etter PD; Atwood TS; Currey MC; Shiver AL; Lewis ZA; Selker EU; Cresko WA; Johnson EA (2008). "Rapid SNP discovery and genetic mapping using sequenced RAD markers". PLOS ONE. 3 (10): e3376. doi:10.1371/journal.pone.0003376. PMC 2557064. PMID 18852878.
- ^ a b Davey JW; Hohenlohe PA; Etter PD; Boone JQ; Catchen JM; Blaxter ML (2011). "Genome-wide genetic marker discovery and genotyping using next-generation sequencing". Nature Reviews Genetics. 12 (7): 499–510. doi:10.1038/nrg3012. PMID 21681211. S2CID 15080731.
- ^ a b Miller MR; Atwood TS; Eames BF; Eberhart JK; Yan YL; Postlethwait JH; Johnson EA (2007). "RAD marker microarrays enable rapid mapping of zebrafish mutations". Genome Biol. 8 (6): R105. doi:10.1186/gb-2007-8-6-r105. PMC 2394753. PMID 17553171.
- ^ a b Lewis ZA; Shiver AL; Stiffler N; Miller MR; Johnson EA; Selker EU (2007). "High density detection of restriction site associated DNA (RAD) markers for rapid mapping of mutated loci in Neurospora". Genetics. 177 (2): 1163–1171. doi:10.1534/genetics.107.078147. PMC 2034621. PMID 17660537.
- ^ a b Hohenlohe PA; Bassham S; Etter PD; Stiffler N; Johnson EA; Cresko WA (2010). "Population genomics of parallel adaptation in threespine stickleback using sequenced RAD tags". PLOS Genetics. 6 (2): e1000862. doi:10.1371/journal.pgen.1000862. PMC 2829049. PMID 20195501.
- ^ a b Shendure J; Ji H (2008). "Next-generation DNA-sequencing". Nature Biotechnology. 26 (10): 1135–1145. doi:10.1038/nbt1486. PMID 18846087. S2CID 6384349.
- ^ Hohenlohe PA; Bassham S; Etter PD; Stiffler N; Johnson EA; Cresko WA (2012). "Population Genomics of Parallel Adaptation in Threespine Stickleback using Sequenced RAD Tags". PLOS ONE. 7 (5): e37135. doi:10.1371/journal.pone.0037135. PMC 3365034. PMID 22675423.
- ^ a b Peterson, Brant K.; Weber, Jesse N.; Kay, Emily H.; Fisher, Heidi S.; Hoekstra, Hopi E. (2012). "Double Digest RADseq: An inexpensive method for de novo SNP discovery and genotyping in model and non-model species". PLOS ONE. 7 (5): e37135. doi:10.1371/journal.pone.0037135. PMC 3365034. PMID 22675423.