키아펫
ChIA-PET페어링-엔드 태그 시퀀싱에 의한 크로마틴 상호작용 분석(ChIA-PET 또는 ChIA-PETS)은 크로마틴 면역복귀화(ChIP) 기반 농축, 크로마틴 근접 레깅, 페어링-엔드 태그, 고투과 시퀀싱 등을 통합해 드 노보 장거리 크로마틴 상호작용 게놈을 결정하는 기법이다.[1]null
유전자는 규제 요소, 절연체, 경계 요소, 전사-인자 결합 사이트(TFBS) 등 추진자와 거리가 먼 지역에 의해 규제될 수 있다.규제 영역과 유전자 코딩 영역 사이의 상호작용을 밝히는 것은 건강과 질병에서 유전자 규제를 지배하는 메커니즘을 이해하는데 필수적이다(Maston et al., 2006).ChIA-PET는 원위부 및 근위부 규제 전사-인자 결합 사이트와 이들이 상호작용하는 유전자의 촉진자 사이의 고유한 기능적 염색질 상호작용을 식별하는 데 사용될 수 있다.null
또한 ChIA-PET는 세포 분화, 증식, 개발 등의 과정 중에 게놈 제어의 메커니즘을 푸는 데 사용될 수 있다.DNA 결합 규제 단백질과 촉진자 지역에 대한 ChIA-PET 인터랙티브 맵을 생성함으로써 치료 개입의 고유한 대상을 더 잘 식별할 수 있다(Fullwood & Yijun, 2009).null
방법론
ChIA-PET 방법은 ChIP 기반 방법과 [2]염색체 순응 캡처(3C) 기반 방법을 결합해 두 가지 접근법의 기능을 확장한다.[3]Chip-Sequencing(ChIP-Seq)은 TFBS를 식별하는 데 사용되는 일반적인 방법이며, 3C는 장거리 염색질 상호작용을 식별하는 데 사용되어 왔다.독립적으로, 두 가지 모두 디-노보 장거리 상호작용 게놈을 광범위하게 식별하는 데 한계가 있다.Chip-Seq는 TFBS 게놈전체를 식별할 수 있는 반면 염색체를 따라 단백질 결합 부위의 선형 정보만 제공하며(그들 간의 상호작용이 아님), 유전학적 배경 잡음(허위 양성)이 심해질 수 있다.[4][5]3C는 비선형, 장거리 염색질 상호작용을 분석할 수 있지만 게놈을 넓게 사용할 수 없고, 치프-젝과 마찬가지로 높은 수준의 배경 잡음에 시달린다.상호작용 영역 사이의 거리(최대 100kb)와 관련하여 소음이 증가하기 때문에, 염색질 상호작용의 정확한 특성화를 위해 힘들고 지루한 통제가 필요하다.[6]로키 고유의 상호작용 프로파일링 방식인 3C와는 달리, Hi-C와 같은 대체 방법이 확립되어 상호작용 게놈을 광범위하게 프로파일링하고 있다.[7]TFBS 및 장거리 상호작용에 대한 전체 게놈 프로파일링 방법에도 불구하고, ChIA-PET 방법과 접근법을 결합하면 관심 단백질이 결합되는 게놈 영역뿐만 아니라 그것이 상호작용하는 게놈 영역을 식별할 수 있다.[8][9]null
ChIA-PET 방법은 Chip 파편을 소닉화하여 Chip-Seq에서 발견된 비특정 상호작용 소음 문제를 성공적으로 해결하여 무작위 부착물을 특정 상호작용 콤플렉스에서 분리한다.농축이라고 하는 다음 단계는 게놈 전체 분석의 복잡성을 줄이고 미리 정해진 TF(전송 인자)로 묶인 염색질 상호작용에 특수성을 더한다.장거리 교호작용을 식별하는 3C 접근법의 능력은 근접 레인지 이론에 기초한다.DNA 상호접합에 대해서는 묽은 조건에서 공통 단백질 복합체에 의해 묶여 있는 파편이 용액에서 자유롭게 확산되거나 서로 다른 복합체에 고정되어 있는 파편보다 운동적 이점이 더 크다.ChIA-PET는 단백질 복합체에 연결된 DNA 조각의 자유 끝에 링크 시퀀스를 통합함으로써 이 개념을 활용한다.규제 단지에 의해 묶여진 파편들의 연결을 구축하기 위해, 링커 시퀀스는 핵 근접 레깅 중에 연결된다.따라서 링커 연결 레깅의 제품은 초고처리 PET 시퀀싱으로 분석하여 참조 게놈에 매핑할 수 있다.치아-PET는 3C와 4C처럼 특정 검출 부위에 의존하지 않기 때문에 염색질 상호작용의 편중, 게놈 폭의 디노보 검출이 가능하다.[8]Hi-C와 비교했을 때, 항체 풀다운을 사용하면 시퀀싱된 파편의 수를 관심 단백질에 의해 결합된 염색체 상호작용으로 제한하여 데이터 분석을 용이하게 할 수 있다.null
워크플로우
워크플로의 습식 랩 부분:
- 포름알데히드는 DNA-단백질 복합체를 교차 연결하는 데 사용된다.소닉은 염색질을 분해하고 비특정 교호작용을 감소시키는 데 사용된다.
- 특정한 선택항체는 관심 단백질 결합 염색체 조각을 풍부하게 하는데 사용된다.항체에 의해 결합되는 ChIP 물질은 ChIA-PET를 구성하는 데 사용된다.
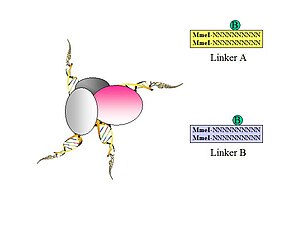
- 그림 1.옆구리 MeI 사이트를 포함하는 바이오티닐화 올리고뉴클레오티드 하프링커를 사용하여 근접 결합 DNA 조각을 연결한다.두 개의 서로 다른 링커는 두 개의 링커 시퀀스 각각에 대해 특정 뉴클레오티드 바코드(CG 또는 AT)를 사용하여 설계된다(그림 5에 설명한 키메라 레깅 제품을 식별할 수 있다).
- 그림 2.연결기는 테더링된 DNA 조각에 묶여 있다.
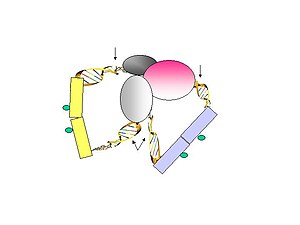
- 그림 3.링커 파편들은 묽은 조건에서 Chip 비드에 묶인다.그런 다음 정제된 DNA는 MeI에 의해 소화되는데, MeI는 인식 사이트에서 멀리 떨어져서 태그링커 태그 구조를 해제한다.
- 그림 4.그런 다음, 바이오티닐이 첨가된 PET는 스트렙타비딘 컨쥬얼 마그네틱 비드에 고정된다.
- 그림 5. AA (CG/CG)와 BB (AT/AT) 링커 바코드 조성이 있는 PET 시퀀스는 복합 내 결찰 제품으로 간주되는 반면, AB (CG/AT) 링커 조성이 있는 PET 시퀀스는 서로 다른 염색체 복합체에서 경계가 되는 DNA 조각들 사이의 키메릭 결찰 제품에서 도출되는 것으로 간주된다.
워크플로의 드라이랩 부분:
PET 추출, 매핑 및 통계 분석
분석.
PET 태그를 추출하여 실리코에서 참조 인간 게놈에 매핑한다.null
ChIP 농축 피크의 식별(베인딩 사이트)
자체 부여된 PET는 참조 게놈에 대한 가장 신뢰할 수 있는 매핑(20 + 20비트/s)을 제공하기 때문에 ChIP 농축 사이트를 식별하는 데 사용된다.null
ChIP 농축 피크 탐색 알고리즘
PET가 겹치는 경우 peak라 불리는 것을 바인딩 사이트로 간주한다.FDR(False Discovery Rate)은 통계 시뮬레이션을 통해 PET에서 파생된 가상 DNA의 무작위 배경과 추정 배경 노이즈를 추정해 결정한다.null
반복 DNA의 필터링(비특정 결합에 영향을 미침)
구조적 편차가 심한 지역에 존재하는 위성 지역 및 바인딩 현장은 제거된다.null
ChIP 농축수
각 사이트마다 자기조율 PET와 상호조율 PET의 수(+250bp 창 이내)가 보고된다.특정 사이트에서 자체 자격 부여 및 상호 자격 부여된 PET의 총 개수를 Chip 농축 카운트라고 한다.null
그림 6. PET 분류:고유하게 정렬된 PET 시퀀스는 한 개의 DNA 조각에서 파생된 것인지 두 개의 DNA 조각에서 파생된 것인지에 따라 분류할 수 있다.
- 자가용 PET
PET의 두 태그가 Chip DNA 파편 범위(3Kb 미만)의 게놈 스팬과 동일한 염색체에 매핑되어 있을 경우, 예상되는 자기동작 방향과 같은 가닥에 있는 경우, 하나의 Chip DNA 파편의 자기동작에서 파생된 것으로 간주되며, 자기동작 PET로 간주된다.null
- 인터릴레이션 PET
만약 PET가 이러한 기준에 맞지 않는다면, PET는 두 개의 DNA 조각 사이에 있는 레깅 제품에서 비롯되었을 가능성이 높고 인터레이징 PET라고 한다.인터랙션 PET의 두 태그는 고정 태그 방향이 없고, 같은 가닥에서 발견되지 않을 수 있으며, 유전학적 스팬이 있을 수 있으며, 동일한 염색체에 매핑되지 않을 수 있다.null
- 세포내 상호 작용 PET
인터랙션 PET의 두 태그가 동일한 염색체에서 매핑되지만 어떤 방향으로든 스팬이 3Kb 미만인 경우, 이러한 PET를 인터랙션 PET라고 한다.null
- 색조간 상호 작용 PET
서로 다른 염색체에 매핑된 PET를 색간 교배 PET라고 한다.null
그림 7. 원위적 규제 요소가 표적 유전자의 촉진 영역을 포함하는 장거리 염색질 상호작용을 어떻게 시작할 수 있는지를 보여주는 제안 메커니즘
이 상호작용은 앵커링 센터에서 복수의 TFBS로 DNA 루프 구조를 형성한다.작은 루프는 고정 센터 근처의 유전자를 촘촘한 서브 컴파트먼트로 포장할 수 있으며, 이것은 강화된 전사 활성화를 위한 규제 단백질의 국소 농도를 증가시킬 수 있다.이 메커니즘은 또한 전사 효율을 높여 RNA pol II가 촘촘한 원형 유전자 템플릿을 순환시킬 수 있게 할 수 있다.큰 상호작용 루프는 조정된 규제를 위해 앵커 사이트 근처에 있는 루프의 양쪽 끝에서 먼 유전자를 연결하거나 긴 루프에서 유전자를 분리하여 그 활성화를 방지할 수 있다.풀우드 등으로부터 개조됨.(2009).null
장단점
ChIA-PET 방식의 장점
- ChIA-PET는 장거리 염색체 상호작용 분석을 위한 편향되지 않은, 전유전자 및 디노보 접근방식이 될 가능성이 있다(Fullwood & Yijun, 2009).
- ChIA-PET 실험은 두 가지 글로벌 데이터셋을 제공할 수 있다.단백질 인자 결합 부위(자율 PET) 및 결합 부위(자율 PET) 간의 상호 작용.
- ChIA-PET는 Genome-wide 분석의 복잡성을 줄이기 위해 Chip을 포함하며 특정 관심 인자에 의해 결합된 염색체 상호작용에 특수성을 추가한다.
- ChIA-PET는 Roche 454 파이로시퀀싱, Illumina GA, ABI SOLiD, 헬리코스와 같은 태그 기반 차세대 시퀀싱 접근법과 호환된다.
- ChIA-PET는 전사적 조절 또는 염색질 구조 순응과 관련된 많은 다른 단백질 인자에 적용된다.
- ChIA-PET 분석은 특정 핵 과정에 포함된 염색체 상호작용에 적용할 수 있다.RNA 중합효소 II와 같은 일반 TF를 사용하면 전사 규제에 관련된 모든 염색질 상호작용을 식별할 수 있을 것이다.또한 DNA 복제 또는 염색질 구조에 관련된 단백질 인자를 사용하면 DNA 복제 및 염색질 구조 변경으로 인한 모든 상호작용을 식별할 수 있다(Fullwood et al., 2009).
약점
- cis와 trans-regulation 콤플렉스는 세포와 조직의 특정 조건에 근거한 고유한 단백질 조합을 포함하고 있다는 것이 잘 확립되어 있다(Dekker et al., 2006).단일 기능 TFBS의 식별은 상당한 진전이지만, 복합체에서 개별 단백질을 식별하기 위해 ChIA-PET를 사용하는 것은 각각의 상호 작용하는 단백질을 식별하기 위한 추측 작업과 여러 실험이 필요할 것이다.이것은 비용이 많이 들고 시간이 많이 드는 노력이 될 것이다.
- ChIA-PET는 사용되는 항체의 품질, 순도 및 특수성에 의해 제한된다(Fullwood et al., 2009).
- ChIA-PET는 기준 시퀀스(ref)에 매핑할 수 있는 시퀀스의 식별에 의존한다.
- ChIA-PET는 PET 읽기를 참조 게놈에 구성하고 매핑하기 위해 피크콜링 컴퓨터 알고리즘을 사용할 것을 요구한다.소프트웨어 플랫폼 간의 차이 때문에 어떤 프로그램을 사용하느냐에 따라 결과가 달라질 수 있다.
- 반복 DNA 영역은 유전자 조절과 연관될 수 있지만(폴락&도마니, 2006), 데이터에 영향을 미칠 수 있으므로 제거해야 한다(풀우드 외, 2009).
- 두 곳의 농축은 동시에 주변 지역에 대한 편견을 불러일으킨다.[10]
역사
풀우드 외(2009) ChIA-PET를 사용하여 인간 암세포에서 에스트로겐 수용체 알파(ER-alpha)에 의해 매개되는 염색질 상호작용 네트워크를 검출하고 지도화했다.그 결과로 나온 글로벌 염색체 상호작용 맵은 원격 ER-알파 바인딩 사이트들이 또한 광범위한 염색체 루프에 의한 ER-알파 함수를 제안하는 장거리 염색체 상호작용을 통해 유전자 촉진기에 고정되었다는 것을 밝혀냈다.null
분석 및 소프트웨어
| 소프트웨어 | 설명 |
|---|---|
| C3PET | ChIA-PET 데이터 처리를 위한 소프트웨어 제품군.비모수적 베이지안 접근방식을 사용하여 단백질 복합체를 상호 작용하는 크로마틴을 예측한다.[1] [2] |
| ChIA-PET 공구 | ChIA-PET 데이터 처리를 위한 소프트웨어 제품군.[3] [4] |
| 키아-PET2 | ChIA-PET 데이터 처리를 위한 소프트웨어 제품군.다양한 프로토콜의 데이터를 지원하고 데이터 분석의 품질 관리 제공.[5] [6] |
| 치아식 | NCHG 모델을 사용하여 ChIA-PET 데이터를 처리하기 위한 소프트웨어 제품군.ChIA-Sig 웹 사이트 [7] [8] |
| 이랜드 | 지도 ChIP는 기준 인간 게놈에 DNA 조각을 농축했다.[9] |
| 망고 | ChIA-PET 데이터 처리를 위한 소프트웨어 제품군.ChIA-PET 데이터셋 처리에 필요한 모든 단계를 완료하고 상호작용을 위한 통계적 신뢰도 추정치를 제공한다.[10] [11] |
| 몬테카를로 시뮬레이션 | 잘못된 발견 비율을 추정하는 데 사용됨.[11]null |
| 게노믹인터액션 | ChIA-PET 또는 Hi-C 데이터를 처리하기 위한 R 패키지.[12] [13] |
| 주다 | ChIA-PET 또는 Hi-C 데이터와 호환되는 사용자 정의 게놈 브라우저를 만들기 위한 프로그래밍 라이브러리 [14] [15] |
| RepeatMasker | 반복적인 요소의 실리코 마스킹.[16] |
대안
Chip(Chromatin 면역소모):
오리지널 Chip 방법은 염색체 상태와 유전자 전사에 관한 정보를 제공하기 위해 단백질을 선별적으로 식별하고 결합하는 항체 기반 기술이다.[17]
게놈 아키텍처 매핑(GAM):
이 기법은 3C 기반 기법과 관련된 여러 가지 단점을 제거하는데, 어떤 게놈 로키 사이의 3차원 근위부를 수집한다.[12]null
태그 확장에 의한 분할-풀 상호작용 인식(SPRITE)
SPRITE는 게놈 전체에 걸쳐 핵의 고차 상호작용 지도를 만드는 기법이다.이 접근방식은 더 큰 공간 거리에 걸쳐 발생하는 상호작용을 탐지하고 동시에 발생하는 수많은 RNA와 DNA 상호작용을 게놈 전체로 탐지할 수 있게 한다.[13]null
ChIA-Drop
ChIA-Drop은 드롭릿 기반과 바코드 연결 시퀀싱을 사용하여 단일 분자 정확도로 멀티플렉스 크로마틴 상호작용을 분석할 수 있는 간단한 방법이다.Hi-C와 ChIA-PET와 같은 이전의 쌍방향 인구 수준 접근법은 이 기술과 구별된다.[14][15]null
참조
- ^ Fullwood, Melissa J.; Ruan, Yijun (2009-05-01). "ChIP-based methods for the identification of long-range chromatin interactions". Journal of Cellular Biochemistry. 107 (1): 30–39. doi:10.1002/jcb.22116. ISSN 1097-4644. PMC 2748757. PMID 19247990.
- ^ Kuo, Min-Hao; Allis, C.David (November 1999). "In Vivo Cross-Linking and Immunoprecipitation for Studying Dynamic Protein:DNA Associations in a Chromatin Environment". Methods. 19 (3): 425–433. doi:10.1006/meth.1999.0879.
- ^ Dekker, Job; Rippe, Karsten; Dekker, Martijn; Kleckner, Nancy (2002-02-15). "Capturing Chromosome Conformation". Science. 295 (5558): 1306–1311. doi:10.1126/science.1067799. ISSN 0036-8075.
- ^ Barski, Artem; Cuddapah, Suresh; Cui, Kairong; Roh, Tae-Young; Schones, Dustin E.; Wang, Zhibin; Wei, Gang; Chepelev, Iouri; Zhao, Keji (May 2007). "High-Resolution Profiling of Histone Methylations in the Human Genome". Cell. 129 (4): 823–837. doi:10.1016/j.cell.2007.05.009.
- ^ Wei, Chia-Lin; Wu, Qiang; Vega, Vinsensius B.; Chiu, Kuo Ping; Ng, Patrick; Zhang, Tao; Shahab, Atif; Yong, How Choong; Fu, YuTao; Weng, Zhiping; Liu, JianJun (January 2006). "A Global Map of p53 Transcription-Factor Binding Sites in the Human Genome". Cell. 124 (1): 207–219. doi:10.1016/j.cell.2005.10.043.
- ^ Dekker, Job (January 2006). "The three 'C' s of chromosome conformation capture: controls, controls, controls". Nature Methods. 3 (1): 17–21. doi:10.1038/nmeth823. ISSN 1548-7091.
- ^ Lieberman-Aiden, Erez; van Berkum, Nynke L.; Williams, Louise; Imakaev, Maxim; Ragoczy, Tobias; Telling, Agnes; Amit, Ido; Lajoie, Bryan R.; Sabo, Peter J.; Dorschner, Michael O.; Sandstrom, Richard (2009-10-09). "Comprehensive Mapping of Long-Range Interactions Reveals Folding Principles of the Human Genome". Science. 326 (5950): 289–293. doi:10.1126/science.1181369. ISSN 0036-8075. PMC 2858594. PMID 19815776.
- ^ a b Fullwood, Melissa J.; Liu, Mei Hui; Pan, You Fu; Liu, Jun; Xu, Han; Mohamed, Yusoff Bin; Orlov, Yuriy L.; Velkov, Stoyan; Ho, Andrea; Mei, Poh Huay; Chew, Elaine G. Y. (November 2009). "An oestrogen-receptor-α-bound human chromatin interactome". Nature. 462 (7269): 58–64. doi:10.1038/nature08497. ISSN 0028-0836. PMC 2774924. PMID 19890323.
- ^ Li, Guoliang; Ruan, Xiaoan; Auerbach, Raymond K.; Sandhu, Kuljeet Singh; Zheng, Meizhen; Wang, Ping; Poh, Huay Mei; Goh, Yufen; Lim, Joanne; Zhang, Jingyao; Sim, Hui Shan (2012-01-20). "Extensive Promoter-Centered Chromatin Interactions Provide a Topological Basis for Transcription Regulation". Cell. 148 (1): 84–98. doi:10.1016/j.cell.2011.12.014. ISSN 0092-8674. PMC 3339270. PMID 22265404.
- ^ Downes, Damien J.; Beagrie, Robert A.; Gosden, Matthew E.; Telenius, Jelena; Carpenter, Stephanie J.; Nussbaum, Lea; De Ornellas, Sara; Sergeant, Martin; Eijsbouts, Chris Q.; Schwessinger, Ron; Kerry, Jon (2021-01-22). "High-resolution targeted 3C interrogation of cis-regulatory element organization at genome-wide scale". Nature Communications. 12 (1): 531. doi:10.1038/s41467-020-20809-6. ISSN 2041-1723. PMC 7822813.
- ^ 몬테카를로법
- ^ Beagrie, Robert A.; Scialdone, Antonio; Schueler, Markus; Kraemer, Dorothee C. A.; Chotalia, Mita; Xie, Sheila Q.; Barbieri, Mariano; de Santiago, Inês; Lavitas, Liron-Mark; Branco, Miguel R.; Fraser, James (March 2017). "Complex multi-enhancer contacts captured by genome architecture mapping". Nature. 543 (7646): 519–524. doi:10.1038/nature21411. ISSN 1476-4687.
- ^ Quinodoz, Sofia A.; Ollikainen, Noah; Tabak, Barbara; Palla, Ali; Schmidt, Jan Marten; Detmar, Elizabeth; Lai, Mason M.; Shishkin, Alexander A.; Bhat, Prashant; Takei, Yodai; Trinh, Vickie (2018-07-26). "Higher-Order Inter-chromosomal Hubs Shape 3D Genome Organization in the Nucleus". Cell. 174 (3): 744–757.e24. doi:10.1016/j.cell.2018.05.024. ISSN 0092-8674. PMID 29887377.
- ^ Zheng, Meizhen; Tian, Simon Zhongyuan; Capurso, Daniel; Kim, Minji; Maurya, Rahul; Lee, Byoungkoo; Piecuch, Emaly; Gong, Liang; Zhu, Jacqueline Jufen; Li, Zhihui; Wong, Chee Hong (February 2019). "Multiplex chromatin interactions with single-molecule precision". Nature. 566 (7745): 558–562. doi:10.1038/s41586-019-0949-1. ISSN 1476-4687. PMC 7001875.
- ^ Koch, Linda (April 2019). "Getting the drop on chromatin interaction". Nature Reviews Genetics. 20 (4): 192–193. doi:10.1038/s41576-019-0103-9. ISSN 1471-0064.
- 바스키 외, (2007)인간 게놈의 히스톤 메틸화에 대한 고해상도 프로파일링.세포 (129); 823–37.
- 데커, (2002년)염색체 일치 캡처.과학(295); 1306–1311.
- 데커,(2006년).염색체 순응 포획의 세 가지 'C': 제어, 제어, 제어.Nat. 방법 (3); 17–21.
- 풀우드 외, (2009년).에스트로겐-수용체-α 결합 인간 염색질이 상호작용한다.자연. (462); 58–64.
- 풀우드&이쥔(2009년).장거리 염색질 상호작용 식별을 위한 ChIP 기반 방법.J 세포 생화학. 107(1); 30–39.
- 존슨 외, (2007)체내 단백질-DNA 상호작용의 게놈 범위 매핑.과학. (316), 1497–502.
- 궈앤알리스(1999년).동적 단백질을 연구하기 위한 in-vivo 교차 링크와 면역복구: 염색질 환경의 DNA 연관성.방법. (19); 425–33.
- Li, G., Fullwood, M.J., Xu, H., Mulawadi, F.H., Velkov, S., Vega, V., Ariyaratne, P.N., Mohamed, Y.B., Ooi, H.S., Tennakoon, C., Wei, C.L., Ruan, Y. and Sung, W.K. ChIA-PET tool for comprehensive chromatin interaction analysis with paired-end tag sequencing.게놈 비올, 11(2)R22.
- 마스턴 외, (2006년).인간 게놈의 전사적 규제 요소.안누 레프: 게노믹스.험 지네 (7); 29–59.
- 폴락&도마니,(2006년).알루 요소는 전사 인자에 대한 많은 결합 부지를 포함하고 있으며 개발 프로세스 규제에 역할을 할 수 있다.BMC Genomics. (7); 133.
- Wei 등, (2006년).인간 게놈에 있는 p53 전사-인자 결합 사이트의 글로벌 맵.세포(124개), 207–19.
외부 링크
- ChIA-PET 게놈 브라우저 - 이 브라우저는 풀우드 등으로부터 데이터를 보기 위한 것이다.(2009), 전체 게놈 지형과 함께 바인딩 사이트 및 상호작용의 거시적 그림을 제공하는 사용자 정의 전체 게놈 상호작용 뷰어를 포함한다.