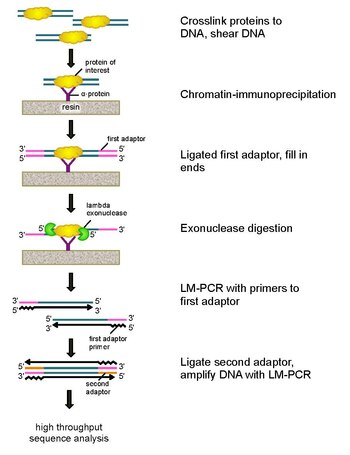
ChIP-exo
ChIP-exoChIP-exo는 관심 단백질(전사인자)이 게놈에 결합하는 위치를 매핑하기 위한 염색질 면역침강 기반의 방법이다.ChIP-seq 프로토콜을 수정하여 바인딩 사이트의 해상도를 수백 개의 기본 쌍에서 거의 하나의 기본 쌍으로 개선합니다.단백질 결합 DNA의 가닥을 단백질 결합 부위의 소수의 뉴클레오티드 내에서 5'-3' 방향으로 분해하기 위해 엑소뉴클라아제 사용을 사용한다.엑소핵산가수분해효소 처리 말단의 뉴클레오티드는 DNA 배열, 마이크로어레이 및 PCR의 조합을 사용하여 결정된다.이 배열들은 단백질이 결합하는 게놈의 위치를 확인하기 위해 게놈에 매핑된다.
이론.
염색질 면역침강(ChIP) 기술은 단백질-DNA 상호작용을 검출하기 위해 1984년부터[1] 사용되어 왔다.CHIP는 결과의 품질을 개선하기 위해 많은 변형이 있었다.이러한 개선점 중 하나인 ChIP-on-chip(ChIP-chip)은 ChIP와 마이크로 어레이 기술을 결합한 것입니다.이 기술은 특히 핵 구획에 존재하는 수천 개의 단백질에 의해 마이크로어레이가 제약되어 높은 [2]오검출률을 초래하는 생체 내 감도와 특이성이 제한된다.다음으로 ChIP 시퀀싱(ChIP-seq)을 도입했습니다.ChIP는 높은 스루풋 [3]시퀀싱과 결합되어 있습니다.그러나 전단 DNA 조각의 이종 특성은 결합 부위를 ±300 염기쌍 이내로 매핑하여 특이성을 제한한다.둘째, DNA를 오염시키는 것은 매우 적은 수의 유전자가 관심 단백질과 가교되어 비특이적인 유전체 DNA가 배경 [4]잡음의 중요한 원천이 되기 때문에 심각한 문제를 일으킨다.
이 문제를 해결하기 위해, 이승만과 푸는 Chip-exo를 [5]개발하기 위해 전형적인 핵산 분해효소 보호 분석을 수정했다.이 새로운 ChIP 기술은 람다 엑소핵산가수분해효소에 의존하며, 이는 5µ-3µ 방향으로 모든 결합 이중 가닥 DNA를 분해한다.간단히 말해서, 관심 단백질(에피토프 태그를 가진 공학적으로 면역 침강에 유용할 수 있음)은 포름알데히드를 사용하여 게놈을 통해 유전체의 자연 결합 위치와 생체 내에서 가교된다.그런 다음 세포를 채취하고, 분해하여 열고, 초음파 처리를 통해 염색질을 절단하고 가용화한다.다음으로 해당 단백질을 가교된 DNA와 함께 면역침투하기 위해 항체를 사용한다.그 후 DNA PCR 어댑터는 말단에 결합되며, 이는 엑소뉴클레아제 소화 후 제2의 가닥 DNA 합성을 위한 프라이밍 포인트 역할을 한다.람다핵산가수분해효소는 단백질-DNA 공유 상호작용의 경계에서 소화가 차단될 때까지 5' 말단에서 이중 DNA 가닥을 소화한다.대부분의 오염 DNA는 두 번째 단일 가닥 특이핵산가수분해효소의 첨가에 의해 분해된다.가교 반전 후 PCR 어댑터에 대한 프라이머를 연장하여 이중가닥 DNA를 형성하고, 두 번째 어댑터를 5'단에 결속하여 엑소뉴클레아제 소화중지의 정확한 위치를 특정한다.라이브러리는 PCR에 의해 증폭되고 제품은 높은 throughput 시퀀싱으로 식별됩니다.이 방법은 ChIP 칩이나 ChIP-seq보다 훨씬 높은 분해능인 게놈 내의 단백질 결합 부위에 대해 단일 염기쌍까지 분해할 수 있습니다.
이점
ChIP-exo는 단백질 결합 위치를 식별하는 데 있어 단일 염기쌍 분해능을 제공하는 것으로 나타났다.이는 단백질의 결합 [4]부위를 ±300 염기쌍으로만 찾을 수 있는 ChIP-seq와는 대조적이다.
비단백질 결합 DNA 조각의 오염은 ChIP 실험에서 높은 비율의 잘못된 양성 및 음성 결과를 초래할 수 있습니다.프로세스에 엑소핵산가수분해효소를 추가하면 결합부위 호출의 분해능이 향상될 뿐만 아니라 염기서열 분석 [4]전에 용액에서 오염 DNA를 제거할 수 있습니다.
뉴클레오티드 단편에 비효율적으로 결합되어 있는 단백질은 ChIP-exo에 의해 검출될 가능성이 높다.이를 통해 예를 들어 이전에 [5]발견된 것보다 더 많은 CTCF 전사 팩터바인딩 사이트를 인식할 수 있게 되었습니다.
ChIP-exo를 [4]사용할 경우 고해상도와 축소된 백그라운드로 인해 시퀀싱 커버리지의 깊이가 줄어듭니다.
제한 사항
단백질-DNA 복합체가 단일 결합 사건 내에서 여러 개의 교차 연결 위치를 갖는 경우, 여러 개의 별개의 결합 사건이 있는 것처럼 보일 수 있다.이는 이러한 단백질이 변성되어 동일한 사건 내에서 사용 가능한 결합 부위 중 하나에서 가교된 결과일 수 있다.엑소핵산가수분해효소는 단백질이 어떤 부위와 [5]가교되어 있는지에 따라 결합된 부위 중 하나에서 멈춥니다.
ChIP 기반 방법과 마찬가지로, 이 기술을 사용하기 위해서는 해당 단백질에 적합한 항체를 사용할 수 있어야 합니다.
적용들
리와 푸는 효모의 Reb1, Gal4, Phd1, Rap1, 인간의 CTCF의 작은 전사 인자를 분석함으로써 Chip-exo를 도입했다.Reb1 사이트는 종종 클러스터에서 발견되었으며, 이러한 클러스터는 예상보다 최대 10배 높은 점유율을 보였다.클러스터의 보조 사이트가 주 바인딩 사이트에서 최대 40bp까지 발견되었습니다.Gal4의 결합 모티브는 4개의 뉴클레오티드 중 3개에 대한 강한 선호도를 보여 Gal4와 제외된 뉴클레오티드 사이의 음의 상호작용을 시사했다.Phd1은 3개의 다른 모티브를 인식하여 Phd1의 바인딩 모티브의 모호성에 대한 이전 보고서를 설명합니다.Rap1은 네 가지 모티브를 인식하는 것으로 밝혀졌다.이 단백질에 의해 결합된 리보솜 단백질 유전자는 보다 강한 합의 서열을 가진 특정 모티브를 사용하는 경향이 있었다.다른 유전자는 유사한 점유율을 달성하기 위해 종종 더 약한 합의 모티브의 클러스터를 사용했다.CTCF의 결합 모티브는 4개의 "모듈"을 사용했다.바인드된 CTCF 사이트의 절반은 모듈1과 모듈2를 사용하고 나머지 절반은 모듈1과 모듈2를 사용하고 있습니다.CTCF는 이러한 모듈의 다른 조합을 인식하기 [5]위해 아연 손가락을 사용하는 것으로 알려져 있습니다.
이 교수와 푸 교수는 사카로미세스 게놈의 사전개시복합체(PIC) 구조와 구조를 분석했다.ChIP-exo를 사용하여 TATA가 [6]없는 것으로 알려진 프로모터에서 TATA와 유사한 기능을 정확하게 식별할 수 있었다.
「 」를 참조해 주세요.
레퍼런스
- ^ Gilmour, DS; JT Lis (1983). "Detecting protein-DNA interactions in vivo: Distribution of RNA polymerase on specific bacterial genes". Proceedings of the National Academy of Sciences. 81 (14): 4275–4279. doi:10.1073/pnas.81.14.4275. PMC 345570. PMID 6379641.
- ^ Albert, I; TN Mavrich; LP Tomsho; J Qi; SJ Zanton; SC Schuster; BF Pugh (2007). "Translational and rotational settings of H2A.Z nucleosomes cross the Saccharomyces cerevisiae genome". Nature. 446 (7135): 572–576. Bibcode:2007Natur.446..572A. doi:10.1038/nature05632. PMID 17392789. S2CID 4416890.
- ^ Ren, B; F Robert; JJ Wyrick; O Aparicio; EG Jennings; I Simon; J Zeitlinger; J Schreiber; N Hannett; E Kan; et al. (2000). "Genome-wide location and function of DNA binding proteins". Science. 290 (5500): 2306–2309. Bibcode:2000Sci...290.2306R. CiteSeerX 10.1.1.123.6772. doi:10.1126/science.290.5500.2306. PMID 11125145.
- ^ a b c d Pugh, Benjamin. "Methods, Systems and Kits for Detecting Protein-Nucleic Acid Interactions". United States Application Publication. United States Patents. Retrieved 17 February 2012.
- ^ a b c d Rhee, Ho Sung; BJ Pugh (2011). "Comprehensive Genome-wide Protein-DNA Interactions Detected at Single-Nucleotide Resolution". Cell. 147 (6): 1408–1419. doi:10.1016/j.cell.2011.11.013. PMC 3243364. PMID 22153082.
- ^ Rhee, Ho Sung; BJ Pugh (2012). "Genome-wide structure and organization of eukaryotic pre-initiation complexes". Nature. 483 (7389): 295–301. Bibcode:2012Natur.483..295R. doi:10.1038/nature10799. PMC 3306527. PMID 22258509.