아열대
Subtelomere이 글은 검증을 위해 인용구가 추가로 필요하다. – · · · · (2016년 4월 (이 |
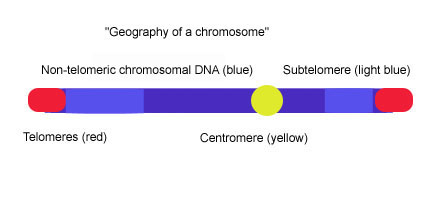
아열대(subelomeres)는 텔로메릭 캡과 염색질 사이의 DNA 부분이다.
구조
텔로미어는 특화된 단백질이다.DNA는 진핵 염색체의 끝부분에 존재하는데, 이것은 그들이 분해되는 것을 막고 끝에서 끝까지 염색체를 융합하는 것을 방지한다. 대부분의 척추동물 말단체 DNA는 가변 길이의 긴 (TTAGGG)n 반복측정(흔히 약 3-20kb)으로 구성된다. 아열대(subelomeres)는 텔로메릭 캡과 염색질 사이의 DNA 부분이다. 각 염색체는 긴(TTAGGG)n 반복에 바로 인접한 두 개의 하위 골격을 가지고 있다. 아열대는 염색체에 있는 고유 DNA의 원위부(중심부로부터 가장 빠른)로 간주되며, 다색체 염기서열 블록의 특이하게 역동적이고 가변적인 모자이크다. 인간, 플라스모디움 팔시파룸, 드로소필라 멜라노가라스터, 사카로메시스 세레비시아아 등 다양한 종의 아열대들은 여러 가지 반복되는 원소로 이루어져 있다는 점에서 구조적으로 유사하지만, 아열대의 범위와 원소의 순서는 유기체마다 크게 다르다.[1] 효모(S. serebisiae)에서 하위종류는 근위부와 원위부(telomeric) 도메인의 두 개의 영역으로 구성된다. 두 영역은 다른 염색체 종단과의 동질학 내용 및 범위가 다르며, 퇴화된 텔로미어 반복(TTAGGG)과 '핵심 X'라는 원소에 의해 분리되는 경우가 많은데, 이 원소는 모든 염색체 종단에서 발견되며 자율적으로 복제되는 염기서열(ARS)과 ABF1 결합 사이트를 포함하고 있다.[2][3] 근위부 영역은 가변 색간 복제(<1-30kb)로 구성된다. 이 영역은 Pho, Mel, Mal과 같은 유전자를 포함할 수 있다.[4] 원위 영역은 보존도가 높은 Y' 원소의 0-4 탠덤 복사본으로 구성된다. Y; 원소의 수와 염색체 분포는 효모 균주에 따라 다르다.[5] 코어 X와 Y' 요소 또는 코어 X와 TTAGGG 순서 사이에는 종종 척추동물 텔로메릭 모티브 TTAGGG의 여러 복사본으로 구성된 4개의 하위 골격 반복 요소(STR: STR-A, STR-B, STR-C, STR-D) 세트가 있다.[6] 이 2-영역 구조는 20p, 4q, 18p의 인간 염색체에서 근위부 및 원위부 하위영역 영역이 퇴화 TTAGGG의 스트레칭에 의해 분리되는 하위영역 구조와 현저히 유사하지만, 다른 인간 염색체의 하위영역 연구에서 나타나는 그림은 2-영역 모델 doe를 나타낸다.s는 보편적으로 적용되지 않는다.[1]
특성.
반복된 시퀀스를 가진 이 구조는 결합 다양성의 기원에서 새로운 유전자를 만들어 내는 빈번한 중복 이벤트와 재결합 사건을 담당한다. 이러한 특성은 개별적인 규모로 다양성을 생성하므로 유기체가 환경에 적응하는 데 기여한다. 예를 들어 적혈구 단계 간상 동안의 플라스모디움 팔시파룸에서는 염색체 사지가 세포핵 주변부에 모여 자주 삭제와 텔로미어 위치 효과(TPE)를 겪는다. 이 사건은 중합체 반복실험의 확대와 삭제에 더해 염색체 크기의 다형성(polymorism)을 발생시켜 후생유전적, 유전적 통제를 받게 된다. 아열대의 특성 때문에 플라스모듐 팔시파룸은 감염된 적혈구의 항원성 및 접착성 특성을 변화시켜 호스트 면역성을 회피한다(아열성 대본 참조).[7][8]
변형
중합체 영역의 변동은 (TTAGGG)n과 같은 반복 시퀀스로 구분된 대규모 스트레칭의 재결합으로 인해 STR에서 대부분 변동이며, 이는 재결합과 전사에서 중요한 역할을 한다. 따라서 개체 간에 Happlotype(DNA 시퀀스 변형)과 길이 차이가 관찰된다.
부차적 대본
중합체 대본은 유사 유전자(단백질로 번역되지 않은 RNA 시퀀스를 생성하는 변환 유전자)와 유전자 계열이다. 인간에서는 후각수용체, 면역글로불린 헤비체인, 아연손가락 단백질 등을 코딩한다. 다른 종에서는 플라모듐, 트라이파노소마 브루시 등 여러 기생충이 면역체계를 벗어나기 위해 가변 표면 항원을 노출하는 등 숙주가 처한 적대적 환경에 적응하기 위한 정교한 회피 메커니즘을 개발했다. 이들 유기체에서 표면 항원을 위한 유전자 코딩은 미분위기에 위치하며, 이 선호되는 위치가 유전자 전환과 발현, 새로운 변종의 발생을 용이하게 하는 것으로 추측되고 있다.[9][10] 예를 들어 플라스모디움 팔시파룸(말라리아산염)의 var과에 속하는 유전자는 대부분 미분위성으로 국부화된다. 항원적 변화는 핵 주변부(핵공)의 별도 공간 영역에서 모노알렐릭 바 전사, 달리 동일하지 않은 바 유전자에 대한 미분 히스톤 마크, 텔로메릭 이질 색소카틴에 의해 매개된 바 음소거 등 후생유전 인자에 의해 조정된다. 부호화 RNA와 같은 다른 요인은 인접한 부분 또는 var 유전자에서 생성되는 부호화 RNA와 같이 항원 변화에도 기여할 수 있다.[11][12] 트라이파노소마 브루시(수면병의 매개체)에서 가변 표면 당단백질(VSG) 항원 변화는 기생충이 숙주 면역체계를 피하기 위해 사용하는 관련 메커니즘이다. VSG 표현은 전적으로 미완성이며 무성 VSG 유전자의 현장 활성화 또는 활성 말단 표현 사이트에 VSG 유전자의 내부 무성 복사본을 삽입하는 DNA 재배열로 발생한다. 플라스토듐 팔시파룸과 대조적으로 트라이파노소마 브루시에서는 후생유전적 및 유전적 요인에 의해 항원적 변화가 조정된다.[13][14]
폐렴구균에서 주요 표면 당단백질(MSG) 유전자 계열은 항원성 변동을 일으킨다. MSG 유전자는 염색체 끝에 있는 상자와 같으며 고유 위치 UCS(상류 보존 시퀀스)에 있는 MSG 유전자만 전사한다. 다른 MSG 유전자가 표현 사이트(UCS)를 점유할 수 있으며, 재결합이 잠재 기증자 풀에서 유전자를 가져다가 표현 사이트에 설치할 수 있으며, 아마도 십자형(Crossovers)을 통해 새로운 MSG 유전자의 전사를 활성화하고, 폐렴구스티스 지로브시스의 표면 항원을 변경할 수 있음을 시사한다. 표현 현장에서의 전환은 표현형 및 침묵형 MSG 유전자의 미분위치에 의해 촉진될 수 있다. 두 번째 미분위 유전자 계열인 MSR은 전사적 차원에서 엄격하게 규제되지 않지만 표현적 다양성에 기여할 수 있다. P. 지로베csi의 항원학적 변화는 유전적 규제에 의해 지배된다.[15][16]
병리학적 함축성
세포분열의 반복적인 주기에 의한 말단백질 DNA의 상실은 노쇠나 체세포 노화와 관련이 있다. 이와는 대조적으로 세균라인과 암세포는 텔로머레이스라는 효소를 가지고 있는데, 텔로메레 열화를 방지하고 텔로미어의 건전성을 유지하여 이러한 유형의 세포가 매우 오래 살도록 한다.
인간에서 아열대 질환의 역할은 근위축성 근위축증(FSHD), 알츠하이머병, 특이 신드롬 질환(피질 및 정신지체)에서 입증된다. 예를 들어, FSHD는 4q 염색체의 하위 골격 부위의 삭제와 관련이 있다. 일련의 10 ~ 100 kb의 반복측정기가 정상 4q 하위절차에 위치하지만, FSHD 환자들은 1 ~ 10의 반복측정 단위에 불과하다. 이 삭제는 반복 배열 자체의 상실을 통해서가 아니라 가까운 유전자의 전사에 영향을 미치는 위치 효과 때문에 질병을 일으키는 것으로 생각된다.[1]
장점과 효과
아열대는 다른 염색체에 위치한 다른 아열대 물질과 동일하며 유전체 주위를 움직일 수 있는 전이성 소자의 한 유형이다. 아열대는 유사유전자로 단백질을 코드화하지 않지만 유전자를 다양화함으로써 진화적 이점을 제공한다. 중합, 재결합, 소결은 새로운 유전자와 새로운 염색체 성질의 생성을 가능하게 한다.[1] 아열대의 장점은 길이와 순서를 따지지 않고 인간과 유전적 요소가 유사하기 때문에 플라스모듐팔치파룸,[1] 드로소필라멜라노가스터,[1] 사카로마이오스 세레비시아아 등 여러 종에서 연구되어 왔다.[1][1] 파스톨루스속초라고 알려진 흔한 콩 식물에서 같은 장점이 발견되었기 때문에, 아열대들은 식물에서 같은 역할을 할 수 있을 것이다.[17]
감수성 재조합과 유사성 재조합을 하는 동안 다른 종류의 미분모세포가 자주 재배열되는데, 이는 미분모세포가 염색체에서 새롭고 빠른 유전적 변화를 일으키는 자주 분열을 일으킨다는 것을 나타낸다.[1] 사카로마이오스 세레비시아에서, 하위종류의 염색체 7L의 15kb 부위는 텔로머레이즈 제거에서 세포 생존성을 유지한 반면, 마지막 15kb의 제거는 염색체 노화를 증가시켰다.[18] 핵분열 효모, 정신분열증균 퐁베, 세포는 유사분열과 감수분열을 방해하지 않아 세포분열을 위해 아열량이 필요하지 않음을 나타낸다.[19] 그들은 아직 유사분열과 감수분열의 행렬에 필요하지 않다. 미분열은 세포 DNA 재조합을 이용한다. 히드록시아, 캠토테신, 자외선, 티아벤다졸을 다량으로 투여했을 때, 정신분열체 퐁베 세포에서 아열대의 녹아웃은 다중 스트레스 반응의 조절에 영향을 미치지 않는다.[19] 정신분열증균 퐁베 세포에서 아벨로메르의 녹아웃은 텔로메르의 길이에 영향을 주지 않았으며, 이는 그들이 길이에 대한 규제로 아무런 역할도 하지 않는다는 것을 보여준다.[19] 그러나 하위종류는 텔로미어의 복제 타이밍에 강하게 영향을 미친다.[20] 텔로머레이즈를 상실한 후 정신분열체 포메세포에서 아열대세포가 녹아웃되는 것은 세포 생존에 영향을 주지 않으며, 이는 아열대세포가 세포 생존에 필요하지 않음을 나타낸다.[19] 텔로머아제 손실 후 미분수체가 필요하지 않은 이유에 대한 설명은 염색체가 염색체 안정화를 유지하기 위해 내부 또는 염색체 간 순환화[21] 또는 HAATI를[22] 사용할 수 있기 때문이다. 그러나 염색체간 원형화의 사용은 단일 염색체에서 2개의 센트롬을 생성하여 염색체가 불안정해져 유사시 염색체 파괴를 일으킨다. 이에 대응하여 염색체는 두 개의 센트롬의 형성을 방해하기 위해 센트롬 비활성화를 유도할 수 있지만, 이것은 센트롬에서 이질색질체 형성을 유도할 것이다. 헤테로크롬화염은 그것이 들어가서는 안 되는 장소에 들어가면 해로울 수 있다. 아열대는 이질 색소 침착이 유로마틴 지역에 들어가는 것을 막는 역할을 한다. 아열대 색소침입은 이질 색소침입의 영향을 완화시킬 수 있으며, 아열대 끝부분에 이질 색소침착을 분산시킨다. 아열대 없이, 이염색체는 중요한 유전자에 너무 가까이 다가가면서 아열대 영역 주위에 퍼질 것이다. 이 거리에서 이질 색소 침착은 근처에 있는 유전자를 잠재울 수 있어 삼투압에 대한 민감도가 높아진다.[19]
아열대는 슈고신 단백질로 필수 기능을 수행한다. 슈고신은 감수분열과 유사분열 시 염색체 분리를 위한 센트롬 단백질이다. 슈고신 단백질에는 SGOL1과 SGOL2 두 종류가 있다. Sgo1은 자매 염색체의 센트럴 응집력에 대한 감수분열 1로만 표현되며,[23] 감수분열과 유사분열로 표현되는 Sgo2는 M상에서의 센트롬에서의 염색체 분리를 담당한다. 핵분열 효모에서, Sgo2는 센트롬뿐만 아니라 미분열 효모에서도 국부화된다. sgo2는 G2상 중간 단계인 중간 단계인 중간 단계인 중간 단계인 아열대와 상호작용하며 고농축 색소체인 '노브'를 형성하는 데 큰 역할을 한다. sgo2는 세포에 말단소립 DNA가 결핍된 미분모세포에 남아 있다.sgo2는 H3K9me3-swi6 매개 이질색소화염과는 다른 통로 안에 있는 미분모 유전자의 발현을 억제한다. sgo2는 복제 시작 시 복제 요인인 [24]Sld3를 억제함으로써 하위 계층 복제 타이밍에 대한 억제 효과도 있다.[25] 따라서 sgo2는 적절한 미분위 유전자 발현과 복제 타이밍을 보장하기 위해 유전자 발현과 복제를 규제한다.
분석
특히 환자 하위 계층의 시퀀싱 및 프로파일링, 하위 계층 분석은 반복된 시퀀스, 스트레칭 길이, 주제에 대한 데이터베이스 부족으로 인해 어렵다.
참조
- ^ a b c d e f g h i Mefford, Heather C.; Trask, Barbara J. (February 2002). "The complex structure and dynamic evolution of human subtelomeres". Nature Reviews Genetics. 3 (2): 91–102. doi:10.1038/nrg727. PMID 11836503.
- ^ Louis, E. J.; Naumova, E. S.; Lee, A.; Naumov, G.; Haber, J. E. (March 1994). "The Chromosome End in Yeast: Its Mosaic Nature and Influence on Recombinational Dynamics". Genetics. 136 (3): 789–802. PMC 1205885. PMID 8005434.
- ^ Walmsley, Richard W.; Chan, Clarence S. M.; Tye, Bik-Kwoon; Petes, Thomas D. (July 1984). "Unusual DNA sequences associated with the ends of yeast chromosomes". Nature. 310 (5973): 157–160. Bibcode:1984Natur.310..157W. doi:10.1038/310157a0. PMID 6377091.
- ^ Coissac, Eric; Maillier, Evelyne; Robineau, Sylviane; Netter, Pierre (December 1996). "Sequence of a 39 411 bp DNA fragment covering the left end of chromosome VII of Saccharomyces cerevisiae". Yeast. 12 (15): 1555–1562. doi:10.1002/(SICI)1097-0061(199612)12:15<1555::AID-YEA43>3.0.CO;2-Q. PMID 8972578.
- ^ Louis, E. J.; Haber, J. E. (July 1992). "The Structure and Evolution of Subtelomeric Y' Repeats in Saccharomyces Cerevisiae". Genetics. 131 (3): 559–574. PMC 1205030. PMID 1628806.
- ^ Louis, Edward J. (December 1995). "The chromosome ends ofSaccharomyces cerevisiae". Yeast. 11 (16): 1553–1573. doi:10.1002/yea.320111604. PMID 8720065.
- ^ Rubio, J P; Thompson, J K; Cowman, A F (1 August 1996). "The var genes of Plasmodium falciparum are located in the subtelomeric region of most chromosomes". The EMBO Journal. 15 (15): 4069–4077. doi:10.1002/j.1460-2075.1996.tb00780.x. PMC 452127. PMID 8670911.
- ^ Su, Xin-zhuan; Heatwole, Virginia M.; Wertheimer, Samuel P.; Guinet, Frangoise; Herrfeldt, Jacqueline A.; Peterson, David S.; Ravetch, Jeffrey A.; Wellems, Thomas E. (July 1995). "The large diverse gene family var encodes proteins involved in cytoadherence and antigenic variation of plasmodium falciparum-infected erythrocytes". Cell. 82 (1): 89–100. doi:10.1016/0092-8674(95)90055-1. PMID 7606788.
- ^ Cano, Maria Isabel N (September 2001). "Telomere biology of Trypanosomatids: more questions than answers". Trends in Parasitology. 17 (9): 425–429. doi:10.1016/S1471-4922(01)02014-1. PMID 11530354.
- ^ Barry, J.D.; Ginger, M.L.; Burton, P.; McCulloch, R. (January 2003). "Why are parasite contingency genes often associated with telomeres?". International Journal for Parasitology. 33 (1): 29–45. doi:10.1016/S0020-7519(02)00247-3. PMID 12547344.
- ^ Scherf, Artur; Lopez-Rubio, Jose Juan; Riviere, Loïc (October 2008). "Antigenic Variation in Plasmodium falciparum". Annual Review of Microbiology. 62 (1): 445–470. doi:10.1146/annurev.micro.61.080706.093134. PMID 18785843.
- ^ Guizetti, Julien; Scherf, Artur (May 2013). "Silence, activate, poise and switch! Mechanisms of antigenic variation in". Cellular Microbiology. 15 (5): 718–726. doi:10.1111/cmi.12115. PMC 3654561. PMID 23351305.
- ^ Cross, George A. M. (April 1996). "Antigenic variation in trypansosomes: Secrets surface slowly". BioEssays. 18 (4): 283–291. doi:10.1002/bies.950180406. PMID 8967896.
- ^ Rudenko, G. (1 October 2000). "The polymorphic telomeres of the African trypanosome Trypanosoma brucei". Biochemical Society Transactions. 28 (5): 536–540. doi:10.1042/bst0280536. PMC 3375589. PMID 11044370.
- ^ Stringer, James R. (2014). "Pneumocystis carinii Subtelomeres". Subtelomeres. pp. 101–115. doi:10.1007/978-3-642-41566-1_5. ISBN 978-3-642-41565-4.
- ^ Portnoy, D. A.; Stringer, James R.; Keely, Scott P. (1 February 2001). "Genetics of Surface Antigen Expression inPneumocystis carinii". Infection and Immunity. 69 (2): 627–639. doi:10.1128/IAI.69.2.627-639.2001. PMC 97933. PMID 11159949.
- ^ Chen, Nicolas W. G.; Thareau, Vincent; Ribeiro, Tiago; Magdelenat, Ghislaine; Ashfield, Tom; Innes, Roger W.; Pedrosa-Harand, Andrea; Geffroy, Valérie (14 August 2018). "Common Bean Subtelomeres Are Hot Spots of Recombination and Favor Resistance Gene Evolution". Frontiers in Plant Science. 9: 1185. doi:10.3389/fpls.2018.01185. PMC 6102362. PMID 30154814.
- ^ Jolivet, Pascale; Serhal, Kamar; Graf, Marco; Eberhard, Stephan; Xu, Zhou; Luke, Brian; Teixeira, Maria Teresa (12 February 2019). "A subtelomeric region affects telomerase-negative replicative senescence in Saccharomyces cerevisiae". Scientific Reports. 9 (1): 1845. Bibcode:2019NatSR...9.1845J. doi:10.1038/s41598-018-38000-9. PMC 6372760. PMID 30755624.
- ^ a b c d e Tashiro, Sanki; Nishihara, Yuki; Kugou, Kazuto; Ohta, Kunihiro; Kanoh, Junko (13 October 2017). "Subtelomeres constitute a safeguard for gene expression and chromosome homeostasis". Nucleic Acids Research. 45 (18): 10333–10349. doi:10.1093/nar/gkx780. PMC 5737222. PMID 28981863.
- ^ Piqueret-Stephan, Laure; Ricoul, Michelle; Hempel, William M.; Sabatier, Laure (2 September 2016). "Replication Timing of Human Telomeres is Conserved during Immortalization and Influenced by Respective Subtelomeres". Scientific Reports. 6 (1): 32510. Bibcode:2016NatSR...632510P. doi:10.1038/srep32510. PMC 5009427. PMID 27587191.
- ^ Wang, Xiaorong; Baumann, Peter (22 August 2008). "Chromosome Fusions following Telomere Loss Are Mediated by Single-Strand Annealing". Molecular Cell. 31 (4): 463–473. doi:10.1016/j.molcel.2008.05.028. PMID 18722173.
- ^ Jain, Devanshi; Hebden, Anna K.; Nakamura, Toru M.; Miller, Kyle M.; Cooper, Julia Promisel (September 2010). "HAATI survivors replace canonical telomeres with blocks of generic heterochromatin". Nature. 467 (7312): 223–227. Bibcode:2010Natur.467..223J. doi:10.1038/nature09374. PMID 20829796.
- ^ Watanabe, Yoshinori (July 2005). "Sister chromatid cohesion along arms and at centromeres". Trends in Genetics. 21 (7): 405–412. doi:10.1016/j.tig.2005.05.009. PMID 15946764.
- ^ Bruck, Irina; Kaplan, Daniel L. (6 November 2015). "The Replication Initiation Protein Sld3/Treslin Orchestrates the Assembly of the Replication Fork Helicase during S Phase". Journal of Biological Chemistry. 290 (45): 27414–27424. doi:10.1074/jbc.M115.688424. PMC 4646389. PMID 26405041.
- ^ Tashiro, Sanki; Handa, Tetsuya; Matsuda, Atsushi; Ban, Takuto; Takigawa, Toru; Miyasato, Kazumi; Ishii, Kojiro; Kugou, Kazuto; Ohta, Kunihiro; Hiraoka, Yasushi; Masukata, Hisao; Kanoh, Junko (25 January 2016). "Shugoshin forms a specialized chromatin domain at subtelomeres that regulates transcription and replication timing". Nature Communications. 7 (1): 10393. Bibcode:2016NatCo...710393T. doi:10.1038/ncomms10393. PMC 4737732. PMID 26804021.