U2 스플라이소솜 RNA
U2 spliceosomal RNA| U2 스플라이소솜 RNA | |
|---|---|
 | |
| 식별자 | |
| 기호 | U2 |
| Rfam | RF00004 |
| 기타자료 | |
| RNA형 | 유전자; snRNA; 스플라이싱 |
| 도메인 | 에우카리오타 |
| 바둑 | GO:0000370 GO:0045131 GO:0005686 |
| 그렇게 | SO:0000392 |
| PDB 구조 | PDBe |
U2 스플라이소솜 snRNA는 사실상 모든 진핵생물의 주요 스플라이소솜(Sm) 기계에서 발견되는 작은 핵 RNA(snRNA) 분자의 한 종이다.체내에서는 U2 snRNA와 연관된 폴리펩타이드들이 모여 주요 이음매 복합체의 필수 성분인 U2 소형 핵 리보핵단백질(snRNP)을 생산한다.[1]주요 이음매 분할 경로는 이음매 조립의 초기 단계에서 U2 snRNP가 독점적으로 인정한 SM 인트론(mRNA primary transcripts)의 클래스에 기초하여 U2 종속 경로라고도 한다.[2]U2 종속 인트론 인식 외에도, U2 snRNA는 사전 RNA 스플라이싱의 화학에서 촉매 역할을 하는 이론이 있다.[3][4]리보솜 RNA(rRNA)와 유사하게, SM snRNA는 RNA:RNA와 RNA:단백질 접점을 모두 중재해야 하며, 따라서 이러한 유형의 상호작용을 촉진하기 위해 전문적이고 보존성이 높은 1차 및 2차 구조 요소를 진화시켰다.[5][6]
mRNA 1차 대본이 샤프와 로버츠에 의해 길고 부호화되지 않는 방해 시퀀스(intron)를 포함하고 있다는 사실이 발견된 직후,[7][8] 조안 스티츠는 인트론 절제의 생화학 메커니즘을 특징짓는 작업을 시작했다.[9]U1 snRNA의 5' 영역에서 발견된 시퀀스가 hnRNA 기록에서 5' 스플라이스 접합부에 걸쳐 보존된 시퀀스와 함께 광범위한 염기쌍 보완성을 보인다는 흥미로운 관찰은 특정 snRNA가 RNA를 통해 스플라이스 사이트 경계를 인식하는 데 관여할 수 있다는 추측을 불러일으켰다.RNA 접점.[9]최근에 와서야 비로소 원자 결정 구조는 비록 이 상호작용의 복잡성이 당시에 완전히 실현되지 않았더라도 원래의 추측이 정말로 옳았다는 것을 증명할 수 있었다.[5][6][10]
U2 snRNA 인식 요소
사카로마이오스 세레비시아에 U2 snRNA는 18개의 폴리펩타이드와 연관되어 있으며, 그 중 7개는 모든 sm급 snRNP에 공통적인 구조 단백질이다.[11]이러한 비특정 구조 단백질은 SM-binding 부위라고 불리는 RNA 내에 위치한 고도로 보존된 인식 시퀀스(AUGn,n = 4-6)를 통해 SM snRNA와 연관된다.[12]다른 두 단백질인 A'와 B'는 U2 특이적이며, snNP 조립을 위해 U2 snRNA 고유의 구조 요소(특히 두 개의 3' 스템 루프)를 요구한다.[11]3개 서브 유닛 SF3a와 6개 서브 유닛 SF3b 단백질 복합체도 U2 snRNA와 연관된다.[13]
U2 snRNA는 지점 시퀀스(BPS)로 알려진 3' 스플라이스 부지의 업스트림 18-40 뉴클레오티드 사이의 7-12 뉴클레오티드 시퀀스를 통해 내부 인식에 관여한다.[1][2]효모에서 컨센서스 BPS는 길이 7 뉴클레오티드 잔류물이며 U2 snRNA 내의 보완적 인식 순서는 6 뉴클레오티드다.이 두 시퀀스 사이의 이중 형성은 BPS의 위치 5에서 보존된 아데노신 잔여물이 불룩하게 된다.불룩해진 아데노신 잔여물은 C3'-endo 순응을[14] 채택하여 스플리싱 인자 Cwc25, Yju2 및 Isy1의 도움으로 5' 스플라이스 부위에서 인 원자 인라인 공격을 위한 2' OH를 정렬한다.[15]핵포화성 공격은 인트론(비정상적인 2-5'-3' 연계 유충 중간을 통해 두 개의 연속적인 성전환 반응 중 첫 번째를 개시한다. 여기서 두 번째 성전환은 두 개의 옆구리 바깥쪽 외과의 교전을 포함한다.
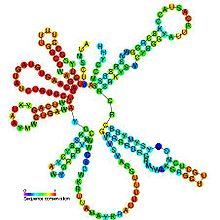
1차 및 2차 구조
U2 snRNA의 시퀀스 길이는 모든 진핵 유기체에 걸쳐 최대 크기 순서로 달라질 수 있지만, 모든 U2 snRNA는 특히 위치의 85%가 보존된 5' 끝의 하류에서 첫 80개 뉴클레오티드 내에 많은 유전학적으로 상수 영역을 포함한다.[16]또한, 스템 루프 I, II, III, IV 및 이들 도메인을 연결하는 고립된 단일 지역을 포함한 몇 가지 2차 구조 요소도 보존된다.[16][17]효모 U2 snRNA의 스템 루프 II에는 특이한 깎은 GA 기본 쌍이 들어 있어 tRNA 안티코돈 루프와 유사한 기하학적 순응을 공유하는 독특한 U턴 루프 모티브로 이어진다.[5]모든 U2 snRNA는 10-16 베이스 쌍 나선을 가진 단자 스템 루프(IV)와 컨센서스 시퀀스 5'-UYGCANUURYN-3"의 보존된 11개의 뉴클레오티드 루프를 가지고 있다.[16]
U2 snRNA는 모든 작은 핵 RNA 중에서 가장 광범위하게 변형된 것이다.[18]이러한 변환 후 수정의 정확한 위치는 유기체에 따라 다를 수 있지만, 새로운 증거는 U2 snRNA 수정과 생물학적 기능 사이에는 강한 상관관계가 있음을 시사한다.[18]수정사항에는 일부 우리딘 잔류물을 유사우리딘으로 변환, 2'-O-메틸화, 뉴클레오베이스 메틸화, 5'모노메틸화 구아노신 캡을 2,7-트리메틸화 구아노신 캡으로 변환하는 것이 포함된다.[18]이러한 변형들 중 많은 것들이 분자의 5' 끝부분에 있는 27 뉴클레오티드 지역에 존재한다.[18]
컨포메이션 다이내믹스
스플라이소솜은 조립과 스플라이싱에 걸쳐 몇 가지 일치 재배열을 거치는 동적 분자 기계다.이음재 재배열을 둘러싼 많은 생화학적 세부사항들은 여전히 불명확하지만, 최근의 연구는 U2와 U6 snRNA 사이에 중요한 접이식 단지의 형성이 즉시 이음새 반응의 첫 단계를 진행하는 것을 시각화했다.[19][6]이 접는 이벤트는 활성 부위며 두 Mg2+이온을 조정하기 이 절차 세인트에서 음전하 형성 안정을 위해 그 5´인라인 공격에 대한 분기점 아데노신과 2에서 접속 사이트´OH제휴를 포함한 필수 요소들을 발판을 제공할 것으로 보이는four-helix 접합의 형성을 촉진한다eps.[19]
진화론적 오리진스
U2-U6 접기의 주목할 만한 특징은 자기 분열 그룹 II intron에서 도메인 V와 구조적 유사성이다.[6][3]U6 snRNA에서 발견된 AGC 3중창은 그룹 II 인트론에서 보존되며, 동일한 3차 적층 상호작용도 선호하는 것으로 밝혀졌다.[6]U2-U6 접이식 이벤트 초기에 GU 흔들림 쌍의 형성은 그룹 II 인트론의 촉매 코어 형성에서도 관찰된다.[19]마지막으로, U2-U6 접이식 내에서 발견된 금속 결합 부위의 구조적 보존을 고려할 때 스플라이소솜은 그룹 II 인트론과 동일한 2-메탈 이온 메커니즘을 사용할 가능성이 높다.[3]그룹 II 인트론과 그룹 II 인토솜의 활성 부위에서 U2-U6 접종 사이의 2차 구조 및 3차 구조 보존 범위는 그룹 II 인트론과 이음매 모두 공통적인 진화 기원을 공유함을 강력히 시사한다.
참고 항목
참조
- ^ a b Alberts B, Johnson A, Lewis J, Raff M, Roberts K, Walter P (2002). Molecular Biology of the Cell (4th ed.). Garland Science. ISBN 978-0815332183.
- ^ a b Nelson DL, Cox MM, Lehninger AL (2013). Lehninger principles of biochemistry (6th ed.). New York: W.H. Freeman and Company. ISBN 9781429234146. OCLC 824794893.
- ^ a b c Fica SM, Tuttle N, Novak T, Li NS, Lu J, Koodathingal P, Dai Q, Staley JP, Piccirilli JA (November 2013). "RNA catalyses nuclear pre-mRNA splicing". Nature. 503 (7475): 229–34. Bibcode:2013Natur.503..229F. doi:10.1038/nature12734. PMC 4666680. PMID 24196718.
- ^ Shi Y (August 2017). "The Spliceosome: A Protein-Directed Metalloribozyme". Journal of Molecular Biology. 429 (17): 2640–2653. doi:10.1016/j.jmb.2017.07.010. PMID 28733144.
- ^ a b c Stallings, Sarah C; Moore, Peter B (1997). "The structure of an essential splicing element: stem loop IIa from yeast U2 snRNA". Structure. 5 (9): 1173–1185. doi:10.1016/s0969-2126(97)00268-2. ISSN 0969-2126. PMID 9331416.
- ^ a b c d e Burke JE, Sashital DG, Zuo X, Wang YX, Butcher SE (April 2012). "Structure of the yeast U2/U6 snRNA complex". RNA. 18 (4): 673–83. doi:10.1261/rna.031138.111. PMC 3312555. PMID 22328579.
- ^ Berget SM, Moore C, Sharp PA (August 1977). "Spliced segments at the 5' terminus of adenovirus 2 late mRNA". Proceedings of the National Academy of Sciences of the United States of America. 74 (8): 3171–5. Bibcode:1977PNAS...74.3171B. doi:10.1073/pnas.74.8.3171. PMC 431482. PMID 269380.
- ^ Chow LT, Gelinas RE, Broker TR, Roberts RJ (September 1977). "An amazing sequence arrangement at the 5' ends of adenovirus 2 messenger RNA". Cell. 12 (1): 1–8. doi:10.1016/0092-8674(77)90180-5. PMID 902310. S2CID 2099968.
- ^ a b Lerner MR, Boyle JA, Mount SM, Wolin SL, Steitz JA (January 1980). "Are snRNPs involved in splicing?". Nature. 283 (5743): 220–4. Bibcode:1980Natur.283..220L. doi:10.1038/283220a0. PMID 7350545. S2CID 4266714.
- ^ Perriman R, Ares M (May 2010). "Invariant U2 snRNA nucleotides form a stem loop to recognize the intron early in splicing". Molecular Cell. 38 (3): 416–27. doi:10.1016/j.molcel.2010.02.036. PMC 2872779. PMID 20471947.
- ^ a b Pan ZQ, Prives C (December 1989). "U2 snRNA sequences that bind U2-specific proteins are dispensable for the function of U2 snRNP in splicing". Genes & Development. 3 (12A): 1887–98. doi:10.1101/gad.3.12a.1887. PMID 2559872.
- ^ Mattaj IW, Habets WJ, van Venrooij WJ (May 1986). "Monospecific antibodies reveal details of U2 snRNP structure and interaction between U1 and U2 snRNPs". The EMBO Journal. 5 (5): 997–1002. doi:10.1002/j.1460-2075.1986.tb04314.x. PMC 1166893. PMID 2941274.
- ^ Dziembowski, Andrzej; Ventura, Ana-Paula; Rutz, Berthold; Caspary, Friederike; Faux, Céline; Halgand, Frédéric; Laprévote, Olivier; Séraphin, Bertrand (2004-12-08). "Proteomic analysis identifies a new complex required for nuclear pre-mRNA retention and splicing". The EMBO Journal. 23 (24): 4847–4856. doi:10.1038/sj.emboj.7600482. ISSN 0261-4189. PMC 535094. PMID 15565172.
- ^ Berglund JA, Rosbash M, Schultz SC (May 2001). "Crystal structure of a model branchpoint-U2 snRNA duplex containing bulged adenosines". RNA. 7 (5): 682–91. doi:10.1017/S1355838201002187. PMC 1370120. PMID 11350032.
- ^ Galej, Wojciech P.; Wilkinson, Max E.; Fica, Sebastian M.; Oubridge, Chris; Newman, Andrew J.; Nagai, Kiyoshi (8 September 2016). "Cryo-EM structure of the spliceosome immediately after branching". Nature. 537 (7619): 197–201. Bibcode:2016Natur.537..197G. doi:10.1038/nature19316. ISSN 1476-4687. PMC 5156311. PMID 27459055.
- ^ a b c Guthrie C, Patterson B (1988). "Spliceosomal snRNAs". Annual Review of Genetics. 22: 387–419. doi:10.1146/annurev.ge.22.120188.002131. PMID 2977088.
- ^ Krämer A (August 1987). "Analysis of RNase-A-resistant regions of adenovirus 2 major late precursor-mRNA in splicing extracts reveals an ordered interaction of nuclear components with the substrate RNA". Journal of Molecular Biology. 196 (3): 559–73. doi:10.1016/0022-2836(87)90032-5. PMID 3681967.
- ^ a b c d Yu YT, Shu MD, Steitz JA (October 1998). "Modifications of U2 snRNA are required for snRNP assembly and pre-mRNA splicing". The EMBO Journal. 17 (19): 5783–95. doi:10.1093/emboj/17.19.5783. PMC 1170906. PMID 9755178.
- ^ a b c Sashital DG, Cornilescu G, McManus CJ, Brow DA, Butcher SE (December 2004). "U2-U6 RNA folding reveals a group II intron-like domain and a four-helix junction". Nature Structural & Molecular Biology. 11 (12): 1237–42. doi:10.1038/nsmb863. PMID 15543154. S2CID 35368436.
추가 읽기
- Newby MI, Greenbaum NL (June 2001). "A conserved pseudouridine modification in eukaryotic U2 snRNA induces a change in branch-site architecture". RNA. 7 (6): 833–45. doi:10.1017/S1355838201002308. PMC 1370140. PMID 11424937.
- Berglund JA, Rosbash M, Schultz SC (May 2001). "Crystal structure of a model branchpoint-U2 snRNA duplex containing bulged adenosines". RNA. 7 (5): 682–91. doi:10.1017/S1355838201002187. PMC 1370120. PMID 11350032.


