ATAC-seq
ATAC-seqATAC-seq(서열 분석을 통한 Transposase-accessible Chromatin)는 분자생물학에서 게놈 전체 크로마틴 접근성을 평가하기 위해 사용되는 기법이다.[1]2013년 MNase-seq, FAIRE-Seq, DNase-Seq의 대안적 고급 방법으로 처음 기술되었다.[1]ATAC-seq는 DNase-seq 또는 MNase-seq보다 후생유전자에 대한 빠르고 민감한 분석이다.[2][3][4]null
설명
ATAC-seq는 게놈의 열린 영역에 시퀀싱 어댑터를 삽입하는 초능동 돌연변이 Tn5 Transposase로 개방형 염색체를 조사하여 접근 가능한 DNA 영역을 식별한다.[2][5] 자연적으로 발생하는 전이들은 활동량이 적은 반면, ATAC-seq는 돌연변이 초능동 전이효소를 사용한다.[6]Tn5 transposase cleaves and tagging dna with sequence adaptors.[7][8] Tn5 transposase cleaves and tagging dna with double str태그가 지정된 DNA 조각은 차세대 염기서열을 사용하여 정제, PCR 증폭 및 염기서열 처리된다.[8]시퀀싱 판독치는 접근성이 향상된 영역을 추론할 뿐만 아니라 전사 계수 결합 부위와 핵소자 위치의 영역을 지도화하는 데 사용할 수 있다.[2]한 영역에 대한 판독 횟수는 단일 뉴클레오티드 분해능에서 염색질이 얼마나 개방되어 있는지와 관련이 있다.[2]ATAC-seq는 FAIRE-seq와 같은 소음이나 페놀-클로로폼 추출이 필요하지 않으며, Chip-seq와 같은 항체가 없으며, MNase-seq나 DNase-seq와 같은 민감한 효소 소화가 필요하지 않다.[9][10][11]ATAC-seq 준비는 3시간 이내에 완료될 수 있다.[12]null
적용들
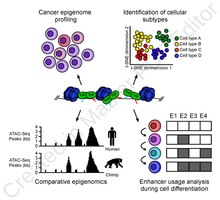
ATAC-Seq 분석은 많은 염색체 접근성 서명을 조사하기 위해 사용된다.가장 일반적인 용도는 뉴클레오솜 매핑 실험이지만,[3] 전사 인자 결합 부위 매핑에 적용하거나,[13] DNA 메틸화 사이트를 매핑하는 데 적용하거나,[14] 시퀀싱 기법과 결합할 수 있다.[15]null
고해상도 진통제 매핑의 효용성은 개발[16] 중 진화된 진화의 차이(예: 침팬지와 사람 사이)를 연구하고, 혈액 세포 분화 중에 사용되는 혈통별 진통제 맵을 발견하는 것으로부터 다양하다.[17]null
또한 ATAC-Seq는 인간 암의 게놈 범위 염색체 접근성 지형을 정의하고 [18]황반변성의 염색체 접근성 전반을 감소시키는 데도 적용되었다.[19]ATAC-seq에서 연산 풋프린팅 방법을 수행하여 셀별 결합 사이트와 셀별 활동이 있는 전사 계수를 찾을 수 있다.[13]null
싱글 셀 ATAC-seq
단일 셀 분석을 수용하도록 ATAC-seq 프로토콜이 수정되었다.미세유체학은 단일핵을 분리하고 개별적으로 ATAC-seq 반응을 수행하는 데 사용될 수 있다.[12]이 접근방식으로, 단일 셀은 태그 지정 전에 미세유체 장치 또는 액체 증착 시스템에 의해 포착된다.[12][20]단일 셀 격리가 필요하지 않은 다른 기술은 결합형 셀룰러 인덱싱이다.[21]이 기술은 수천 개의 개별 세포에서 염색질 접근성을 측정하기 위해 바코드를 사용한다; 그것은 실험당 10,000-10,000개의 세포로부터 후생유전학적 프로파일을 생성할 수 있다.[22]그러나 결합형 셀룰러 인덱싱은 추가적인 맞춤형 설계 장비 또는 다량의 맞춤형 수정 Tn5를 필요로 한다.[23]최근에는 sci-CAR이라는 풀링된 바코드 방식이 개발되어 단일 세포의 크로마팅 접근성과 유전자 발현에 대한 공동 프로파일링이 가능해졌다.[24]null
SCATAC-seq의 연산 분석은 개방된 염색질 영역당 판독 횟수를 갖는 카운트 매트릭스 구성을 기반으로 한다.예를 들어, 의사 대량 ATAC-seq 데이터의 표준 피크 호출에 의해 개방 염색질 영역을 정의할 수 있다.추가 단계에는 PCA를 통한 데이터 감소와 셀의 클러스터링이 포함된다.[20] SCATAC-seq 행렬은 매우 클 수 있으며(수십만 개의 지역) 매우 희박하다. 즉, 항목 중 3% 미만이 0이 아니다.[25]따라서 카운트 매트릭스의 귀속은 비음행 매트릭스 인자화로서 방법을 사용하는 또 다른 중요한 단계다.대량 ATAC-seq와 마찬가지로, SCATAC-seq는 세포의 유전자 발현을 제어하는 전사 인자 같은 규제자를 찾을 수 있다.이는 TF 모티브를[26] 중심으로 한 읽기 수나 발자국을 분석해 보면 알 수 있다.[25]null
참조
- ^ a b Buenrostro JD, Giresi PG, Zaba LC, Chang HY, Greenleaf WJ (December 2013). "Transposition of native chromatin for fast and sensitive epigenomic profiling of open chromatin, DNA-binding proteins and nucleosome position". Nature Methods. 10 (12): 1213–8. doi:10.1038/nmeth.2688. PMC 3959825. PMID 24097267.
- ^ a b c d Buenrostro JD, Wu B, Chang HY, Greenleaf WJ (January 2015). "ATAC-seq: A Method for Assaying Chromatin Accessibility Genome-Wide". Current Protocols in Molecular Biology. 109: 21.29.1–21.29.9. doi:10.1002/0471142727.mb2129s109. PMC 4374986. PMID 25559105.
- ^ a b Schep AN, Buenrostro JD, Denny SK, Schwartz K, Sherlock G, Greenleaf WJ (November 2015). "Structured nucleosome fingerprints enable high-resolution mapping of chromatin architecture within regulatory regions". Genome Research. 25 (11): 1757–70. doi:10.1101/gr.192294.115. PMC 4617971. PMID 26314830.
- ^ Song L, Crawford GE (February 2010). "DNase-seq: a high-resolution technique for mapping active gene regulatory elements across the genome from mammalian cells". Cold Spring Harbor Protocols. 2010 (2): pdb.prot5384. doi:10.1101/pdb.prot5384. PMC 3627383. PMID 20150147.
- ^ Bajic M, Maher KA, Deal RB (2018). "Identification of Open Chromatin Regions in Plant Genomes Using ATAC-Seq". Plant Chromatin Dynamics. Methods in Molecular Biology. Vol. 1675. pp. 183–201. doi:10.1007/978-1-4939-7318-7_12. ISBN 978-1-4939-7317-0. ISSN 1064-3745. PMC 5693289. PMID 29052193.
- ^ Reznikoff WS (2008). "Transposon Tn5". Annual Review of Genetics. 42 (1): 269–86. doi:10.1146/annurev.genet.42.110807.091656. PMID 18680433.
- ^ Adey, Andrew (December 2010). "Rapid, low-input, low-bias construction of shotgun fragment libraries by high-density in vitro transposition". Genome Biology. 11 (12): R119. doi:10.1186/gb-2010-11-12-r119. PMC 3046479. PMID 21143862.
- ^ a b Picelli S, Björklund AK, Reinius B, Sagasser S, Winberg G, Sandberg R (December 2014). "Tn5 transposase and tagmentation procedures for massively scaled sequencing projects". Genome Research. 24 (12): 2033–40. doi:10.1101/gr.177881.114. PMC 4248319. PMID 25079858.
- ^ Simon JM, Giresi PG, Davis IJ, Lieb JD (January 2012). "Using formaldehyde-assisted isolation of regulatory elements (FAIRE) to isolate active regulatory DNA". Nature Protocols. 7 (2): 256–67. doi:10.1038/nprot.2011.444. PMC 3784247. PMID 22262007.
- ^ Savic D, Partridge EC, Newberry KM, Smith SB, Meadows SK, Roberts BS, et al. (October 2015). "CETCh-seq: CRISPR epitope tagging ChIP-seq of DNA-binding proteins". Genome Research. 25 (10): 1581–9. doi:10.1101/gr.193540.115. PMC 4579343. PMID 26355004.
- ^ Hoeijmakers WA, Bártfai R (2018). "Characterization of the Nucleosome Landscape by Micrococcal Nuclease-Sequencing (MNase-seq)". Chromatin Immunoprecipitation. Methods in Molecular Biology. Vol. 1689. pp. 83–101. doi:10.1007/978-1-4939-7380-4_8. ISBN 978-1-4939-7379-8. ISSN 1064-3745. PMID 29027167.
- ^ a b c Buenrostro JD, Wu B, Litzenburger UM, Ruff D, Gonzales ML, Snyder MP, et al. (July 2015). "Single-cell chromatin accessibility reveals principles of regulatory variation". Nature. 523 (7561): 486–90. Bibcode:2015Natur.523..486B. doi:10.1038/nature14590. PMC 4685948. PMID 26083756.
- ^ a b Li, Zhijian; Schulz, Marcel H.; Look, Thomas; Begemann, Matthias; Zenke, Martin; Costa, Ivan G. (26 February 2019). "Identification of transcription factor binding sites using ATAC-seq". Genome Biology. 20 (1): 45. doi:10.1186/s13059-019-1642-2. PMC 6391789.
- ^ Spektor R, Tippens ND, Mimoso CA, Soloway PD (June 2019). "methyl-ATAC-seq measures DNA methylation at accessible chromatin". Genome Research. 29 (6): 969–977. doi:10.1101/gr.245399.118. PMC 6581052. PMID 31160376.
- ^ Hendrickson DG, Soifer I, Wranik BJ, Botstein D, Scott McIsaac R (2018), Simultaneous Profiling of DNA Accessibility and Gene Expression Dynamics with ATAC-Seq and RNA-Seq, Methods in Molecular Biology, vol. 1819, Springer New York, pp. 317–333, doi:10.1007/978-1-4939-8618-7_15, ISBN 9781493986170, PMID 30421411
- ^ Prescott SL, Srinivasan R, Marchetto MC, Grishina I, Narvaiza I, Selleri L, et al. (September 2015). "Enhancer divergence and cis-regulatory evolution in the human and chimp neural crest". Cell. 163 (1): 68–83. doi:10.1016/j.cell.2015.08.036. PMC 4848043. PMID 26365491.
- ^ Lara-Astiaso D, Weiner A, Lorenzo-Vivas E, Zaretsky I, Jaitin DA, David E, et al. (August 2014). "Immunogenetics. Chromatin state dynamics during blood formation". Science. 345 (6199): 943–9. doi:10.1126/science.1256271. PMC 4412442. PMID 25103404.
- ^ Corces MR, Granja JM, Shams S, Louie BH, Seoane JA, Zhou W, et al. (October 2018). "The chromatin accessibility landscape of primary human cancers". Science. 362 (6413): eaav1898. Bibcode:2018Sci...362.1898C. doi:10.1126/science.aav1898. PMC 6408149. PMID 30361341.
- ^ Wang J, Zibetti C, Shang P, Sripathi SR, Zhang P, Cano M, et al. (April 2018). "ATAC-Seq analysis reveals a widespread decrease of chromatin accessibility in age-related macular degeneration". Nature Communications. 9 (1): 1364. Bibcode:2018NatCo...9.1364W. doi:10.1038/s41467-018-03856-y. PMC 5893535. PMID 29636475.
- ^ a b Mezger A, Klemm S, Mann I, Brower K, Mir A, Bostick M, et al. (September 2018). "High-throughput chromatin accessibility profiling at single-cell resolution". Nature Communications. 9 (1): 3647. Bibcode:2018NatCo...9.3647M. doi:10.1038/s41467-018-05887-x. PMC 6128862. PMID 30194434.
- ^ Cusanovich, Darren (May 2015). "Multiplex single cell profiling of chromatin accessibility by combinatorial cellular indexing". Science. 348 (6237): 910–914. Bibcode:2015Sci...348..910C. doi:10.1126/science.aab1601. PMC 4836442. PMID 25953818.
- ^ Lareau CA, Duarte FM, Chew JG, Kartha VK, Burkett ZD, Kohlway AS, Pokholok D, Aryee MJ, et al. (2019). "Droplet-based combinatorial indexing for massive scale single-cell epigenomics". bioRxiv. doi:10.1101/612713.
- ^ Chen X, Miragaia RJ, Natarajan KN, Teichmann SA (December 2018). "A rapid and robust method for single cell chromatin accessibility profiling". Nature Communications. 9 (1): 5345. Bibcode:2018NatCo...9.5345C. doi:10.1038/s41467-018-07771-0. PMC 6297232. PMID 30559361.
- ^ Cao, Junyue; Cusanovich, Darren A.; Ramani, Vijay; Aghamirzaie, Delasa; Pliner, Hannah A.; Hill, Andrew J.; Daza, Riza M.; McFaline-Figueroa, Jose L.; Packer, Jonathan S.; Christiansen, Lena; Steemers, Frank J. (2018-09-28). "Joint profiling of chromatin accessibility and gene expression in thousands of single cells". Science. 361 (6409): 1380–1385. doi:10.1126/science.aau0730. ISSN 0036-8075. PMID 30166440.
- ^ a b Li Z, Kuppe C, Cheng M, Menzel S, Zenke M, Kramann R, et al. (2021). "Chromatin-accessibility estimation from single-cell ATAC-seq data with scOpen". Nature Communications: 865931. doi:10.1038/s41467-021-26530-2.
- ^ Schep AN, Wu B, Buenrostro JD, Greenleaf WJ (October 2017). "chromVAR: inferring transcription-factor-associated accessibility from single-cell epigenomic data". Nature Methods. 14 (10): 975–978. doi:10.1038/nmeth.4401. PMC 5623146. PMID 28825706.