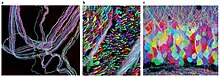
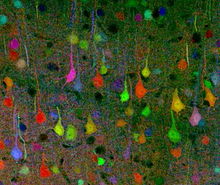
브레인보
Brainbow브레인보우는 형광 단백질을 이용해 뇌의 개별 뉴런을 이웃 뉴런과 구별할 수 있는 과정이다. 개별 뉴런에서 녹색 형광 단백질의 적색, 녹색, 청색 유도체의 비율을 임의로 표현함으로써 각 뉴런에 독특한 색상으로 플래그를 표시할 수 있다. 이 과정은 신경 코넥토믹스 분야에 큰 기여를 해왔다.
이 기술은 원래 2007년 제프 W. 리히트만과 조슈아 R이 이끄는 팀에 의해 개발되었다. Sanes, 둘 다 하버드 대학교에서.[1] 원래의 기술은 최근 드로소필라 멜라노가스터, 다니오 레리오[2], 아라비도시스 탈리아나 등 다른 모델 유기체들과 함께 사용하도록 변형되었다.[3]
이전의 라벨링 기법이 소수의 뉴런만 매핑할 수 있도록 허용한 반면, 이 새로운 방법은 100개 이상의 서로 다른 매핑된 뉴런을 동시에 그리고 이러한 방식으로 차등 조명할 수 있게 한다. 이는 영상물에 대한 특색 있는 다색의 외관으로 이어지며, 과학 사진 대회에서 이름을 떨치고 상을 수상하는 결과를 낳는다.[citation needed]
역사와 발전
브레인보우는 처음에 Jeff W. Lichtman과 Joshua R에 의해 개발되었다. 세인트 워싱턴 대학교의 샌즈 루이스, 비록 그들은 그 이후로 하버드 대학교로 이사했다.[1] 연구팀은 2단계 공정을 이용해 브레인보우를 구축했다. 첫째, 구현 중인 특정 형광 단백질(XFP)을 기반으로 3-4가지 색상 중 하나를 생산하기 위해 여러 가지 배열로 재조합할 수 있는 특정 유전자 구조가 생성됐다.[4] 다음으로 대상 종의 게놈에 동일한 유전자변형 구조를 여러 부 삽입하여 서로 다른 XFP 비율을 무작위로 발현하고, 그 결과 서로 다른 세포가 다양한 색조를 나타내도록 하였다.[4]
브레인보우는 원래 골지 점착술과 염료주입술과 같은 전통적인 신경영상화 기법보다 개선된 것으로, 두 가지 모두 연구자들이 뇌에서 복잡한 신경회로의 구조를 시각화하는 능력에 있어 심각한 한계를 보였다.[1] 기존의 기법들은 제한된 범위의 색상으로만 세포를 착색할 수 있었지만, 종종 2색 및 3색 유전체 생쥐를 이용하여 신경 구조에 관한 제한된 정보를 공개하는 데 활용되었지만, 브레인보우는 최대 100개의 다른 색조로 개별 뉴런에 형광 표시를 할 수 있다는 점에서 훨씬 유연하다. 과학자들이 단음극과 축 과정을 식별하고 심지어 구별할 수 있도록 하기 위해서입니다.[4] 때로는 생체내에서도 뉴런 연결성과 패턴에 대한 그러한 상세한 정보를 공개함으로써 과학자들은 종종 뉴런 상호작용과 뉴런 상호작용에 대한 정보와 그 이후의 행동과 기능에 대한 영향을 추론할 수 있다. 따라서 브레인보우는 이전의 신경영상화 방법들이 남긴 공백을 메웠다.
최근 신경과학에서 브레인보우의 출현과 함께, 연구자들은 신경회로의 특정 지도를 제작할 수 있게 되었고, 이러한 지도들이 다양한 정신활동과 그들의 연결된 행동들과 어떻게 관련되는지 더 잘 조사할 수 있게 되었다(즉, 브레인보우는 뉴런과 뉴런들의 상호연결에 대한 정보를 드러낸다).모든 뇌 기능). 따라서 브레인보우는 이 방법의 추가적인 추론으로서 신경지도의 차이를 분석함으로써 신경장애와 심리장애를 동시에 연구하는데도 사용될 수 있다.[4]
방법들
브레인보우 기술은 Cre-Lox 재조합에 의존하는데, Cre rebinaase 단백질은 loxP 사이트들 사이에서 DNA의 반전이나 분리를 유도한다. 원래의 브레인보우 방식은 브레인보우-1과 브레인보우-2를 모두 포함하며, 서로 다른 형태의 크레/로크 재조합을 활용한다. 브레인보우-1의 변형판인 브레인보우-3는 2013년에 개발되었다.[5] 모든 Brainbow 하위 유형에서, 주어진 XFP의 표현은 확률적 또는 무작위 사건이다.
브레인보우-1은 돌연변이 및 표준 형태의 loxP로 분리된 서로 다른 형광 단백질 유전자(XFP)를 가진 DNA 구조를 사용한다. 이것은 크레이밍 재조합은 동일한 loxP 사이트 사이에서만 발생하기 때문에 상호 배타적인 분리 가능성을 만들어낸다.[1] 재조합이 발생한 후 프로모터 직후에 남아 있는 형광성 단백질이 독특하게 표현된다. 따라서, 4개의 XFP가 서로 다른 3개의 loxP 사이트로 분리된 구조, 3개의 분리 이벤트, 그리고 원래 구조는 4개의 다른 형광 단백질을 생산할 수 있다.[4]
브레인보우-2는 크레 절제와 뒤집기를 사용하여 주어진 구성에서 복수의 표현 가능성을 허용한다. 두 개의 반대방향의 XFP를 가진 하나의 DNA 부분에서 크레는 발현을 위한 적절한 방향으로 하나의 형광 단백질을 남겨두는 임의의 반전 이벤트를 유도할 것이다. 이러한 반전성 시퀀스 중 두 개가 정렬되면 세 가지 반전 이벤트가 가능하다. 절개 이벤트도 고려할 때, 4개의 형광 단백질 중 1개는 크레 절개 및 뒤집기의 주어진 조합에 대해 발현될 것이다.
Brainbow-3 retains the Brainbow-1 loxP format, but replaces the RFP, YFP, and CFP genes with mOrange2, EGFP, and mKate2. mO2, EGFP, and mK2 were chosen both because their fluorescent excitation and emission spectra overlap minimally, and because they share minimal sequence homology, allowing for the design of selective antibodies that can be used to는 면역화학 규약에서 그것들을 검출한다. 브레인보우-3는 또한 뉴런막으로 보다 고르게 밀거래되는 XFP의 farnesylated 파생물을 사용하여 XFP로 뉴런을 불균일하게 채우는 문제를 해결한다.[5]
브레인보우는 두 가지 유전자 변형 유기체 변종을 교차시켜 체내 구현된다. 하나는 크레 단백질을 표현하는 것이고 다른 하나는 여러 버전의 loxP/X로 전이된 것이다.FP 구성. 트랜스젠의 여러 사본을 사용하면 XFP가 약 100가지 다른 색 중 하나를 제공할 수 있는 방식으로 결합할 수 있다.[4] 따라서, 각각의 뉴런은 형광 단백질의 주어진 결합체 및 확률적 발현에 기초하여 다른 색으로 표시된다.
미분 XFP 표현 패턴을 가시적인 형태로 설명하기 위해, 뇌 조각은 공초점 현미경으로 이미징된다. 특정 흥분 파장을 가진 광자에 노출되면 각 불소자는 적색, 녹색 또는 청색 채널로 수집되는 신호를 방출하고, 그 결과 빛의 조합을 데이터 분석 소프트웨어로 분석한다.[1] 색상이 다른 뉴런의 중첩은 복잡한 신경회로를 시각적으로 분리할 수 있다.
브레인보우는 지금까지 주로 쥐를 대상으로 시험해 왔지만, 위에서 설명한 기본 기법도 2007년에 도입된 원래의 방법의 등장 이후 더 최근의 연구에서 사용하기 위해 수정되었다.
쥐

생쥐의 뇌는 7500,000,000개의 뉴런을 가지고 있으며, 드로소필라와 이 기술을 모형화하기 위해 흔히 사용되는 다른 유기체들보다 인간의 뇌와 더 비슷하다. 생쥐는 브레인보우(Brainbow) 방식의 신경영상촬영(Neuro Mimaging)이 성공한 최초의 유기체였다.[1] 라이브트 외 (2007)은 위에서 설명한 브레인보우-1과 브레인보우-2를 이용하여 브레인보우 생쥐의 두 가지 버전을 개발하였다.[1] 이러한 방법을 사용하여 완전한 지도를 만들고 마우스 근육의 축을 추적하려면 수만 개의 이미지를 수집하여 스택으로 컴파일하여 완전한 도식을 만들어야 한다.[4] 그리고 나서 근육의 완전한 코넥텀을 형성하기 위해 각 모터 액손과 그것의 시냅스 접점을 추적할 수 있다.
유전자이전 생쥐에서 브레인보우 기법을 사용하여 검사한 더 많은 뉴런의 예는 운동 신경 내복 귀근육, 뇌계의 액손 트러스트, 해마 틀니트 회오리 등에 있다.[4]
드로소필라
약 10만 개의 뉴런으로 구성된 드로소필라 뇌의 복잡성은 브레인보우처럼 신경생리학과 신경과학 기법을 시행하기에 탁월한 후보라 할 수 있다. 실제로 스테파니 햄펠 외 연구진(2011년)은 브레인보우를 유전자 표적 도구와 연계해 드로필라 뇌 내의 개별 뉴런과 다양한 뉴런 선들을 식별했다.[6] 유전자 표적 도구 중 하나는 UAS-Brainbow의 발현을 제어하고 그 발현을 작은 뉴런 그룹에 표적으로 하는 GAL4/UAS 이진표현 시스템이었다. 플립 아웃(Flip Out) 방법을 활용함으로써 리포터 구조물의 셀룰러 해상도가 높아졌다. 형광 단백질의 표현은 원래 브레인보우와 마찬가지로 일치하는 롭스 부위와 일치하는 크레 재조합에 의존했다. 함펠 외 연구진(2011년)도 내인성 형광보다는 상피에 대한 항체 라벨링을 바탕으로 브레인보우(dBrainbow)의 변형을 자체 개발했다.[6] 그들의 건축물을 두 장 복사하면 6개의 밝고 분리할 수 있는 색상이 나온다. 이것은 색 배정의 단순화와 함께, 그들이 장거리에서 각 뉴런의 궤적을 관찰할 수 있게 해주었다. 구체적으로는 더듬이엽에서 신경근접합부까지 운동 뉴런을 추적해 개별 뉴런의 특정 근육 대상을 식별할 수 있게 했다.
궁극적으로, 이 기술은 연구자들이 이 무척추동물의 뇌 구조와 그것이 어떻게 이어지는 행동과 관련이 있는지에 대한 더 많은 정보를 발견할 수 있도록 드로필라의 신경회로를 효과적으로 지도하는 능력을 제공한다.
제한 사항
어떤 신경영상 기법과 마찬가지로 브레인보우에도 그것을 수행하는 데 필요한 방법에서 비롯되는 여러 가지 한계가 있다. 예를 들어, 배아줄기세포로부터 적어도 두 종류의 유전자이전동물을 번식시키는 과정은 시간이 많이 걸리고 복잡하다. 두 개의 유전자 변형 종을 성공적으로 만들어내더라도, 그들의 자손이 모두 재조합을 보여주지는 않을 것이다. 따라서 이는 실험을 수행하기 전에 광범위한 계획을 세워야 한다.[4]
또한 형광성 단백질의 발현에 나타나는 임의의 성질 때문에 과학자들은 신경 회로의 표지를 정밀하게 조절할 수 없어 특정 뉴런의 식별이 잘 되지 않을 수도 있다.
포유류 집단에서 뇌궁의 사용은 중앙 신경계의 믿을 수 없을 정도로 다양한 뉴런들에 의해 방해받는다. 액손의 긴 트랙이 있는 것과 결합된 뉴런의 순수한 밀도는 높은 해상도로 CNS의 더 큰 영역을 보는 것을 어렵게 만든다. 뇌궁은 복잡한 다세포 환경을 배경으로 단일 세포 분해능을 검사할 때 가장 유용하다. 그러나 광학 현미경의 분해능 한계로 인해 뉴런 간의 시냅스 연결에 대한 결정적인 식별은 쉽게 이루어지지 않는다. 이 문제는 시냅스 마커를 사용하여 시냅스 연결에 광학 현미경 사용을 보완함으로써 다소 피할 수 있다.[7]
참고 항목
참조
- ^ a b c d e f g Livet, J.; Weissman, T. A.; Kang, H.; Draft, R. W.; Lu, J.; Bennis, R. A.; Sanes, J. R.; Lichtman, J. W. (2007). "Transgenic strategies for combinatorial expression of fluorescent proteins in the nervous system". Nature. 450 (7166): 56–62. Bibcode:2007Natur.450...56L. doi:10.1038/nature06293. PMID 17972876.
- ^ Pan, Y. A.; Livet, J.; Sanes, J. R.; Lichtman, J. W.; Schier, A. F. (2011-01-01). "Multicolor Brainbow Imaging in Zebrafish". Cold Spring Harbor Protocols. 2011 (1): pdb.prot5546–pdb.prot5546. doi:10.1101/pdb.prot5546. ISSN 1559-6095. PMC 3082469. PMID 21205846.
- ^ Mach, Jennifer (2011-07-01). "Clonal Analysis with the Brother of Brainbow System". The Plant Cell. 23 (7): 2471. doi:10.1105/tpc.111.230710. ISSN 1532-298X. PMC 3226220.
- ^ a b c d e f g h i Lichtman, Jeff; Jean Livet; Joshua Sanes (June 2008). "A technicolour approach to the connectome". Nature Reviews Neuroscience. 9 (6): 417–422. doi:10.1038/nrn2391. PMC 2577038. PMID 18446160.
- ^ a b Cai, D.; Cohen, K. B.; Luo, T.; Lichtman, J. W.; Sanes, J. R. (2013). "Improved tools for the Brainbow toolbox". Nature Methods. 10 (6): 540–547. doi:10.1038/nmeth.2450. PMC 3713494. PMID 23817127.
- ^ a b Stefanie Hampel; Phuong Chung; Claire McKellar; Donald Hall; Loren Looger; Julie Simpson (February 2011). "Drosophila Brainbow: a recombinase-based fluorescence labeling technique to subdivide neural expression patterns". Nature Methods. 8 (3): 253–260. doi:10.1038/nmeth.1566. PMC 3077945. PMID 21297621.
- ^ Dhawale, A; Bhalla (2008). "The network and the synapse: 100 years after Cajal". HFSP Journal. 2 (1): 12–16. doi:10.2976/1.2835214. PMC 2640997. PMID 19404449.
외부 링크
- NPR의 사이언스 프라이데이 팟캐스트
- "브레인보우" GFP의 시원한 사용법