FAM98A
FAM98A| FAM98A | |||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 식별자 | |||||||||||||||||||||||||
| 별칭 | FAM98A, 시퀀스 유사도 98 멤버 A | ||||||||||||||||||||||||
| 외부 ID | MGI: 1919972 호몰로진: 41042 GeneCard: FAM98A | ||||||||||||||||||||||||
| |||||||||||||||||||||||||
| |||||||||||||||||||||||||
| |||||||||||||||||||||||||
| |||||||||||||||||||||||||
| 직교체 | |||||||||||||||||||||||||
| 종 | 인간 | 마우스 | |||||||||||||||||||||||
| 엔트레스 | |||||||||||||||||||||||||
| 앙상블 | |||||||||||||||||||||||||
| 유니프로트 | |||||||||||||||||||||||||
| RefSeq(mRNA) | |||||||||||||||||||||||||
| RefSeq(단백질) | |||||||||||||||||||||||||
| 위치(UCSC) | Cr 2: 33.53 – 33.6Mb | Cr 17: 75.84 – 75.86Mb | |||||||||||||||||||||||
| PubMed 검색 | [3] | [4] | |||||||||||||||||||||||
| 위키다타 | |||||||||||||||||||||||||
| |||||||||||||||||||||||||
서열 유사성 98, 멤버 A, 또는 FAM98A를 가진 패밀리는 인간 게놈에서 FAM98A 단백질을 암호화하는 유전자다.FAM98A는 인간에게 FAM98B와 FAM98C라는 두 개의 파라로그를 가지고 있다.이 세 가지 모두 RNA와 결합하는 것으로 보이는 보존된 도메인인 DUF2465가 특징이며,[5] FAM98A는 글리신 함량이 높은 C-단자 영역도 특징이다.[6]FAM98A는 척추동물과 무척추동물에도 동음이의어가 있고 choanoflagellates와 녹조에도 동음이의어가 있다.
유전자
로커스
FAM98A 유전자는 인간에서 "-" (마이너스) 가닥의 2p22.3에 위치한다.5와 3'의 UTR을 포함하여, 이 유전자는 15,634 염기에 이르며 8 exon을 포함하고 있다.[7]
mRNA
mRNA는 2745bp로 8 exon으로 구성된다.코딩 시퀀스는 베이스 75에서 시작하여 베이스 1631까지 계속된다.polyA tail signal sequence는 base 2725-2730에 있는 대본의 3' 끝에서 6 nucleotide sequence 20 base이며, polyA 사이트는 base 2745에 있다.[8]
단백질
기본 시퀀스
FAM98A는 분자량 55.3kDa의 길이 518개의 아미노산으로 수정 없이 분자량이다.잔류물 10-329는 DUF2465로 구성되며, 나머지 단백질은 디글리신(diglycine)이 풍부한 C 종단이다.글리신은 단백질의 약 20%를 차지하며, 이 중 대다수가 마지막 200개 잔류물에 포함되어 있다.[9]
변환 후 수정
FAM98A에는 DUF2465에 6개의 강력한 예측 인산화 부위가 있다.이들 부위는 단백질 키나제 C에 의해 인산염 S169, T178, S236, T243, S276, S285로 예측된다.[10] GPS도 S285와 T178에서 단백질 키나제 C에 의한 인산화를 예측한다.[11]FAM98A는 K183과 K195로 요약되어 있을 것이다.[12]섬모일화는 세포핵과 세포질 사이에 FAM98A를 다시 국소화할 수 있게 할 수 있다.[13]글리신 함량이 많은 C 종단에는 GRG 시퀀스가 반복적으로 나타나며, 이는 대칭 또는 비대칭적으로 아르기닌의 메틸화에 취약한 것으로 나타났다.[14]또 다른 논문은 아르기닌 메틸화가 전사의 활성화와 억압, mRNA 스플라이싱, 핵-사이토솔릭 폐쇄, DNA 수리 등 생화학적 기능에 미치는 영향에 대해 설명한다.[15]
이차 구조
N 종단부는 복수의 알파 나선형을 가질 것으로 예측되지만, C 종단부는 코일화되었을 가능성이 있다.[16]알파 나선은 어떤 채널도 형성하지 않으며, FAM98A는 투과성 단백질이 아니다.
3차 및 2차 구조
FAM98A의 구조는 Pyre2 프로그램으로 예측되었다.N단자 영역에는 여러 개의 알파 헬리컬과 글리신 농후 C 단자에 해당하는 C단자 코일 영역이 있다.단백질의 이 두 부위는 잔류물 200-256에서 약 50개의 잔류물을 알파 나선형으로 연결된다.피레2는 가장 유사한 단백질이 미세관(micro tubule)에 결합하는 핵 단백질인 인간 단백질 NDC80 키네토코레 복합 성분임을 발견했다.[17]
도메인 및 모티프
FAM98A는 아미노산 10-329에서 2465(DUF2465)를 알 수 없는 기능을 가진 도메인을 가지고 있다.DUF2465 내에는 시험한 모든 종에 보존된 C-단자 끝 근처에 헵타이드(VPDRGR)가 있다.C-단자 끝은 GGRGR 반복이 있는 글리신 풍부 도메인(글리신이 C 단자의 약 40%를 차지한다)이다.[9]At residues 149-155, there is a predicted nuclear export signal, with the sequence ICIALGM (generally [LIVFM]-X-[LIVFM]-X-[LIVFM]-X-[LIVFM]).[18] Residues 173-176 are predicted to be a nuclear localization signal KKLK (K-[K/R]-X-[K/R]).[19]
호몰로지
파라로그
FAM98A에는 두 개의 파라로그가 있다.FAM98B와 FAM98C.FAM98A는 518개의 아미노산을 함유한 3개의 파라로그 단백질 제품 중 가장 긴 제품이다.그것은 글리신이 풍부한 C 종단점이 FAM98A보다 훨씬 짧은 FAM98B와 더 비슷하다.FAM98C는 FAM98B와 FAM98A와 덜 유사하며, DUF2465 이후의 C 종단부에 결여되어 있을 뿐 아니라 DUF2465 내의 아미노산 염기서열에서 더 많은 차이를 포함하고 있다.세 가지 단백질 제품 모두 RNA와 비특이적으로 연관되는 것으로 실험적으로 나타났다: FAM98A는 mRNA에 결합하고 FAM98B는 tRNA 분할 콤플렉스에 통합된다.[5]
직교체
FAM98A용 직교구는 척추동물에서 발견되었다.곤충과 연체동물에는 FAM98A 유전자에 대한 예측 단백질이 있다.인간에게는 세 개의 FAM98의 파라로그가 있기 때문에 이러한 유전자의 공통 조상이 있다.FAM98A 전체 가문이 아닌 FAM98A와 직교하는 유전자인 엄격한 직교법은 명확하지 않다.FAM98A는 아직 철저히 연구되지 않았으며, 아직 기록되지 않은 게놈들이 많다는 사실과 복합되어 모기의 예측된 FAM98A 유전자가 엄격한 맞춤법인지(종이 갈라지기 전에 FAM98A,B,C로 분할) 또는 모기의 호몰로어인지("FAM98A"는 조상의 조상이다)를 판단하기가 더 어려워진다.FAM98 유전자).
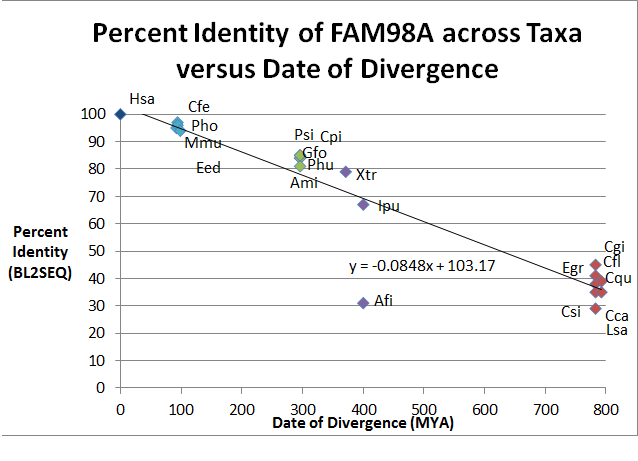
| 시퀀스 번호 | 속종(Gsp) | 공용 이름 | 분기 날짜(MYA)(타임 트리에서) | 등록 번호(NCBI에서) | 시퀀스 길이(AA) | 아이덴티티 | 유사성 |
| 1 | 호모 사피엔스 (HSA) | 인간 | 0 | 518 | 100 | 100 | |
| 2 | 무스 무스쿨루스 (Mmu) | 마우스 | 92.3 | 515 | 95 | 96 | |
| 3 | 카멜루스 페루스 (Cfe) | 박트리안 카멜 | 94.2 | 517 | 97 | 98 | |
| 4 | 판톨롭스호지소니 (포) | 티베트 영양 | 94.2 | 521 | 96 | 97 | |
| 5 | Elephantulus Edwardii (Eed) | 새장 코끼리 말괄량이 | 98.7 | 517 | 94 | 96 | |
| 6 | 거스피자 포티스 (Gfo) | 중간 지반 핀치 | 296 | 648 | 84 | 88 | |
| 7 | 유사포도체 역모증(Phu) | 그라운드 티트 | 296 | 545 | 84 | 88 | |
| 8 | 앨리게이터 미시시피엔시스 (아미) | 아메리카 악어 | 296 | 556 | 81 | 86 | |
| 9 | 펠로디스커스 시넨시스(psi) | 부드러운 표지의 거북이 | 296 | 549 | 85 | 88 | |
| 10 | 크리세미스 픽타벨리 (Cpi) | 웨스턴 페인팅 터틀 | 296 | 549 | 85 | 88 | |
| 11 | 제노푸스 열대성(Xtr) | 서양발톱개구리 | 371.2 | 520 | 79 | 86 | |
| 12 | 아노플라포마핌브리아(아피) | 사향어 | 400.1 | 353 | 31 | 48 | |
| 13 | Iftalurus pintatus (Ipu) | 채널 메기 | 400.1 | 543 | 67 | 75 | |
| 14 | 캄포노투스 플로리다누스 (Cfl) | 플로리다 카펜터 개미 | 782.7 | 516 | 41 | 53 | |
| 15 | 큐렉스 퀸케파시아투스(Cqu) | 모기 | 782.7 | 498 | 38 | 52 | |
| 16 | 세라티염 카피타타타(Cca) | 메드플라이 | 782.7 | 454 | 35 | 51 | |
| 17 | 레페오페바이러스 살모니스 (Lsa) | 연어 루즈 | 782.7 | 467 | 29 | 45 | |
| 18 | 크라소트레아 기가스(Cgi) | 퍼시픽 오이스터 | 782.7 | 422 | 45 | 59 | |
| 19 | 클로노르치스 시넨시스(Csi) | 중국 간 요정 | 792.4 | 378 | 35 | 47 | |
| 20 | 에치노코쿠스 그라눌로수스 (EGR) | 개 테이프벌레 | 792.4 | 1177 | 39 | 56 |
원거리 호몰로지스
FAM98A와 동일한 유전자는 Analytia 내 많은 세사에서 발생할 것으로 예측되지만 Analytia 외 다른 세자는 그들의 게놈에 동일한 FAM98 유전자를 가지고 있을 수 있다.Opisthokonts Monosiga brevicolis(XP_00174505.1)와 Capraspora Owczarzaki(XP_004346371.1)와 같은 eukaryotes와 녹색 알가인 Clorela variabilis(XP_005845167.1)도 유전자에 FAM98을 포함할 수 있다.[20]
호몰로고 도메인
FAM98A의 동질 도메인은 DUF2465(Domain of Unknown Function 2465) 도메인이다.이 영역의 기능은 유전자 자체와 마찬가지로 FAM98A에서는 mRNA, FAM98B에서는 tRNA를 대상으로 RNA에 우선적으로 결합한다고 보고되었지만 대체로 알려져 있지 않다.[5]
표현
프로모터
인간 FAM98A 대본(GXT_24436545)[21]에 할당된 발기인(GXP_90934)은 길이가 915bp이며, mRNA 대본 243bp를 포함하도록 대본과 겹친다.원자력 호흡 계수 1(NRF1)은 촉진자에게 바인딩될 것으로 예측되는 7개의 사이트가 있는 전사 인자로, 그 중 4개는 매트릭스 유사성 - 최적 점수는 0.085점 이상이거나 같으며 가장 높은 점수 전사는 0.204점과 0.199점으로 NRF1이었다.[22]
표현
GEO 대규모 인간 성적표에서 FAM98A는 균일하게 표현되지는 않았지만 보편적으로 표현되었다.가장 많이 발현된 세포 종류는 뇌의 많은 부분(피질, 편도, 쇄골, 뇌하수체), 고환, 자궁, 그리고 평활근이었다.[23]Aceview에 따르면 FAM98A는 평균 유전자 발현량의 3.9배로 표현된다.11개의 대본이 에이스뷰에 의해 확인되었는데, 그 중 5개는 "좋은" 완전 단백질이었다(N과 C termini 완전 번역).녹취록에서 보면 FAM98A의 주요 부분은 처음 4개의 엑손과 두 번째 4개의 엑손, 이 부분은 대략 단백질의 3차 구조 즉 1-4번 엑손에 N-터미네랄 알파헬리케이트, 그리고 긴 알파헬리알과 C 터미네이션 코일은 5-8번 엑손에 해당한다.[24]
함수 및 생화학
FAM98A의 기능은 MRNA로 DUF2465를 묶는 것으로 나타났지만 실험적으로 결정되지는 않았다.[5] 키라가 외.기본적인 단백질은 핵산과 결합한다는 것을 알아냈다.[25]실제로 FAM98A(및 직교)는 약 9의 수정되지 않은 등전점을 가지고 있다.[26]
알려진 상호 작용
FAM98A는 UBC, DDX1, C14orf166, SMO3와 상호작용하는 것으로 실험적으로 나타났으며 DDX1, C14orf166, RBM25와 공존한다.[27]이 후자의 세 단백질은 mRNA와 상호작용을 하는데, FAM98A 또한 그렇게 될 것으로 예측된다.DDX1은 스플라이소솜에 있는 투입성 ATP 의존성 RNA 헬리코아제로서, RNA를 스플라이싱 콤플렉스에서 방출할 가능성이 높다.[28] C14orf166은 폴리머아제 II 결합인자로,[29] RBM25는 대체 스플라이싱을 조절한다.[30]이 모든 상호작용은 FAM98A가 핵 단백질이라는 것을 암시한다.FAM98A는 또한 Sumoylllates가 핵막과 세포솔 사이의 핵막을 가로질러 이동하기 쉽도록 단백질 내의 리신(Lysines)을 함유한 SOMO3와 상호작용한다.[13]또한 FAM98A는 핵에서 잠재적 mRNA 셔틀을 리보솜에 결합한다는 것을 나타내는 비특정 mRNA도 결합한다.[5]
임상적 중요성
단백질 식단이 높거나 낮은 젊은 남성과 노년층 모두에서 특정 유전자(FAM98A 포함)의 발현량 차이를 살펴본 연구에서 발현량은 남성 그룹(청년 및 노인)의 각 저/고단백질 식단의 비율로 측정됐다.FAM98A는 각각 1.01과 1.20으로 젊은 남성과 노인 모두에서 저단백질 식단에서 발현이 증가했다.이 연구에서 유일한 다른 유전자는 두 그룹 모두에서 저단백질 식단에서 발현이 증가하는 동일한 경향을 보였다.THOC4.[31] THOC4, THO Complex 4 또는 Aly/REF 수출 인자는 더 작은 복합체를 형성하도록 조광하며, 샤퍼론이 분할된 mRNA를 형성하여 mRNA의 가공 및 수출을 돕는다.[32]이 논문은 노년층에서 mRNA의 상향 조절은 RNA 결합/분쇄, 신호 단백질, 단백질 분해와 관련이 있다고 언급하고 있다. 실제로 노년층은 젊은 남성들보다 저단백질 식단에서 FAM98A의 발현이 더 높다.[31]
질병 협회
대만의 한 인구에 대한 연구는 로쿠스 2p22.3에서 4개 유전자의 업스트림에서 젊은 발병 고혈압과 두 개의 SNP 사이의 연관성을 발견했다.이 네 개의 유전자 중 하나는 FAM98A였지만 고혈압의 원인이 된 유전자가 FAM98A인지 더 많은 연구가 이루어져야 한다.[33]실제로 FAM98A는 평활근과 심장근육에서 적당히 높은 수치(대략 75번째 백분위수)로 표현된다.[23]
참조
- ^ a b c GRCh38: 앙상블 릴리스 89: ENSG00000119812 - 앙상블, 2017년 5월
- ^ a b c GRCm38: 앙상블 출시 89: ENSMUSG00000002017 - 앙상블, 2017년 5월
- ^ "Human PubMed Reference:". National Center for Biotechnology Information, U.S. National Library of Medicine.
- ^ "Mouse PubMed Reference:". National Center for Biotechnology Information, U.S. National Library of Medicine.
- ^ a b c d e Dürnberger G, Bürckstümmer T, Huber K, Giambruno R, Doerks T, Karayel E, Burkard TR, Kaupe I, Müller AC, Schönegger A, Ecker GF, Lohninger H, Bork P, Bennett KL, Superti-Furga G, Colinge J (July 2013). "Experimental characterization of the human non-sequence-specific nucleic acid interactome". Genome Biology. 14 (7): R81. doi:10.1186/gb-2013-14-7-r81. PMC 4053969. PMID 23902751.
- ^ "Pfam: Family: DUF2465 (PF10239)". Pfam. EMBL-EBI. Retrieved 5 May 2014.
- ^ "Human Gene FAM98A (uc002rpa.1)". Genome. NCBI. Retrieved 5 May 2014.
- ^ NCBI(National Center for Biology Information) mRNA 시퀀스 FAM98A NM_015475.3 https://www.ncbi.nlm.nih.gov/nuccore/NM_015475.3
- ^ a b Brendel, V.; Bucher, P.; Nourbakhsh, I.R.; Blaisdell, B.E.; Karlin, S. (1992). "Methods and algorithms for statistical analysis of protein sequences". Proc. Natl. Acad. Sci. U.S.A. 89 (6): 2002–2006. Bibcode:1992PNAS...89.2002B. doi:10.1073/pnas.89.6.2002. PMC 48584. PMID 1549558.
- ^ Blom N, Sicheritz-Pontén T, Gupta R, Gammeltoft S, Brunak S (June 2004). "Prediction of post-translational glycosylation and phosphorylation of proteins from the amino acid sequence". Proteomics. 4 (6): 1633–49. doi:10.1002/pmic.200300771. PMID 15174133. S2CID 18810164.
- ^ Xue Y, Ren J, Gao X, Jin C, Wen L, Yao X (September 2008). "GPS 2.0, a tool to predict kinase-specific phosphorylation sites in hierarchy". Molecular & Cellular Proteomics. 7 (9): 1598–608. doi:10.1074/mcp.m700574-mcp200. PMC 2528073. PMID 18463090.
- ^ Abgent, WuXi App Tec 회사.스모 플롯TM 분석 프로그램.2013. http://www.abgent.com/tools
- ^ a b Matunis MJ, Coutavas E, Blobel G (December 1996). "A novel ubiquitin-like modification modulates the partitioning of the Ran-GTPase-activating protein RanGAP1 between the cytosol and the nuclear pore complex". The Journal of Cell Biology. 135 (6 Pt 1): 1457–70. doi:10.1083/jcb.135.6.1457. PMC 2133973. PMID 8978815.
- ^ Hyun YL, Lew DB, Park SH, Kim CW, Paik WK, Kim S (June 2000). "Enzymic methylation of arginyl residues in -gly-arg-gly- peptides". The Biochemical Journal. 348 (3): 573–8. doi:10.1042/0264-6021:3480573. PMC 1221099. PMID 10839988.
- ^ Bedford MT, Clarke SG (January 2009). "Protein arginine methylation in mammals: who, what, and why" (PDF). Molecular Cell. 33 (1): 1–13. doi:10.1016/j.molcel.2008.12.013. PMC 3372459. PMID 19150423.
- ^ 펠레(BPS, D_R, DSC, GGR, GGR, G_G, H_K, K_S, L_G, Q_S, JOI)SDSC 워크벤치.1999년 일리노이 대학의 이사회
- ^ Kelley LA, Sternberg MJ (2009). "Protein structure prediction on the Web: a case study using the Phyre server" (PDF). Nature Protocols. 4 (3): 363–71. doi:10.1038/nprot.2009.2. hdl:10044/1/18157. PMID 19247286. S2CID 12497300.
- ^ Fu SC, Imai K, Horton P (September 2011). "Prediction of leucine-rich nuclear export signal containing proteins with NESsential". Nucleic Acids Research. 39 (16): e111. doi:10.1093/nar/gkr493. PMC 3167595. PMID 21705415.
- ^ Timmers AC, Stuger R, Schaap PJ, van 't Riet J, Raué HA (June 1999). "Nuclear and nucleolar localization of Saccharomyces cerevisiae ribosomal proteins S22 and S25". FEBS Letters. 452 (3): 335–40. doi:10.1016/s0014-5793(99)00669-9. PMID 10386617. S2CID 1933493.
- ^ Altschul SF, Madden TL, Schäffer AA, Zhang J, Zhang Z, Miller W, Lipman DJ (September 1997). "Gapped BLAST and PSI-BLAST: a new generation of protein database search programs". Nucleic Acids Research. 25 (17): 3389–402. doi:10.1093/nar/25.17.3389. PMC 146917. PMID 9254694.
- ^ 대본 GXT_2827489.제노마틱스 소프트웨어.2014. http://www.genomatix.de/cgi-bin/[permanent dead link]/vmorado/eldorado.pl?s=2ab9d4751cbd873358acdd746c629f61;TRANS=1;TRANSCRIPTID=2827489;ELDORADO_VERSION=E28R1306
- ^ GXP_90934.제노마틱스 소프트웨어.2014. http://www.genomatix.de/cgi-bin/[permanent dead link]/vmorado/eldorado.pl?s=99a7e4da5d3118fa8a93fb9a283d710f;PROM_ID=GXP_90934;GROUP=vertebrates;GROUP=others;ELDORADO_VERSION=E28R1306
- ^ a b 국립 생명공학 정보 센터, 미국 국립 의학 도서관GEO(Gene Expression 옴니버스) 프로파일"HG-U133A(Human Transcriptom)에 대한 대규모 분석"https://www.ncbi.nlm.nih.gov/geo/tools/profileGraph.cgi?ID=GDS596:212333_at
- ^ "Homo sapiens complex locus FAM98A, encoding family with sequence similarity 98, member A." AceView. NCBI.
- ^ Kiraga J, Mackiewicz P, Mackiewicz D, Kowalczuk M, Biecek P, Polak N, Smolarczyk K, Dudek MR, Cebrat S (June 2007). "The relationships between the isoelectric point and: length of proteins, taxonomy and ecology of organisms". BMC Genomics. 8: 163. doi:10.1186/1471-2164-8-163. PMC 1905920. PMID 17565672.
- ^ http://www.embl-heidelberg.de에서 개발한 Luca Tedo 박사의 프로그램.Bjoen Kindler에 의해 인쇄로 변경되었으며, 또한 가장 낮은 순전하를 발견했다.EMBL WWW Gateway에서 Isoelectric Point Service로 이용 가능 : CS1 maint: 제목으로 보관된 복사본(링크)
- ^ String 9.1. FAM98A.http://string-db.org/newstring_cgi/show_network_section.pl
- ^ "DEAD (Asp-Glu-Ala-Asp) Box Helicase 1". GeneCards.
- ^ "Chromosome 14 Open Reading Frame 166". GeneCards.
- ^ "RNA Binding Motif Protein 25". GeneCards.
- ^ a b Thalacker-Mercer AE, Fleet JC, Craig BA, Campbell WW (November 2010). "The skeletal muscle transcript profile reflects accommodative responses to inadequate protein intake in younger and older males". The Journal of Nutritional Biochemistry. 21 (11): 1076–82. doi:10.1016/j.jnutbio.2009.09.004. PMC 2891367. PMID 20149619.
- ^ "Aly/REF Export Factor". GeneCards.
- ^ Yang HC, Liang YJ, Wu YL, Chung CM, Chiang KM, Ho HY, Ting CT, Lin TH, Sheu SH, Tsai WC, Chen JH, Leu HB, Yin WH, Chiu TY, Chen CI, Fann CS, Wu JY, Lin TN, Lin SJ, Chen YT, Chen JW, Pan WH (2009). "Genome-wide association study of young-onset hypertension in the Han Chinese population of Taiwan". PLOS ONE. 4 (5): e5459. Bibcode:2009PLoSO...4.5459Y. doi:10.1371/journal.pone.0005459. PMC 2674219. PMID 19421330.