Ff 페이저
Ff phagesFf phages(F 특이 필라멘트 파지의 경우)는 F 가임인자를 [1][2][3][4][5][6][7]가진 박테리아에 감염되는 f1, fd, M13 및 ZJ/2 파지를 포함한 거의 동일한 필라멘트 파지의 그룹이다.비리온(바이러스 입자)은 약 6x900nm 크기의 유연한 필라멘트로, 중심부에서 단가닥 원형 DNA 분자를 보호하는 원통형 단백질 튜브로 구성되어 있습니다.파지는 11개의 유전자 제품만을 코드하며 알려진 가장 단순한 바이러스 중 하나이다.그것은 분자생물학의 근본적인 측면을 연구하는 데 널리 사용되어 왔다.George Smith와 Greg Winter는 파지 디스플레이에 대한 연구로 f1과 fd를 사용하여 2018년 노벨 [8]화학상을 수상했다.Ff 파지에 대한 초기 실험은 유전자 [9][10]기능을 식별하기 위해 M13을 사용했고, M13도 복제 [11]매개체로 개발되었기 때문에 M13이라는 이름은 Ff 파지의 전체 그룹의 비공식적인 동의어로 사용되기도 한다.
구조.
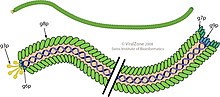
비리온은 직경 6nm, 길이 900nm의 유연한 필라멘트이다.겹치는 대상포진상 배열의 작은(50개의 아미노산 잔류물) 가늘고 긴 알파 나선형 메이저 코트 단백질 서브유닛(유전자 8 또는 p8) 수천 개가 원형 단일사슬 DNA 게놈을 둘러싼 중공 원통을 형성한다.각 p8 서브유닛은 가늘고 긴 단백질의 C말단 부근에 염기성 잔류물 집합과 N말단 부근에 산성 잔류물을 가지고 있으며, 이들 두 영역은 약 20개의 소수성(비극성) 잔류물에 의해 분리되어 있다.대상포진의 배열은 p8의 산성 잔류물을 실린더 외부 표면 근처에 배치하여 바이러스 입자가 음전하를 띠게 하고, 비극성 상호작용이 바이러스 입자의 현저한 물리적 안정성에 기여하는 인접 p8 서브유닛의 비극성 영역 근처의 비극성 영역 및 기본 잔류물을 배치한다.원통 중앙 부근에서 음전하를 띠는 DNA 인산염과 상호작용을 합니다.더[12] 많은 (또는 더 적은) p8 서브유닛이 DNA를 보호하기 위해 필요한 만큼 조립 중에 첨가될 수 있기 때문에, 더 긴 (또는[13] 더 짧은) DNA 분자가 포장될 수 있고, 파지는 유전자 연구에 유용하다.(이 효과는 여러 개의 분리된 별개의 DNA 분자를 포장할 수 있는 폴리파지와 혼동해서는 안 됩니다).약 5개의 작은 단백질 각각이 비리온의 [14]양끝을 덮는다.
Virion Capsid(p8 서브유닛 단백질의 집합체)의 분자 구조는 X선 섬유 회절에 의해 결정되었으며, 구조 모델은 단백질 데이터 뱅크에 축적되었다.특히, 수십 년에 걸쳐 PDB에 축적된 일련의 fd 및 Pf1 비리온 구조는 섬유 회절 데이터 수집 및 계산 분석을 위한 방법의 개선을 보여준다.p3 capsid 단백질 및 p5 복제/조립 단백질의 구조도 X선 결정학에서 결정되어 PDB에 축적되었다.
유전학
fd 게놈의 DNA 배열은 9개의 유전자로 구성된 6408개의 뉴클레오티드를 가지고 있지만 유전자 2와 유전자 1의 내부 번역이 시작돼 p10과 p11이라는 두 개의 단백질이 추가로 생성되기 때문에 게놈은 11개의 단백질을 생성하는 11개의 열린 읽기 프레임을 가지고 있다.게놈은 또한 짧은 비코드 유전자 [15]간 염기서열을 포함한다.M13 및 f1 시퀀스는 fd와 약간 다릅니다.둘 다 6407개의 뉴클레오티드를 가지고 있다; f1은 180개의 위치에서 fd와 다르고, M13은 [16]f1과 59개의 뉴클레오티드 차이만 가지고 있다.많은 목적으로 Ff 그룹의 페이지는 교환 가능한 것으로 간주할 수 있다.
5개의 유전자 생성물은 바이러스온의 일부이다: 주요 외피 단백질과 양끝을 덮는 작은 단백질, 한쪽 끝은 p3와 p6, 다른 끝은 p7과 p9.세 가지 유전자 생성물(p2, p5, p10)은 DNA 합성에 필요한 세포질 단백질이며, 나머지는 비리온 [17]조립에 관여하는 막 단백질이다.
| 이노바이러스 | |
|---|---|
| 바이러스 분류 | |
| (순위 미지정): | 바이러스 |
| 영역: | 모노나비리아속 |
| 왕국: | 뢰브바이러스속 |
| 문: | 호프네이비리코타 |
| 클래스: | 파세르비케테스 |
| 주문: | 투불라비랄레스 |
| 패밀리: | 이노바이러스과 |
| 속: | 이노바이러스 |
p1을 코드하는 유전자는 미생물의 [18]게놈에서 10000개 이상의 이노바이러스 유사 서열을 식별하는 자동 기계 학습 접근에서 이노바이러스 게놈에 특정한 세 가지 특징과 함께 보존된 마커 유전자로 사용되어 왔다.
라이프 사이클
감염
p3 단백질은 p3의 C 말단 도메인에 의해 비리온의 한쪽 끝에 고정된다.숙주 박테리아의 감염은 p3의 두 개의 다른 N 말단 영역과 숙주 박테리아의 두 개의 다른 부위의 상호작용을 포함한다.우선 p3의 N2 도메인이 F필러스의 외측에 부착되어 필러스가 세포 내로 후퇴한다.이 후퇴는 필러스 성장 및 중합 과정의 [1][19][20]역전에 의해 필러스 서브유닛 어셈블리의 필러스 베이스의 세포막으로의 탈중합이 수반될 수 있습니다.p3를 가진 필러스의 끝이 세포벽에 가까워지면 p3의 N1 도메인은 세균성 TolQRA 단백질과 상호작용하여 감염을 완료하고 게놈을 [21][22]숙주의 세포질로 방출한다.
레플리케이션
단가닥 바이러스 DNA가 세포질에 들어간 후 상보적인 DNA 가닥의 합성을 위한 템플릿으로 기능한다.이 합성은 숙주 RNA 중합효소에 의해 DNA 배열의 유전자 간 영역에서 시작되며, 이것은 템플릿으로 감염 DNA에 짧은 RNA 프라이머를 합성한다.숙주 DNA 중합효소 III는 이 프라이머를 사용하여 DNA의 완전한 상보적 가닥을 합성하고, 때로는 복제 형태(RF) DNA라고 불리는 이중 가닥 원을 생성합니다.RF의 상보적인 가닥은 추가적인 DNA 복제에 필요한 파지 코드 단백질, 특히 p2와 p10에 대한 전사 템플릿이다.
p2 단백질은 RF DNA의 바이러스 가닥을 절단하고 숙주 DNA 중합효소 III는 새로운 바이러스 가닥을 합성합니다.오래된 바이러스 사슬은 새로운 바이러스 사슬이 합성되면서 대체된다.원이 완성되면 공유결합된 p2는 오래된 DNA와 새로 합성된 DNA의 접합부에서 치환된 바이러스 가닥을 절단하고 양끝을 재결합시켜 p2를 해방시킨다.이 롤링 서클메커니즘에 의해 RF가 복제되어 수십 개의 RF 복사가 생성됩니다.
파지 단백질의 농도가 증가하면 새로운 바이러스 가닥은 상보적인 DNA 가닥이 아닌 복제/조립 단백질 p5로 코팅된다.p5는 또한 p2의 번역을 억제하여 자손 바이러스 ssDNA의 생산과 패키징이 동시에 [6]이루어지도록 한다.
조립 및 압출
대부분의 다른 파지 계열과는 달리, 감염은 숙주 [23]박테리아를 죽이지 않는다.자손의 파지는 아마도 안쪽과 바깥쪽 막을 연결하는 접착 부위에서 자라나는 박테리아의 막을 통해 돌출될 때 조립됩니다.완성된 파지의 외피를 형성하는 5개의 파지 단백질은 내부 막으로 들어간다. p8과 p3의 경우, N-termini가 세포질에서 주변으로 향하는 방향과 함께 N-termini가 박테리아 막으로 들어가는 것을 돕는다.파지에 존재하지 않는 세 가지 다른 파지막 단백질인 p1, p11 및 p4도 조립에 관여한다.RF DNA의 복제는 p5로 DNA를 코팅함으로써 page ssDNA의 생산으로 변환되어 가늘고 긴 p5/DNA 복제/조립 복합체를 형성하고, 이어서 막결합 파지 단백질과 상호작용한다.압출 과정은 자손 파지의 바깥쪽 끝을 형성하는 p7과 p9 단백질을 집어 올린다.p5가 DNA에서 제거됨에 따라 자손 DNA가 막에서 압출되어 p8의 나선형 케이싱에 싸여 조립 마지막에 p3, p6가 첨가된다.p4 단백질은 [14]외막에서 압출 기공을 형성할 수 있다.
호스트 내막에서의 p1-티오레독신 착체와 이중사슬 포장 DNA 신호의 상호작용에 의해 모공 형성이 트리거된다.p1 단백질은 파지 [24]조립에 필수적인 워커 모티브를 포함하고 있어 p1이 파지 조립에 관여하는 분자 모터임을 시사합니다.p1 단백질은 세포질의 N-말단 부분과 periplasm의 C-말단 부분(p8의 방향의 반대)과 함께 막 스팬 소수성 도메인을 가진다.막스패닝 도메인의 세포질 측에는 p8의 C 말단 부근에 있는 염기성 잔기의 패턴과 밀접하게 일치하는 염기성 잔기의 패턴을 가진 p1의 13-잔기 배열이 있지만, [25]그 배열에 대해서는 반전되어 있다.
파지를 [26][27][28]클로로포름으로 처리함으로써 p8의 중간조립체를 생성할 수 있다.이러한 중간 형태에서 p8의 나선 함량은 파지와 유사하며, 조립 중 구조 변화는 조립 [29][30]내 인접 p8 서브유닛에 대한 미끄러짐만 수반할 수 있음을 시사한다.
적용들
생명과학 및 의학
Ff phages는 생물학 및 의학 분야 응용을 위해 설계되었습니다.많은 어플리케이션은 항생제 카나마이신에 대한 내성을 결정하는 DNA 배열을 기능적 형태로 fd page DNA의 비부호화 유전자 배열에 삽입할 수 있다는 것을 보여주는 실험에[12] 기초하고 있다.이와 같이 변형된 파지는 야생형 필라멘트 fd보다 상대적으로 긴데, 이는 DNA가 대응하는 더 많은 유전자 8로 코팅되어 단백질을 코팅하고 있기 때문이다.그러나 파지의 라이프 사이클은 다른 방법으로 중단되지 않는다.반면, 제한된 크기의 캡시드를 가진 전통적인 "태드폴" 또는 등각선 모양의 화상은 더 큰 DNA 분자를 캡슐화하는데 그렇게 쉽게 사용될 수 없습니다.변형된 파지는 카나마이신에 대한 내성을 도입하기 위해 변형된 파지로 카나마이신 감수성 박테리아를 감염시키고, 감염된 박테리아를 카나마이신 치사 농도를 포함한 배지에서 성장시킴으로써 선택될 수 있다.
이 결과는 외래펩타이드를 발현하는 외래DNA를 유전자 간 배열이 아닌 fd phage 유전자 3에 삽입함으로써 확장되어 외래펩타이드가 유전자 3 흡착 [31][32][33]단백질의 일부로 파지 표면에 나타나도록 하였다.다음으로 적절한 항체를 사용하여 외래펩타이드를 운반하는 파지를 검출할 수 있다.이 접근법의 반대는 항체를 코드하는 DNA를 유전자 3에 삽입하고 적절한 [34]항원에 의해 항체의 존재를 검출하는 것이다.
이러한 기술은 예를 들어 유전자 3 이외의 파지코트 단백질을 코드하는 유전자에 외래 DNA를 삽입하거나 관심 유전자를 복제하여 대응하는 유전자 생성물의 일부만 수정함으로써 여러 가지 방법으로 확장되어 왔다.파지 디스플레이 기술은 다양한 용도로 널리 사용되어 왔다.[35][36][37]
재료과학 및 나노테크놀로지
Ff 페이지는 교정, 전기화학, 태양광 발전, 촉매, 감지 및 디지털 메모리 장치, 특히 Angela Belcher와 동료들에 의해 설계되었습니다.[6][38][39][40][41][42][43][44][45]
「 」를 참조해 주세요.
레퍼런스
- ^ a b Rasched I, Oberer E (December 1986). "Ff coliphages: structural and functional relationships". Microbiological Reviews. 50 (4): 401–27. doi:10.1128/MR.50.4.401-427.1986. PMC 373080. PMID 3540571.
- ^ Mai-Prochnow A, Hui JG, Kjelleberg S, Rakonjac J, McDougald D, Rice SA (July 2015). "'Big things in small packages: the genetics of filamentous phage and effects on fitness of their host'". FEMS Microbiology Reviews. 39 (4): 465–87. doi:10.1093/femsre/fuu007. PMID 25670735.
- ^ Rakonjac J, Bas B, Derda R, eds. (2017). Filamentous Bacteriophage in Bio/Nano/Technology, Bacterial Pathogenesis and Ecology. Frontiers Research Topics. Frontiers Media SA. doi:10.3389/978-2-88945-095-4. ISBN 978-2-88945-095-4.
- ^ Morag O, Abramov G, Goldbourt A (December 2011). "Similarities and differences within members of the Ff family of filamentous bacteriophage viruses". The Journal of Physical Chemistry B. 115 (51): 15370–9. doi:10.1021/jp2079742. PMID 22085310.
- ^ Hay ID, Lithgow T (June 2019). "Filamentous phages: masters of a microbial sharing economy". EMBO Reports. 20 (6). doi:10.15252/embr.201847427. PMC 6549030. PMID 30952693.
- ^ a b c Rakonjac J, Bennett NJ, Spagnuolo J, Gagic D, Russel M (2011). "Filamentous bacteriophage: biology, phage display and nanotechnology applications". Current Issues in Molecular Biology. 13 (2): 51–76. PMID 21502666.
- ^ Rakonjac J, Russel M, Khanum S, Brooke SJ, Rajič M (2017). Lim TS (ed.). "Filamentous Phage: Structure and Biology". Advances in Experimental Medicine and Biology. Cham: Springer International Publishing. 1053: 1–20. doi:10.1007/978-3-319-72077-7_1. ISBN 978-3-319-72076-0. PMID 29549632.
- ^ "The Nobel Prize in Chemistry 2018". NobelPrize.org. Retrieved 2021-04-10.
- ^ Pratt D, Tzagoloff H, Erdahl WS (November 1966). "Conditional lethal mutants of the small filamentous coliphage M13. I. Isolation, complementation, cell killing, time of cistron action". Virology. 30 (3): 397–410. doi:10.1016/0042-6822(66)90118-8. PMID 5921643.
- ^ Pratt D, Tzagoloff H, Beaudoin J (September 1969). "Conditional lethal mutants of the small filamentous coliphage M13. II. Two genes for coat proteins". Virology. 39 (1): 42–53. doi:10.1016/0042-6822(69)90346-8. PMID 5807970.
- ^ Messing J (April 1991). "Cloning in M13 phage or how to use biology at its best". Gene. 100: 3–12. doi:10.1016/0378-1119(91)90344-b. PMID 2055478.
- ^ a b Herrmann R, Neugebauer K, Zentgraf H, Schaller H (February 1978). "Transposition of a DNA sequence determining kanamycin resistance into the single-stranded genome of bacteriophage fd". Molecular & General Genetics. 159 (2): 171–8. doi:10.1007/BF00270890. PMID 345091. S2CID 22923713.
- ^ Sattar S, Bennett NJ, Wen WX, Guthrie JM, Blackwell LF, Conway JF, Rakonjac J (2015). "Ff-nano, short functionalized nanorods derived from Ff (f1, fd, or M13) filamentous bacteriophage". Frontiers in Microbiology. 6: 316. doi:10.3389/fmicb.2015.00316. PMC 4403547. PMID 25941520.
- ^ a b Straus SK, Bo HE (2018). "Filamentous Bacteriophage Proteins and Assembly". Sub-Cellular Biochemistry. 88: 261–279. doi:10.1007/978-981-10-8456-0_12. ISBN 978-981-10-8455-3. PMID 29900501.
- ^ Beck E, Sommer R, Auerswald EA, Kurz C, Zink B, Osterburg G, et al. (December 1978). "Nucleotide sequence of bacteriophage fd DNA". Nucleic Acids Research. 5 (12): 4495–503. doi:10.1093/nar/5.12.4495. PMC 342768. PMID 745987.
- ^ Beck E, Zink B (December 1981). "Nucleotide sequence and genome organisation of filamentous bacteriophages fl and fd". Gene. 16 (1–3): 35–58. doi:10.1016/0378-1119(81)90059-7. PMID 6282703.
- ^ Russel M, Linderoth NA, Sali A (June 1997). "Filamentous phage assembly: variation on a protein export theme". Gene. 192 (1): 23–32. doi:10.1016/s0378-1119(96)00801-3. PMID 9224870.
- ^ Roux S, Krupovic M, Daly RA, Borges AL, Nayfach S, Schulz F, et al. (November 2019). "Cryptic inoviruses revealed as pervasive in bacteria and archaea across Earth's biomes". Nature Microbiology. 4 (11): 1895–1906. doi:10.1038/s41564-019-0510-x. PMC 6813254. PMID 31332386.
- ^ Lawley TD, Klimke WA, Gubbins MJ, Frost LS (July 2003). "F factor conjugation is a true type IV secretion system". FEMS Microbiology Letters. 224 (1): 1–15. doi:10.1016/S0378-1097(03)00430-0. PMID 12855161.
- ^ Craig L, Forest KT, Maier B (July 2019). "Type IV pili: dynamics, biophysics and functional consequences". Nature Reviews. Microbiology. 17 (7): 429–440. doi:10.1038/s41579-019-0195-4. PMID 30988511. S2CID 115153017.
- ^ Bennett NJ, Rakonjac J (February 2006). "Unlocking of the filamentous bacteriophage virion during infection is mediated by the C domain of pIII". Journal of Molecular Biology. 356 (2): 266–73. doi:10.1016/j.jmb.2005.11.069. PMID 16373072.
- ^ Hoffmann-Thoms S, Jakob RP, Schmid FX (April 2014). "Energetic communication between functional sites of the gene-3-protein during infection by phage fd". Journal of Molecular Biology. 426 (8): 1711–22. doi:10.1016/j.jmb.2014.01.002. PMID 24440124.
- ^ Hoffmann Berling H, Maze R (March 1964). "Release of male-specific bacteriophages from surviving host bacteria BACTERIA". Virology. 22 (3): 305–13. doi:10.1016/0042-6822(64)90021-2. PMID 14127828.
- ^ Loh B, Haase M, Mueller L, Kuhn A, Leptihn S (April 2017). "The Transmembrane Morphogenesis Protein gp1 of Filamentous Phages Contains Walker A and Walker B Motifs Essential for Phage Assembly". Viruses. 9 (4): 73. doi:10.3390/v9040073. PMC 5408679. PMID 28397779.
- ^ Rapoza MP, Webster RE (May 1995). "The products of gene I and the overlapping in-frame gene XI are required for filamentous phage assembly". Journal of Molecular Biology. 248 (3): 627–38. doi:10.1006/jmbi.1995.0247. PMID 7752229.
- ^ Griffith J, Manning M, Dunn K (March 1981). "Filamentous bacteriophage contract into hollow spherical particles upon exposure to a chloroform-water interface". Cell. 23 (3): 747–53. doi:10.1016/0092-8674(81)90438-4. PMID 7226228. S2CID 46531024.
- ^ Manning M, Griffith J (January 1985). "Association of M13 I-forms and spheroids with lipid vesicles". Archives of Biochemistry and Biophysics. 236 (1): 297–303. doi:10.1016/0003-9861(85)90629-0. PMID 3966795.
- ^ Stopar D, Spruijt RB, Wolfs CJ, Hemminga MA (July 1998). "Mimicking initial interactions of bacteriophage M13 coat protein disassembly in model membrane systems". Biochemistry. 37 (28): 10181–7. doi:10.1021/bi9718144. PMID 9665724.
- ^ Roberts LM, Dunker AK (October 1993). "Structural changes accompanying chloroform-induced contraction of the filamentous phage fd". Biochemistry. 32 (39): 10479–88. doi:10.1021/bi00090a026. PMID 8399194.
- ^ Xue, Bin; Blocquel, David; Habchi, Johnny; Uversky, Alexey V.; Kurgan, Lukasz; Uversky, Vladimir N.; Longhi, Sonia (2014). "Structural Disorder in Viral Proteins". Chemical Reviews. 114 (13): 6880–6911. doi:10.1021/cr4005692. ISSN 0009-2665. PMID 24823319.
- ^ Smith, G. (1985). "Filamentous fusion phage: novel expression vectors that display cloned antigens on the virion surface". Science. 228 (4705): 1315–1317. Bibcode:1985Sci...228.1315S. doi:10.1126/science.4001944. ISSN 0036-8075. PMID 4001944.
- ^ Parmley, Stephen F.; Smith, George P. (1988). "Antibody-selectable filamentous fd phage vectors: affinity purification of target genes". Gene. 73 (2): 305–318. doi:10.1016/0378-1119(88)90495-7. ISSN 0378-1119. PMID 3149606.
- ^ 웹스터, R.E., 2001.필라멘트 파지 생물학.입력: Barbas III, C.F., Burton, D.R., Scott, J.K, Silverman, G.J. (Ed.) , Page Display: 실험실 매뉴얼.콜드 스프링 하버 연구소 프레스, 뉴욕, 콜드 스프링 하버, 1.1-1.37페이지.
- ^ Winter, Greg; Griffiths, Andrew D.; Hawkins, Robert E.; Hoogenboom, Hennie R. (1994). "Making Antibodies by Phage Display Technology". Annual Review of Immunology. 12 (1): 433–455. doi:10.1146/annurev.iy.12.040194.002245. ISSN 0732-0582. PMID 8011287.
- ^ Prisco A, De Berardinis P (2012). "Filamentous bacteriophage fd as an antigen delivery system in vaccination". International Journal of Molecular Sciences. 13 (4): 5179–94. doi:10.3390/ijms13045179. PMC 3344273. PMID 22606037.
- ^ Henry KA, Arbabi-Ghahroudi M, Scott JK (2015). "Beyond phage display: non-traditional applications of the filamentous bacteriophage as a vaccine carrier, therapeutic biologic, and bioconjugation scaffold". Frontiers in Microbiology. 6: 755. doi:10.3389/fmicb.2015.00755. PMC 4523942. PMID 26300850.
- ^ Sioud M (April 2019). "Phage Display Libraries: From Binders to Targeted Drug Delivery and Human Therapeutics". Molecular Biotechnology. 61 (4): 286–303. doi:10.1007/s12033-019-00156-8. PMID 30729435. S2CID 73434013.
- ^ Dogic Z (2016). "Filamentous Phages As a Model System in Soft Matter Physics". Frontiers in Microbiology. 7: 1013. doi:10.3389/fmicb.2016.01013. PMC 4927585. PMID 27446051.
- ^ Oh D, Qi J, Lu YC, Zhang Y, Shao-Horn Y, Belcher AM (2013). "Biologically enhanced cathode design for improved capacity and cycle life for lithium-oxygen batteries". Nature Communications. 4 (1): 2756. Bibcode:2013NatCo...4.2756O. doi:10.1038/ncomms3756. PMC 3930201. PMID 24220635.
- ^ Dorval Courchesne NM, Klug MT, Huang KJ, Weidman MC, Cantú VJ, Chen PY, et al. (2015). "Constructing Multifunctional Virus-Templated Nanoporous Composites for Thin Film Solar Cells: Contributions of Morphology and Optics to Photocurrent Generation". The Journal of Physical Chemistry C. 119 (25): 13987–14000. doi:10.1021/acs.jpcc.5b00295. hdl:1721.1/102981. ISSN 1932-7447.
- ^ Lee SW, Belcher AM (2004). "Virus-Based Fabrication of Micro- and Nanofibers Using Electrospinning". Nano Letters. 4 (3): 387–390. Bibcode:2004NanoL...4..387L. doi:10.1021/nl034911t. ISSN 1530-6984.
- ^ Casey JP, Barbero RJ, Heldman N, Belcher AM (November 2014). "Versatile de novo enzyme activity in capsid proteins from an engineered M13 bacteriophage library". Journal of the American Chemical Society. 136 (47): 16508–14. doi:10.1021/ja506346f. PMID 25343220.
- ^ Zhang G, Wei S, Belcher AM (2018). "Biotemplated Zinc Sulfide Nanofibers as Anode Materials for Sodium-Ion Batteries". ACS Applied Nano Materials. 1 (10): 5631–5639. doi:10.1021/acsanm.8b01254. hdl:1721.1/126086. ISSN 2574-0970. S2CID 104742577.
- ^ Li L, Belcher AM, Loke DK (December 2020). "Simulating selective binding of a biological template to a nanoscale architecture: a core concept of a clamp-based binding-pocket-favored N-terminal-domain assembly". Nanoscale. 12 (47): 24214–24227. doi:10.1039/D0NR07320B. PMID 33289758. S2CID 227950477.
- ^ Brogan AP, Heldman N, Hallett JP, Belcher AM (September 2019). "Thermally robust solvent-free biofluids of M13 bacteriophage engineered for high compatibility with anhydrous ionic liquids". Chemical Communications. 55 (72): 10752–10755. doi:10.1039/C9CC04909F. hdl:1721.1/125988. PMID 31432818. S2CID 201115233.