페이지 디스플레이
Phage display이 기사는 대부분의 독자들이 이해하기에는 너무 전문적일 수 있다.하지 않고 도쉽게 할 수 있도록 해 . (2018년 10월 (이 및 ) |
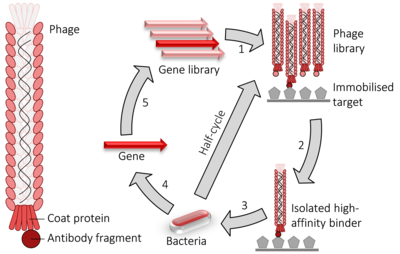
파지 디스플레이는 단백질-단백질, 단백질-펩타이드 및 단백질 연구를 위한 실험실 기술이다.박테리오파지를 사용하여 단백질을 [1]암호화하는 유전자 정보와 연결하는 DNA 상호작용.본 발명은 관심단백질을 코드하는 유전자를 파지코트단백질유전자에 삽입함으로써 파지코트단백질유전자가 내부에 포함된 상태에서 파지가 그 단백질을 외부에 '표시'하게 함으로써 유전자형과 표현형과의 연관성을 가져온다.표시된 단백질과 다른 분자 간의 상호작용을 검출하기 위해 이러한 표시된 페이지는 다른 단백질, 펩타이드 또는 DNA 배열에 대해 선별될 수 있다.이러한 방법으로, 단백질의 큰 라이브러리는 체외 선택이라고 불리는 과정에서 선별되고 증폭될 수 있는데, 이것은 자연 선택과 유사하다.
파지 디스플레이에 가장 많이 사용되는 박테리오파지는 M13 및 fd 필라멘트 파지이며,[2][3] [4]T4, T7, δ 파지도 사용되었습니다.
역사
파지 디스플레이는 George P에 의해 처음 설명되었습니다. 1985년 스미스 박사는 펩타이드 배열 [1]중 하나의 펩타이드로 바이러스의 캡시드 단백질을 융합시켜 필라멘트 파지(세균을 감염시키는 길고 얇은 바이러스)에 펩타이드의 표시를 시연했다.이를 통해 바이러스 클론 수집의 외부 표면에 서로 다른 펩타이드가 표시되었으며, 이 과정에서 프로세스의 스크리닝 단계는 결합 친화력이 가장 높은 펩타이드를 분리하였다.1988년, 스티븐 팜리와 조지 스미스는 친화력 선택을 위한 바이오패닝을 설명했고 재귀적 선택 라운드가 10억분의 [5]1 이하로 존재하는 클론에 대해 풍부하게 할 수 있다는 것을 증명했다.1990년에 제이미 스콧과 조지 스미스는 필라멘트 파지에 표시된 [6]큰 랜덤 펩타이드 라이브러리의 생성을 설명했다.파지 디스플레이 기술은 그렉 윈터, 존 맥카퍼티와 함께 분자생물학 연구소, 리처드 러너, 카를로스 바바스와 함께 스크립스 연구소, 항체 치료용 단백질 디스플레이를 위해 프랭크 브레이틀링과 스테판 뒤벨과 함께 독일 암 연구 센터의 그룹에 의해 더욱 개발되고 개선되었습니다.c단백질공학스미스와 윈터는 파지 [7]디스플레이 개발에 기여한 공로로 2018년 노벨 화학상 절반의 상을 받았다.1985년에 우선권을 주장하는 조지 픽제닉의 특허도 펩타이드 [8]라이브러리의 생성을 기술하고 있습니다.
원칙
2-하이브리드 시스템과 마찬가지로 파지 디스플레이는 단백질 상호작용의 높은 처리량 스크리닝에 사용됩니다.M13 필라멘트 파지 디스플레이의 경우 관심 단백질 또는 펩타이드를 코드하는 DNA가 pII 또는 pVII 유전자에 결합되어 각각 마이너 또는 메이저 코트 단백질을 코드한다.cDNA fragment가 적절한 프레임으로 변환되도록 fragment가 3개의 판독 프레임 모두에 삽입되도록 하기 위해 여러 복제 사이트가 사용되는 경우가 있습니다.파지 유전자와 삽입 DNA 하이브리드는 TG1, SS320, ER2738 또는 XL1-Blue E. coli와 같은 대장균 세포에 삽입됩니다."phagemid" 벡터를 사용하는 경우(단순한 디스플레이 구성 벡터) 파지 입자는 도우미 파지에 감염될 때까지 대장균 세포에서 방출되지 않으며, 이는 파지 DNA의 포장 및 마이너(pII) 또는 메이저 외피의 일부인 관련 단백질 단편과 성숙한 비리온의 조립을 가능하게 한다.(pVII) 외피 단백질.해당 DNA 또는 단백질 타깃을 마이크로티터 플레이트 표면에 고정함으로써 이들 타깃 중 하나에 결합하는 단백질을 표면에 표시하는 파지가 유지되고 다른 타깃은 세척으로 제거된다.남은 파지는 용출되어 더 많은 파지를 생산하고(도우미 파지에 의한 세균 감염에 의해) 관련 파지로 농축된 파지 혼합물을 생성하는 데 사용될 수 있다.이러한 단계를 반복하여 순환하는 것을 '패닝'이라고 하며, 이는 바람직하지 않은 물질을 제거하여 금 샘플을 농축하는 것을 말합니다.마지막 단계에서 용출된 파지는 적절한 세균 숙주를 감염시키기 위해 사용될 수 있으며, 파지미드는 여기에서 수집될 수 있으며 관련 DNA 배열을 제거 및 배열하여 관련된 상호작용 단백질 또는 단백질 단편을 식별할 수 있다.
'세균포장세포주' [9]기술을 이용하면 도우미 파지의 사용을 없앨 수 있습니다.
용출은 저pH 용출 완충제와 소닌화를 조합하여 이루어지며, 이는 펩타이드-타겟 상호작용을 완화하는 것 외에 표적 분자를 고정화 표면에서 분리하는 역할을 한다.이 초음파 기반 방법은 고친화성 [10]펩타이드의 단일 단계 선택을 가능하게 한다.
적용들
파지 표시 기술의 적용에는 단백질의 기능 또는 기능의 메커니즘을 [11]결정할 수 있도록 단백질의 상호작용 파트너(세포, 조직 또는 유기체의 모든 코드 배열로 구성된 DNA 라이브러리와 고정화된 파지 "bait"으로 사용됨)의 결정이 포함된다.파지 디스플레이는 또한 시험관 내 단백질 진화(단백질 공학이라고도 함)에 널리 사용되는 방법이다.따라서 파지 디스플레이는 약물 발견에 유용한 도구이다.단백질 [12][13][14]표적에 대한 새로운 리간드(효소 억제제, 수용체 작용제 및 길항제)를 찾는 데 사용됩니다.또한 이 기술은 종양 항원을 결정하고(진단 및 치료 대상화),[15] 무작위화된 세그먼트를 가진 특수하게 구성된 DNA 라이브러리를 사용하여 단백질-DNA 상호작용을[16] 탐색하는 데에도 사용된다.최근에는 입양 세포 이동 [17]접근법과 같은 암 치료의 맥락에서도 파지 디스플레이가 사용되고 있다.이러한 경우, 파지 디스플레이는 종양 표면 단백질을 [17]표적으로 하는 합성 항체를 생성하고 선택하는 데 사용됩니다.이것들은 환자로부터 수집된 T세포를 위한 합성 수용체로 만들어지며,[18] 이는 질병과 싸우기 위해 사용된다.
시험관내 단백질 진화를 위한 경쟁 방법으로는 효모 표시, 세균 표시, 리보솜 표시 및 mRNA 표시 등이 있다.
시험관내 항체 성숙
항체 파지의 발명은 혁신적인 항체 약물 발견을 보여준다.초기 작업은 MRC 분자생물학 연구소(Greg Winter and John McCafferty), 스크립스 연구소(Richard Lerner and Carlos F)의 실험실들에 의해 수행되었다.Barbas) 및 독일 암 연구 센터(Frank Breitling 및 Stefan Durbel).[19][20][21]1991년, Scripps 그룹은 [22]파지에 인간 항체의 첫 표시와 선택을 보고했다.이 초기 연구는 파상풍 독소를 결합하는 인간 항체 Fab의 신속한 분리를 기술한 후 백신 설계 및 [23][24][25][26][27]치료를 위해 인간 항HIV-1 항체를 신속하게 복제하도록 방법을 확장하였다.
항체 라이브러리의 파지 표시는 면역 반응을 연구하는 동시에 치료를 위해 인간 항체를 신속하게 선택하고 진화시키는 강력한 방법이 되었다.항체 파지 디스플레이는 나중에 Carlos F.에 의해 사용되었다.The Scripps Institute의 Barbas는 Breitling과 동료들에 의해 1990년에 처음 특허받은 원리인 합성 인간 항체 라이브러리를 만들어, 합성 다양성 [28][29][30][31]요소로부터 시험관내로 인간 항체를 만들 수 있게 했다.
파지에 수백만 개의 다른 항체를 표시하는 항체 라이브러리는 주로 항암 또는 항염증 치료제로 항체 약물로 개발하기 위해 고도로 특정한 치료용 항체 리드를 분리하기 위해 제약 산업에서 종종 사용됩니다.가장 성공적인 것 중 하나는 Adalimumab으로, Cambridge Antivid Technology에 의해 D2E7로 발견되고 Abbott Laboraties에 의해 개발 및 판매되었습니다.TNF 알파에 대한 항체인 아달리미맵은 세계 최초로 연간 매출 [33]10억 달러를 돌파한 완전 인간[32] 항체였다.
일반 프로토콜
다음은 원하는 표적 단백질 또는 DNA 염기서열에 높은 친화력으로 결합하는 폴리펩타이드를 식별하기 위해 파지 디스플레이 스크리닝에서 이어지는 일련의 사건이다.
- 표적 단백질 또는 DNA 배열을 마이크로미터 플레이트의 웰에 고정시킨다.
- 많은 유전자 배열이 박테리오파지 라이브러리에서 박테리오파지 피막 단백질과의 융합 형태로 발현되어 바이러스 입자의 표면에 표시된다.표시된 단백질은 파지 내 유전자 염기서열과 일치합니다.
- 이 파지 표시 라이브러리는 접시에 추가되어 파지 시간을 두고 접시를 씻습니다.
- 표적 분자와 상호작용하는 파지를 나타내는 단백질은 접시에 붙어 있는 반면, 다른 모든 것들은 씻겨 내려갑니다.
- 부착된 파지는 용출되어 적절한 세균 숙주의 감염에 의해 더 많은 파지를 생성하기 위해 사용될 수 있다.새로운 파지는 농축된 혼합물을 구성하며, 초기 혼합물에 존재했던 것보다 관련 없는 파지(즉, 비결합성)를 상당히 적게 포함한다.
- 스텝 3~5를 선택적으로 1회 이상 반복함으로써 결합단백질의 파지 라이브러리를 더욱 풍부하게 한다.
- 또한 세균 기반의 증폭에 이어 상호작용하는 파지 내의 DNA를 배열하여 상호작용하는 단백질 또는 단백질 단편을 동정한다.
피복단백질 선택
필라멘트 페이저
pII
pII는 비리온의 감염성을 결정하는 단백질로, pII는 글리신이 풍부한 [34]링커에 의해 연결된 3개의 도메인(N1, N2, CT)으로 구성되어 있다.N2 도메인은 바이러스 감염 동안 F필러스에 결합하고 N1 도메인은 [34]박테리아의 표면에서 TolA 단백질과 상호작용하는 N1 도메인을 해방시킵니다.이 단백질 내의 삽입은 보통 위치 249(CT와 N2 사이의 링커 영역 내), 위치 198(N2 도메인 내) 및 N 말단(N 말단 분비 배열과 [34]PII의 N 말단 사이에 삽입됨)에 첨가된다.단, Bam을 사용하는 경우위치 198에 위치한 HI 사이트는 pIII의 [34]비절단 버전을 사용하는 경우 파지 디스플레이 중에 문제를 일으킬 수 있는 비페어 시스테인 잔류물(C201)에 주의해야 합니다.
pVII가 아닌 pII를 사용하는 장점은 pagemid(Ff phages에서 파생된 플라스미드)를 도우미 파지와 조합하여 사용할 때 pII가 단일값 표시를 허용한다는 것이다.또한 pII는 더 큰 단백질 배열(>100 아미노산)[35]을 삽입할 수 있으며 pVII보다 더 내성이 있다.단, p를 사용하여III 융합 파트너로서 파지 감염성의 저하로 인해 파지 성장률의[36] 차이로 인한 선택편향 등의 문제가 발생할 수 있으며,[34] 나아가서는 숙주를 감염시킬 수 없다.야생형 및 융접 [34]pII를 모두 포함하도록 파지미드 플라스미드와 도우미 파지를 사용함으로써 파지 감염성의 손실을 회피할 수 있다.
cDNA는 또한 p를 사용하여 분석되었다.두 개의 상보적인 류신 지퍼 [37]시스템인 Direct Interaction[38] Rescue를 통해 또는 C [39]말단에서 cDNA와 pII 사이에 8-10 아미노산 결합제를 추가하여 III.
pVII
pVii는 Ff phages의 주피막 단백질이다.펩타이드는 보통 pVII의 [34]N 말단에 융합된다.보통 pVII에 융합될 수 있는 펩타이드는 [34]6~8개의 아미노산 길이이다.크기 제한은 추가된[40] 섹션으로 인한 구조적 장애보다는 코팅 단백질 [40]수출 시 pIV로 인한 크기 제외와 더 관련이 있는 것으로 보인다.전형적인 화지에 약 2700개의 단백질 복사가 있기 때문에, 파제미드가 [34]사용되더라도 관심 단백질이 다가적으로 발현될 가능성이 더 높다.이것은 이 단백질의 사용을 고친화성 결합 [34]파트너의 발견에 불리하게 만든다.
pVII의 크기 문제를 극복하기 위해 인공피복단백질이 [41]설계되었다.예를 들어 바이스와 시두의 역인공피막단백질(ACP)[41]은 C 말단에 큰 단백질을 표시할 수 있게 해준다.그러나 ACP는 낮은 수준(대부분 [41]단가만)에서만 20kDa의 단백질을 표시할 수 있었다.
pVI
pVI는 cDNA [34]라이브러리의 표시에 널리 사용되고 있습니다.상호 작용 단백질과 펩티드의 높은 처리량 능력 등의 탐색을 위한 가장yeast-2-hybrid 메서드에cDNA 도서관의 파지 디스플레이를 통해 진열품은 매력적인 대안이다.[34]pVI를 우선적으로 때문에 누군가는 시말단에 대한 대상 단백질을 보충할 수 있고 pIII 상보적 DNA도서관을 표현하기 위하pVIII로 이용되어 왔다.pVI가 page 어셈블리에서 차지하는 역할에 큰 영향을 주지 않고 pVI를 사용할 수 있습니다.즉, cDNA의 stop codon이 문제가 [42]되지 않게 되었습니다.그러나, cDNA의 파지 표시는 진핵세포에 존재하는 번역 후 변형을 생성하는데 있어 대부분의 원핵생물들의 무능력 또는 다중 도메인 단백질의 잘못된 접힘에 의해 항상 제한된다.
pVI는 cDNA 라이브러리 분석에 유용했지만, pIII와 pVIII는 파지 표시에 [34]가장 많이 사용되는 코팅 단백질로 남아 있다.
pVII 및 pIX
1995년 실험에서 pVII와 pIX 모두에서 글루타치온 S-전달효소 표시가 시도되었으나 실패했다.[43]그러나 N 말단에 [44]periplasmic 신호 배열(pelB 또는 ompA)을 추가한 후 이 단백질의 파지 표시가 성공적으로 완료되었다.최근 연구에서 AviTag, FLAG 및 His가 신호 시퀀스 없이도 pVII에 표시될 수 있는 것으로 나타났습니다.그 후 단일 사슬 Fv(scFv)와 단일 사슬 T세포 수용체(scTCR)의 발현을 신호 [45]배열 유무에 관계없이 발현시켰다.
PelB(신호 펩티드가 분해되는 Periplasm에 단백질을 표적으로 하는 아미노산 신호 배열)는 신호 배열이 없는 pVi 및 pIX 융합에 비해 파지 표시 수준을 향상시켰다.그러나 이것은 파제미드 게놈보다는 도우미 파지 게놈을 더 많이 포함시켰다.모든 경우에서 파지 표시 수준은 pII 융접을 사용하는 것보다 낮았다.그러나 낮은 디스플레이는 진정한 1가 디스플레이에 가깝기 때문에 낮은 디스플레이가 바인더 선택에 더 유리할 수 있습니다.6번 중 5번에서 펠B가 없는 pVii 및 pIX 융합이 친화력 선택 분석에서 pII 융합보다 더 효율적이었다.이 문서에서는 pVII 및 pIX 디스플레이 플랫폼이 장기적으로 [45]pII를 능가할 수도 있다고 기술하고 있습니다.
사용된 pII가 야생형인 경우 비리온-항원 결합을 끊지 않고 비리온 구조를 수행할 수 있기 때문에 pII 대신 pVII와 pIX를 사용하는 것도 장점이 될 수 있다.대신 구슬과 항원 사이의 부분을 쪼개 용출시킬 수 있다.pII는 온전하기 때문에 항원이 파지에 [45]결합되어 있는지 여부는 중요하지 않다.
T7 페이징
Ff 페이지를 사용하여 파지를 표시하는 문제는 해당 [46]단백질이 파지로 조립되기 전에 세균 내막을 가로질러 배치되어야 한다는 것입니다.일부 단백질은 이 과정을 거치지 못하기 때문에 Ff phages의 표면에 표시할 수 없다.이 경우 대신 [46]T7 페이지 디스플레이가 사용됩니다.T7 파지 표시에서는 T7의 [46]유전자 10 캡시드 단백질의 C 말단에 표시해야 할 단백질이 부착되어 있다.
T7을 사용하는 것의 단점은 표면에서 발현될 수 있는 단백질의 크기가 더 짧은 펩타이드로 제한된다는 것이다. 왜냐하면 T7 게놈의 큰 변화는 M13에서처럼 수용될 수 없기 때문이다. M13에서는 파지가 더 큰 게놈에 맞도록 외피를 더 길게 만들어 주기 때문이다.단, M13 파지에 scFV가 발현되고 T7 [47]파지의 표면에 항원이 발현되는 scFV 선택용 대형 단백질 라이브러리 작성에는 유용할 수 있다.
생물정보학 자원 및 도구
mimotope용 데이터베이스와 계산 도구는 파지 디스플레이 연구에서 [48]중요한 부분을 차지해 왔다.데이터베이스,[49] 프로그램 및 웹 서버는[50] 표적-관련되지 않은 [51]펩타이드를 배제하고, 작은 분자-단백질 상호작용을 특성화하고, 단백질-단백질 상호작용을 매핑하기 위해 널리 사용되어 왔다.사용자는 단백질의 3차원 구조와 파지 디스플레이 실험에서 선택된 펩타이드를 사용하여 입체구조 에피토프를 매핑할 수 있습니다.빠르고 효율적인 계산 방법 중 일부는 온라인에서 [50]이용할 수 있습니다.
「 」를 참조해 주세요.
경쟁 기술:
레퍼런스
- ^ a b Smith GP (June 1985). "Filamentous fusion phage: novel expression vectors that display cloned antigens on the virion surface". Science. 228 (4705): 1315–7. Bibcode:1985Sci...228.1315S. doi:10.1126/science.4001944. PMID 4001944.
- ^ Smith GP, Petrenko VA (April 1997). "Phage Display". Chem. Rev. 97 (2): 391–410. doi:10.1021/cr960065d. PMID 11848876.
- ^ Kehoe JW, Kay BK (November 2005). "Filamentous phage display in the new millennium". Chem. Rev. 105 (11): 4056–72. doi:10.1021/cr000261r. PMID 16277371.
- ^ Malys N, Chang DY, Baumann RG, Xie D, Black LW (2002). "A bipartite bacteriophage T4 SOC and HOC randomized peptide display library: detection and analysis of phage T4 terminase (gp17) and late sigma factor (gp55) interaction". J Mol Biol. 319 (2): 289–304. doi:10.1016/S0022-2836(02)00298-X. PMID 12051907.
- ^ Parmley SF, Smith GP (1988). "Antibody-selectable filamentous fd phage vectors: affinity purification of target genes". Gene. 73 (2): 305–318. doi:10.1016/0378-1119(88)90495-7. PMID 3149606.
- ^ Scott, J.; Smith, G. (1990). "Searching for peptide ligands with an epitope library". Science. 249 (4967): 386–390. Bibcode:1990Sci...249..386S. doi:10.1126/science.1696028. PMID 1696028.
- ^ "The Nobel Prize in Chemistry 2018". NobelPrize.org. Retrieved 2018-10-03.
- ^ 미국 특허 5866363, Pieczenik G, "생물정보를 분류하고 식별하는 방법 및 수단", 1999-02-02 발행
- ^ Chasteen L, Ayriss J, Pavlik P, Bradbury AR (2006). "Eliminating helper phage from phage display". Nucleic Acids Res. 34 (21): e145. doi:10.1093/nar/gkl772. PMC 1693883. PMID 17088290.
- ^ Lunder M, Bratkovic T, Urleb U, Kreft S, Strukelj B (June 2008). "Ultrasound in phage display: a new approach to nonspecific elution". BioTechniques. 44 (7): 893–900. doi:10.2144/000112759. PMID 18533899.
- ^ 웰컴 트러스트의 '단백질 상호작용 맵핑' 설명
- ^ Lunder M, Bratkovic T, Doljak B, Kreft S, Urleb U, Strukelj B, Plazar N (November 2005). "Comparison of bacterial and phage display peptide libraries in search of target-binding motif". Appl. Biochem. Biotechnol. 127 (2): 125–31. doi:10.1385/ABAB:127:2:125. PMID 16258189. S2CID 45243314.
- ^ Bratkovic T, Lunder M, Popovic T, Kreft S, Turk B, Strukelj B, Urleb U (July 2005). "Affinity selection to papain yields potent peptide inhibitors of cathepsins L, B, H, and K". Biochem. Biophys. Res. Commun. 332 (3): 897–903. doi:10.1016/j.bbrc.2005.05.028. PMID 15913550.
- ^ Lunder M, Bratkovic T, Kreft S, Strukelj B (July 2005). "Peptide inhibitor of pancreatic lipase selected by phage display using different elution strategies". J. Lipid Res. 46 (7): 1512–6. doi:10.1194/jlr.M500048-JLR200. PMID 15863836.
- ^ Hufton SE, Moerkerk PT, Meulemans EV, de Bruïne A, Arends JW, Hoogenboom HR (December 1999). "Phage display of cDNA repertoires: the pVI display system and its applications for the selection of immunogenic ligands". J. Immunol. Methods. 231 (1–2): 39–51. doi:10.1016/S0022-1759(99)00139-8. PMID 10648926.
- ^ Gommans WM, Haisma HJ, Rots MG (December 2005). "Engineering zinc finger protein transcription factors: the therapeutic relevance of switching endogenous gene expression on or off at command". J. Mol. Biol. 354 (3): 507–19. doi:10.1016/j.jmb.2005.06.082. PMID 16253273.
- ^ a b "CAR T Cells: Engineering Patients' Immune Cells to Treat Their Cancers". National Cancer Institute. 2013-12-06. Retrieved 9 February 2018.
- ^ Løset GÅ, Berntzen G, Frigstad T, Pollmann S, Gunnarsen KS, Sandlie I (12 January 2015). "Phage Display Engineered T Cell Receptors as Tools for the Study of Tumor Peptide-MHC Interactions". Frontiers in Oncology. 4 (378): 378. doi:10.3389/fonc.2014.00378. PMC 4290511. PMID 25629004.
- ^ McCafferty J, Griffiths AD, Winter G, Chiswell DJ (December 1990). "Phage antibodies: filamentous phage displaying antibody variable domains". Nature. 348 (6301): 552–4. Bibcode:1990Natur.348..552M. doi:10.1038/348552a0. PMID 2247164. S2CID 4258014.
- ^ Scott JS, Barbas CF III, Burton DA (2001). Phage Display: A Laboratory Manual. Plainview, N.Y: Cold Spring Harbor Laboratory Press. ISBN 978-0-87969-740-2.
- ^ Breitling F, Dübel S, Seehaus T, Klewinghaus I, Little M (August 1991). "A surface expression vector for antibody screening". Gene. 104 (2): 147–53. doi:10.1016/0378-1119(91)90244-6. PMID 1916287.
- ^ Barbas CF, Kang AS, Lerner RA, Benkovic SJ (September 1991). "Assembly of combinatorial antibody libraries on phage surfaces: the gene III site". Proceedings of the National Academy of Sciences of the United States of America. 88 (18): 7978–82. Bibcode:1991PNAS...88.7978B. doi:10.1073/pnas.88.18.7978. PMC 52428. PMID 1896445.
- ^ Burton DR, Barbas CF, Persson MA, Koenig S, Chanock RM, Lerner RA (November 1991). "A large array of human monoclonal antibodies to type 1 human immunodeficiency virus from combinatorial libraries of asymptomatic seropositive individuals". Proceedings of the National Academy of Sciences of the United States of America. 88 (22): 10134–7. Bibcode:1991PNAS...8810134B. doi:10.1073/pnas.88.22.10134. PMC 52882. PMID 1719545.
- ^ Barbas CF, Björling E, Chiodi F, Dunlop N, Cababa D, Jones TM, Zebedee SL, Persson MA, Nara PL, Norrby E (October 1992). "Recombinant human Fab fragments neutralize human type 1 immunodeficiency virus in vitro". Proceedings of the National Academy of Sciences of the United States of America. 89 (19): 9339–43. Bibcode:1992PNAS...89.9339B. doi:10.1073/pnas.89.19.9339. PMC 50122. PMID 1384050.
- ^ Burton DR, Pyati J, Koduri R, Sharp SJ, Thornton GB, Parren PW, Sawyer LS, Hendry RM, Dunlop N, Nara PL (November 1994). "Efficient neutralization of primary isolates of HIV-1 by a recombinant human monoclonal antibody". Science. 266 (5187): 1024–7. Bibcode:1994Sci...266.1024B. doi:10.1126/science.7973652. PMID 7973652.
- ^ Yang WP, Green K, Pinz-Sweeney S, Briones AT, Burton DR, Barbas CF (December 1995). "CDR walking mutagenesis for the affinity maturation of a potent human anti-HIV-1 antibody into the picomolar range". Journal of Molecular Biology. 254 (3): 392–403. doi:10.1006/jmbi.1995.0626. PMID 7490758.
- ^ Barbas CF, Hu D, Dunlop N, Sawyer L, Cababa D, Hendry RM, Nara PL, Burton DR (April 1994). "In vitro evolution of a neutralizing human antibody to human immunodeficiency virus type 1 to enhance affinity and broaden strain cross-reactivity". Proceedings of the National Academy of Sciences of the United States of America. 91 (9): 3809–13. Bibcode:1994PNAS...91.3809B. doi:10.1073/pnas.91.9.3809. PMC 43671. PMID 8170992.
- ^ Barbas CF, Bain JD, Hoekstra DM, Lerner RA (May 1992). "Semisynthetic combinatorial antibody libraries: a chemical solution to the diversity problem". Proc. Natl. Acad. Sci. U.S.A. 89 (10): 4457–61. Bibcode:1992PNAS...89.4457B. doi:10.1073/pnas.89.10.4457. PMC 49101. PMID 1584777.
- ^ Barbas CF, Languino LR, Smith JW (November 1993). "High-affinity self-reactive human antibodies by design and selection: targeting the integrin ligand binding site". Proc. Natl. Acad. Sci. U.S.A. 90 (21): 10003–7. Bibcode:1993PNAS...9010003B. doi:10.1073/pnas.90.21.10003. PMC 47701. PMID 7694276.
- ^ Barbas CF, Wagner J (October 1995). "Synthetic Human Antibodies: Selecting and Evolving Functional Proteins". Methods. 8 (2): 94–103. doi:10.1006/meth.1995.9997.
- ^ Barbas CF (August 1995). "Synthetic human antibodies". Nat. Med. 1 (8): 837–9. doi:10.1038/nm0895-837. PMID 7585190. S2CID 6983649.
- ^ Lawrence S (April 2007). "Billion dollar babies--biotech drugs as blockbusters". Nat. Biotechnol. 25 (4): 380–2. doi:10.1038/nbt0407-380. PMID 17420735. S2CID 205266758.
- ^ 케임브리지 항체: 판매 업데이트 회사 발표 텔레그래프
- ^ a b c d e f g h i j k l m Lowman HB, Clackson T (2004). "1.3". Phage display: a practical approach. Oxford [Oxfordshire]: Oxford University Press. pp. 10–11. ISBN 978-0-19-963873-4.
- ^ Sidhu SS, Weiss GA, Wells JA (February 2000). "High copy display of large proteins on phage for functional selections". J. Mol. Biol. 296 (2): 487–95. doi:10.1006/jmbi.1999.3465. PMID 10669603.
- ^ Derda R, Tang SK, Whitesides GM (July 2010). "Uniform amplification of phage with different growth characteristics in individual compartments consisting of monodisperse droplets". Angew. Chem. Int. Ed. Engl. 49 (31): 5301–4. doi:10.1002/anie.201001143. PMC 2963104. PMID 20583018.
- ^ Crameri R, Jaussi R, Menz G, Blaser K (November 1994). "Display of expression products of cDNA libraries on phage surfaces. A versatile screening system for selective isolation of genes by specific gene-product/ligand interaction". Eur. J. Biochem. 226 (1): 53–8. doi:10.1111/j.1432-1033.1994.tb20025.x. PMID 7957259.
- ^ Gramatikoff K, Georgiev O, Schaffner W (December 1994). "Direct interaction rescue, a novel filamentous phage technique to study protein-protein interactions". Nucleic Acids Res. 22 (25): 5761–2. doi:10.1093/nar/22.25.5761. PMC 310144. PMID 7838733.
- ^ Fuh G, Sidhu SS (September 2000). "Efficient phage display of polypeptides fused to the carboxy-terminus of the M13 gene-3 minor coat protein". FEBS Lett. 480 (2–3): 231–4. doi:10.1016/s0014-5793(00)01946-3. PMID 11034335. S2CID 23009887.
- ^ a b Malik P, Terry TD, Bellintani F, Perham RN (October 1998). "Factors limiting display of foreign peptides on the major coat protein of filamentous bacteriophage capsids and a potential role for leader peptidase". FEBS Lett. 436 (2): 263–6. doi:10.1016/s0014-5793(98)01140-5. PMID 9781692. S2CID 19331069.
- ^ a b c Weiss GA, Sidhu SS (June 2000). "Design and evolution of artificial M13 coat proteins". J. Mol. Biol. 300 (1): 213–9. doi:10.1006/jmbi.2000.3845. PMID 10864510.
- ^ Jespers LS, Messens JH, De Keyser A, Eeckhout D, Van den Brande I, Gansemans YG, Lauwereys MJ, Vlasuk GP, Stanssens PE (April 1995). "Surface expression and ligand-based selection of cDNAs fused to filamentous phage gene VI". Bio/Technology. 13 (4): 378–82. doi:10.1038/nbt0495-378. PMID 9634780. S2CID 6171262.
- ^ Endemann H, Model P (July 1995). "Location of filamentous phage minor coat proteins in phage and in infected cells". J. Mol. Biol. 250 (4): 496–506. doi:10.1006/jmbi.1995.0393. PMID 7616570.
- ^ Gao C, Mao S, Lo CH, Wirsching P, Lerner RA, Janda KD (May 1999). "Making artificial antibodies: a format for phage display of combinatorial heterodimeric arrays". Proc. Natl. Acad. Sci. U.S.A. 96 (11): 6025–30. Bibcode:1999PNAS...96.6025G. doi:10.1073/pnas.96.11.6025. PMC 26829. PMID 10339535.
- ^ a b c Løset GÅ, Roos N, Bogen B, Sandlie I (2011). "Expanding the versatility of phage display II: improved affinity selection of folded domains on protein VII and IX of the filamentous phage". PLOS ONE. 6 (2): e17433. Bibcode:2011PLoSO...617433L. doi:10.1371/journal.pone.0017433. PMC 3044770. PMID 21390283.
- ^ a b c Danner S, Belasco JG (November 2001). "T7 phage display: a novel genetic selection system for cloning RNA-binding proteins from cDNA libraries". Proc. Natl. Acad. Sci. U.S.A. 98 (23): 12954–9. Bibcode:2001PNAS...9812954D. doi:10.1073/pnas.211439598. PMC 60806. PMID 11606722.
- ^ Castillo J, Goodson B, Winter J (November 2001). "T7 displayed peptides as targets for selecting peptide specific scFvs from M13 scFv display libraries". J. Immunol. Methods. 257 (1–2): 117–22. doi:10.1016/s0022-1759(01)00454-9. PMID 11687245.
- ^ Huang J, Ru B, Dai P (2011). "Bioinformatics resources and tools for phage display". Molecules. 16 (1): 694–709. doi:10.3390/molecules16010694. PMC 6259106. PMID 21245805.
- ^ Huang J, Ru B, Zhu P, Nie F, Yang J, Wang X, Dai P, Lin H, Guo FB, Rao N (January 2012). "MimoDB 2.0: a mimotope database and beyond". Nucleic Acids Res. 40 (Database issue): D271–7. doi:10.1093/nar/gkr922. PMC 3245166. PMID 22053087.
- ^ a b Negi SS, Braun W (2009). "Automated Detection of Conformational Epitopes Using Phage Display Peptide Sequences". Bioinform Biol Insights. 3: 71–81. doi:10.4137/BBI.S2745. PMC 2808184. PMID 20140073.
- ^ Huang J, Ru B, Li S, Lin H, Guo FB (2010). "SAROTUP: scanner and reporter of target-unrelated peptides". J. Biomed. Biotechnol. 2010: 101932. doi:10.1155/2010/101932. PMC 2842971. PMID 20339521.
추가 정보
- Ledsgaard L, Kilstrup M, Karatt-Vellatt A, McCafferty J, Laustsen AH (2018). "Basics of antibody phage display technology" (PDF). Toxins. 10 (6): 236. doi:10.3390/toxins10060236. PMC 6024766. PMID 29890762.
- 화학공학에서의 선택과 설계
- ETH-2 인간 항체 파지 라이브러리
- Sidhu SS, Lowman HB, Cunningham BC, Wells JA (2000). "Phage display for selection of novel binding peptides". Meth. Enzymol. Methods in Enzymology. 328: 333–63. doi:10.1016/S0076-6879(00)28406-1. ISBN 9780121822293. PMID 11075354.
외부 링크
| 라이브러리 리소스 정보 페이지 디스플레이 |