중복 유전자
Overlapping gene중첩 유전자(또는 OLG)[1][2]는 발현 가능한 뉴클레오티드 배열이 [3]다른 유전자의 발현 가능한 뉴클레오티드 배열과 부분적으로 중복되는 유전자이다.이와 같이 뉴클레오티드 배열은 하나 이상의 유전자 생성물의 기능에 기여할 수 있다.중복되는 유전자가 존재하며 세포와 바이러스 [2]게놈의 기본적인 특징이 있다.중복 유전자에 대한 현재의 정의는 진핵생물, 원핵생물,[2] 그리고 바이러스 사이에 상당히 다르다.원핵생물과 바이러스는 반드시 코드 배열 사이에 존재해야 하지만 mRNA 전사체 사이에 존재해서는 안 되며, 이러한 코드 배열이 동일하거나 반대되는 가닥에서 뉴클레오티드를 공유할 때 정의된다.진핵생물에서 유전자 중첩은 거의 항상 mRNA 전사 중첩으로 정의된다.구체적으로는 진핵생물 중 적어도 하나의 뉴클레오티드가 2개 이상의 유전자의 1차 mRNA 전사 경계 사이에 공유될 때 유전자의 중복이 정의되며, 중복 영역의 임의의 점에서의 DNA 염기 돌연변이가 관련된 모든 유전자의 전사물에 영향을 준다.이 정의에는 인트론과 함께 5' 및 3' 비번역 영역(UTR)이 포함됩니다.
오버프린트란 한 유전자의 배열의 전부 또는 일부를 같은 [4]궤적에 있는 다른 유전자의 대체 판독범위에서 읽는 중복을 말한다.대체 오픈 리딩 프레임(ORF)은 발현 가능한 기존 유전자 내에서 중요한 뉴클레오티드 치환에 의해 생성된 것으로 생각되며, 원래 [5]유전자의 기능을 유지하면서 새로운 단백질을 발현하도록 유도될 수 있다.과잉인쇄는 기존의 유전자 배열에서 새로운 유전자의 새로운 출현을 위한 메커니즘으로 가설화 되었다. 즉,[6] 이전의 유전자의 코드화되지 않은 영역이다.대부분의 중복되는 유전자, 즉 발현 가능한 뉴클레오티드 배열이 부분적으로 서로 겹치는 유전자는 부분적으로 이 메커니즘 때문에 진화한 것으로 믿어지며, 각각의 중복이 하나의 조상 유전자와 하나의 새로운 [7]유전자로 구성되어 있음을 암시한다.그 후,[8] 이러한 새로운 유전자에 의해 코드화된 de novo 단백질이 일반적으로 데이터베이스에서 원격 호몰로지가 부족하기 때문에, 오버프린트는 또한 새로운 단백질의 원천으로 여겨진다.과잉 인쇄된 유전자는 특히 바이러스의 게놈 구조의 일반적인 특징으로, 작은 바이러스 유전 정보 [9]세트로부터 발현 가능한 유전자의 수를 크게 증가시킬 가능성이 있다.과잉인쇄는 바이러스의 진화 과정에서 수많은 새로운 단백질 생성의 원인이 될 수 있다.
분류
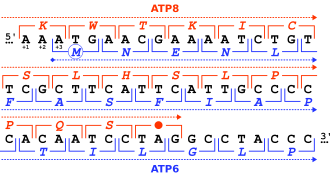
유전자는 다양한 방식으로 중복될 수 있고 서로 [3][11][12][13][14]상대적인 위치에 따라 분류될 수 있다.
- 단방향 또는 탠덤 오버랩: 한 유전자의 3' 끝은 같은 가닥의 다른 유전자의 5' 끝과 오버랩됩니다.이 배열은 →→ 표기로 기호화할 수 있습니다. 여기서 화살표는 처음부터 끝까지 판독 프레임을 나타냅니다.
- 수렴성 또는 엔드온 중첩: 두 유전자의 3' 끝은 서로 다른 가닥에서 겹칩니다.→←라고 쓸 수 있습니다.
- 다이버전트 또는 테일온 오버랩: 두 유전자의 5' 끝은 서로 다른 가닥에 오버랩됩니다.←→라고 쓸 수 있습니다.
중복되는 유전자는 단계별로 분류할 수 있으며, 이는 상대적인 판독 [3][11][12][13][14]프레임을 설명한다.
- 동상 오버랩은 공유 시퀀스가 동일한 판독 프레임을 사용하는 경우 발생합니다.이것을 「단계 0」이라고도 합니다.단계 0이 겹치는 단방향 유전자는 별개의 유전자로 간주되지 않고 동일한 유전자의 대체 시작 부위로 간주됩니다.
- 위상 외 오버랩은 공유 시퀀스가 서로 다른 판독 프레임을 사용하는 경우 발생합니다.판독 프레임이 1뉴클레오티드 또는 2뉴클레오티드에 의해 오프셋되는지 여부에 따라 "단계 1" 또는 "단계 2"에서 발생할 수 있습니다.코돈은 3개의 뉴클레오티드의 길이이기 때문에 3개의 뉴클레오티드의 오프셋은 동상의 0상 프레임이다.
중복되는 유전자에 대한 연구는 그들의 진화가 두 [4]가지 가능한 모델로 요약될 수 있다는 것을 암시한다.하나의 모델에서 각각의 중복 유전자에 의해 부호화된 두 단백질은 유사한 선택 압력 하에서 진화한다.아미노산 변화에 대한 강력한 선택이 선호될 때 단백질과 겹치는 영역은 매우 보존됩니다.단일 뉴클레오티드 치환이 두 단백질의 구조와 기능을 동시에 변화시킬 수 있기 때문에 중복 유전자는 엄격한 제약 조건 하에서 진화한다.DNA 게놈에 수많은 중복 유전자가 포함된 B형 간염 바이러스(HBV)에 대한 연구는 중복 부호화 영역에서 부위당 동의어 뉴클레오티드 치환의 평균 수가 중복되지 않는 [15]영역보다 유의미하게 낮았다.같은 연구에서 아미노산 변화에 대한 선택이 약할 때 이러한 중복되는 영역과 단백질이 원래의 영역과 크게 분리되는 것이 가능하다는 것을 보여주었습니다.예를 들어 중합효소의 스페이서 도메인과 HBV 표면단백질의 프리 S1 영역은 각각 [15]30%, 40%의 보존 아미노산 비율을 보였다.그러나 이러한 중첩 영역은 프로세스에 절대적으로 필요한 서로 다른 HBV 균주 간에 보존성이 높았던 중첩 영역에 비해 복제에 덜 중요한 것으로 알려져 있다.
두 번째 모델은 두 단백질과 각각의 중복 유전자가 반대되는 선택 압력 하에서 진화하는 것을 시사한다: 한 프레임은 양성 선택을 경험하고 다른 프레임은 정제 선택을 경험한다.톰버스바이러스는 549nt 부호화 영역을 형성하는 중복 유전자에 의해 단백질 p19와 p22가 부호화되며, p19는 양성 선택,[16] p22는 정제 선택 하에 있음을 나타낸다.센다이 바이러스,[17] 감자 잎말이 바이러스,[18] 인간 파르보 바이러스 [19]B19의 유전자 중복에 관한 연구에서는, 그 외의 예가 언급되고 있다.서로 다른 선택 압력을 경험하는 중복 유전자의 이러한 현상은 두 프레임에 서로 다른 영향을 미치는 높은 비율의 뉴클레오티드 치환의 결과일 수 있다. 치환은 주로 한 프레임에 대해 동의어가 아닌 반면, 다른 [4]프레임에 대해서는 동의어가 될 수 있다.
진화
중복 유전자는 바이러스, 박테리아, 미토콘드리아와 같이 빠르게 진화하는 게놈에서 특히 흔하다.다음의 3가지 [20]방법이 있습니다.
- 정지 코돈의 상실로 인해 기존의 개방 판독 프레임(ORF)을 연속 유전자로 확장하여
- 개시 코돈의 상실로 인해 기존 ORF를 연속 유전자로 확장한다.
- 포인트 돌연변이에 의한 기존 ORF 내에서 신규 ORF 생성.
여러 유전자를 부호화하기 위해 동일한 뉴클레오티드 배열을 사용하는 것은 게놈 크기의 감소와 중복 [12][21][22][23]유전자의 전사 및 번역적 공동 조절의 기회 때문에 진화상의 이점을 제공할 수 있다.유전자 중복은 중복 [14][24]영역의 배열에 새로운 진화적 제약을 일으킨다.
새로운 유전자의 기원

1977년, Pierre-Paul Grassé는 쌍 중 하나의 유전자가 돌연변이에 의해 새로운 ORF를 대체 판독 프레임에 도입함으로써 de novo를 발생시킬 수 있다고 제안했다; 그는 그 메커니즘을 [25]: 231 오버프린트라고 설명했다.그것은 나중에 [26]스스무 오노에 의해 입증되었고, 그는 이 메커니즘에 의해 발생할 수 있는 후보 유전자를 확인했다.이러한 방식으로 유래한 일부 de novo 유전자는 중복되지 않고 유전자 [6]복제에 따라 하위 기능을 수행하며, 고아 유전자의 확산에 기여한다.중복되는 유전자 쌍의 어떤 멤버가 더 어린지는 보다 제한된 계통발생학적 분포 또는 덜 최적화된 [9][27][28]코돈 사용에 의해 생물정보학적으로 식별될 수 있다.이 쌍의 젊은 구성원들은 나이든 구성원들보다 더 높은 내재적 구조적 장애를 가지는 경향이 있지만, 나이든 구성원들은 다른 단백질들보다 더 무질서하며,[27] 아마도 겹침에 의해 야기되는 증가된 진화적 제약을 완화하는 방법일 것이다.중복은 이미 높은 [27]장애를 가진 단백질에서 발생할 가능성이 더 높다.
분류학적 분포

중복되는 유전자는 다양한 빈도로 생명체의 모든 영역에서 발생한다.그것들은 바이러스 게놈에서 특히 흔하다.
바이러스
중복 유전자의 존재는 1977년 [29]프레데릭 생어에 의해 배열된 최초의 DNA 게놈이었던 바이러스 δX174에서 처음 확인되었다.대장균을 감염시킨 작은 단가닥 DNA 박테리오파지 δX174의 이전 분석에서는 감염 중 생성된 단백질이 [31]게놈의 측정된 길이보다 더 긴 코드 배열을 필요로 한다는 것을 시사했다.완전히 배열된 5386 뉴클레오티드 게놈의 분석은 바이러스가 코딩 영역 사이에 광범위한 중복을 가지고 있다는 것을 보여주었고, 일부 유전자(유전자 D와 E와 같은)는 같은 DNA 배열에서 번역되었지만 다른 판독 [29][31]프레임에서 번역되었다는 것을 보여주었다.gene. 그래서 게놈 내에 대체 시작 사이트 복제 한 ΦX174의 시말단은 원래 A단백질에 대한 동일한 코딩 순서에 절단 단백질을 표현하지만 다른 function[32][33]을 소지하는 것이 폴리 펩타이드 합성의 다른 발견되지 않은 사이트들이 게놈을 통해 숨길 수 있는 것으로 결론지었다 o.으로 인해 공개되었다verlapp유전자를 주입하다또 다른 중복 유전자 궤적의 확인된 de novo 유전자는 대장균 세포벽의 생합성을 저해하여 대장균의 용해를 유도하는 새로운 단백질을 발현하는 것으로 나타나 오버프린트 과정을 통한 de novo 단백질 생성이 [4]바이러스의 병원성 진화에 중요한 요인이 될 수 있음을 시사한다.또 다른 예는 사스-CoV 2 [1][34]바이러스의 ORF3d 유전자이다.중복 유전자는 바이러스 [9]게놈에서 특히 흔하다.일부 연구는 이러한 관찰이 바이러스 캡시드로 게놈을 포장하는 물리적 제약, 특히 정십면체 [35]기하학 중 하나에 의해 매개되는 작은 게놈 크기에 대한 선택적 압력 때문이라고 보고 있다.그러나 다른 연구들은 이 결론에 이의를 제기하며 바이러스 유전자의 중복 분포가 중복되는 바이러스 [36]유전자의 진화적 기원으로서 과잉 인쇄를 반영할 가능성이 높다고 주장한다.과잉인쇄는 [28]바이러스의 새로운 유전자의 흔한 원천이다.
게놈 내에서 코드 배열이 겹치는 바이러스의 비율은 다양합니다.[2]이중가닥 RNA 바이러스는 4분의 3 이하인 반면 레트로바이러스과와 단일가닥 DNA 게놈을 가진 바이러스는 중복되는 코드 [37]서열을 가지고 있다.특히 세분화된 바이러스, 또는 게놈이 다른 조각으로 분할되어 모두 동일한 캡슐화 또는 별도의 캡슐화 중 하나로 포장된 바이러스는 세분화되지 않은 [37]바이러스보다 중복되는 시퀀스를 포함할 가능성이 높습니다.RNA 바이러스는 낮은 돌연변이율과 덜 [37][38]제한적인 게놈 크기를 가진 DNA 바이러스보다 중복되는 유전자가 적다.DNA 바이러스의 낮은 돌연변이율은 구조적으로 제약된 게놈 내에서 더 큰 게놈 신규성과 진화적 탐구를 촉진하고 중복 유전자의 [39][40]진화의 주요 동인이 될 수 있다.
과도하게 인쇄된 바이러스 유전자에 대한 연구는 그들의 단백질 생산물이 바이러스 증식에 필수적인 것은 아니지만 병원성에 기여하는 보조 단백질인 경향이 있다는 것을 암시한다.과잉 인쇄된 단백질은 종종 특이한 아미노산 분포와 높은 수준의 내인성 [41]장애를 가지고 있다.일부 경우에 과잉 인쇄된 단백질은 잘 정의되었지만 참신한 3차원 [42]구조를 가지고 있다. 한 가지 예는 Tombusvirus에서 발견된 RNA 사일링 억제제 p19이며, 이는 새로운 단백질 접힘과 siRNA [28][30][43]인식에 있어 새로운 결합 모드를 모두 가지고 있다.
원핵생물
박테리아 게놈에서 유전자가 중복되는 추정치는 보통 박테리아 유전자의 약 3분의 1이 중복된다는 것을 발견하는데, 이는 보통 몇 개의 [12][44][45]염기쌍에 의해서만 나타난다.박테리아 게놈의 중복에 대한 대부분의 연구는 중복이 유전자 조절의 기능을 하고, 중복된 유전자들이 전사적으로 그리고 번역적으로 함께 조절되도록 [12][23]하는 증거를 발견합니다.원핵생물 게놈에서, 단방향 중복은 가장 흔하며,[12][14][11] 아마도 인접한 원핵생물 유전자들이 방향을 공유하는 경향 때문일 것이다.단방향 오버랩 중 긴 오버랩은 읽기 프레임(즉, 단계 1)에서 1뉴클레오티드 오프셋과 함께 더 일반적으로 읽히고 짧은 오버랩은 단계 [45][46]2에서 더 일반적으로 읽힌다.60개 이상의 염기쌍의 긴 겹침은 수렴 유전자에 더 흔하지만, 추정 긴 겹침은 [47]오주율이 매우 높습니다.박테리아 게놈의 긴 중복의 강력 검증된 예는 드물다; 잘 연구된 모델 유기체 대장균에서, 오직 4쌍의 유전자 쌍만이 긴 중복을 [48]가진 것으로 잘 검증되었다.
진핵생물
원핵 게놈과 비교했을 때, 진핵 게놈은 종종 주석이 잘 되어 있지 않기 때문에 진짜 중복을 확인하는 것은 상대적으로 어렵다.[28]그러나 검증된 유전자 중복의 예는 생쥐와 [49][50][51][52]인간과 같은 포유류를 포함한 다양한 진핵 생물에서 입증되었다.진핵생물은 겹치는 형태의 분포에서 원핵생물과 다르다: 단방향 (같은 가닥) 겹치는 것은 원핵생물에서 가장 흔하지만, 반대 또는 반평행- 겹치는 것은 진핵생물에서 더 흔하다.반대쪽 스트랜드 오버랩 중 수렴방향이 가장 [50]일반적입니다.진핵생물 유전자의 중복에 대한 대부분의 연구는 중복 유전자가 밀접하게 관련된 종에서도 광범위하게 유전자 재조합의 대상이 된다는 것을 발견했고, 따라서 중복의 존재가 항상 잘 [51][53]보존되는 것은 아니다.오래된 또는 분류학적으로 제한되지 않은 유전자와 중복되는 것은 또한 주어진 진핵생물 [51][54][55]계통에서 de novo가 발생했을 가능성이 있는 유전자의 공통적인 특징이다.
기능.
중복되는 유전자의 정확한 기능은 생명체의 영역에 따라 달라 보이지만, 몇몇 실험들은 그것들이 적절한 단백질 발현과 화학측정법을 통해 적절한 단백질 [57]접힘의 역할을 할 뿐만 아니라 바이러스의 수명 주기에 중요하다는 것을 보여주었다.박테리오파지 δX174의 버전도 생성되었으며, 중복되는 모든 유전자가 제거되어 복제에 필요하지 않음을 증명하였다.
바이러스 내에서 중복되는 유전자의 유지와 진화는 캡시드의 크기 [59]제한 때문일지도 모른다.야생형 [60]게놈보다 길도록 조작된 게놈을 가진 바이러스에서 극적인 생존력 손실이 관찰되었다.δX174의 단가닥 DNA 게놈 길이를 1% 이상 증가시키면 유한 캡시드 [61]부피에 의해 부과된 엄격한 물리적 제약의 결과로 여겨지는 감염성의 거의 완전한 상실을 초래한다.유전자 전달 벡터로서의 아데노 관련 바이러스에 대한 연구는 바이러스 포장이 유전자 화물 크기 제한에 의해 제한된다는 것을 보여주었고, CFTR81과 [62][63]같은 큰 인간 유전자를 전달하기 위해 다중 벡터를 사용해야 한다.따라서 중복 유전자는 이러한 물리적 제약을 극복하기 위한 수단으로 진화해 게놈 길이를 늘리기보다는 기존 염기서열만을 활용함으로써 유전적 다양성을 증가시켰다고 볼 수 있다.
중복유전자 및 ORF 식별방법
게놈 주석과 같은 표준화된 방법은 중복 유전자의 검출에 부적절할 수 있다.중복 유전자는 일반적으로 비정형 배열조성을 포함하고 [2][64][65][66]있는 반면 중복 유전자는 이미 큐레이션된 유전자에 의존하기 때문이다.게놈 주석 표준은 또한 종종 [67]다른 유전자 안에 완전히 포함된 유전자와 같은 특징 중복에 대해 편파적이다.또한 RAST 파이프라인과 같은 일부 생물정보학 파이프라인은 예측된 ORF [68]간의 중복을 현저하게 처벌한다.그러나 게놈 규모의 단백질과 RNA 측정 툴의 급속한 발전과 함께 점점 더 발전하는 예측 알고리즘은 수많은 [2]게놈에서 중복되는 유전자와 ORF의 눈사태를 드러내고 있다.단백질 유전학 방법은 수많은 중복 유전자를 발견하는 데 필수적이며 상향식 단백질학, 리보솜 프로파일링, DNA 배열 분석, 섭동과 같은 기술의 조합을 포함한다.RNA 염기서열처리는 또한 중복되는 전사를 포함하는 게놈 영역을 식별하는 데 사용됩니다.그것은 [69]사람에게서 발견된 이전에 주석이 달린 코딩 영역 내에서 180,000개의 대체 ORF를 식별하는 데 이용되었다.이와 같이 새롭게 발견된 ORF는 CRISPR-Cas9 및 촉매적으로 죽은 Cas9([70][71][72]dCas9) 교란과 같은 다양한 역유전 기술을 사용하여 검증됩니다.발견되지 않은 중복 [73]유전자의 부재를 확실히 보여주기 위해 합성에 의한 증명 시도도 행해진다.
레퍼런스
- ^ a b Nelson, Chase W; et al. (1 October 2020). "Dynamically evolving novel overlapping gene as a factor in the SARS-CoV-2 pandemic". eLife. 9. doi:10.7554/eLife.59633. PMC 7655111. PMID 33001029. Retrieved 11 November 2020.
- ^ a b c d e f Wright, Bradley W.; Molloy, Mark P.; Jaschke, Paul R. (5 October 2021). "Overlapping genes in natural and engineered genomes". Nature Reviews Genetics. 23 (3): 154–168. doi:10.1038/s41576-021-00417-w. ISSN 1471-0064. PMC 8490965. PMID 34611352.
- ^ a b c Y. Fukuda, M. Tomita et T. Washio (1999). "Comparative study of overlapping genes in the genomes of Mycoplasma genitalium and Mycoplasma pneumoniae". Nucleic Acids Res. 27 (8): 1847–1853. doi:10.1093/nar/27.8.1847. PMC 148392. PMID 10101192.
- ^ a b c d Pavesi, Angelo (26 May 2021). "Origin, Evolution and Stability of Overlapping Genes in Viruses: A Systematic Review". Genes. 12 (6): 809. doi:10.3390/genes12060809. ISSN 2073-4425. PMC 8227390. PMID 34073395.
- ^ Normark, Staffan; Bergström, Sven; Edlund, Thomas; Grundström, Thomas; Jaurin, Bengtake; Lindberg, Frederik P.; Olsson, Olof (December 1983). "Overlapping Genes". Annual Review of Genetics. 17 (1): 499–525. doi:10.1146/annurev.ge.17.120183.002435. ISSN 0066-4197. PMID 6198955.
- ^ a b Keese, PK; Gibbs, A (15 October 1992). "Origins of genes: "big bang" or continuous creation?". Proceedings of the National Academy of Sciences of the United States of America. 89 (20): 9489–93. Bibcode:1992PNAS...89.9489K. doi:10.1073/pnas.89.20.9489. PMC 50157. PMID 1329098.
- ^ Keese, P. K.; Gibbs, A. (15 October 1992). "Origins of genes: "big bang" or continuous creation?". Proceedings of the National Academy of Sciences. 89 (20): 9489–9493. Bibcode:1992PNAS...89.9489K. doi:10.1073/pnas.89.20.9489. ISSN 0027-8424. PMC 50157. PMID 1329098.
- ^ Gibbs, Adrian; Keese, Paul K. (19 October 1995), "In search of the origins of viral genes", Molecular Basis of Virus Evolution, Cambridge University Press, pp. 76–90, doi:10.1017/cbo9780511661686.008, ISBN 9780521455336, retrieved 3 December 2021
- ^ a b c d Pavesi, Angelo; Magiorkinis, Gkikas; Karlin, David G.; Wilke, Claus O. (15 August 2013). "Viral Proteins Originated De Novo by Overprinting Can Be Identified by Codon Usage: Application to the "Gene Nursery" of Deltaretroviruses". PLOS Computational Biology. 9 (8): e1003162. Bibcode:2013PLSCB...9E3162P. doi:10.1371/journal.pcbi.1003162. PMC 3744397. PMID 23966842.
- ^ Anderson S, Bankier AT, Barrell BG, de Bruijn MH, Coulson AR, Drouin J, Eperon IC, Nierlich DP, Roe BA, Sanger F, Schreier PH, Smith AJ, Staden R, Young IG (April 1981). "Sequence and organization of the human mitochondrial genome". Nature. 290 (5806): 457–465. Bibcode:1981Natur.290..457A. doi:10.1038/290457a0. PMID 7219534. S2CID 4355527.
- ^ a b c Fukuda, Yoko; Nakayama, Yoichi; Tomita, Masaru (December 2003). "On dynamics of overlapping genes in bacterial genomes". Gene. 323: 181–187. doi:10.1016/j.gene.2003.09.021. PMID 14659892.
- ^ a b c d e f Johnson Z, Chisholm S (2004). "Properties of overlapping genes are conserved across microbial genomes". Genome Res. 14 (11): 2268–72. doi:10.1101/gr.2433104. PMC 525685. PMID 15520290.
- ^ a b Normark S.; Bergstrom S.; Edlund T.; Grundstrom T.; Jaurin B.; Lindberg F.P.; Olsson O. (1983). "Overlapping genes". Annual Review of Genetics. 17: 499–525. doi:10.1146/annurev.ge.17.120183.002435. PMID 6198955.
- ^ a b c d Rogozin, Igor B.; Spiridonov, Alexey N.; Sorokin, Alexander V.; Wolf, Yuri I.; Jordan, I.King; Tatusov, Roman L.; Koonin, Eugene V. (May 2002). "Purifying and directional selection in overlapping prokaryotic genes". Trends in Genetics. 18 (5): 228–232. doi:10.1016/S0168-9525(02)02649-5. PMID 12047938.
- ^ a b Mizokami, Masashi; Orito, Etsuro; Ohba, Ken-ichi; Ikeo, Kazuho; Lau, Johnson Y. N.; Gojobori, Takashi (January 1997). "Constrained evolution with respect to gene overlap of hepatitis B virus". Journal of Molecular Evolution. 44 (S1): S83–S90. Bibcode:1997JMolE..44S..83M. doi:10.1007/pl00000061. ISSN 0022-2844. PMID 9071016. S2CID 22644652.
- ^ Allison, Jane R.; Lechner, Marcus; Hoeppner, Marc P.; Poole, Anthony M. (12 February 2016). "Positive Selection or Free to Vary? Assessing the Functional Significance of Sequence Change Using Molecular Dynamics". PLOS ONE. 11 (2): e0147619. Bibcode:2016PLoSO..1147619A. doi:10.1371/journal.pone.0147619. ISSN 1932-6203. PMC 4752228. PMID 26871901.
- ^ Fujii, Yutaka; Kiyotani, Katsuhiro; Yoshida, Tetsuya; Sakaguchi, Takemasa (2001). "Conserved and non-conserved regions in the Sendai virus genome: Evolution of a gene possessing overlapping reading frames". Virus Genes. 22 (1): 47–52. doi:10.1023/a:1008130318633. ISSN 0920-8569. PMID 11210938. S2CID 12869504.
- ^ Guyader, Sébastien; Ducray, Danièle Giblot (1 July 2002). "Sequence analysis of Potato leafroll virus isolates reveals genetic stability, major evolutionary events and differential selection pressure between overlapping reading frame products". Journal of General Virology. 83 (7): 1799–1807. doi:10.1099/0022-1317-83-7-1799. ISSN 0022-1317. PMID 12075102.
- ^ Stamenković, Gorana G.; Ćirković, Valentina S.; Šiljić, Marina M.; Blagojević, Jelena V.; Knežević, Aleksandra M.; Joksić, Ivana D.; Stanojević, Maja P. (24 October 2016). "Substitution rate and natural selection in parvovirus B19". Scientific Reports. 6 (1): 35759. Bibcode:2016NatSR...635759S. doi:10.1038/srep35759. ISSN 2045-2322. PMC 5075947. PMID 27775080.
- ^ Krakauer, David C. (June 2000). "Stability and Evolution of Overlapping Genes". Evolution. 54 (3): 731–739. doi:10.1111/j.0014-3820.2000.tb00075.x. PMID 10937248. S2CID 8818055.
- ^ Delaye, Luis; DeLuna, Alexander; Lazcano, Antonio; Becerra, Arturo (2008). "The origin of a novel gene through overprinting in Escherichia coli". BMC Evolutionary Biology. 8 (1): 31. doi:10.1186/1471-2148-8-31. PMC 2268670. PMID 18226237.
- ^ Saha, Deeya; Podder, Soumita; Panda, Arup; Ghosh, Tapash Chandra (May 2016). "Overlapping genes: A significant genomic correlate of prokaryotic growth rates". Gene. 582 (2): 143–147. doi:10.1016/j.gene.2016.02.002. PMID 26853049.
- ^ a b Luo, Yingqin; Battistuzzi, Fabia; Lin, Kui; Gibas, Cynthia (29 November 2013). "Evolutionary Dynamics of Overlapped Genes in Salmonella". PLOS ONE. 8 (11): e81016. Bibcode:2013PLoSO...881016L. doi:10.1371/journal.pone.0081016. PMC 3843671. PMID 24312259.
- ^ Wei, X.; Zhang, J. (31 December 2014). "A Simple Method for Estimating the Strength of Natural Selection on Overlapping Genes". Genome Biology and Evolution. 7 (1): 381–390. doi:10.1093/gbe/evu294. PMC 4316641. PMID 25552532.
- ^ Grassé, Pierre-Paul (1977). Evolution of Living Organisms: Evidence for a New Theory of Transformation. Academic Press. ISBN 9781483274096.
- ^ Ohno, S (April 1984). "Birth of a unique enzyme from an alternative reading frame of the preexisted, internally repetitious coding sequence". Proceedings of the National Academy of Sciences of the United States of America. 81 (8): 2421–5. Bibcode:1984PNAS...81.2421O. doi:10.1073/pnas.81.8.2421. PMC 345072. PMID 6585807.
- ^ a b c Willis, Sara; Masel, Joanna (19 July 2018). "Gene Birth Contributes to Structural Disorder Encoded by Overlapping Genes". Genetics. 210 (1): 303–313. doi:10.1534/genetics.118.301249. PMC 6116962. PMID 30026186.
- ^ a b c d Sabath, N.; Wagner, A.; Karlin, D. (19 July 2012). "Evolution of Viral Proteins Originated De Novo by Overprinting". Molecular Biology and Evolution. 29 (12): 3767–3780. doi:10.1093/molbev/mss179. PMC 3494269. PMID 22821011.
- ^ a b c Sanger, F.; Air, G. M.; Barrell, B. G.; Brown, N. L.; Coulson, A. R.; Fiddes, J. C.; Hutchison, C. A.; Slocombe, P. M.; Smith, M. (1977). "Nucleotide sequence of bacteriophage ΦX174 DNA". Nature. 265 (5596): 687–95. Bibcode:1977Natur.265..687S. doi:10.1038/265687a0. PMID 870828. S2CID 4206886.
- ^ a b Ye, Keqiong; Malinina, Lucy; Patel, Dinshaw J. (3 December 2003). "Recognition of small interfering RNA by a viral suppressor of RNA silencing". Nature. 426 (6968): 874–878. Bibcode:2003Natur.426..874Y. doi:10.1038/nature02213. PMC 4694583. PMID 14661029.
- ^ a b Barrell, B. G.; Air, G. M.; Hutchison, C. A. (November 1976). "Overlapping genes in bacteriophage φX174". Nature. 264 (5581): 34–41. Bibcode:1976Natur.264...34B. doi:10.1038/264034a0. ISSN 1476-4687. PMID 1004533. S2CID 4264796.
- ^ LINNEY, ELWOOD; HAYASHI, MASAKI (May 1974). "Intragenic regulation of the synthesis of ΦX174 gene A proteins". Nature. 249 (5455): 345–348. Bibcode:1974Natur.249..345L. doi:10.1038/249345a0. ISSN 0028-0836. PMID 4601823. S2CID 4175651.
- ^ Roznowski, Aaron P.; Doore, Sarah M.; Kemp, Sundance Z.; Fane, Bentley A. (6 January 2020). "Finally, a Role Befitting A star : Strongly Conserved, Unessential Microvirus A* Proteins Ensure the Product Fidelity of Packaging Reactions". Journal of Virology. 94 (2). doi:10.1128/jvi.01593-19. ISSN 0022-538X. PMC 6955274. PMID 31666371.
- ^ Dockrill, Peter (11 November 2020). "Scientists Just Found a Mysteriously Hidden 'Gene Within a Gene' in SARS-CoV-2". ScienceAlert. Retrieved 11 November 2020.
- ^ Chirico, N.; Vianelli, A.; Belshaw, R. (7 July 2010). "Why genes overlap in viruses". Proceedings of the Royal Society B: Biological Sciences. 277 (1701): 3809–3817. doi:10.1098/rspb.2010.1052. PMC 2992710. PMID 20610432.
- ^ Brandes, Nadav; Linial, Michal (21 May 2016). "Gene overlapping and size constraints in the viral world". Biology Direct. 11 (1): 26. doi:10.1186/s13062-016-0128-3. PMC 4875738. PMID 27209091.
- ^ a b c Schlub, Timothy E; Holmes, Edward C (1 January 2020). "Properties and abundance of overlapping genes in viruses". Virus Evolution. 6 (1): veaa009. doi:10.1093/ve/veaa009. ISSN 2057-1577. PMC 7017920. PMID 32071766.
- ^ Chirico, Nicola; Vianelli, Alberto; Belshaw, Robert (7 July 2010). "Why genes overlap in viruses". Proceedings of the Royal Society B: Biological Sciences. 277 (1701): 3809–3817. doi:10.1098/rspb.2010.1052. ISSN 0962-8452. PMC 2992710. PMID 20610432.
- ^ Brandes, Nadav; Linial, Michal (21 May 2016). "Gene overlapping and size constraints in the viral world". Biology Direct. 11 (1): 26. doi:10.1186/s13062-016-0128-3. ISSN 1745-6150. PMC 4875738. PMID 27209091.
- ^ Pavesi, Angelo (July 2020). "New insights into the evolutionary features of viral overlapping genes by discriminant analysis". Virology. 546: 51–66. doi:10.1016/j.virol.2020.03.007. ISSN 0042-6822. PMC 7157939. PMID 32452417.
- ^ Rancurel, C.; Khosravi, M.; Dunker, A. K.; Romero, P. R.; Karlin, D. (29 July 2009). "Overlapping Genes Produce Proteins with Unusual Sequence Properties and Offer Insight into De Novo Protein Creation". Journal of Virology. 83 (20): 10719–10736. doi:10.1128/JVI.00595-09. PMC 2753099. PMID 19640978.
- ^ Abroi, Aare (1 December 2015). "A protein domain-based view of the virosphere–host relationship". Biochimie. 119: 231–243. doi:10.1016/j.biochi.2015.08.008. PMID 26296474.
- ^ Vargason, Jeffrey M; Szittya, György; Burgyán, József; Hall, Traci M.Tanaka (December 2003). "Size Selective Recognition of siRNA by an RNA Silencing Suppressor". Cell. 115 (7): 799–811. doi:10.1016/S0092-8674(03)00984-X. PMID 14697199. S2CID 12993441.
- ^ Huvet, Maxime; Stumpf, Michael PH (1 January 2014). "Overlapping genes: a window on gene evolvability". BMC Genomics. 15 (1): 721. doi:10.1186/1471-2164-15-721. ISSN 1471-2164. PMC 4161906. PMID 25159814.
- ^ a b Cock, Peter J. A.; Whitworth, David E. (19 March 2007). "Evolution of Gene Overlaps: Relative Reading Frame Bias in Prokaryotic Two-Component System Genes". Journal of Molecular Evolution. 64 (4): 457–462. Bibcode:2007JMolE..64..457C. doi:10.1007/s00239-006-0180-1. PMID 17479344. S2CID 21612308.
- ^ Fonseca, M. M.; Harris, D. J.; Posada, D. (5 November 2013). "Origin and Length Distribution of Unidirectional Prokaryotic Overlapping Genes". G3: Genes, Genomes, Genetics. 4 (1): 19–27. doi:10.1534/g3.113.005652. PMC 3887535. PMID 24192837.
- ^ Pallejà, Albert; Harrington, Eoghan D; Bork, Peer (2008). "Large gene overlaps in prokaryotic genomes: result of functional constraints or mispredictions?". BMC Genomics. 9 (1): 335. doi:10.1186/1471-2164-9-335. PMC 2478687. PMID 18627618.
- ^ Fellner, Lea; Simon, Svenja; Scherling, Christian; Witting, Michael; Schober, Steffen; Polte, Christine; Schmitt-Kopplin, Philippe; Keim, Daniel A.; Scherer, Siegfried; Neuhaus, Klaus (18 December 2015). "Evidence for the recent origin of a bacterial protein-coding, overlapping orphan gene by evolutionary overprinting". BMC Evolutionary Biology. 15 (1): 283. doi:10.1186/s12862-015-0558-z. PMC 4683798. PMID 26677845.
- ^ McLysaght, Aoife; Guerzoni, Daniele (31 August 2015). "New genes from non-coding sequence: the role of de novo protein-coding genes in eukaryotic evolutionary innovation". Philosophical Transactions of the Royal Society B: Biological Sciences. 370 (1678): 20140332. doi:10.1098/rstb.2014.0332. PMC 4571571. PMID 26323763.
- ^ a b C. Sanna, W. Li et L. Zhang (2008). "Overlapping genes in the human and mouse genomes". BMC Genomics. 9 (169): 169. doi:10.1186/1471-2164-9-169. PMC 2335118. PMID 18410680.
- ^ a b c Makałowska, Izabela; Lin, Chiao-Feng; Hernandez, Krisitina (2007). "Birth and death of gene overlaps in vertebrates". BMC Evolutionary Biology. 7 (1): 193. doi:10.1186/1471-2148-7-193. PMC 2151771. PMID 17939861.
- ^ Veeramachaneni, V. (1 February 2004). "Mammalian Overlapping Genes: The Comparative Perspective". Genome Research. 14 (2): 280–286. doi:10.1101/gr.1590904. PMC 327103. PMID 14762064.
- ^ Behura, Susanta K; Severson, David W (2013). "Overlapping genes of Aedes aegypti: evolutionary implications from comparison with orthologs of Anopheles gambiae and other insects". BMC Evolutionary Biology. 13 (1): 124. doi:10.1186/1471-2148-13-124. PMC 3689595. PMID 23777277.
- ^ Murphy, Daniel N.; McLysaght, Aoife; Carmel, Liran (21 November 2012). "De Novo Origin of Protein-Coding Genes in Murine Rodents". PLOS ONE. 7 (11): e48650. Bibcode:2012PLoSO...748650M. doi:10.1371/journal.pone.0048650. PMC 3504067. PMID 23185269.
- ^ Knowles, D. G.; McLysaght, A. (2 September 2009). "Recent de novo origin of human protein-coding genes". Genome Research. 19 (10): 1752–1759. doi:10.1101/gr.095026.109. PMC 2765279. PMID 19726446.
- ^ Wright, Bradley W.; Ruan, Juanfang; Molloy, Mark P.; Jaschke, Paul R. (20 November 2020). "Genome Modularization Reveals Overlapped Gene Topology Is Necessary for Efficient Viral Reproduction". ACS Synthetic Biology. 9 (11): 3079–3090. doi:10.1021/acssynbio.0c00323. ISSN 2161-5063. PMID 33044064. S2CID 222300240.
- ^ Pradhan, Prajakta; Li, Wen; Kaur, Parjit (January 2009). "Translational Coupling Controls Expression and Function of the DrrAB Drug Efflux Pump". Journal of Molecular Biology. 385 (3): 831–842. doi:10.1016/j.jmb.2008.11.027. PMID 19063901.
- ^ Jaschke, Paul R.; Lieberman, Erica K.; Rodriguez, Jon; Sierra, Adrian; Endy, Drew (December 2012). "A fully decompressed synthetic bacteriophage øX174 genome assembled and archived in yeast". Virology. 434 (2): 278–284. doi:10.1016/j.virol.2012.09.020. ISSN 0042-6822. PMID 23079106.
- ^ Krakauer, D. C.; Plotkin, J. B. (29 January 2002). "Redundancy, antiredundancy, and the robustness of genomes". Proceedings of the National Academy of Sciences. 99 (3): 1405–1409. Bibcode:2002PNAS...99.1405K. doi:10.1073/pnas.032668599. ISSN 0027-8424. PMC 122203. PMID 11818563.
- ^ Feiss, Michael; Fisher, R.A.; Crayton, M.A.; Egner, Carol (March 1977). "Packaging of the bacteriophage λ chromosome: Effect of chromosome length". Virology. 77 (1): 281–293. doi:10.1016/0042-6822(77)90425-1. ISSN 0042-6822. PMID 841861.
- ^ Aoyama, A; Hayashi, M (September 1985). "Effects of genome size on bacteriophage phi X174 DNA packaging in vitro". Journal of Biological Chemistry. 260 (20): 11033–11038. doi:10.1016/s0021-9258(17)39144-5. ISSN 0021-9258. PMID 3161888.
- ^ Wu, Zhijian; Yang, Hongyan; Colosi, Peter (January 2010). "Effect of Genome Size on AAV Vector Packaging". Molecular Therapy. 18 (1): 80–86. doi:10.1038/mt.2009.255. ISSN 1525-0016. PMC 2839202. PMID 19904234.
- ^ Vaidyanathan, Sriram; Baik, Ron; Chen, Lu; Bravo, Dawn T.; Suarez, Carlos J.; Abazari, Shayda M.; Salahudeen, Ameen A.; Dudek, Amanda M.; Teran, Christopher A.; Davis, Timothy H.; Lee, Ciaran M. (March 2021). "Targeted replacement of full-length CFTR in human airway stem cells by CRISPR-Cas9 for pan-mutation correction in the endogenous locus". Molecular Therapy. 30 (1): 223–237. doi:10.1016/j.ymthe.2021.03.023. PMC 8753290. PMID 33794364. S2CID 232761334.
- ^ Willis, Sara; Masel, Joanna (19 July 2018). "Gene Birth Contributes to Structural Disorder Encoded by Overlapping Genes". Genetics. 210 (1): 303–313. doi:10.1534/genetics.118.301249. ISSN 1943-2631. PMC 6116962. PMID 30026186.
- ^ Pavesi, Angelo; Vianelli, Alberto; Chirico, Nicola; Bao, Yiming; Blinkova, Olga; Belshaw, Robert; Firth, Andrew; Karlin, David (19 October 2018). "Overlapping genes and the proteins they encode differ significantly in their sequence composition from non-overlapping genes". PLOS ONE. 13 (10): e0202513. Bibcode:2018PLoSO..1302513P. doi:10.1371/journal.pone.0202513. ISSN 1932-6203. PMC 6195259. PMID 30339683.
- ^ Pavesi, Angelo; Magiorkinis, Gkikas; Karlin, David G. (15 August 2013). "Viral Proteins Originated De Novo by Overprinting Can Be Identified by Codon Usage: Application to the "Gene Nursery" of Deltaretroviruses". PLOS Computational Biology. 9 (8): e1003162. Bibcode:2013PLSCB...9E3162P. doi:10.1371/journal.pcbi.1003162. ISSN 1553-7358. PMC 3744397. PMID 23966842.
- ^ "Supplemental Information 2: NCBI genome database accession information (PDF file)". dx.doi.org. doi:10.7717/peerj.6447/supp-2. Retrieved 3 December 2021.
- ^ Ahmed, Niyaz (27 March 2009). "Faculty Opinions recommendation of The RAST Server: rapid annotations using subsystems technology". Faculty Opinions – Post-Publication Peer Review of the Biomedical Literature. doi:10.3410/f.1157743.618965. Retrieved 3 December 2021.
- ^ Ben-Tal, Nir, ed. (23 June 2017). "Decision letter: Deep transcriptome annotation enables the discovery and functional characterization of cryptic small proteins". doi:10.7554/elife.27860.082.
{{cite journal}}:Cite 저널 요구 사항journal=(도움말) - ^ Bazzini, Ariel; Wu, Qiushuang (6 March 2020). "Faculty Opinions recommendation of Pervasive functional translation of noncanonical human open reading frames". Faculty Opinions – Post-Publication Peer Review of the Biomedical Literature. doi:10.3410/f.737484924.793572056. S2CID 215850701. Retrieved 3 December 2021.
- ^ Prensner, John R.; Enache, Oana M.; Luria, Victor; Krug, Karsten; Clauser, Karl R.; Dempster, Joshua M.; Karger, Amir; Wang, Li; Stumbraite, Karolina; Wang, Vickie M.; Botta, Ginevra (28 January 2021). "Noncanonical open reading frames encode functional proteins essential for cancer cell survival". Nature Biotechnology. 39 (6): 697–704. doi:10.1038/s41587-020-00806-2. ISSN 1087-0156. PMC 8195866. PMID 33510483.
- ^ Cao, Xiongwen; Khitun, Alexandra; Luo, Yang; Na, Zhenkun; Phoodokmai, Thitima; Sappakhaw, Khomkrit; Olatunji, Elizabeth; Uttamapinant, Chayasith; Slavoff, Sarah A. (5 March 2020). "Alt-RPL36 downregulates the PI3K-AKT-mTOR signaling pathway by interacting with TMEM24". Nature Communications. 12 (1): 508. bioRxiv 10.1101/2020.03.04.977314. doi:10.1038/s41467-020-20841-6. PMC 7820019. PMID 33479206. Retrieved 3 December 2021.
- ^ Jaschke, Paul R.; Dotson, Gabrielle A.; Hung, Kay S.; Liu, Diane; Endy, Drew (12 November 2019). "Definitive demonstration by synthesis of genome annotation completeness". Proceedings of the National Academy of Sciences. 116 (48): 24206–24213. doi:10.1073/pnas.1905990116. ISSN 0027-8424. PMC 6883844. PMID 31719208.