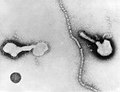
RNA바이러스
RNA virus이 문서의 일부(볼티모어 섹션의 분류법과 관련된 부분 [ICTV release 2018b→2019])를 업데이트해야 한다.(2021년 1월) |
RNA 바이러스는 레트로바이러스가 아닌 리보핵산을 유전 [1]물질로 하는 바이러스이다.핵산은 보통 단일 가닥 RNA(ssRNA)이지만 이중 가닥 RNA(dsRNA)[2]일 수 있습니다.RNA 바이러스에 의해 야기되는 주목할 만한 인간 질병은 일반적인 감기, 인플루엔자, 사스, 메르스, Covid-19, 뎅기 바이러스, C형 간염, E형 간염, 웨스트 나일 열, 에볼라 바이러스, 광견병, 소아마비, 유행성 이하선염, 홍역을 포함한다.
ICTV(International Committee on Taxonomy of Virus)는 RNA 바이러스를 볼티모어 분류 시스템의 그룹 III, 그룹 IV 또는 그룹 V에 속하는 바이러스로 분류합니다.이 범주는 RNA 유전 물질을 가지고 있지만 생명 주기에 DNA 중간체를 사용하는 VI 그룹 바이러스는 제외한다: 이것들은 에이즈를 일으키는 HIV-1과 HIV-2를 포함한 [3]레트로바이러스라고 불린다.
2020년 5월 현재, RNA 유도 RNA 중합효소를 코드하는 모든 알려진 RNA 바이러스는 리보비리아로 [4]알려진 단통군을 형성하는 것으로 믿어진다.그러한 RNA 바이러스의 대부분은 Orthornavirae 왕국에 속하고 나머지는 아직 [5]정의되지 않은 위치를 가지고 있다.영역에 일부 RNA 바이러스가 포함되어 있지 않습니다.Deltavirus, Asunviroidae, Pospiviroidae는 RNA [a]바이러스의 분류군으로 2019년에 잘못 포함되었으나 [6]2020년에 수정되었다.
특성.
단일 가닥 RNA 바이러스 및 RNA 감지
RNA 바이러스는 RNA의 감각이나 극성에 따라 음의 감각과 양의 감각 또는 양성의 RNA 바이러스로 더 분류될 수 있습니다.양성 감각 바이러스 RNA는 mRNA와 유사하기 때문에 숙주 세포에 의해 즉시 번역될 수 있다.음감 바이러스 RNA는 mRNA와 상보적이므로 번역하기 전에 RNA 의존성 RNA 중합효소에 의해 양감 RNA로 전환되어야 한다.양성 바이러스의 정제 RNA는 전체 바이러스 입자에 비해 감염성이 낮을 수 있지만 직접적으로 감염을 일으킬 수 있다.반면 음감 바이러스의 정제 RNA는 양감 RNA로 전사될 필요가 있기 때문에 그 자체로 감염되지 않는다. 각각의 비리온은 여러 양감 RNA로 전사될 수 있다.앰비센스 RNA 바이러스는 음의 가닥과 양의 [7]가닥에서 유전자를 변환한다는 점을 제외하면 음의 감각 RNA 바이러스와 유사하다.
이중가닥 RNA바이러스
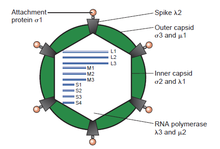
이중 가닥(ds)RNA 바이러스는 숙주 범위(인간, 동물, 식물, 곰팡이,[b] 박테리아), 게놈 세그먼트 번호(1~12개), 바이러스 조직(삼각형 수, 캡시드 층, 스파이크, 포탑 등)에서 매우 다양한 바이러스군을 나타냅니다.이 그룹의 구성원은 어린 아이들의 위장염의 가장 흔한 원인인 로타바이러스와 설사의 징후가 있거나 없는 인간과 동물의 배설물 샘플에서 가장 흔한 바이러스인 피코비르나바이러스를 포함한다.블루통그 바이러스는 소와 양을 감염시키는 경제적으로 중요한 병원체이다.최근 몇 년 동안 여러 dsRNA 바이러스의 주요 바이러스 단백질과 비리온 캡시드의 원자 및 서브나노미터 분해능 구조를 결정하는 데 진전이 있었으며, 이러한 [2][page needed]바이러스의 구조와 복제 과정에서 중요한 유사성이 강조되었다.
돌연변이율
RNA 바이러스는 일반적으로 DNA [9]바이러스에 비해 매우 높은 돌연변이율을 가지고 있는데, 왜냐하면 바이러스 RNA 중합효소는 DNA [10]중합효소의 교정 능력이 부족하기 때문이다.RNA 바이러스의 유전적 다양성은 [11]효과적인 백신을 만들기 어려운 이유 중 하나이다.레트로바이러스는 또한 DNA 중간체가 숙주 게놈에 통합됨에도 불구하고 높은 돌연변이율을 가지고 있다. 왜냐하면 역전사 중의 오류는 [12]통합되기 전에 양쪽 DNA 가닥에 내장되기 때문이다.RNA 바이러스의 일부 유전자는 바이러스 복제 주기에 중요하기 때문에 돌연변이는 용납되지 않는다.예를 들어 코어 단백질을 코드하는 C형 간염 바이러스 게놈 영역은 내부 리보솜 진입 부위에 [14]관여하는 RNA 구조를 포함하고 있기 때문에 [13]보존성이 높다.
레플리케이션
동물 RNA 바이러스는 ICTV에 의해 분류된다.RNA 바이러스에는 게놈과 복제 모드에 따라 세 가지 그룹이 있습니다.
- 이중 가닥 RNA 바이러스(그룹 III)는 각각 하나 이상의 바이러스 단백질을 코딩하는 1개에서 12개의 서로 다른 RNA 분자를 포함합니다.
- 양성 감각의 ssRNA 바이러스(그룹 IV)는 그들의 게놈을 mRNA로 직접 활용하며, 숙주 리보솜은 복제를 위해 필요한 다양한 단백질을 형성하기 위해 숙주에 의해 변형되는 단일 단백질과 바이러스 단백질로 변환한다.이들 중 하나는 RNA의존성 RNA중합효소(RNA 리플리케이트라아제)를 포함하고 있으며, 이는 바이러스 RNA를 복제하여 이중가닥 복제 형태를 형성한다.차례로, 이 dsRNA는 새로운 바이러스 RNA의 형성을 지시한다.
- 음의 ssRNA 바이러스(그룹 V)는 양성의 RNA를 형성하기 위해 RNA 복제 효소에 의해 게놈을 복제해야 합니다.이것은 바이러스가 RNA 복제 효소를 함께 가져와야 한다는 것을 의미한다.양감각 RNA 분자는 바이러스 mRNA로 작용하며, 이는 숙주 리보솜에 의해 단백질로 변환됩니다.
레트로바이러스(Group VI)는 단가닥 RNA 게놈을 가지고 있지만, 일반적으로 복제하기 위해 DNA 중간체를 사용하기 때문에 RNA 바이러스로 간주되지 않는다.바이러스가 코팅되지 않은 후 바이러스 자체에서 나오는 바이러스 효소인 역전사효소는 바이러스 RNA를 DNA의 상보적인 가닥으로 변환하고, DNA는 복제되어 이중 가닥의 바이러스 DNA 분자를 생성한다.이 DNA가 바이러스 효소 인테그레이스를 사용하여 숙주 게놈에 통합된 후, 암호화된 유전자의 발현은 새로운 바이러스 이온의 형성을 초래할 수 있다.
재결합
수많은 RNA 바이러스는 적어도 두 개의 바이러스 게놈이 같은 숙주 [15]세포에 존재할 때 유전자 재조합이 가능하다.매우 드물게 바이러스 RNA가 [16]숙주 RNA와 재결합할 수 있다. RNA 재조합은 피코나바이러스과(+) ssRNA)[17] 간의 게놈 구조와 바이러스 진화 과정을 결정하는 주요 원동력으로 보인다.예를 들어 HIV와 같은 Retroviridae((+)ssRNA)에서 RNA 게놈의 손상은 [18][19][20]재조합의 한 형태인 가닥 전환에 의한 역전사 중에 회피되는 것으로 보인다.재조합은 Reoviridae(dsRNA), Orthomyxoviridae(-)ssRNA(인플루엔자 [20]바이러스), Coronaviridae(+)ssRNA(SARS)[21] 등에서도 발생합니다.RNA 바이러스의 재조합은 게놈 [15]손상에 대처하기 위한 적응으로 보인다.재조합은 같은 종이지만 혈통이 다른 동물 바이러스들 사이에서 드물게 발생할 수 있다.그 결과 발생하는 재조합 바이러스는 때때로 사람에게 [21]감염의 발생을 일으킬 수 있다.
분류
RNA 바이러스의 분류는 어렵다.이것은 부분적으로 이 게놈들이 겪는 높은 돌연변이율 때문이다.분류는 주로 게놈 유형(이중가닥, 음성 또는 양성 단일가닥)과 유전자 번호 및 구성에 기초한다.현재 확인된 RNA 바이러스는 5개 목과 47개 계열이다.또한 많은 미지정 종과 속들이 있다.
RNA 바이러스와 관련이 있지만 구별되는 것은 비로이드와 RNA 위성 바이러스입니다.이들은 현재 RNA 바이러스로 분류되지 않으며 자체 페이지에 설명되어 있습니다.
수천 개의 RNA 바이러스에 대한 연구는 적어도 5개의 주요 분류군의 존재를 보여주었다: 레비 바이러스 및 친척 그룹, 피코나 바이러스 슈퍼 그룹, 알파 바이러스 슈퍼 그룹 및 플라비 바이러스 슈퍼 그룹, dsRNA 바이러스 및 -ve 스트랜드 [22]바이러스.렌티바이러스 그룹은 남아있는 모든 RNA 바이러스의 기초가 되는 것으로 보인다.다음 주요 분할은 피코르나수프라군과 나머지 바이러스 사이에 있습니다.dsRNA 바이러스는 +ve RNA 조상에서, -ve RNA 바이러스는 dsRNA 바이러스 내에서 진화한 것으로 보입니다.-가닥 RNA 바이러스와 가장 가까운 관계는 Reoviridae이다.
양성 가닥 RNA 바이러스
이것은 30개의 패밀리를 가진 가장 큰 RNA[23] 바이러스 그룹입니다.이들 가족을 더 높은 순서로 분류하려는 시도가 있었다.이러한 제안들은 RNA 중합효소의 분석에 기초했으며 여전히 검토 중이다.지금까지 제안된 제안들은 단일 유전자가 분지 분류법을 결정하는 데 적합한지에 대한 의구심 때문에 널리 받아들여지지 않았다.
제안된 양성 가닥 RNA 바이러스의 분류는 RNA의존성 RNA 중합효소에 기초한다.세 가지 그룹이 [24]인식되었습니다.
- 바이모바이러스, 코모바이러스, 네포바이러스, 노다바이러스, 피코나바이러스, 포티바이러스, 소베모바이러스 및 루테오바이러스(비트 웨스턴 옐로우스 바이러스와 감자 잎말이 바이러스)의 서브셋인 피코나 유사군(피코나비라타).
- 카르모바이러스, 뎬토바이러스, 플라비바이러스, 페스티바이러스, 스타토바이러스, 톰버스바이러스, 외가닥 RNA 박테리오파지, C형 간염 바이러스 및 루테오바이러스(배럴형 황색 왜소 바이러스)의 하위 집합인 플라비바이러스(플라비바이러스)
- 알파바이러스, 칼라바이러스, 푸로바이러스, 호다이바이러스, 포텍스바이러스, 루비바이러스, 토브라바이러스, 트리코르나바이러스, 타이모바이러스, 사과클로로틱잎스팟바이러스, 비트황색바이러스 및 E형 간염바이러스(루비바이러스)는 알파 유사군이다.
바이러스 복제에 관여하는 단백질의 N 말미니 근처에 위치한 새로운 도메인에 기초한 알파 유사(신드비스 유사) 슈퍼그룹의 분할이 [25]제안되었다.제안된 두 그룹은 '알토바이러스' 그룹(알파바이러스, 푸로바이러스, E형 간염 바이러스, 호다이바이러스, 토바모바이러스, 토브라바이러스, 트리코나바이러스 그리고 아마도 루비바이러스)과 '티포바이러스' 그룹(사과 엽록소 바이러스, 칼라바이러스, 포테바이러스, 티무비루스)이다.
알파 유사 슈퍼그룹은 루비 유사 바이러스, 토바모 유사 바이러스,[26] 타이모 유사 바이러스의 세 가지 군으로 더 나눌 수 있습니다.
추가 연구를 통해 [27]각각 4, 3, 3, 3, 3 및 1개의 순서를 포함하는 5개의 양성 가닥 RNA 바이러스 그룹이 확인되었다.이들 14개 목은 31개 바이러스 계열(17개 식물 바이러스 계열 포함)과 48개 속(30개 식물 바이러스 포함)을 포함하고 있다.이 분석에 따르면 알파바이러스와 플라비바이러스는 각각 토가바이러스과와 플라비바이러스과라는 두 과로 분리될 수 있지만 페스티바이러스, C형 간염 바이러스, 루비바이러스, E형 간염 바이러스, 동맥 바이러스와 같은 다른 분류학적 할당은 정확하지 않을 수 있다.코로나바이러스와 토로바이러스는 서로 다른 과로 분류되며 현재 분류되는 서로 다른 과의 다른 속은 아닌 것으로 보인다.루테오바이러스는 한 과가 아닌 두 과로 보이며 사과 엽록소성 반점 바이러스는 클로스테로바이러스가 아니라 포텍스바이러스과의 새로운 속인 것으로 보인다.
진화
피코나바이러스의 진화는 그들의 RNA 중합효소 및 헬리케이스의 분석에 기초한 것으로 진핵생물의 [28]발산까지 거슬러 올라간다.그들의 추정 조상은 박테리아 그룹 II 레트로 요소, HtrA 단백질 분해효소 및 DNA 박테리오파지를 포함합니다.
파르티바이러스는 토티바이러스 [29]조상과 관련이 있고 토티바이러스 조상에서 진화했을 수도 있다.
저바이러스와 바나바이러스는 각각 [29]포티바이러스 계열과 소베모바이러스 계열과 조상을 공유하는 것으로 보인다.
이중가닥 RNA바이러스
또한 이 분석에 따르면 dsRNA 바이러스는 서로 밀접하게 관련되어 있지 않으며 대신 Birnaviridae, Castoviridae, Partitiviridae 및 Reoviridae의 4개 추가 등급과 양성 SSRNA 바이러스 중 하나의 추가 등급(Totiviridae)에 속해 있습니다.
한 연구에 따르면 두 개의 큰 군락이 있습니다.하나는 칼리시바이러스과, 플라비바이러스과, 피코나바이러스과를 포함하고, 다른 하나는 알파테트라바이러스과, 비르나바이러스과, 시스토바이러스과, 노다바이러스과 [30]및 페름토트레트라바이러스과를 포함한다.
음성 가닥 RNA 바이러스
이 바이러스들은 단일 RNA 분자에서 8개의 세그먼트에 이르는 다양한 종류의 게놈을 가지고 있다.그들의 다양성에도 불구하고, 그들은 절지동물에서 유래했고 [31]거기서부터 다양해진 것으로 보인다.
위성 바이러스
라이프 사이클을 완료하기 위해 다른 바이러스의 지원이 필요한 바이러스인 위성 바이러스도 다수 알려져 있습니다.그들의 분류법은 아직 정해지지 않았다.식물을 감염시키는 양성 단일 가닥 RNA 위성 바이러스인 알베토바이러스, 아우마이바이러스, 파파니바이러스, 비르토바이러스에 [32]대한 다음 4개 속들이 제안되었다.절지동물을 감염시키는 양성 단일 가닥 RNA 위성 바이러스에 대해 마크롱 바이러스속을 포함하는 사트로바이러스과(Sarthroviridae)가 제안되었다.
그룹 III – dsRNA 바이러스
이 그룹에는 12개 과와 다수의 미지정 속과 종이 알려져 있다.[10]
- 아말가바이러스과
- 비르나바이러스과
- 크리소바이러스과
- 시스토바이러스과
- 엔도르나바이러스과
- 하이포바이러스과
- 메가비르나바이러스과
- 파르티비루스과
- 피코비르나바이러스과
- 로타바이러스과 – 로타바이러스 포함
- 토티바이러스과
- 콰드리바이러스과
- 보티비르나바이러스속
- 미지정종
그룹 IV – 양성 감지 ssRNA 바이러스
이 그룹에는 3개의 오더와 34개의 패밀리가 있습니다.게다가, 많은 미분류 종과 속들이 있다.
- 니도비랄레스목
- 피코나비랄레스목
- 디키스트로바이러스과
- 이플라비루스과
- 마나비루스과
- Picornaviridae 패밀리– 폴리오바이러스, 라이노바이러스(일반 감기 바이러스), A형 간염 바이러스 포함
- 세코바이러스과(Secoviridae)는 코모바이러스아과(Comovirinae)를 포함한다.
- 바실라리오르나바이러스속
- 종 다시마파리바이러스
- Tymovirales 주문
- 미할당
- 알파테트라바이러스과
- 알베르나비루스과
- 아스트로바이러스과
- 바르나비루스과
- 베니바이러스과
- 보투르미아바이러스과
- 브로모바이러스과
- 갈리시바이러스과 – Norwalk 바이러스 포함
- 카모테트라바이러스과
- 클로스테로바이러스과
- 플라비바이러스과 패밀리– 황열 바이러스, 웨스트 나일 바이러스, C형 간염 바이러스, 뎅기열 바이러스, 지카 바이러스 포함
- 후사리바이러스과
- 헤페바이러스과
- 하이포바이러스과
- 레비바이러스과
- 루테오바이러스과 – 보리황색왜소바이러스 포함
- 폴리시피바이러스과
- 나나비루스과
- 노다바이러스과
- 페르무토테트라바이러스과
- 포티바이러스과
- 사트로바이러스과
- 스타토바이러스과
- 토가바이러스과 – 풍진바이러스, 로스리버바이러스, 신비스바이러스, 치쿤구냐바이러스 포함
- 톰부스바이러스과
- 버가바이러스과[33]
- 미지정속
- 미지정종
- 아시르토시폰피썸바이러스
- 바스트로바이러스
- 블랙포드 바이러스
- 블루베리괴사환반점바이러스
- 카디시스트로바이러스
- 카라오스트레일리아바이러스
- 초소형 바이러스
- 고지베리클로로시스바이러스
- 하르모니아악시리디스바이러스1
- 헤펠리바이러스
- 징먼진드기바이러스
- 르블랑바이러스
- 네디시스트로바이러스
- 네시디오코리스텐우이스바이러스1
- 니플라바이러스
- 니란데리아풀바바이러스1
- 오르세 바이러스
- 오세닥자포닉스RNA바이러스1
- 삐칼리바이러스
- 플라나리아분비세포니도바이러스
- 플라스모파라할스테디바이러스
- 로젤리니아네카트릭스후사리바이러스1
- 산테유바이러스
- 세칼리바이러스
- 솔레놉시스 인빅타 바이러스 3
- 우한대돼지회충바이러스
위성 바이러스
미분류 아스트로바이러스/헤페바이러스 유사 바이러스도 [34]기술되었다.
그룹 V – 음의 ssRNA 바이러스
D형 간염 바이러스를 제외하고, 이 바이러스군은 단일 문인 Negarnaviricota에 배치되어 있습니다.이 문(문)은 두 개의 아종인 하플로빌리코티나와 폴리플로빌리코티나로 나뉘었다.하플로빌리코티나아문 내에서는 현재 4개의 클래스가 인식되고 있다.춘추비리세테스, 밀네비리세테스, 몬지비리세테스, 윤창비리세테스.폴리플로빌리코티나아문에서는 다음 두 가지 클래스가 인식됩니다.엘리오바이러스제와 인스토바이러스제.
현재 이 그룹에는 6개 클래스, 7개 주문, 24개 가족이 등록되어 있습니다.많은 미지정 종과 속은 아직 [10]분류되지 않았다.
- 네가르나비리코타문[35]
- 하플로빌리코티나아문
- 폴리플로바이러스아문
- 엘리오바이러스류
- 클래스 Insthoviricetes
- Articulavirales 주문
- 타스트럽바이러스과 (Amnoonviridae)– Taastrup 바이러스 포함
- 오르토믹소바이러스과– 인플루엔자 바이러스 포함
- Articulavirales 주문
- 할당되지 않은 속:
갤러리
유행성 이하선염바이러스과(Paramyxoviridae)
「 」를 참조해 주세요.
메모들
레퍼런스
- ^ Wagner, Edward K.; Hewlett, Martinez J. (1999). Basic virology. Malden, MA: Blackwell Science, Inc. p. 249. ISBN 0-632-04299-0. Retrieved 30 March 2020.
- ^ "Listing in Taxonomic Order – Index to ICTV Species Lists". Retrieved 11 April 2008.
- ^ International Committee on Taxonomy of Viruses Executive Committee (May 2020). "The new scope of virus taxonomy: partitioning the virosphere into 15 hierarchical ranks". Nature Microbiology. 5 (5): 668–674. doi:10.1038/s41564-020-0709-x. PMC 7186216. PMID 32341570.
- ^ TaxoProp 2019.006G
- ^ TaxoProp 2019.009G
- ^ Nguyen M, Haenni AL (June 2003). "Expression strategies of ambisense viruses". Virus Research. 93 (2): 141–50. doi:10.1016/S0168-1702(03)00094-7. PMID 12782362.
- ^ Kondo H, Chiba S, Toyoda K, Suzuki N (January 2013). "Evidence for negative-strand RNA virus infection in fungi". Virology. 435 (2): 201–09. doi:10.1016/j.virol.2012.10.002. PMID 23099204.
- ^ Sanjuán R, Nebot MR, Chirico N, Mansky LM, Belshaw R (October 2010). "Viral mutation rates". Journal of Virology. 84 (19): 9733–48. doi:10.1128/JVI.00694-10. PMC 2937809. PMID 20660197.
- ^ a b c Klein DW, Prescott LM, Harley J (1993). Microbiology. Dubuque, Iowa: Wm. C. Brown. ISBN 978-0-697-01372-9.
- ^ Steinhauer DA, Holland JJ (1987). "Rapid evolution of RNA viruses". Annual Review of Microbiology. 41: 409–33. doi:10.1146/annurev.mi.41.100187.002205. PMID 3318675.
- ^ Boutwell CL, Rolland MM, Herbeck JT, Mullins JI, Allen TM (October 2010). "Viral evolution and escape during acute HIV-1 infection". The Journal of Infectious Diseases. 202 Suppl 2 (Suppl 2): S309–14. doi:10.1086/655653. PMC 2945609. PMID 20846038.
- ^ Bukh J, Purcell RH, Miller RH (August 1994). "Sequence analysis of the core gene of 14 hepatitis C virus genotypes". Proceedings of the National Academy of Sciences of the United States of America. 91 (17): 8239–43. Bibcode:1994PNAS...91.8239B. doi:10.1073/pnas.91.17.8239. PMC 44581. PMID 8058787.
- ^ Tuplin A, Evans DJ, Simmonds P (October 2004). "Detailed mapping of RNA secondary structures in core and NS5B-encoding region sequences of hepatitis C virus by RNase cleavage and novel bioinformatic prediction methods". The Journal of General Virology. 85 (Pt 10): 3037–47. doi:10.1099/vir.0.80141-0. PMID 15448367.
- ^ a b Barr JN, Fearns R (June 2010). "How RNA viruses maintain their genome integrity". The Journal of General Virology. 91 (Pt 6): 1373–87. doi:10.1099/vir.0.020818-0. PMID 20335491.
- ^ Stedman, Kenneth M. (2015). "Deep Recombination: RNA and ssDNA Virus Genes in DNA Virus and Host Genomes". Annual Review of Virology. 2 (1): 203–217. doi:10.1146/annurev-virology-100114-055127. ISSN 2327-0578. PMID 26958913.
- ^ Muslin C, Mac Kain A, Bessaud M, Blondel B, Delpeyroux F (September 2019). "Recombination in Enteroviruses, a Multi-Step Modular Evolutionary Process". Viruses. 11 (9): 859. doi:10.3390/v11090859. PMC 6784155. PMID 31540135.
- ^ Hu WS, Temin HM (November 1990). "Retroviral recombination and reverse transcription". Science. 250 (4985): 1227–33. Bibcode:1990Sci...250.1227H. doi:10.1126/science.1700865. PMID 1700865.
- ^ Rawson JM, Nikolaitchik OA, Keele BF, Pathak VK, Hu WS (November 2018). "Recombination is required for efficient HIV-1 replication and the maintenance of viral genome integrity". Nucleic Acids Research. 46 (20): 10535–45. doi:10.1093/nar/gky910. PMC 6237782. PMID 30307534.
- ^ a b Bernstein H, Bernstein C, Michod RE (January 2018). "Sex in microbial pathogens". Infection, Genetics and Evolution. 57: 8–25. doi:10.1016/j.meegid.2017.10.024. PMID 29111273.
- ^ a b Su S, Wong G, Shi W, Liu J, Lai AC, Zhou J, et al. (June 2016). "Epidemiology, Genetic Recombination, and Pathogenesis of Coronaviruses". Trends in Microbiology. 24 (6): 490–502. doi:10.1016/j.tim.2016.03.003. PMC 7125511. PMID 27012512.
- ^ Wolf YI, Kazlauskas D, Iranzo J, Lucía-Sanz A, Kuhn JH, Krupovic M, Dolja VV, Koonin EV (November 2018). "Origins and Evolution of the Global RNA Virome". mBio. 9 (6). doi:10.1128/mBio.02329-18. PMC 6282212. PMID 30482837.
- ^ Francki RI, Fauquet CM, Knudson DL, Brown F (1991). Classification and nomenclature of viruses. Fifth report of the International Committee on Taxonomy of Viruses, Archives of Virology (Suppl. 2). ISBN 978-3-7091-9163-7.
- ^ Koonin EV (September 1991). "The phylogeny of RNA-dependent RNA polymerases of positive-strand RNA viruses". The Journal of General Virology. 72 (Pt 9): 2197–206. doi:10.1099/0022-1317-72-9-2197. PMID 1895057.
- ^ Rozanov MN, Koonin EV, Gorbalenya AE (August 1992). "Conservation of the putative methyltransferase domain: a hallmark of the 'Sindbis-like' supergroup of positive-strand RNA viruses". The Journal of General Virology. 73 (Pt 8): 2129–34. CiteSeerX 10.1.1.532.7367. doi:10.1099/0022-1317-73-8-2129. PMID 1645151.
- ^ Koonin EV, Dolja VV (1993). "Evolution and taxonomy of positive-strand RNA viruses: implications of comparative analysis of amino acid sequences". Critical Reviews in Biochemistry and Molecular Biology. 28 (5): 375–430. doi:10.3109/10409239309078440. PMID 8269709.
- ^ Ward CW (1993). "Progress towards a higher taxonomy of viruses". Research in Virology. 144 (6): 419–53. doi:10.1016/S0923-2516(06)80059-2. PMC 7135741. PMID 8140287.
- ^ Koonin EV, Wolf YI, Nagasaki K, Dolja VV (December 2008). "The Big Bang of picorna-like virus evolution antedates the radiation of eukaryotic supergroups". Nature Reviews. Microbiology. 6 (12): 925–39. doi:10.1038/nrmicro2030. PMID 18997823.
- ^ a b Ghabrial SA (1998). "Origin, adaptation and evolutionary pathways of fungal viruses". Virus Genes. 16 (1): 119–31. doi:10.1023/a:1007966229595. PMC 7089520. PMID 9562896.
- ^ Gibrat JF, Mariadassou M, Boudinot P, Delmas B (July 2013). "Analyses of the radiation of birnaviruses from diverse host phyla and of their evolutionary affinities with other double-stranded RNA and positive strand RNA viruses using robust structure-based multiple sequence alignments and advanced phylogenetic methods". BMC Evolutionary Biology. 13: 154. doi:10.1186/1471-2148-13-154. PMC 3724706. PMID 23865988.
- ^ Li CX, Shi M, Tian JH, Lin XD, Kang YJ, Chen LJ, et al. (January 2015). "Unprecedented genomic diversity of RNA viruses in arthropods reveals the ancestry of negative-sense RNA viruses". eLife. 4. doi:10.7554/eLife.05378. PMC 4384744. PMID 25633976.
- ^ Krupovic M, Kuhn JH, Fischer MG (January 2016). "A classification system for virophages and satellite viruses". Archives of Virology. 161 (1): 233–47. doi:10.1007/s00705-015-2622-9. PMID 26446887.
- ^ Adams MJ, Antoniw JF, Kreuze J (2009). "Virgaviridae: a new family of rod-shaped plant viruses". Archives of Virology. 154 (12): 1967–72. doi:10.1007/s00705-009-0506-6. PMID 19862474.
- ^ 판코빅스 P, 보로스 AH, 키스 T, 엥겔만 P, 로이터 G(2019) 육식 조류인 유럽 롤러(Coracias garrulus)에서 아스트로바이러스 유사(5'-end) 및 헤페바이러스 유사(3'-end) 게놈 구성을 가진 유전적으로 매우 발산되는 RNA 바이러스.감염 유전자 진화
- ^ "Virus Taxonomy: 2018 Release". International Committee on Taxonomy of Viruses. Retrieved 13 November 2018.
- ^ Mihindukulasuriya KA, Nguyen NL, Wu G, Huang HV, da Rosa AP, Popov VL, et al. (May 2009). "Nyamanini and midway viruses define a novel taxon of RNA viruses in the order Mononegavirales". Journal of Virology. 83 (10): 5109–16. doi:10.1128/JVI.02667-08. PMC 2682064. PMID 19279111.