RNA온도계
RNA thermometerRNA 온도계(또는 RNA 열감소기)는 유전자 발현을 조절하는 온도에 민감한 비코딩 RNA 분자다.RNA 온도계는 종종 열충격이나 냉충격 반응 동안에 필요한 유전자를 조절하지만, 병원성이나 기아와 같은 다른 규제 역할에 관여해왔다.[1]
일반적으로 RNA 온도계는 온도 변동에 대응하여 2차 구조를 변경하여 동작한다.이 구조적 변화는 리보솜 결합 사이트와 같은 RNA의 중요한 영역을 노출하거나 방해할 수 있으며, 이는 근처의 단백질 코딩 유전자의 변환율에 영향을 미친다.
RNA 온도계는 리보스와 함께 RNA 세계 가설을 뒷받침하는 예로 사용된다.이 이론은 RNA가 한때 세포에 존재하는 유일한 핵산이었으며, 현재의 DNA → RNA → 단백질 시스템으로 대체되었다고 제안한다.[2]
RNA 온도계의 예로는 FourU,[3] Hsp90 cis 조절 원소, [4]ROSE 원소,[5] Lig RNA 온도계,[6] Hsp17 온도계가 있다.[7]
디스커버리
첫 번째 온도에 민감한 RNA 원소는 1989년에 보고되었다.[8]본 연구에 앞서 람다(λ) 페이징 cIII mRNA의 전사 시작 지점 업스트림에서 돌연변이가 cIII 단백질의 번역 수준에 영향을 미치는 것으로 밝혀졌다.[9]이 단백질은 λ 페이지의 라이틱 또는 리소겐 라이프 사이클의 선택에 관여하며, 고농도의 cIII가 리소겐을 촉진한다.[9]이 업스트림 RNA 영역에 대한 추가 연구는 두 가지 대안적인 2차 구조를 확인했다; 실험 연구에서는 구조물이 상호 교환이 가능하고 마그네슘 이온 농도와 온도 모두에 의존한다는 것을 발견했다.[8][10]이 RNA 온도계는 이제 박테리오파지가 숙주 세포에서 빠르게 복제하고 탈출하기 위해 열 스트레스 하에서 리틱 사이클에 진입하도록 장려하는 것으로 생각된다.[1]
'RNA 온도계'라는 용어는 대장균에서 확인된 rPOH RNA 원소에 적용되기 전인 1999년까지 만들어지지 않았다.[11][12]좀 더 최근에, 생물정보학 연구는 몇몇 새로운 후보 RNA 온도계를 밝혀내기 위해 채택되었다.[13]그러나 원소의 2차 구조가 핵산 염기서열보다 훨씬 보존되어 있기 때문에 전통적인 염기서열 기반 검색은 비효율적이다.[13]
분배
대부분의 알려진 RNA 온도계는 메신저 RNA 인코딩 열충격 단백질의 5㎛ 미통역 영역(UTR)에 위치하지만, 이 사실은 부분적으로 유전체 데이터에서 짧고 서비스되지 않는 RNA 시퀀스를 검출하는 데 있어 샘플링 바이어스와 내재된 어려움 때문일 수 있다.[14][15]
주로 원핵생물에서 발견되지만, 인간을 포함한 포유류에서 잠재적 RNA 온도계가 발견되었다.[16]후보 열센서 열충격 RNA-1(HSR1)은 열충격전사 인자 1(HSF1)을 활성화하고 세포 온도가 37°C(체온)를 초과할 때 보호 단백질을 유도해 세포가 과열되는 것을 방지한다.[16]
구조
RNA 온도계는 구조적으로 단순하며 짧은 RNA 시퀀스로 만들어질 수 있다; 가장 작은 것은 44개의 뉴클레오티드일 뿐이며 PCC 6803의 열충격 단백질인 hsp17의 mRNA에서 발견된다.[18][19]일반적으로 이러한 RNA 원소들은 길이가 60에서 110까지[20] 다양하며 일반적으로 구조물의 안정성을 감소시켜 온도 상승에 반응하여 쉽게 펼 수 있는 불일치 베이스 쌍이 적은 헤어핀을 포함하고 있다.[21]
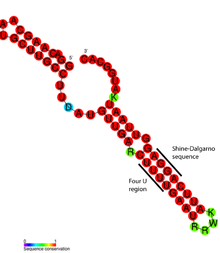
ROSE RNA 온도계의 상세한 구조 분석 결과 일치하지 않는 베이스가 RNA의 나선 구조를 보존하는 비표준 베이스페어링에 실제로 관여하고 있는 것으로 밝혀졌다(그림 참조).특이한 베이스페어는 G-G, U-U, UC-U 쌍으로 구성된다.이러한 비수평적 염기쌍은 상대적으로 불안정하기 때문에 온도 상승으로 인해 이 지역의 RNA 구조가 국소적으로 녹으면서 샤인-달가르노 순서가 노출된다.[17]
일부 RNA 온도계는 하나의 머리핀보다 훨씬 더 복잡하다. CspA mRNA에서 발견되는 부위의 경우처럼 가성비뿐만 아니라 여러 개의 머리핀도 포함하고 있다.[22][23]
합성 RNA 온도계는 단순한 단일 헤어핀 구조로 설계되었다.[24]그러나 단 하나의 기저 변화로 머리핀이 체내에서 비활성화될 수 있기 때문에 그러한 짧은 RNA 온도계의 2차 구조는 돌연변이에 민감할 수 있다.[25]
메커니즘
RNA 온도계는 단백질 코딩 유전자의 업스트림인 메신저 RNA의 5 5 UTR에서 발견된다.[1]여기서 그들은 리보솜 결합 사이트(RBS)를 차단하고 mRNA가 단백질로 변환되는 것을 막을 수 있다.[14]온도가 상승하면 헤어핀 구조가 '멜트'되어 RBS나 샤인달가노 시퀀스를 노출시켜 소형 리보솜 소단위(30S)의 결합을 허용하고, 이후 다른 번역기계를 조립한다.[1]일반적으로 샤인 달가노 수열의 하류에서 8개의 뉴클레오티드가 발견되는 시작 코돈은 리보솜에 의해 펩타이드 제품으로 번역되는 단백질 코딩 유전자의 시작을 나타낸다.[14]이 시스 작용 메커니즘 외에도, 단식 반응에 관여한다고 여겨지는 RpoS mRNA에서 트랜스액션 RNA 온도계의 유일한 예가 발견되었다.[1]
RNA 온도계 모티브의 구체적인 예로는 살모넬라 엔티카에서 발견된 FourU 온도계가 있다.[3]45 °C 이상의 온도에 노출되면 샤인-달가르노 수열 반대편에 있는 베이스퍼인 스템 루프가 손상되지 않고 mRNA가 리보솜에 들어가 번역을 할 수 있게 된다.[25]Mg2+ 이온 농도는 FourU의 안정성에도 영향을 미치는 것으로 나타났다.[26]가장 잘 연구된 RNA 온도계는 대장균의 rpoH 유전자에서 발견된다.[27]이 열감소기는 고온에서 열충격 단백질을 전문 열충격 시그마 인자인 σ을32 통해 상향 조절한다.[11]
일반적으로 열에 의한 단백질 발현과 관련이 있지만, RNA 온도계는 또한 냉충격 단백질을 조절할 수 있다.[22]예를 들어, 두 개의 7kDa 단백질의 발현이 열성 박테리아 테르무스 열성체에서[28] RNA 온도계에 의해 조절되며 엔테로박테리아에서도 이와 유사한 메커니즘이 확인되었다.[23]
37 °C의 온도에 민감한 RNA 온도계는 병원균에 의해 감염 특이 유전자를 활성화하는데 사용될 수 있다.[14]예를 들어, 리스테리아 모노키토제네스에 있는 독성 유전자의 주요 전사 조절기를 인코딩하는 prfA의 상향 조절은 prfA의 5′ DNA를 녹색 형광 단백질 유전자에 융합시킴으로써 증명되었다. 그런 다음 유전자 융합을 대장균의 T7 촉진제로부터 복사했고, 형광은 37℃에서 30℃에서 관찰되지 않았다.[29]
RNA 세계 가설에 대한 의미
RNA 세계 가설은 RNA가 한때 유전적 정보의 매개체였고 효소적으로 활성했으며, 서로 다른 시퀀스가 생체 촉매, 조절기 및 센서의 역할을 했다고 명시하고 있다.[30]그 가설은 그 후 현대의 DNA, RNA, 단백질 기반의 생명체가 진화했고 선택으로 RNA의 역할 대부분을 다른 생체 분자와 대체했다고 제안한다.[2]
RNA 온도계와 리보스 와치는 먼 곳과 관련된 유기체에서 광범위한 분포로 인해 진화적으로 오래된 것으로 생각된다.[31]RNA 세계에서는 RNA 열감지기가 다른 RNA 분자의 온도 의존적 조절을 담당했을 것이라는 제안이 제기되었다.[2][32]현대 유기체의 RNA 온도계는 이전에 RNA 세계에서 더 널리 퍼져있던 중요성을 암시할 수 있는 분자 화석일 수 있다.[2]
기타 예
- Hsp90 시스 조절 원소는 드로소필라에서 hsp90을 조절해 고온에서 열충격 단백질의 번역률을 높인다.[4]
- 대장균의 IbpAB 피연산자는 두 개의 협동 RNA 온도계, 즉 ROSE 원소와 IbpB 온도계를 포함할 것으로 예측된다.[33]
- ROSE와1 ROSE는AT2 각각 Hyphomicrobiales Bradyrhizobium japonicum과 Agrobatterium tomepaciens에서 발견된다.그것들은 HspA mRNA의 5′ UTR에 존재하며, 생리적 온도에서 열충격 단백질 번역을 억제한다.[5][34]
- 시아노박테리아 RNA 온도계
- 유전자간 RNA온도계
- 네이세리아 RNA 온도계
- 리그 RNA 온도계
참조
- ^ a b c d e f Narberhaus F, Waldminghaus T, Chowdhury S (January 2006). "RNA thermometers". FEMS Microbiol. Rev. 30 (1): 3–16. doi:10.1111/j.1574-6976.2005.004.x. PMID 16438677.
- ^ a b c d Atkins JF, Gesteland RF, Cech T (2006). The RNA world: the nature of modern RNA suggests a prebiotic RNA world. Plainview, N.Y: Cold Spring Harbor Laboratory Press. ISBN 978-0-87969-739-6.
- ^ a b Waldminghaus T, Heidrich N, Brantl S, Narberhaus F (July 2007). "FourU: a novel type of RNA thermometer in Salmonella". Mol. Microbiol. 65 (2): 413–424. doi:10.1111/j.1365-2958.2007.05794.x. PMID 17630972.
- ^ a b Ahmed R, Duncan RF (2004). "Translational regulation of Hsp90 mRNA. AUG-proximal 5′-untranslated region elements essential for preferential heat shock translation". J Biol Chem. 279 (48): 49919–49930. doi:10.1074/jbc.M404681200. PMID 15347681.
- ^ a b Nocker A, Hausherr T, Balsiger S, Krstulovic NP, Hennecke H, Narberhaus F (2001). "A mRNA-based thermosensor controls expression of rhizobial heat shock genes". Nucleic Acids Res. 29 (23): 4800–4807. doi:10.1093/nar/29.23.4800. PMC 96696. PMID 11726689.
- ^ Matsunaga J, Schlax PJ, Haake DA (2013-11-15). "Role for cis-Acting RNA Sequences in the Temperature-Dependent Expression of the Multiadhesive Lig Proteins in Leptospira interrogans". Journal of Bacteriology. 195 (22): 5092–5101. doi:10.1128/jb.00663-13. ISSN 0021-9193. PMC 3811586. PMID 24013626.
- ^ Kortmann J, Sczodrok S, Rinnenthal J, Schwalbe H, Narberhaus F (2011). "Translation on demand by a simple RNA-based thermosensor". Nucleic Acids Res. 39 (7): 2855–2868. doi:10.1093/nar/gkq1252. PMC 3074152. PMID 21131278.
- ^ a b Altuvia S, Kornitzer D, Teff D, Oppenheim AB (1989-11-20). "Alternative mRNA structures of the cIII gene of bacteriophage lambda determine the rate of its translation initiation". Journal of Molecular Biology. 210 (2): 265–280. doi:10.1016/0022-2836(89)90329-X. PMID 2532257.
- ^ a b Altuvia S, Oppenheim AB (July 1986). "Translational regulatory signals within the coding region of the bacteriophage lambda cIII gene". Journal of Bacteriology. 167 (1): 415–419. doi:10.1128/jb.167.1.415-419.1986. PMC 212897. PMID 2941413.
- ^ Altuvia S, Kornitzer D, Kobi S, Oppenheim AB (1991-04-20). "Functional and structural elements of the mRNA of the cIII gene of bacteriophage lambda". Journal of Molecular Biology. 218 (4): 723–733. doi:10.1016/0022-2836(91)90261-4. PMID 1827163.
- ^ a b Storz G (1999-03-15). "An RNA thermometer". Genes & Development. 13 (6): 633–636. doi:10.1101/gad.13.6.633. PMID 10090718.
- ^ Morita MT, Tanaka Y, Kodama TS, Kyogoku Y, Yanagi H, Yura T (1999-03-15). "Translational induction of heat shock transcription factor sigma32: evidence for a built-in RNA thermosensor". Genes & Development. 13 (6): 655–665. doi:10.1101/gad.13.6.655. PMC 316556. PMID 10090722.
- ^ a b Waldminghaus T, Gaubig LC, Narberhaus F (November 2007). "Genome-wide bioinformatic prediction and experimental evaluation of potential RNA thermometers". Molecular Genetics and Genomics. 278 (5): 555–564. doi:10.1007/s00438-007-0272-7. PMID 17647020. S2CID 24747327.
- ^ a b c d Narberhaus F (2010). "Translational control of bacterial heat shock and virulence genes by temperature-sensing mRNAs". RNA Biol. 7 (1): 84–89. doi:10.4161/rna.7.1.10501. PMID 20009504. Retrieved 2011-04-23.
- ^ Johansson J (2009). "RNA thermosensors in bacterial pathogens". Bacterial Sensing and Signaling. Contrib Microbiol. Contributions to Microbiology. Vol. 16. Basel. pp. 150–160. doi:10.1159/000219378. ISBN 978-3-8055-9132-4. PMID 19494584.
- ^ a b Shamovsky I, Ivannikov M, Kandel ES, Gershon D, Nudler E (March 2006). "RNA-mediated response to heat shock in mammalian cells". Nature. 440 (7083): 556–560. Bibcode:2006Natur.440..556S. doi:10.1038/nature04518. PMID 16554823. S2CID 4311262.
- ^ a b Chowdhury S, Maris C, Allain FH, Narberhaus F (2006-06-07). "Molecular basis for temperature sensing by an RNA thermometer". The EMBO Journal. 25 (11): 2487–2497. doi:10.1038/sj.emboj.7601128. PMC 1478195. PMID 16710302.
- ^ Kortmann J, Sczodrok S, Rinnenthal J, Schwalbe H, Narberhaus F (April 2011). "Translation on demand by a simple RNA-based thermosensor". Nucleic Acids Research. 39 (7): 2855–2868. doi:10.1093/nar/gkq1252. PMC 3074152. PMID 21131278.
- ^ Kortmann J, Sczodrok S, Rinnenthal J, Schwalbe H, Narberhaus F (April 2011). "Translation on demand by a simple RNA-based thermosensor". Nucleic Acids Res. 39 (7): 2855–2868. doi:10.1093/nar/gkq1252. PMC 3074152. PMID 21131278.
- ^ Waldminghaus T, Fippinger A, Alfsmann J, Narberhaus F (December 2005). "RNA thermometers are common in alpha- and gamma-proteobacteria". Biol. Chem. 386 (12): 1279–1286. doi:10.1515/BC.2005.145. PMID 16336122. S2CID 84557068.
- ^ Narberhaus F (January–February 2010). "Translational control of bacterial heat shock and virulence genes by temperature-sensing mRNAs". RNA Biology. 7 (1): 84–89. doi:10.4161/rna.7.1.10501. PMID 20009504.
- ^ a b Breaker RR (January 2010). "RNA switches out in the cold". Mol. Cell. 37 (1): 1–2. doi:10.1016/j.molcel.2009.12.032. PMC 5315359. PMID 20129048.
- ^ a b Giuliodori AM, Di Pietro F, Marzi S, Masquida B, Wagner R, Romby P, Gualerzi CO, Pon CL (January 2010). "The cspA mRNA is a thermosensor that modulates translation of the cold-shock protein CspA". Mol. Cell. 37 (1): 21–33. doi:10.1016/j.molcel.2009.11.033. PMID 20129052.
- ^ Neupert J, Karcher D, Bock R (November 2008). "Design of simple synthetic RNA thermometers for temperature-controlled gene expression in Escherichia coli". Nucleic Acids Research. 36 (19): e124. doi:10.1093/nar/gkn545. PMC 2577334. PMID 18753148.
- ^ a b Nikolova EN, Al-Hashimi HM (September 2010). "Thermodynamics of RNA melting, one base pair at a time". RNA. 16 (9): 1687–1691. doi:10.1261/rna.2235010. PMC 2924531. PMID 20660079.
- ^ Rinnenthal J, Klinkert B, Narberhaus F, Schwalbe H (2011-07-04). "Modulation of the stability of the Salmonella fourU-type RNA thermometer". Nucleic Acids Research. 39 (18): 8258–8270. doi:10.1093/nar/gkr314. PMC 3185406. PMID 21727085.
- ^ Shah P, Gilchrist MA (2010). Spirin AS (ed.). "Is thermosensing property of RNA thermometers unique?". PLOS ONE. 5 (7): e11308. Bibcode:2010PLoSO...511308S. doi:10.1371/journal.pone.0011308. PMC 2896394. PMID 20625392.
- ^ Mega R, Manzoku M, Shinkai A, Nakagawa N, Kuramitsu S, Masui R (August 2010). "Very rapid induction of a cold shock protein by temperature downshift in Thermus thermophilus". Biochem. Biophys. Res. Commun. 399 (3): 336–340. doi:10.1016/j.bbrc.2010.07.065. PMID 20655297.
- ^ Johansson J, Mandin P, Renzoni A, Chiaruttini C, Springer M, Cossart P (September 2002). "An RNA thermosensor controls expression of virulence genes in Listeria monocytogenes". Cell. 110 (5): 551–561. doi:10.1016/S0092-8674(02)00905-4. PMID 12230973.
- ^ Gilbert W (February 1986). "The RNA World". Nature. 319 (6055): 618. Bibcode:1986Natur.319..618G. doi:10.1038/319618a0.
- ^ Serganov A, Patel DJ (October 2007). "Ribozymes, riboswitches and beyond: regulation of gene expression without proteins". Nature Reviews Genetics. 8 (10): 776–790. doi:10.1038/nrg2172. PMC 4689321. PMID 17846637.
- ^ Bocobza SE, Aharoni A (October 2008). "Switching the light on plant riboswitches". Trends in Plant Science. 13 (10): 526–533. doi:10.1016/j.tplants.2008.07.004. PMID 18778966.
- ^ Gaubig LC, Waldminghaus T, Narberhaus F (January 2011). "Multiple layers of control govern expression of the Escherichia coli ibpAB heat-shock operon". Microbiology. 157 (Pt 1): 66–76. doi:10.1099/mic.0.043802-0. PMID 20864473.
- ^ Balsiger S, Ragaz C, Baron C, Narberhaus F (2004). "Replicon-specific regulation of small heat shock genes in Agrobacterium tumefaciens". J Bacteriol. 186 (20): 6824–6829. doi:10.1128/JB.186.20.6824-6829.2004. PMC 522190. PMID 15466035.