스핀들 장치
Spindle apparatus세포생물학에서 방추장치는 딸세포 사이에 자매 염색체를 분리하기 위해 세포 분열 중에 형성되는 진핵세포의 세포골격 구조를 말한다.그것은 유전적으로 동일한 딸세포를 생성하는 과정인 유사분열 중의 유사분열 방추체 또는 부모 세포의 염색체 수의 절반인 생식체를 생성하는 과정인 감수분열 중의 감수분열 방추체라고 불립니다.
염색체 외에도 방추 장치는 수백 개의 [1][2]단백질로 구성되어 있다.마이크로튜브는 기계에서 가장 풍부한 구성 요소로 구성됩니다.
스핀들 구조
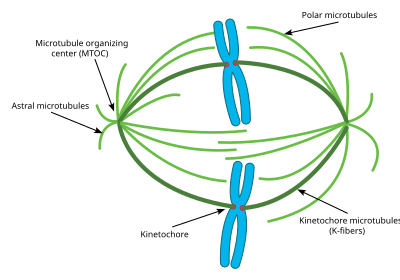
염색체에 대한 미소관의 부착은 스핀들 형성을 능동적으로 감시하고 조기 무지외상 발병을 방지하는 키네토코어에 의해 매개된다.미소관 중합과 탈중합은 염색체 결합을 촉진한다.미소관의 탈중합은 키네토코어에서 [3]장력을 발생시킨다; 반대편 세포극에서 나오는 미소관에 자매 키네토코어의 양극성 부착은 장력에 대항하는 힘을 결합하여 세포 적도에 염색체를 정렬하고 딸 세포로 분리하기 위해 그것들을 포이즈한다.모든 염색체가 쌍방향 염색체일 때, 아나phase가 시작되고 자매 염색체를 결합하는 응집체가 절단되어 자매 염색체가 반대 극으로 전달될 수 있습니다.
세포방추장치는 방추미소관, 키네신 및 다이네인 분자모터, 축합염색체 및 세포유형에 [4]따라 방추극에 존재할 수 있는 모든 중심체 또는 아스터를 포함한다.스핀들 장치의 단면은 약간 타원형이며 끝부분은 테이퍼형입니다.스핀들 미드존으로 알려진 광중간부에서 역평행 미세관은 키네신에 의해 묶인다.방추극으로 알려진 뾰족한 끝부분에서, 미소관은 대부분의 동물 세포에서 중심체에 의해 핵으로 형성된다.아센트로솜 또는 문합 방추체에는 각각 방추극에 중심체 또는 아스테르가 없으며, 예를 들어 대부분의 [5]동물에서 암컷 감수분열 중에 발생합니다.이 경우 Ran GTP 구배는 스핀들 미세관 구성 및 조립의 주요 조절 장치입니다.균류에서 방추체는 핵 외피 안에 박힌 방추체 사이에 형성되며, 이는 유사분열 중에 분해되지 않는다.
미소관관련단백질및방추역학
동적 불안정성이라고 알려진 과정을 통해 스핀들 미세관의 동적 연장 및 단축은 상당 부분 유사분열 방추의 형태를 결정하고 스핀들 중간 영역에서 염색체의 적절한 정렬을 촉진한다.미세관 관련 단백질(MAPs)은 중간 영역 및 방추 극에서 미세관과 결합하여 역학을 조절합니다.γ-tubulin은 α/β tubulin 헤테로디머의 핵중합을 미세관으로 만드는 γ-TuRC라고 하는 고리 복합체로 조립되는 특수 튜불린 변종이다.γ-TuRC를 페리센트로솜 영역에 투입함으로써 미소관 마이너스 엔드를 안정화시켜 미소관 조직 중심 부근에 고정한다.미세관 관련 단백질 Augmin은 γ-TURC와 함께 작용하여 기존 [6]미세관에서 새로운 미세관을 생성한다.
미소관의 성장 말단은 중간 영역의 키네토코어와의 연관성을 촉진하기 위해 단백질 추적(+TIPs)의 작용에 의해 재앙으로부터 보호된다.CLIP170은 Hela 세포에서 미세관 플러스 엔드 부근에 위치시키고 프로메타베이스 [8]동안 키네토코어에 축적되는 것으로 나타났다.CLIP170이 플러스 엔드를 인식하는 방법은 아직 불분명하지만, 그 호몰로지는 재해로부터 보호하고 구조를 [9][10]촉진하는 것으로 나타나며, 플러스 엔드를 안정화하고 아마도 키네토코어에 [11]직접 접속하는 데 CLIP170의 역할을 시사하고 있습니다.인간의 CLAP1과 같은 CLAP 관련 단백질은 또한 키네토코어 미세관의 역학을 조절하는 것뿐만 아니라 플러스 엔드 및 외부 키네토코어에 국재하는 것으로 나타났다(Maiato 2003).적절한 스핀들 조립을 위해서는 Drosophila, Xenopus 및 효모의 CLASP 호몰로그가 필요합니다.포유동물에서는 CLASP1과 CLASP2가 모두 [12]Anaphase에서 적절한 스핀들 조립과 미세관 역학에 기여합니다.플러스 엔드 중합은 EB1 단백질에 의해 더욱 완화될 수 있으며, EB1 단백질은 마이크로튜브의 성장 말단을 직접 결합하고 다른 +TIP의 [13][14]결합을 조정한다.
이들 미소관 안정화 단백질의 작용에 반대되는 것은 염색체 결합 및 쌍극성 달성을 촉진하기 위해 유사분열 방추체의 동적 리모델링을 가능하게 하는 다수의 미소관 탈중합 인자이다.MAPs의 키네신-13 슈퍼패밀리는 잘 연구된 포유동물 MCAK와 Xenopus XKCM1을 포함한 관련 미세관 탈중합 활성을 가진 플러스 엔드 지향 운동단백질을 포함하고 있다. MCAK는 AC 안정화와 직접적인 경쟁으로 대재앙을 촉발할 수 있는 키네토코어의 미소관의 성장 팁에 국소화한다.이 [15]단백질들은 ATP 가수분해 에너지를 이용하여 키네신 방출과 미세관 [16]탈중합의 원인이 되는 프로토필라멘트 구조의 불안정한 입체구조 변화를 유도한다.그들의 활동 상실은 수많은 유사분열성 [15]결함을 초래한다.추가적인 미소관 불안정 단백질로는 유사분열 방추의 리모델링과 아나기 [17]중의 염색체 분리를 촉진하는 역할을 하는 Op18/stathmin 및 카타닌이 포함된다.
이러한 MAP의 활동은 스핀들 조립 중에 적절한 미세관 역학을 유지하기 위해 세심하게 조절되며, 이러한 단백질의 대부분은 Aurora 및 Polo-like kinase [17][18]기질 역할을 한다.
스핀들 장비 정리
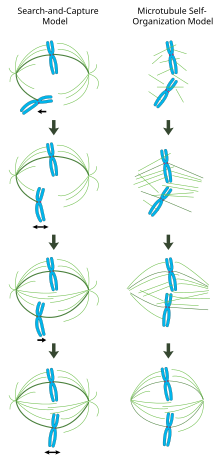
적절하게 형성된 유사분열방추에서 쌍방향 염색체는 염색체에 대해 거의 수직인 방추 미소관과 함께 세포의 적도를 따라 배열되며, 그 끝부분은 키네토코어에 박혀 있고, 끝부분은 세포극에 고정되어 있다.이 복합체의 정확한 배향은 정확한 염색체 분리를 보장하고 세포 분열 평면을 지정하기 위해 필요하다.그러나 스핀들이 어떻게 구성되는지는 여전히 불분명합니다.두 모델이 이 분야를 지배하고 있는데, 이 두 모델은 시너지 효과가 있고 상호 배타적이지 않습니다.탐색 캡처 모델에서 스핀들은 주로 중심체 미세관 조직중심(MTOC)의 극방향 분리에 의해 구성된다.방추 미세관은 중심체에서 나와 운동체를 찾아내고 운동체를 결합하면 안정되어 염색체에 장력을 가한다.다른 자기조립모델에서는 마이크로튜브가 축합염색체 사이에서 아세트로좀핵생성을 한다.세포 치수, 운동단백질을 통한 반평행 미세관과의 측면 연관성, 그리고 키네토코어에 대한 엔드온 부착에 의해 구속된 미세관은 세포 적도를 따라 배열된 염색체와 함께 자연스럽게 방추와 같은 구조를 채택합니다.
중심체 매개 '검색 및 캡처' 모델
이 모델에서 미소관은 미소관 조직 중심에서 핵을 형성하고 빠른 성장과 대재앙을 겪으며 동심원 세포질을 '검색'한다.동심원을 결합하면 안정되고 동력이 저하됩니다.새로 발견된 모노 지향 염색체는 반대편 극에서 온 미세관이 자매 운동체를 결합할 때까지 부착되어 있는 극 근처 공간에서 진동합니다.이 두 번째 부착물은 유사분열 스핀들에 대한 키네토코어 부착을 더욱 안정화시킵니다.서서히 쌍방향 염색체는 미소관 장력이 동원체의 양쪽에 평형을 이룰 때까지 세포의 중심을 향해 당겨지고, 이어서 분쇄된 염색체는 아나phase 개시 시 자매 염색체의 응집력을 방출할 때까지 중기판에서 진동한다.
이 모델에서 미소관 조직 중심은 세포 극에 국소화되어 미소관 중합에 의해 분리되며 양극성 플러스 엔드 지향 키네신에 [19][20]의해 매개되는 스핀들 중간존에서 서로에 대한 반평행 스핀들 미세관의 '슬라이드'에 의해 구동된다.이러한 슬라이딩력은 유사분열 초기에 스핀들 극의 분리뿐만 아니라 후기의 Anaphase 동안 스핀들 신장을 설명할 수 있다.
유사분열방추의 염색질 매개 자기조직화
중심체가 주로 유사분열 방추체의 구성을 지시하는 탐색 및 포착 메커니즘과 대조적으로, 이 모델은 미소관이 염색체 근처에서 핵으로 형성되어 자발적으로 반평행 다발로 결합되어 방추체와 유사한 [21]구조를 채택하는 것을 제안한다.힐드와 카르센티에 의한 고전적인 실험에 따르면 Xenopus 난자 추출물에 배양된 DNA 코팅된 구슬 주위에 기능성 분추와 핵이 형성되고 중심체와 키네토코어가 [22]없는 상태에서 극성 미세관 배열이 형성된다.실제로 척추동물 세포에서 중심체의 레이저 어블레이션은 방추 조립이나 염색체 [23]분리를 억제하지 않는 것으로 나타났다.이 체계 하에서 유사분열 방추의 모양과 크기는 가교 운동 [24]단백질의 생물물리학적 성질의 함수이다.
Ran GTP 구배에 의한 염색질 매개 미세관 핵생성
작은 GTPase Ran(염색체 응축 조절기 1 또는 RCC1)에 대한 구아닌 뉴클레오티드 교환 인자는 코어 히스톤 H2A 및 H2B를 [25]통해 뉴클레오솜에 결합된다.따라서 GTP 결합 Ran의 구배는 유사분열 크로마틴 부근에서 생성된다.RCC1로 코팅된 유리 비즈는 Xenopus 난자 추출물에서 미세관 핵형성과 양극성 스핀들 형성을 유도하여 Ran GTP 구배만으로도 스핀들 [26]조립에 충분함을 알 수 있다.구배는 운반 단백질 β/α를 통해 억제 상호작용으로부터 스핀들 조립 인자(SAF)의 방출을 트리거한다.결합 SAF는 이어서 유사분열 크로마틴 주변에서 미세관 핵 형성과 안정화를 촉진하고, 방추 2극성은 미세관 운동 [27]단백질에 의해 구성된다.
스핀들 조립의 조절
스핀들 어셈블리는 주로 유사분열 키나제에 의해 촉매되는 인산화 현상에 의해 조절된다.사이클린 의존성 키나아제 복합체(CDK)는 유사분열 중에 번역이 증가하는 유사분열 사이클린에 의해 활성화된다.CDK1(CDC2)은 포유동물 세포에서 주요 유사분열 키나제로 간주되며 사이클린 B1에 의해 활성화된다.스핀들 조립 및 [28]분리를 위해서는 오로라 키나아제(Ourora kinase오로라 A는 중심체와 연관되어 유사분열 입구를 조절하는 것으로 믿어진다.Aurora B는 염색체 승객 복합체의 일원으로 염색체-마이크로튜브 부착과 자매 염색체 응집을 매개한다.PLK로도 알려진 폴로 유사 키나제, 특히 PLK1은 미세관 [29]역학을 조절함으로써 방추 유지에 중요한 역할을 합니다.
유사분열염색체구조
DNA 복제가 끝날 때까지 자매 염색체는 엉킨 DNA와 단백질의 비정질 덩어리로 결합된다.유사분열 진입은 복제된 게놈의 극적인 재구성을 유발하며, 자매 염색체가 서로 분리되고 분리되는 결과를 초래한다.염색체는 또한 축합이라고 불리는 과정에서 길이가 동물의 [30]세포에서 10,000배까지 짧아진다.응축은 전상에서 시작되고 염색체는 중기에서 방추의 중앙에 정렬될 때 막대 모양의 구조로 최대 압축된다.이것은 각각의 응축 자매 염색체가 응집 단백질에 의해 그들의 길이에 따라 연결되고 종종 중심 [30][31][32]부근에서, 중심에서 결합되는 핵형에서 볼 수 있는 전형적인 "X" 형태를 제공합니다.
이러한 동적 재배치는 게놈의 정확하고 충실한 분리를 보장하기 위해 매우 중요하지만, 유사분열 염색체 구조에 대한 우리의 이해는 대부분 불완전하다.그러나 몇 가지 특정 분자 주체가 확인되었다.토포이소머라아제 II는 ATP 가수분해를 이용하여 DNA 얽힘의 분해를 촉매하여 자매 염색체 [33]분해를 촉진한다.응축소는 또한 염색체 [34]축합을 촉진하기 위해 ATP 가수분해를 사용하는 5개의 서브유닛 복합체이다.Xenopus 난자 추출물에 대한 실험은 링커 히스톤 H1을 유사분열 염색체 [35]압축의 중요한 조절자로 포함시켰다.
유사분열 스핀들 어셈블리 체크 포인트
스핀들 형성 완료는 스핀들 어셈블리 체크 포인트라고 불리는 셀 사이클의 중요한 전환 포인트입니다.이 체크포인트까지 염색체가 유사분열방추에 제대로 부착되지 않으면 무지외상 발병이 [36]지연됩니다.이 스핀들 조립체 체크포인트가 고장나면 배수가 잘 되지 않을 수 있으며 노화 및 [37]암 형성에 관여할 수 있습니다.
스핀들 장치 방향
세포 분열 방향은 조직 구조, 세포 운명 및 형태 형성에 있어 매우 중요합니다.세포는 이른바 허트윅 법칙에 따라 긴 축을 따라 분열하는 경향이 있다.셀 분할의 축은 스핀들 장치의 방향에 따라 결정됩니다.셀은 스핀들 장치의 두 개의 중심체를 연결하는 선을 따라 분할됩니다.스핀들 장치는 형성 후 셀 내부에서 회전한다.중심체에서 유래한 아스트랄 미세관은 세포막에 도달하여 특정한 피질의 단서를 향해 당겨진다.시험관내에서는 점착 [38]패턴에 의해 피질 단서의 분포를 설정한다.생체 내 극성 신호는 세포 [39]정점에 국재화된 삼세포 접합부의 국재화에 의해 결정된다.피질 단서의 공간 분포는 최종 스핀들 장치 방향과 후속 세포 분열 방향을 결정하는 힘장으로 이어집니다.
「 」를 참조해 주세요.
레퍼런스
- ^ C. E. Walczak; R. Heald (2008). "Mechanisms of Mitotic Spindle Assembly and Function". International Review of Cytology. 265: 111–158. doi:10.1016/s0074-7696(07)65003-7. ISBN 9780123743329. PMID 18275887.
- ^ Helmke KJ, Heald R, Wilbur JD (2013). "Interplay between spindle architecture and function" (PDF). Int. Rev. Cell Mol. Biol. International Review of Cell and Molecular Biology. 306: 83–125. doi:10.1016/B978-0-12-407694-5.00003-1. ISBN 9780124076945. PMID 24016524.
- ^ E. Nogales; V. H. Ramey (1 November 2009). "Structure-function insights into the yeast Dam1 kinetochore complex". J Cell Sci. 122 (21): 3831–3836. doi:10.1242/jcs.004689. PMC 2773187. PMID 19889968.
- ^ Campbell, Neil A.; Jane B. Reece (2005). Biology, 7th Edition. San Francisco: Benjamin Cummings. pp. 221–224. ISBN 0-8053-7171-0.
- ^ Manandhar Gf; Schatten H; Sutovsky P (2005). "Centrosome reduction during gametogenesis and its significance". Biol. Reprod. 72 (1): 2–13. doi:10.1095/biolreprod.104.031245. PMID 15385423. S2CID 37305534.
- ^ Petry S, et al. (2013). "Branching microtubule nucleation in Xenopus egg extracts mediated by augmin and TPX2". Cell. 152 (4): 768–777. doi:10.1016/j.cell.2012.12.044. PMC 3680348. PMID 23415226.
- ^ J.E. Rickard; T.E. Kreis (1990). "Identification of a novel nucleotide-sensitive microtubule-binding protein in HeLa cells". J Cell Biol. 110 (5): 1623–1633. doi:10.1083/jcb.110.5.1623. PMC 2200191. PMID 1970824.
- ^ D. Dujardin; U.I. Wacker; A. Moreau; T.A. Schroer; J.E. Rickard; J.R. DeMey (1998). "Evidence for a role of CLIP-170 in the establishment of metaphase chromosome alignment". J Cell Biol. 141 (4): 849–862. doi:10.1083/jcb.141.4.849. PMC 2132766. PMID 9585405.
- ^ D. Brunner; P. Nurse (2000). "CLIP-170-like tip1p spatially organizes microtubular dynamics in fission yeast". Cell. 102 (5): 695–704. doi:10.1016/S0092-8674(00)00091-X. PMID 11007487. S2CID 11948950.
- ^ Y.A. Komarova; A.S. Kojima; et al. (2002). "Cytoplasmic linker proteins promote microtubule rescue in vivo". J Cell Biol. 159 (4): 589–599. doi:10.1083/jcb.200208058. PMC 2173097. PMID 12446741.
- ^ S. Goldstone; C. Reyes; G. Gay; T. Courthéoux; M. Dubarry; et al. (2010). "Tip1/CLIP-170 Protein Is Required for Correct Chromosome Poleward Movement in Fission Yeast". PLOS ONE. 5 (5): e10634. Bibcode:2010PLoSO...510634G. doi:10.1371/journal.pone.0010634. PMC 2869355. PMID 20498706.
- ^ A.L. Pereira; A.J. Pereira; A.R.R. Maia; et al. (1 October 2006). "Mammalian CLASP1 and CLASP2 Cooperate to Ensure Mitotic Fidelity by Regulating Spindle and Kinetochore Function". Mol Biol Cell. 17 (10): 4526–4542. doi:10.1091/mbc.E06-07-0579. PMC 1635371. PMID 16914514.
- ^ A. Akhmanova; M.O. Steinmetz (April 2008). "Tracking the ends: a dynamic protein network controls the fate of microtubule tips". Nat Rev Mol Cell Biol. 9 (4): 309–322. doi:10.1038/nrm2369. PMID 18322465. S2CID 24977579.
- ^ J.S. Tirnauer; S. Grego; E.D. Salmon; T.J. Mitchison (1 October 2002). "EB1-microtubule interactions in Xenopus egg extracts: Role of EB1 in microtubule stabilization and mechanisms of targeting to microtubules". Mol Biol Cell. 13 (10): 3614–3626. doi:10.1091/mbc.02-04-0210. PMC 129970. PMID 12388761.
- ^ a b M.E. Tanenbaum; R.H. Medema; A. Akhmanova (2011). "Regulation of localization and activity of the microtubule depolymerase MCAK". Bioarchitecture. 1 (2): 80–87. doi:10.4161/bioa.1.2.15807. PMC 3158623. PMID 21866268.
- ^ H. Niederstrasser; H. Salehi-Had; E.C. Gan; C. Walczak; E. Nogales (2002). "XKCM1 acts on a single protofilament and requires the C terminus of tubulin". J Mol Biol. 316 (3): 817–828. doi:10.1006/jmbi.2001.5360. PMID 11866534.
- ^ a b H. Maiato; P Sampaio; C.E. Sunkel (2004). "Microtubule-associated proteins and their essential roles during mitosis". Int Rev Cytol. International Review of Cytology. 241: 53–153. doi:10.1016/S0074-7696(04)41002-X. hdl:10216/53621. ISBN 9780123646453. PMID 15548419.
- ^ R. Tournebize; A. Popov; K. Kinoshita; A.J. Ashford; et al. (2000). "Control of microtubule dynamics by the antagonistic activities of XMAP215 and XKCM1 in Xenopus egg extracts". Nat Cell Biol. 2 (1): 13–19. doi:10.1038/71330. PMID 10620801. S2CID 10732643.
- ^ J. McIntosh; S.C. Landis (1971). "The distribution of spindle microtubules during mitosis in cultured human cells". J Cell Biol. 49 (2): 468–497. doi:10.1083/jcb.49.2.468. PMC 2108320. PMID 19866774.
- ^ D.J. Sharp; K.L. McDonald; H.M. Brown; et al. (1999). "The bipolar kinesin, CLP61F, cross-links microtubules within interpolar microtubule bundles of Drosophila embryonic mitotic spindles". J Cell Biol. 144 (1): 125–138. doi:10.1083/jcb.144.1.125. PMC 2148119. PMID 9885249.
- ^ M.A. Hallen; S.A. Endow (2009). "Anastral spindle assembly: a mathematical model". Biophys J. 97 (8): 2191–2201. Bibcode:2009BpJ....97.2191H. doi:10.1016/j.bpj.2009.08.008. PMC 2764103. PMID 19843451.
- ^ R. Heald; R. Tournebize; et al. (1996). "Self-organization of microtubules into bipolar spindles around artificial chromosomes in Xenopus egg extracts". Nature. 382 (6590): 420–425. Bibcode:1996Natur.382..420H. doi:10.1038/382420a0. PMID 8684481. S2CID 4238425.
- ^ A. Khodjakov; R.W. Cole; B.R. Oakley; C.L. Rieder (2000). "Centrosome-independent mitotic spindle formation in vertebrates". Curr Biol. 10 (2): 59–67. doi:10.1016/S0960-9822(99)00276-6. PMID 10662665. S2CID 9976687.
- ^ K.S. Burbank; T.J. Mitchison; D.S. Fisher (2007). "Slide-and-cluster models for spindle assembly". Curr Biol. 17 (16): 1373–1383. doi:10.1016/j.cub.2007.07.058. PMID 17702580.
- ^ Makde R, England J, Yennawar H, Tan S (2010). "Structure of the RCC1 chromatin factor bound to the nucleosome core particle". Nature. 467 (7315): 562–566. Bibcode:2010Natur.467..562M. doi:10.1038/nature09321. PMC 3168546. PMID 20739938.
- ^ Halpin D, Kalab P, Wang J, Weis K, Heald R (2011). "Mitotic spindle assembly around RCC1-coated beads in Xenopus egg extracts". PLOS Biol. 9 (12): e1001225. doi:10.1371/journal.pbio.1001225. PMC 3246454. PMID 22215983.
- ^ Fu J, Jiang Q, Zhang C (2010). "Coordination of Cell Cycle Events by Ran GTPase". Nature Education. 3 (9): 32.
- ^ A.R. Barr; F. Gergely (2007). "Aurora A: The maker and breaker of spindle poles". J Cell Sci. 120 (17): 2987–2996. doi:10.1242/jcs.013136. PMID 17715155.
- ^ Peters, U., J. Cherian; et al. (2006). "Probing cell-division phenotype space and Polo-like kinase function using small molecules". Nat Chem Biol. 2 (11): 618–26. doi:10.1038/nchembio826. PMID 17028580. S2CID 22213611.
{{cite journal}}: CS1 maint: 여러 이름: 작성자 목록(링크) - ^ a b Morgan DO: 세포 주기: 제어 원리(Primers in Biology) 런던:New Science Press Ltd; 2007:297.ISBN 978-0-9539181-2-6
- ^ Belmont AS (2010). "Large-scale chromatin organization: The good, the surprising, and the still perplexing". Curr Opin Cell Biol. 26: 69–78. doi:10.1016/j.ceb.2013.10.002. PMC 3927141. PMID 24529248.
- ^ 마코, JF유사분열 염색체: 구조와 역학. 2012년.세포핵의 게놈 구성과 기능.Wiley-VCH, Ch. 18, 449-485.doi:10.1002/9783527639991.ch18
- ^ Champoux JJ (2001). "DNA TOPOISOMERASES: Structure, Function, and Mechanism". Annu Rev Biochem. 70 (1): 369–413. doi:10.1146/annurev.biochem.70.1.369. PMID 11395412.
- ^ Hirano T (2012). "Condensins: universal organizers of chromosomes with diverse functions". Genes Dev. 26 (15): 1659–1678. doi:10.1101/gad.194746.112. PMC 3418584. PMID 22855829.
- ^ Maresca TJ, Freedman BS, Heald R (2005). "Histone H1 is essential for mitotic chromosome architecture and segregation in Xenopus laevis egg extracts". J. Cell Biol. 169 (6): 859–69. doi:10.1083/jcb.200503031. PMC 2171634. PMID 15967810.
- ^ Raven, Peter H.; Ray F. Evert; Susan E. Eichhorn (2005). Biology of Plants, 7th Edition. New York: W.H. Freeman and Company Publishers. p. 59. ISBN 0-7167-1007-2.
- ^ Baker DJ, Chen J, van Deursen JM (2005). "The mitotic checkpoint in cancer and aging: what have mice taught us?". Curr. Opin. Cell Biol. 17 (6): 583–9. doi:10.1016/j.ceb.2005.09.011. PMID 16226453.
- ^ Thery M, Jimenez-Dalmaroni A, Racine V, Bornens M, Julicher F (2007). "Experimental and theoretical study of mitotic spindle orientation". Nature. 447 (7143): 493–6. Bibcode:2007Natur.447..493T. doi:10.1038/nature05786. PMID 17495931. S2CID 4391685.
- ^ Bosveld F, Markova O, Guirao B, Martin C, Wang Z, Pierre A, Balakireva M, Gaugue I, Ainslie A, Christophorou N, Lubensky DK, Minc N, Bellaïche Y (2016). "Epithelial tricellular junctions act as interphase cell shape sensors to orient mitosis". Nature. 530 (7591): 496–8. Bibcode:2016Natur.530..495B. doi:10.1038/nature16970. PMC 5450930. PMID 26886796.
외부 링크
 Wikimedia Commons 스핀들 관련 매체
Wikimedia Commons 스핀들 관련 매체