구조 변동
Structural variation게놈 구조 변화는 유기체의 염색체 구조의 변화이다.그것은 한 종의 게놈의 많은 종류의 변이로 구성되며, 보통 결실, 복제, 복사 번호 변형, 삽입, 반전 및 전이와 같은 현미경 및 준현미경 유형을 포함합니다.원래, 구조 변화는 SNP보다 크고 염색체 이상보다 작은 약 1kb - 3Mb의 배열 길이에 영향을 미친다(정의가 일부 [1]중복되기는 하지만).그러나 구조 변형의 작동 범위는 이벤트 > 50bp로 [2]넓어졌다.구조적 변동의 정의는 빈도나 표현형 효과에 관한 어떤 것도 내포하지 않는다.많은 구조적 변종들이 유전적인 질병과 연관되어 있지만,[3][4] 많은 것들이 그렇지 않다.최근 SV에 대한 연구에 따르면 SV는 SNP보다 검출이 더 어려운 것으로 나타났다.인간 게놈의 약 13%는 정상 집단에서 구조적으로 변이체로 정의되며, 인간 집단에는 호모 접합 결실 다형성으로 존재하는 최소 240개의 유전자가 존재하며, 이는 이러한 유전자가 [4]인간에서 불필요하다는 것을 시사한다.빠르게 축적되는 증거는 구조적 변화가 모든 게놈 내에서 이질성의 수백만 개의 뉴클레오티드를 구성할 수 있고, 인간의 다양성과 질병 감수성에 중요한 기여를 할 가능성이 있다는 것을 보여준다.
미세구조변화
현미경은 유배체, 표식염색체, 총 재배열, 염색체 [5][6]크기 변화 등과 같은 광학 현미경으로 검출할 수 있다는 것을 의미한다.이들 중 일부는 실제로 식별이 쉽지 않기 때문에 인구 발생 빈도는 과소평가된 것으로 생각된다.이러한 구조적 이상은 추정 [7]정보에 의해 375명의 정상 출산 중 1명꼴로 존재한다.
준현미경 구조 변화
준현미경 구조 변종은 크기가 작기 때문에 검출하기가 훨씬 어렵다.2004년 DNA 마이크로어레이를 사용한 첫 번째 연구는 인간 [8]게놈에서 100킬로베이스가 넘는 복제 번호의 변화, 결실, 중복을 보이는 수십 개의 유전자 위치를 검출할 수 있었다.그러나 2015년까지 전체 게놈 염기서열 분석 [3][4]연구는 각 개별 게놈에서 약 20메가비스를 포함하는 100개의 염기쌍만큼 작은 5,000개의 구조적 변형을 발견할 수 있을 것이다.이러한 구조 변형에는 삭제, 탠덤 복제, 반전, 이동 요소 삽입 등이 포함됩니다.또한 돌연변이율은 두 연구에서 각각 16%와 20%로 추정된 미시적 구조 변이체보다 훨씬 높으며, 두 연구 모두 구조 [3][9]변이를 정확하게 감지하는 어려움 때문에 과소평가된 것일 수 있다.또한 자발적 구조적 변이체의 생성은 구조적 변이 [3]사건의 100킬로베이스 내에서 추가적인 자발적 단일 뉴클레오티드 변이체 또는 인델을 생성할 가능성을 유의하게 증가시키는 것으로 나타났다.
복사 번호의 변동
복사 번호 변동(CNV)은 삽입, 삭제 및 복제를 포함하는 큰 범주의 구조 변동입니다.최근 연구에서 복제 번호 변이는 유전병이 없는 사람에게 정량적 SNP 유전자형식에 사용되는 방법을 사용하여 테스트된다.그 결과, 개개의 의심되는 영역의 28%가 실제로 카피 번호의 [10][11]변형을 포함하고 있는 것을 알 수 있습니다.또한 인간 게놈의 CNV는 단일 뉴클레오티드 다형성(SNP)보다 더 많은 뉴클레오티드에 영향을 미친다.많은 CNV가 코딩 영역에 있지 않다는 점도 주목할 필요가 있습니다.CNV는 보통 불균등한 재조합에 의해 발생하므로 LINE이나 SINE과 같은 광범위한 유사한 시퀀스가 CNV [12][13]생성의 일반적인 메커니즘일 수 있습니다.
반전
인간의 질병과 관련된 몇 가지 반전이 알려져 있다.예를 들어, 인자 VIII에서 400kb의 반복적인 반전은 혈우병 [14]A의 일반적인 원인이며, 이단산염 2-설파타아제(IDS)에 영향을 미치는 작은 반전은 헌터 [15]증후군을 일으킨다.더 많은 예로는 엔젤만 증후군과 소토스 증후군이 있다.하지만, 최근의 연구는 한 사람이 56개의 추정 역전을 가질 수 있다는 것을 보여주며, 따라서 비질병 역전은 이전에 생각했던 것보다 더 흔하다.또한 이 연구에서는 반전 중단점이 일반적으로 세그먼트 [16]복제와 관련이 있는 것으로 나타났습니다.17번 염색체의 900kb 반전은 긍정적인 선택 하에 있으며 유럽 [17]인구에서 그 빈도가 증가할 것으로 예상된다.
기타 구조 변형
보다 복잡한 구조적 변형이 발생할 수 있으며, 단일 [3]사건에 위의 조합이 포함될 수 있습니다.가장 일반적인 유형의 복잡한 구조적 변화는 비탄뎀 복제이며,[3] 여기서 염기서열이 복제되고 게놈의 다른 부분에 역방향 또는 직접 방향으로 삽입됩니다.복잡한 구조 배리언트의 다른 클래스에는 삭제-반전-삭제, 복제-반전-복제, 중첩된 [3]삭제가 있는 탠덤 복제 등이 있습니다.또한 불가해한 전이와 분절단순성분절제술(UPD)도 있다.이러한 변동에 대한 보고가 증가하고 있지만 기존 변동보다 검출이 어렵습니다.이러한 변동은 밸런스가 잡혀 있고 어레이 기반 또는 PCR 기반 방법으로는 [18]검출할 수 없기 때문입니다.
구조변형 및 표현형
일부 유전 질환은 구조적 변이에 의한 것으로 의심되지만, 그 관계는 그다지 확실하지 않다.이러한 변형을 "정상" 또는 "질병"으로 분류하는 것은 타당하지 않다. 왜냐하면 동일한 변형의 실제 출력도 다양하기 때문이다.또, 몇개의 변형은 실제로 (상기)에 대해서 긍정적으로 선택되고 있다.일련의 연구는 자발적(de novo) CNVs를 교란하는 유전자가 대조군보다 자폐증에서 유전자를 약 4배 더 자주 교란시키고 사례의 [3][19][20][21][22]약 5~10%에 기여한다는 것을 보여주었다.유전 변종 또한 자폐증 [3]사례의 약 5~10%에 기여한다.
구조적 변화는 또한 집단 유전학에서 그 기능을 가지고 있다.동일한 변동의 다른 빈도는 다른 영역의 모집단 간의 관계를 추론하기 위한 유전적 표시로 사용될 수 있다.인간과 침팬지의 구조적 변이를 완전히 비교한 결과, 적응 [23]기능 때문에 이들 중 일부가 한 종에 고정될 수 있다는 사실도 밝혀졌다.말라리아와 [24][25]에이즈에 대한 내성과 관련된 결실도 있다.또한, 일부 고변동 세그먼트는 균형 선택에 의해 발생하는 것으로 생각되지만, 이 [26]가설에 대한 연구도 있다.
구조변동 데이터베이스
일부 게놈 브라우저 및 생물정보 데이터베이스에는 CNV에 중점을 둔 인간 게놈의 구조적 변이 목록이 있으며, 이를 UCSC 게놈 [27]브라우저와 같은 게놈 브라우징 페이지에 표시할 수 있습니다.게놈의 일부를 보는 페이지 아래에는 활성화 할 수 있는 "Common Cell CNVs"와 "Structural Var"가 있습니다.NCBI에는 구조변동에 대한 특별한 페이지가 있습니다.이 시스템에서는 "내부" 및 "외부" 좌표가 모두 표시되며, 둘 다 실제 중단점이 아니라 구조 변동의 영향을 받는 최소 및 최대 범위로 추정된다.유형은 삽입, 손실, 게인, 반전, LOH, 에버드, 트랜스치르 및 [citation needed]UPD로 분류됩니다.
검출 방법
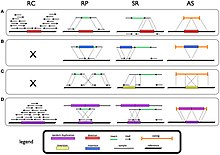
인간의 유전자 구조 변화를 고해상도로 분석하기 위한 새로운 방법들이 개발되었습니다.게놈을 테스트하기 위해 사용되는 방법은 특정 표적 방식 또는 게놈 전체 방식 중 하나입니다.게놈 전체 테스트의 경우 어레이 기반 비교 게놈 하이브리드화 접근방식은 새로운 복사 번호 [30]변형을 찾기 위해 최적의 게놈 전체 스캔을 제공합니다.이 기술들은 관심 있는 게놈에서 라벨이 붙은 DNA 조각들을 사용하며, 다른 게놈과 다른 라벨이 붙은 DNA 조각들이 복제된 DNA 조각으로 발견된 배열들과 교배된다.이것은 두 [30]게놈 사이의 복사 번호 차이를 드러냅니다.
표적 게놈 검사의 경우, 게놈의 특정 영역을 확인하기 위한 최선의 분석은 주로 PCR 기반입니다.PCR 기반 방법 중 가장 잘 확립된 것은 실시간 정량 중합효소 연쇄 반응(qPCR)[30]이다.또 다른 접근법은 기존의 세그먼트 복제를 둘러싸고 있는 특정 영역을 확인하는 것입니다.이는 일반적으로는 복사 번호의 [30]변동이 있는 영역이기 때문입니다.2개의 대립 유전자에 대해 독립적인 형광 강도를 제공하는 SNP 유전자형식법은 분할 [30]복제의 두 복사본 사이의 뉴클레오티드를 목표로 하는 데 사용될 수 있다.이것에 의해, 어느 한쪽의 대립 유전자에 의한 다른 한쪽의 대립 유전자에 대한 강도 증가를 볼 수 있다.
차세대 염기서열분석(NGS) 기술의 발달로 NGS 데이터에 의한 구조 변이 검출을 위한 4가지 등급의 전략이 보고되었으며, 각각 다른 등급의 SV를 진단하는 패턴에 기초하고 있다.
- 읽기 깊이 또는 읽기 카운트 방법은 짧은 읽기 시퀀싱에서 읽기의 랜덤 분포(예: 포아송 분포)를 가정합니다.중복과 삭제를 발견하기 위해 이 분포로부터의 차이를 조사한다.중복된 영역은 읽기 깊이가 높은 반면 삭제된 영역은 읽기 깊이가 낮아집니다.
- 스플릿 리드 방식을 사용하면 삽입(모바일 요소 삽입 포함) 및 삭제를 단일 베이스 페어 해상도로 검출할 수 있습니다.SV의 존재는 참조 게놈에 대한 불연속 정렬에서 식별된다.판독치의 갭은 삭제를 나타내고 참조는 삽입을 나타냅니다.
- 읽기 쌍 구성 방법은 짧은 읽기 시퀀스 데이터에서 쌍으로 구성된 엔드 판독의 길이와 방향을 검사합니다.예를 들어, 읽기 쌍이 예상보다 멀리 떨어져 있으면 삭제되었음을 나타냅니다.마찬가지로 읽기 쌍을 사용하여 변환, 반전 및 탠덤 복제를 검색할 수 있습니다.
- de novo 시퀀스 어셈블리는 충분히 정확한 판독을 적용할 수 있습니다.실제로 이 방법의 사용은 시퀀스 판독 길이에 따라 제한되지만, 장기 판독 기반의 게놈 어셈블리는 다른 [34]방법을 사용할 때 검출되지 않는 삽입과 같은 클래스에 대한 구조적 변이 발견을 제공합니다.
「 」를 참조해 주세요.
레퍼런스
- ^ Feuk, Lars; Carson, Andrew R.; Scherer, Stephen W. (2006). "Structural variation in the human genome". Nature Reviews Genetics. 7 (2): 85–97. doi:10.1038/nrg1767. PMID 16418744. S2CID 17255998.
- ^ Alkan, Can; Coe, Bradley P.; Eichler, Evan E. (2011-03-01). "Genome structural variation discovery and genotyping". Nature Reviews Genetics. 12 (5): 363–376. doi:10.1038/nrg2958. ISSN 1471-0056. PMC 4108431. PMID 21358748.
- ^ a b c d e f g h i Brandler, William M.; Antaki, Danny; Gujral, Madhusudan; Noor, Amina; Rosanio, Gabriel; Chapman, Timothy R.; Barrera, Daniel J.; Lin, Guan Ning; Malhotra, Dheeraj; Watts, Amanda C.; Wong, Lawrence C.; Estabillo, Jasper A.; Gadomski, Therese E.; Hong, Oanh; Fajardo, Karin V. Fuentes; Bhandari, Abhishek; Owen, Renius; Baughn, Michael; Yuan, Jeffrey; Solomon, Terry; Moyzis, Alexandra G.; Maile, Michelle S.; Sanders, Stephan J.; Reiner, Gail E.; Vaux, Keith K.; Strom, Charles M.; Zhang, Kang; Muotri, Alysson R.; Akshoomoff, Natacha; Leal, Suzanne M.; Pierce, Karen; Courchesne, Eric; Iakoucheva, Lilia M.; Corsello, Christina; Sebat, Jonathan (24 March 2016). "Frequency and Complexity of De Novo Structural Mutation in Autism". The American Journal of Human Genetics. 98 (4): 667–79. doi:10.1016/j.ajhg.2016.02.018. PMC 4833290. PMID 27018473.
- ^ a b c Sudmant, Peter H.; Rausch, Tobias; Gardner, Eugene J.; Handsaker, Robert E.; Abyzov, Alexej; Huddleston, John; Zhang, Yan; Ye, Kai; Jun, Goo; Hsi-Yang Fritz, Markus; Konkel, Miriam K.; Malhotra, Ankit; Stütz, Adrian M.; Shi, Xinghua; Paolo Casale, Francesco; Chen, Jieming; Hormozdiari, Fereydoun; Dayama, Gargi; Chen, Ken; Malig, Maika; Chaisson, Mark J. P.; Walter, Klaudia; Meiers, Sascha; Kashin, Seva; Garrison, Erik; Auton, Adam; Lam, Hugo Y. K.; Jasmine Mu, Xinmeng; Alkan, Can; Antaki, Danny; Bae, Taejeong; Cerveira, Eliza; Chines, Peter; Chong, Zechen; Clarke, Laura; Dal, Elif; Ding, Li; Emery, Sarah; Fan, Xian; Gujral, Madhusudan; Kahveci, Fatma; Kidd, Jeffrey M.; Kong, Yu; Lameijer, Eric-Wubbo; McCarthy, Shane; Flicek, Paul; Gibbs, Richard A.; Marth, Gabor; Mason, Christopher E.; Menelaou, Androniki; Muzny, Donna M.; Nelson, Bradley J.; Noor, Amina; Parrish, Nicholas F.; Pendleton, Matthew; Quitadamo, Andrew; Raeder, Benjamin; Schadt, Eric E.; Romanovitch, Mallory; Schlattl, Andreas; Sebra, Robert; Shabalin, Andrey A.; Untergasser, Andreas; Walker, Jerilyn A.; Wang, Min; Yu, Fuli; Zhang, Chengsheng; Zhang, Jing; Zheng-Bradley, Xiangqun; Zhou, Wanding; Zichner, Thomas; Sebat, Jonathan; Batzer, Mark A.; McCarroll, Steven A.; Mills, Ryan E.; Gerstein, Mark B.; Bashir, Ali; Stegle, Oliver; Devine, Scott E.; Lee, Charles; Eichler, Evan E.; Korbel, Jan O. (30 September 2015). "An integrated map of structural variation in 2,504 human genomes". Nature. 526 (7571): 75–81. Bibcode:2015Natur.526...75.. doi:10.1038/nature15394. PMC 4617611. PMID 26432246.
- ^ Reich, David E.; Schaffner, Stephen F.; Daly, Mark J.; McVean, Gil; Mullikin, James C.; Higgins, John M.; Richter, Daniel J.; Lander, Eric S.; Altshuler, David (2002). "Human genome sequence variation and the influence of gene history, mutation and recombination". Nature Genetics. 32 (1): 135–42. doi:10.1038/ng947. PMID 12161752. S2CID 16822751.
- ^ Gripenberg, Ulla (1964). "Size variation and orientation of the human Y chromosome". Chromosoma. 15 (5): 618–29. doi:10.1007/BF00319995. PMID 14333154. S2CID 26549548.
- ^ Wyandt, H. E.; Tonk, V. S. (2004). Atlas of Human Chromosome Heteromorphisms. Netherlands: Kluwer Academic. ISBN 978-90-481-6296-3.[페이지 필요]
- ^ Sebat, J. (23 July 2004). "Large-Scale Copy Number Polymorphism in the Human Genome". Science. 305 (5683): 525–528. Bibcode:2004Sci...305..525S. doi:10.1126/science.1098918. PMID 15273396. S2CID 20357402.
- ^ Kloosterman, Wigard P.; Francioli, Laurent C.; Hormozdiari, Fereydoun; Marschall, Tobias; Hehir-Kwa, Jayne Y.; Abdellaoui, Abdel; Lameijer, Eric-Wubbo; Moed, Matthijs H.; Koval, Vyacheslav; Renkens, Ivo; van Roosmalen, Markus J.; Arp, Pascal; Karssen, Lennart C.; Coe, Bradley P.; Handsaker, Robert E.; Suchiman, Eka D.; Cuppen, Edwin; Thung, Djie Tjwan; McVey, Mitch; Wendl, Michael C.; Uitterlinden, André; van Duijn, Cornelia M.; Swertz, Morris A.; Wijmenga, Cisca; van Ommen, GertJan B.; Slagboom, P. Eline; Boomsma, Dorret I.; Schönhuth, Alexander; Eichler, Evan E.; de Bakker, Paul I.W.; Ye, Kai; Guryev, Victor (June 2015). "Characteristics of de novo structural changes in the human genome". Genome Research. 25 (6): 792–801. doi:10.1101/gr.185041.114. PMC 4448676. PMID 25883321.
- ^ Sebat, J.; Lakshmi, B; Troge, J; Alexander, J; Young, J; Lundin, P; Månér, S; Massa, H; et al. (2004). "Large-Scale Copy Number Polymorphism in the Human Genome". Science. 305 (5683): 525–8. Bibcode:2004Sci...305..525S. doi:10.1126/science.1098918. PMID 15273396. S2CID 20357402.
- ^ Iafrate, A John; Feuk, Lars; Rivera, Miguel N; Listewnik, Marc L; Donahoe, Patricia K; Qi, Ying; Scherer, Stephen W; Lee, Charles (2004). "Detection of large-scale variation in the human genome". Nature Genetics. 36 (9): 949–51. doi:10.1038/ng1416. PMID 15286789.
- ^ Lupski, James R. (2010). "Retrotransposition and Structural Variation in the Human Genome". Cell. 141 (7): 1110–2. doi:10.1016/j.cell.2010.06.014. PMID 20602993. S2CID 2047696.
- ^ Lam, Hugo YK; Mu, Xinmeng Jasmine; Stutz, Adrian M; Tanzer, Andrea; Cayting, Philip D; Snyder, Michael; Kim, Philip M; Korbel, Jan O; Gerstein, Mark B (2010). "Nucleotide-resolution analysis of structural variants using BreakSeq and a breakpoint library". Nature Biotechnology. 28 (1): 47–55. doi:10.1038/nbt.1600. PMC 2951730. PMID 20037582.
- ^ Lakich, Delia; Kazazian, Haig H.; Antonarakis, Stylianos E.; Gitschier, Jane (1993). "Inversions disrupting the factor VIII gene are a common cause of severe haemophilia A". Nature Genetics. 5 (3): 236–41. doi:10.1038/ng1193-236. PMID 8275087. S2CID 25636383.
- ^ Bondeson, Maire-Louise; Dahl, Niklas; Malmgren, Helena; Kleijer, Wim J.; Tönnesen, Tönne; Carlberg, Britt-Marie; Pettersson, Ulf (1995). "Inversion of the IDS gene resulting from recombination with IDS-related sequences in a common cause of the Hunter syndrome". Human Molecular Genetics. 4 (4): 615–21. doi:10.1093/hmg/4.4.615. PMID 7633410.
- ^ Tuzun, Eray; Sharp, Andrew J; Bailey, Jeffrey A; Kaul, Rajinder; Morrison, V Anne; Pertz, Lisa M; Haugen, Eric; Hayden, Hillary; et al. (2005). "Fine-scale structural variation of the human genome". Nature Genetics. 37 (7): 727–32. doi:10.1038/ng1562. PMID 15895083. S2CID 14162962.
- ^ Stefansson, Hreinn; Helgason, Agnar; Thorleifsson, Gudmar; Steinthorsdottir, Valgerdur; Masson, Gisli; Barnard, John; Baker, Adam; Jonasdottir, Aslaug; et al. (2005). "A common inversion under selection in Europeans". Nature Genetics. 37 (2): 129–37. doi:10.1038/ng1508. PMID 15654335. S2CID 120515.
- ^ Sung, Wing-Kin. Algorithms for next-generation sequencing. Boca Raton. p. 215. ISBN 978-1-4665-6551-7. OCLC 987790994.
- ^ Sebat, J.; Lakshmi, B.; Malhotra, D.; Troge, J.; Lese-Martin, C.; Walsh, T.; Yamrom, B.; Yoon, S.; Krasnitz, A.; Kendall, J.; Leotta, A.; Pai, D.; Zhang, R.; Lee, Y.-H.; Hicks, J.; Spence, S. J.; Lee, A. T.; Puura, K.; Lehtimaki, T.; Ledbetter, D.; Gregersen, P. K.; Bregman, J.; Sutcliffe, J. S.; Jobanputra, V.; Chung, W.; Warburton, D.; King, M.-C.; Skuse, D.; Geschwind, D. H.; Gilliam, T. C.; Ye, K.; Wigler, M. (20 April 2007). "Strong Association of De Novo Copy Number Mutations with Autism". Science. 316 (5823): 445–449. Bibcode:2007Sci...316..445S. doi:10.1126/science.1138659. PMC 2993504. PMID 17363630.
- ^ Pinto, Dalila; Delaby, Elsa; Merico, Daniele; Barbosa, Mafalda; Merikangas, Alison; Klei, Lambertus; Thiruvahindrapuram, Bhooma; Xu, Xiao; Ziman, Robert; Wang, Zhuozhi; Vorstman, Jacob A.S.; Thompson, Ann; Regan, Regina; Pilorge, Marion; Pellecchia, Giovanna; Pagnamenta, Alistair T.; Oliveira, Bárbara; Marshall, Christian R.; Magalhaes, Tiago R.; Lowe, Jennifer K.; Howe, Jennifer L.; Griswold, Anthony J.; Gilbert, John; Duketis, Eftichia; Dombroski, Beth A.; De Jonge, Maretha V.; Cuccaro, Michael; Crawford, Emily L.; Correia, Catarina T.; et al. (May 2014). "Convergence of Genes and Cellular Pathways Dysregulated in Autism Spectrum Disorders". The American Journal of Human Genetics. 94 (5): 677–694. doi:10.1016/j.ajhg.2014.03.018. PMC 4067558. PMID 24768552.
- ^ Levy, Dan; Ronemus, Michael; Yamrom, Boris; Lee, Yoon-ha; Leotta, Anthony; Kendall, Jude; Marks, Steven; Lakshmi, B.; Pai, Deepa; Ye, Kenny; Buja, Andreas; Krieger, Abba; Yoon, Seungtai; Troge, Jennifer; Rodgers, Linda; Iossifov, Ivan; Wigler, Michael (June 2011). "Rare De Novo and Transmitted Copy-Number Variation in Autistic Spectrum Disorders". Neuron. 70 (5): 886–897. doi:10.1016/j.neuron.2011.05.015. PMID 21658582. S2CID 11132936.
- ^ Sanders, Stephan J.; Ercan-Sencicek, A. Gulhan; Hus, Vanessa; Luo, Rui; Murtha, Michael T.; Moreno-De-Luca, Daniel; Chu, Su H.; Moreau, Michael P.; Gupta, Abha R.; Thomson, Susanne A.; Mason, Christopher E.; Bilguvar, Kaya; Celestino-Soper, Patricia B.S.; Choi, Murim; Crawford, Emily L.; Davis, Lea; Davis Wright, Nicole R.; Dhodapkar, Rahul M.; DiCola, Michael; DiLullo, Nicholas M.; Fernandez, Thomas V.; Fielding-Singh, Vikram; Fishman, Daniel O.; Frahm, Stephanie; Garagaloyan, Rouben; Goh, Gerald S.; Kammela, Sindhuja; Klei, Lambertus; Lowe, Jennifer K.; Lund, Sabata C.; McGrew, Anna D.; Meyer, Kyle A.; Moffat, William J.; Murdoch, John D.; O'Roak, Brian J.; Ober, Gordon T.; Pottenger, Rebecca S.; Raubeson, Melanie J.; Song, Youeun; Wang, Qi; Yaspan, Brian L.; Yu, Timothy W.; Yurkiewicz, Ilana R.; Beaudet, Arthur L.; Cantor, Rita M.; Curland, Martin; Grice, Dorothy E.; Günel, Murat; Lifton, Richard P.; Mane, Shrikant M.; Martin, Donna M.; Shaw, Chad A.; Sheldon, Michael; Tischfield, Jay A.; Walsh, Christopher A.; Morrow, Eric M.; Ledbetter, David H.; Fombonne, Eric; Lord, Catherine; Martin, Christa Lese; Brooks, Andrew I.; Sutcliffe, James S.; Cook, Edwin H.; Geschwind, Daniel; Roeder, Kathryn; Devlin, Bernie; State, Matthew W. (June 2011). "Multiple Recurrent De Novo CNVs, Including Duplications of the 7q11.23 Williams Syndrome Region, Are Strongly Associated with Autism". Neuron. 70 (5): 863–885. doi:10.1016/j.neuron.2011.05.002. PMC 3939065. PMID 21658581.
- ^ Johnson, Matthew E.; Viggiano, Luigi; Bailey, Jeffrey A.; Abdul-Rauf, Munah; Goodwin, Graham; Rocchi, Mariano; Eichler, Evan E. (2001). "Positive selection of a gene family during the emergence of humans and African apes". Nature. 413 (6855): 514–9. Bibcode:2001Natur.413..514J. doi:10.1038/35097067. PMID 11586358. S2CID 4327069.
- ^ Redon, Richard; Ishikawa, Shumpei; Fitch, Karen R.; Feuk, Lars; Perry, George H.; Andrews, T. Daniel; Fiegler, Heike; Shapero, Michael H.; et al. (2006). "Global variation in copy number in the human genome". Nature. 444 (7118): 444–54. Bibcode:2006Natur.444..444R. doi:10.1038/nature05329. PMC 2669898. PMID 17122850.
- ^ Gonzalez, E.; Kulkarni, H; Bolivar, H; Mangano, A; Sanchez, R; Catano, G; Nibbs, RJ; Freedman, BI; et al. (2005). "The Influence of CCL3L1 Gene-Containing Segmental Duplications on HIV-1/AIDS Susceptibility". Science. 307 (5714): 1434–40. Bibcode:2005Sci...307.1434G. doi:10.1126/science.1101160. PMID 15637236. S2CID 8815153.
- ^ Bubb, K. L.; Bovee, D; Buckley, D; Haugen, E; Kibukawa, M; Paddock, M; Palmieri, A; Subramanian, S; et al. (2006). "Scan of Human Genome Reveals No New Loci Under Ancient Balancing Selection". Genetics. 173 (4): 2165–77. doi:10.1534/genetics.106.055715. PMC 1569689. PMID 16751668.
- ^ "Human hg38 chr1:11,102,837-11,267,747 UCSC Genome Browser v374".
- ^ "Overview of Structural Variation".
- ^ a b Tattini, Lorenzo; D'Aurizio, Romina; Magi, Alberto (2015). "Detection of genomic structural variants from next-generation sequencing data". Frontiers in Bioengineering and Biotechnology. 3 (92): 92. doi:10.3389/fbioe.2015.00092. PMC 4479793. PMID 26161383.
- ^ a b c d e Feuk, L.; Carson, A.R.; Schere, S.W. (2006). "Structural variation in the human genome". Nature Reviews Genetics. 7 (2): 85–97. doi:10.1038/nrg1767. PMID 16418744. S2CID 17255998.
- ^ Korbel JO, Urban AE, Affourtit JP, Godwin B, Grubert F, Simons JF, Kim PM, Palejev D, Carriero NJ, Du L, Taillon BE, Chen Z, Tanzer A, Saunders AC, Chi J, Yang F, Carter NP, Hurles ME, Weissman SM, Harkins TT, Gerstein MB, Egholm M, Snyder M (October 2007). "Paired-end mapping reveals extensive structural variation in the human genome". Science. 318 (5849): 420–6. Bibcode:2007Sci...318..420K. doi:10.1126/science.1149504. PMC 2674581. PMID 17901297.
- ^ Alkan, Can; Coe, Bradley P.; Eichler, Evan E. (2011). "Genome structural variation discovery and genotyping". Nature Reviews Genetics. 12 (5): 363–376. doi:10.1038/nrg2958. PMC 4108431. PMID 21358748.
- ^ Kuzniar, Arnold; Maassen, Jason; Verhoeven, Stefan; Santuari, Luca; Shneider, Carl; Kloosterman, Wigard P.; de Ridder, Jeroen (2020). "sv-callers: a highly portable parallel workflow for structural variant detection in whole-genome sequence data". PeerJ. 8 (5): 2167–8359. doi:10.7717/peerj.8214. PMC 6951283. PMID 31934500.
- ^ Ebert P, Audano PA, Zhu Q, Rodriguez-Martin B, Porubsky D, Bonder MJ, Sulovari A, Ebler J, Zhou W, Serra Mari R, Yilmaz F, Zhao X, Hsieh P, Lee J, Kumar S, Lin J, Rausch T, Chen Y, Ren J, Santamarina M, Höps W, Ashraf H, Chuang NT, Yang X, Munson KM, Lewis AP, Fairley S, Tallon LJ, Clarke WE, Basile AO, Byrska-Bishop M, Corvelo A, Evani US, Lu TY, Chaisson MJ, Chen J, Li C, Brand H, Wenger AM, Ghareghani M, Harvey WT, Raeder B, Hasenfeld P, Regier AA, Abel HJ, Hall IM, Flicek P, Stegle O, Gerstein MB, Tubio JM, Mu Z, Li YI, Shi X, Hastie AR, Ye K, Chong Z, Sanders AD, Zody MC, Talkowski ME, Mills RE, Devine SE, Lee C, Korbel JO, Marschall T, Eichler EE (April 2021). "Haplotype-resolved diverse human genomes and integrated analysis of structural variation". Science. 372 (6537). doi:10.1126/science.abf7117. PMC 8026704. PMID 33632895.


