전사 활성제 유사 이펙터
Transcription activator-like effector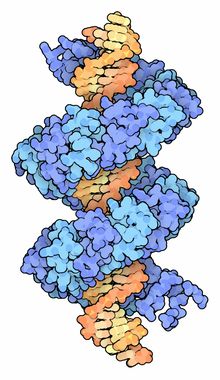 | |||||||
| 식별자 | |||||||
|---|---|---|---|---|---|---|---|
| 유기체 | |||||||
| 기호 | pthXo1 | ||||||
| RefSeq(프로토콜) | WP_041182630.1 | ||||||
| 유니프로트 | B2SU53 | ||||||
| 기타자료 | |||||||
| 염색체 | 게놈: 1.65 - 1.65Mb | ||||||
| |||||||
TAL(변환 활성제 유사) 이펙터(흔히 TALE라고 하지만 단백질의 세 가지 아미노산 루프 확장 홈오박스 등급과 혼동되지 않아야 함)는 일부 β-와 β-단백질균에 의해 분비되는 단백질이다.[1]이 중 대부분은 크산토모나드다.식물병원성 크산토모나스 박테리아는 특히 그들의 타입 III 분비 시스템을 통해 생산되는 TAL로 알려져 있다.이 단백질들은 숙주 식물에서 촉진자 배열을 묶을 수 있고 박테리아 감염을 돕는 식물 유전자의 발현을 활성화시킬 수 있다.그들은 ~34개의 아미노산 반복의 가변 숫자로 구성된 중심 반복 영역을 통해 식물 DNA 서열을 인식한다.각각의 반복에서 두 개의 임계 아미노산의 정체성과 목표 수열의 각 DNA 염기 사이에는 일대일 일치성이 있는 것으로 보인다.이 단백질들은 중요한 작물 종의 질병에 대한 그들의 역할과 새로운 DNA 서열을 결합하기 위해 그것들을 상대적으로 쉽게 재포장하기 때문에 연구자들 모두에게 흥미롭다.병원성 세균인 랄스토니아 솔라나이스아룸과[2][3][1] 부르크홀더리아 리즈옥신카에서도 비슷한 단백질을 발견할 수 있을 뿐만 아니라 [4][1]아직 확인되지 않은 해양 미생물에서도 비슷한 단백질을 발견할 수 있다.[5]TAL-likes라는 용어는 TAL과 이와 관련된 단백질을 포괄하는 putative 단백질 계열을 지칭하기 위해 사용된다.
식물 병원체에서의 기능
크산토모나스속
크산토모나스는 그램 음성 박테리아로 후추/캅시쿰, 쌀, 감귤, 면, 토마토, 콩 등 다양한 식물 종을 감염시킬 수 있다.[6]크산토모나스의 어떤 종류는 국부적인 잎자국이나 잎자국을 유발하는 반면, 다른 종류는 시스템적으로 퍼져 검은 썩거나 잎사귀 병증을 유발한다.그들은 타입 III 분비 시스템을 통해 TAL 이펙터를 포함한 다수의 이펙터 단백질을 발전소에 주입한다.TAL 이펙터는 일반적으로 여러 개의 핵 지역화 신호와 산성 활성화 영역을 포함하여 진핵물질과 관련된 몇 가지 모티브를 가지고 있다.이 단백질들은 식물에 주입되면 식물 세포의 핵으로 들어가 식물 촉진자 서열을 묶고 박테리아 감염을 돕는 식물 유전자의 전사를 활성화시킬 수 있다.[6]식물들은 이들 이펙터에 의해 촉발된 R(저항) 유전자를 포함하는 타입 III 이펙터에 대한 방어 메커니즘을 개발했다.이러한 R 유전자 중 일부는 의도된 표적 유전자의 부위와 유사한 TAL-이펙터 결합 부지를 포함하도록 진화한 것으로 보인다.병원성 박테리아와 숙주 공장 사이의 이러한 경쟁은 TAL 이펙터 DNA 결합 영역의 명백한 유동성을 설명하기 위해 가정되었다.[7]
| X. 종 | 피해 작물 |
|---|---|
| X. 캠페스트리스 pv.usacerum, X. vasicola pv. musacerum | 바나나와 엔제테 |
| X. Axonopodis pv. phaseoli, X. Axonopodis pv. glycines | 보통 콩과 콩 |
| X. 액소노포디스 pv. 비니콜라 | 카우페아 |
| X. campestris pv. campestris, X. campestris pv. armaracieae. | 모든 브라시카스 |
| X. 액소노포디스 pv. 마니호티스 | 카사바 |
| X. 반투명 pv. 반투명 | 보리, 호밀, 밀, 정치 |
| X. Axonopodis pv. citri, X. citri pv. aurantifolii. | 감귤류 |
| X. 캠페스트리스 pv. 말바세아룸 | 면화 |
| X. euvesicatoria, X. gardneri | 후추/후추 |
| X. 오리재 pv. 오리재 | 쌀밥 |
| X. 아보리콜라 pv.프루니 | 자두, 복숭아, 과즙 |
| X. 천공, X. 캠페스트리스 pv. vesicatoria. | 토마토 |
| X. 캠페스트리스 pv. 망지페레인디카에 | 망고 |
| X. 아보리콜라 pv. juglandis | 호두의 |
논산토모나스속
R. Solanacearum, B. Rizoxinica, 바나나 혈액병(R. Solanacearum 종군에서 아직 확실히 식별되지 않은 박테리아).[1]
DNA인식
TALEN에서 사용되는[8] 1차 역할 및 쌍에 대한 정보가 누락된 문서입니다. 이 (1919년 5월) |
TAL 이펙터의 가장 독특한 특징은 길이 34개의 잔류물인 1.5~33.5번의 반복을 포함하는 중심 반복 영역이다(C-단자 반복은 일반적으로 더 짧고 "반복"이라고 한다).[6]일반적인 반복 시퀀스는LTPEQVVVAIASHDGGGKQALETVRLPVLCQAHG 12번째 및 13번째 위치의 잔류물은 고변량(이 두 아미노산은 반복 변수 이레시듀 또는 RVD라고도 한다)이다.TAL 이펙터의 대상 사이트에 있는 순차적 반복에서 이 두 가지 잔류물의 정체성과 순차적 DNA 베이스 사이에 간단한 관계가 있다.[7]DNA에 바인딩된 TAL 이펙터의 결정 구조는 각 반복이 두 개의 알파 나선형과 짧은 RVD 포함 루프로 구성되며, RVD의 두 번째 잔류물은 시퀀스 고유의 DNA 접촉을 하고 RVD 포함 루프는 첫 번째 잔류물이 RVD 포함 루프를 안정화한다는 것을 나타낸다.[9][10]TAL 임팩터의 대상 사이트는 또한 첫 번째 반복이 목표로 하는 5' 베이스 측면의 티민도 포함하는 경향이 있다. 이는 이 T와 중심 반복 영역의 N단계의 보존된 트립토판 사이의 접촉 때문인 것으로 보인다.[9]그러나 이 "제로" 자세는 일부 비계가 더 관용적이기 때문에 항상 티민(theymin)을 함유하고 있지는 않다.[11]
TAL-DNA 코드는 2010년에 두 개의 별도 그룹에 의해 깨졌다.[7]아담 보그다노브(Adam Bogdanove)가 이끄는 첫 번째 그룹은 TAL에 의해 조절된 유전자의 데이터베이스에서 파생된 단백질 시퀀스 정렬의 패턴과 표적 촉진자의 DNA 시퀀스를 검색함으로써 이 코드를 연산적으로 해독했다.[12]두 번째 그룹(Boch)은 AvrBs3가 활성화한 후추 유전자의 촉진자에서 TAL 이펙터 AvrBs3의 분자 분석과 표적 DNA 시퀀스를 통해 코드를 추론했다.[13]RVD 시퀀스와 대상 DNA 베이스 사이의 실험적으로 검증된 코드는 다음과 같이 표현할 수 있다.
| 잔류물 | 베이스 | 메모들 | 참조 |
|---|---|---|---|
| NI | A | [13] | |
| HD | C | 5-메틸-C가 아님 | [13] |
| NG | T, 5mC | [13][14] | |
| NN | R | 푸린: G 또는 A | [13] |
| NS | N | 아무거나 | [13] |
| NK | G | 단독으로 사용할 경우 TALEN 활동 감소 | [15][16] |
| NH | G | [8] |
표적유전자
TAL 이펙터는 NODULIN3(N3) 유전자 계열의 구성원인 감수성 유전자를 유도할 수 있다.이러한 유전자들은 질병의 발달을 위해 필수적이다.쌀에서는 Os-8N3과 Os-11N3라는 두 유전자가 TAL 이펙터에 의해 유도된다.Os-8N3은 PthXo1에 의해, Os-11N3은 PthXo3와 AvrXa7에 의해 유도된다.N3 단백질의 가능한 기능에 대한 두 가지 가설이 존재한다.
- 그들은 구리 운송에 관여하여 박테리아 환경의 해독을 초래한다.구리 수치의 감소는 박테리아의 성장을 촉진한다.
- 그들은 포도당 운반에 관여하여 포도당 흐름을 용이하게 한다.이 메커니즘은 박테리아에게 영양분을 공급하고 병원체의 성장과 정력을[citation needed] 자극한다.
엔지니어링 TAL 이펙터
TAL 임팩터의 아미노산과 그들의 표적 부위의 DNA 염기들 사이의 간단한 대응은 그들을 단백질 공학 용도에 유용하게 만든다.수많은 그룹들이 다양한 실험 시스템에서 새로운 DNA 시퀀스를 인식할 수 있는 인공 TAL 임팩터를 설계했다.[13][15][16][17][18][19]이러한 공학적 TAL 이펙터는 토마토,[15] 아라비도시스 탈리아나,[15] 인간 세포의 내생 유전자를 표적화하고 활성화하거나 억제하는 데 사용할 수 있는 인공 전사 인자를 만드는 데 사용되어 왔다.[16][18][8][20]
TAL 이펙터 기반 단백질을 인코딩하는 유전자 구조는 기존의 유전자 합성이나 모듈 조립을 사용하여 만들 수 있다.[18][20][21][22][23][24][25][26]사용자 지정 TALEN 및 기타 TAL 이펙터 구조를 조립하기 위한 플라스미드 키트는 비영리 저장소인 Addgene을 통해 사용할 수 있다.TAL 이펙터-DNA 타겟팅 애플리케이션을 위한 공용 소프트웨어, 프로토콜 및 기타 리소스에 대한 액세스를 제공하는 웹 페이지에는 TAL 이펙터-뉴클레오티드 타겟터 및 taleffectors.com가 있다.
적용들
엔지니어링된 TAL 이펙터를 FokI의 갈라짐 영역과 융합하여 TALEN(TAL 이펙터 핵)이나 메가뉴클레아제(인식 부위가 긴 핵)를 만들어 "메가"를 만들 수도 있다.TALs."[27] 그러한 결합은 아연 핑거핵과 일부 성질을 공유하며 유전공학 및 유전자 치료 응용에 유용할 수 있다.[28]
TALEN 기반 접근법은 유전자 편집과 게놈 공학이라는 신흥 분야에서 사용된다.TALEN 퓨즈는 효모 기반 검사,[17][29] 내생 효모 유전자,[21] 식물 리포터 검사,[19] 내생 식물 유전자,[22] 내생 제브라피쉬 유전자,[30][31] 내생 쥐 유전자,[32] 내생 인간 유전자에서 활동을 보여준다.[16][22][33]인간 HPRT1 유전자는 검출이 가능하지만 정량화되지 않은 수준을 목표로 하고 있다.[22]또한, 여전히 DNA 결합 도메인을 포함하는 TAL 이펙터의 작은 부분에 융합된 FokI 분할 영역을 포함하는 TALEN 구조물은 최대 [16]25%의 효율성으로 인간 세포의 내생성 NTF3와 CCR5 유전자를 목표로 하는 데 사용되어 왔다.TAL 이펙터 핵은 또한 인간 배아줄기세포와 유도 만능줄기세포(IPSC)[33]를 만들고, C. 선충의 내생성 벤-1 유전자를 박살내는데 사용되었다.[34]
TAL에 의해 유발된 비호몰성 엔드 결합 수정은 쌀에서 새로운 질병 저항성을 생성하기 위해 사용되어 왔다.[1]
참고 항목
참조
- ^ a b c d e f Schornack S, Moscou MJ, Ward ER, Horvath DM (2013-08-04). "Engineering plant disease resistance based on TAL effectors". Annual Review of Phytopathology. Annual Reviews. 51 (1): 383–406. doi:10.1146/annurev-phyto-082712-102255. PMID 23725472.
- ^ Heuer H, Yin YN, Xue QY, Smalla K, Guo JH (July 2007). "Repeat domain diversity of avrBs3-like genes in Ralstonia solanacearum strains and association with host preferences in the field". Applied and Environmental Microbiology. 73 (13): 4379–84. Bibcode:2007ApEnM..73.4379H. doi:10.1128/AEM.00367-07. PMC 1932761. PMID 17468277.
- ^ Li L, Atef A, Piatek A, Ali Z, Piatek M, Aouida M, et al. (July 2013). "Characterization and DNA-binding specificities of Ralstonia TAL-like effectors". Molecular Plant. 6 (4): 1318–30. doi:10.1093/mp/sst006. PMC 3716395. PMID 23300258.
- ^ de Lange O, Wolf C, Dietze J, Elsaesser J, Morbitzer R, Lahaye T (June 2014). "Programmable DNA-binding proteins from Burkholderia provide a fresh perspective on the TALE-like repeat domain". Nucleic Acids Research. 42 (11): 7436–49. doi:10.1093/nar/gku329. PMC 4066763. PMID 24792163.
- ^ de Lange O, Wolf C, Thiel P, Krüger J, Kleusch C, Kohlbacher O, Lahaye T (November 2015). "DNA-binding proteins from marine bacteria expand the known sequence diversity of TALE-like repeats". Nucleic Acids Research. 43 (20): 10065–80. doi:10.1093/nar/gkv1053. PMC 4787788. PMID 26481363.
- ^ a b c Boch J, Bonas U (September 2010). "Xanthomonas AvrBs3 family-type III effectors: discovery and function". Annual Review of Phytopathology. 48: 419–36. doi:10.1146/annurev-phyto-080508-081936. PMID 19400638.
- ^ a b c Voytas DF, Joung JK (December 2009). "Plant science. DNA binding made easy". Science. 326 (5959): 1491–2. Bibcode:2009Sci...326.1491V. doi:10.1126/science.1183604. PMC 7814878. PMID 20007890. S2CID 33257689.
- ^ a b c Cong L, Zhou R, Kuo YC, Cunniff M, Zhang F (July 2012). "Comprehensive interrogation of natural TALE DNA-binding modules and transcriptional repressor domains". Nature Communications. 968. 3 (7): 968. Bibcode:2012NatCo...3..968C. doi:10.1038/ncomms1962. PMC 3556390. PMID 22828628.
- ^ a b Mak AN, Bradley P, Cernadas RA, Bogdanove AJ, Stoddard BL (February 2012). "The crystal structure of TAL effector PthXo1 bound to its DNA target". Science. 335 (6069): 716–9. Bibcode:2012Sci...335..716M. doi:10.1126/science.1216211. PMC 3427646. PMID 22223736.
- ^ Deng D, Yan C, Pan X, Mahfouz M, Wang J, Zhu JK, et al. (February 2012). "Structural basis for sequence-specific recognition of DNA by TAL effectors". Science. 335 (6069): 720–3. Bibcode:2012Sci...335..720D. doi:10.1126/science.1215670. PMC 3586824. PMID 22223738.
- ^ Stella S, Molina R, Yefimenko I, Prieto J, Silva G, Bertonati C, et al. (September 2013). "Structure of the AvrBs3-DNA complex provides new insights into the initial thymine-recognition mechanism". Acta Crystallographica. Section D, Biological Crystallography. 69 (Pt 9): 1707–16. doi:10.1107/S0907444913016429. PMC 3760130. PMID 23999294.
- ^ Moscou MJ, Bogdanove AJ (December 2009). "A simple cipher governs DNA recognition by TAL effectors". Science. 326 (5959): 1501. Bibcode:2009Sci...326.1501M. doi:10.1126/science.1178817. PMID 19933106. S2CID 6648530.
- ^ a b c d e f g Boch J, Scholze H, Schornack S, Landgraf A, Hahn S, Kay S, et al. (December 2009). "Breaking the code of DNA binding specificity of TAL-type III effectors". Science. 326 (5959): 1509–12. Bibcode:2009Sci...326.1509B. doi:10.1126/science.1178811. PMID 19933107. S2CID 206522347.
- ^ Deng D, Yin P, Yan C, Pan X, Gong X, Qi S, et al. (October 2012). "Recognition of methylated DNA by TAL effectors". Cell Research. 22 (10): 1502–4. doi:10.1038/cr.2012.127. PMC 3463267. PMID 22945353.
- ^ a b c d Morbitzer R, Römer P, Boch J, Lahaye T (December 2010). "Regulation of selected genome loci using de novo-engineered transcription activator-like effector (TALE)-type transcription factors". Proceedings of the National Academy of Sciences of the United States of America. 107 (50): 21617–22. Bibcode:2010PNAS..10721617M. doi:10.1073/pnas.1013133107. PMC 3003021. PMID 21106758.
- ^ a b c d e Miller JC, Tan S, Qiao G, Barlow KA, Wang J, Xia DF, et al. (February 2011). "A TALE nuclease architecture for efficient genome editing". Nature Biotechnology. 29 (2): 143–8. doi:10.1038/nbt.1755. PMID 21179091. S2CID 53549397.
- ^ a b Christian M, Cermak T, Doyle EL, Schmidt C, Zhang F, Hummel A, et al. (October 2010). "Targeting DNA double-strand breaks with TAL effector nucleases". Genetics. 186 (2): 757–61. doi:10.1534/genetics.110.120717. PMC 2942870. PMID 20660643.
- ^ a b c Zhang F, Cong L, Lodato S, Kosuri S, Church GM, Arlotta P (February 2011). "Efficient construction of sequence-specific TAL effectors for modulating mammalian transcription". Nature Biotechnology. 29 (2): 149–53. doi:10.1038/nbt.1775. PMC 3084533. PMID 21248753.
- ^ a b Mahfouz MM, Li L, Shamimuzzaman M, Wibowo A, Fang X, Zhu JK (February 2011). "De novo-engineered transcription activator-like effector (TALE) hybrid nuclease with novel DNA binding specificity creates double-strand breaks". Proceedings of the National Academy of Sciences of the United States of America. 108 (6): 2623–8. Bibcode:2011PNAS..108.2623M. doi:10.1073/pnas.1019533108. PMC 3038751. PMID 21262818.
- ^ a b Geissler R, Scholze H, Hahn S, Streubel J, Bonas U, Behrens SE, Boch J (2011). Shiu S (ed.). "Transcriptional activators of human genes with programmable DNA-specificity". PLOS ONE. 6 (5): e19509. Bibcode:2011PLoSO...619509G. doi:10.1371/journal.pone.0019509. PMC 3098229. PMID 21625585.
- ^ a b Li T, Huang S, Zhao X, Wright DA, Carpenter S, Spalding MH, et al. (August 2011). "Modularly assembled designer TAL effector nucleases for targeted gene knockout and gene replacement in eukaryotes". Nucleic Acids Research. 39 (14): 6315–25. doi:10.1093/nar/gkr188. PMC 3152341. PMID 21459844.
- ^ a b c d Cermak T, Doyle EL, Christian M, Wang L, Zhang Y, Schmidt C, et al. (July 2011). "Efficient design and assembly of custom TALEN and other TAL effector-based constructs for DNA targeting". Nucleic Acids Research. 39 (12): e82. doi:10.1093/nar/gkr218. PMC 3130291. PMID 21493687.
- ^ Morbitzer R, Elsaesser J, Hausner J, Lahaye T (July 2011). "Assembly of custom TALE-type DNA binding domains by modular cloning". Nucleic Acids Research. 39 (13): 5790–9. doi:10.1093/nar/gkr151. PMC 3141260. PMID 21421566.
- ^ Weber E, Gruetzner R, Werner S, Engler C, Marillonnet S (2011). Bendahmane M (ed.). "Assembly of designer TAL effectors by Golden Gate cloning". PLOS ONE. 6 (5): e19722. Bibcode:2011PLoSO...619722W. doi:10.1371/journal.pone.0019722. PMC 3098256. PMID 21625552.
- ^ Sanjana NE, Cong L, Zhou Y, Cunniff MM, Feng G, Zhang F (January 2012). "A transcription activator-like effector toolbox for genome engineering". Nature Protocols. 7 (1): 171–92. doi:10.1038/nprot.2011.431. PMC 3684555. PMID 22222791.
- ^ Briggs AW, Rios X, Chari R, Yang L, Zhang F, Mali P, Church GM (August 2012). "Iterative capped assembly: rapid and scalable synthesis of repeat-module DNA such as TAL effectors from individual monomers". Nucleic Acids Research. 40 (15): e117. doi:10.1093/nar/gks624. PMC 3424587. PMID 22740649.
- ^ Boissel S, Jarjour J, Astrakhan A, Adey A, Gouble A, Duchateau P, et al. (February 2014). "megaTALs: a rare-cleaving nuclease architecture for therapeutic genome engineering". Nucleic Acids Research. 42 (4): 2591–601. doi:10.1093/nar/gkt1224. PMC 3936731. PMID 24285304.
- ^ DeFrancesco L (August 2011). "Move over ZFNs". Nature Biotechnology. 29 (8): 681–4. doi:10.1038/nbt.1935. PMID 21822235. S2CID 29925336.
- ^ Li T, Huang S, Jiang WZ, Wright D, Spalding MH, Weeks DP, Yang B (January 2011). "TAL nucleases (TALNs): hybrid proteins composed of TAL effectors and FokI DNA-cleavage domain". Nucleic Acids Research. 39 (1): 359–72. doi:10.1093/nar/gkq704. PMC 3017587. PMID 20699274.
- ^ Huang P, Xiao A, Zhou M, Zhu Z, Lin S, Zhang B (August 2011). "Heritable gene targeting in zebrafish using customized TALENs". Nature Biotechnology. 29 (8): 699–700. doi:10.1038/nbt.1939. PMID 21822242. S2CID 28802632.
- ^ Sander JD, Cade L, Khayter C, Reyon D, Peterson RT, Joung JK, Yeh JR (August 2011). "Targeted gene disruption in somatic zebrafish cells using engineered TALENs". Nature Biotechnology. 29 (8): 697–8. doi:10.1038/nbt.1934. PMC 3154023. PMID 21822241.
- ^ Tesson L, Usal C, Ménoret S, Leung E, Niles BJ, Remy S, et al. (August 2011). "Knockout rats generated by embryo microinjection of TALENs". Nature Biotechnology. 29 (8): 695–6. doi:10.1038/nbt.1940. PMID 21822240. S2CID 13525337.
- ^ a b Hockemeyer D, Wang H, Kiani S, Lai CS, Gao Q, Cassady JP, et al. (July 2011). "Genetic engineering of human pluripotent cells using TALE nucleases". Nature Biotechnology. 29 (8): 731–4. doi:10.1038/nbt.1927. PMC 3152587. PMID 21738127.
- ^ Wood AJ, Lo TW, Zeitler B, Pickle CS, Ralston EJ, Lee AH, et al. (July 2011). "Targeted genome editing across species using ZFNs and TALENs". Science. 333 (6040): 307. Bibcode:2011Sci...333..307W. doi:10.1126/science.1207773. PMC 3489282. PMID 21700836.
추가 읽기
- Schornack S, Boch J (2010). "Unraveling a 20-year enigma". IS-MPMI Reporter. International Society for Molecular Plant-Microbe Interactions (1): 3–4.
- Goodsell D (December 2014). "TAL Effectors". Protein Data Bank (PDB) Molecule of the Month. Research Collaboratory for Structural Bioinformatics (RCSB). doi:10.2210/rcsb_pdb/mom_2014_12.
외부 링크
- "A comprehensive, publicly available resource for engineered TAL effector technology". TALengineering.org.
- "TALengineering newsgroup".
Newsgroup for discussion of engineered TAL effector technology
- "An open resource for TAL effector constructs". www.taleffectors.com.


