탠덤 질량 분석
Tandem mass spectrometry
MS/MS 또는2 MS로도 알려진 탠덤 질량 분석기는 화학 [1]샘플 분석 능력을 높이기 위해 추가 반응 단계를 사용하여 두 개 이상의 질량 분석기가 함께 결합되는 기기 분석 기법입니다.탠덤 MS의 일반적인 용도는 단백질과 펩타이드와 같은 생체 분자의 분석이다.
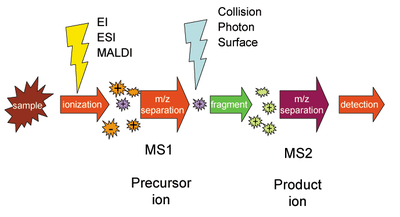
주어진 샘플의 분자는 이온화되며, 첫 번째 분광계(MS1)는 질량 대 전하비(종종 m/z 또는 m/Q로 지정)로 이온을 분리한다.MS1로부터의 특정 m/z비의 이온을 선택하고, 예를 들면 충돌에 의한 해리, 이온 분자 반응, 광분해 등에 의해 보다 작은 단편 이온으로 분할한다.그런 다음 이 조각들이 두 번째 질량 분석계(MS2)에 도입되고, MS2는 조각들을 m/z 비율로 분리하여 검출합니다.파편화 단계를 통해 일반 질량 분석기에서 매우 유사한 m/z 비율을 가진 이온을 식별하고 분리할 수 있습니다.
구조.
탠덤 질량 분석에는 삼중 4극 질량 분석계(QQ), 다중 섹터 질량 분석계, 4극 비행 시간(Q-TOF) 및 하이브리드 질량 분석계가 포함된다.
삼중 사극 질량 분석계
트리플 4극 질량 분석기는 첫 번째와 세 번째 4극 질량 분석기를 질량 필터로 사용합니다.분석 물질이 두 번째 4극점을 통과할 때, 조각화는 기체와의 충돌을 통해 진행됩니다.
4극 비행 시간(Q-TOF)
Q-TOF 질량분석계는 TOF와 4극 계측기를 조합하여 제품 이온의 질량 정확도가 높고 정량 능력이 정확하며 단편화 실험의 적용성이 우수합니다.이는 비행 시간 측정을 통해 이온 조각화(m/z) 비율이 결정되는 질량 분석 방법입니다.
하이브리드 질량 분석계
하이브리드 질량 분석기는 두 개 이상의 질량 분석기로 구성됩니다.
인스트루먼트
개별 질량 분석계 요소를 공간에서 분리하거나 MS 단계를 시간적으로 분리한 단일 질량 분석계를 사용하여 여러 단계의 질량 분석 분리를 수행할 수 있습니다.공간에서의 탠덤 질량 분석의 경우, 사용된 질량 선택기의 유형을 제공하는 다른 요소들이 종종 간략하게 기록된다.
공간에서의 탠덤
공간에서의 탠덤 질량 분석에서 분리 요소는 물리적으로 분리되고 구별되지만 높은 진공 상태를 유지하기 위해 요소 간에 물리적 연결이 있다.이러한 요소는 섹터, 전송 4극 또는 비행 시간일 수 있습니다.여러 개의 쿼드루폴을 사용할 경우 질량 분석기와 충돌 챔버 역할을 모두 수행할 수 있습니다.
질량 분석기의 일반적인 표기법은 Q – 4극 질량 분석기, q – 무선 주파수 충돌 4극, TOF – 비행 시간 질량 분석기, B – 자기 영역 및 E – 전기 영역입니다.이 표기법은 다양한 하이브리드 기기(예: QQ' – 삼중 4극 질량 분석계, QTOF – 4극 비행 시간 질량 분석계(QqTOF) 및 BEBE – 4섹터(역 기하학) 질량 분석계))를 나타내기 위해 조합될 수 있다.
탠덤 인 타임
시간 내에 탠덤 질량 분석법을 실시함으로써 같은 장소에 갇힌 이온에 의한 분리를 실현하고, 시간이 지남에 따라 복수의 분리 공정을 실시한다.이러한 [2]분석에는 4극 이온 트랩 또는 푸리에 변환 이온 사이클로트론 공명(FTICR) 기기를 사용할 수 있다.트랩 계측기는 여러 단계의 분석을 수행할 수 있으며, MS(MS to n)[3]라고도n 합니다.대부분의 경우 스텝 수 n은 표시되지 않지만 값이 지정되는 경우가 있습니다.예를3 들어 MS는 3단계의 분리를 나타냅니다.탠덤 인 타임 MS 계측기는 다음에 설명된 모드를 사용하지 않지만, 일반적으로 전체 스펙트럼의 전구체 이온 스캔 및 부모 이온 스캔에서 모든 정보를 수집합니다.각 계측기 구성에서는 고유한 질량 식별 모드를 사용합니다.
스페이스 MS/MS 모드에서의 탠덤
공간 내 설계로 탠덤 MS를 수행하는 경우 계측기는 다양한 모드 중 하나로 작동해야 합니다.다양한 탠덤 MS/MS 실험 설정이 있으며 각 모드에는 고유한 응용 프로그램이 있으며 서로 다른 정보를 제공합니다.공간의 탠덤 MS는 동일한 질량 스펙트럼 범위를 측정하지만 공간에서는 이들 사이의 분화가 제어된 두 계측기 구성요소의 결합을 사용하는 반면, 시간의 탠덤 MS는 이온 트랩을 사용합니다.
MS/MS를 사용하면 전구체 이온 스캔, 제품 이온 스캔, 중성 손실 스캔 및 선택 반응 모니터링의 네 가지 주요 스캔 실험이 가능합니다.
전구체 이온 스캔은 제2질량분석기에서 생성물 이온을 선택하고, 제1질량분석기에서 전구체 질량을 스캔한다.전구체[4] 이온은 부모[5] 이온과 동의어이고 생성물[6] 이온은 딸 [7]이온과 동의어이다. 그러나 이러한 의인형 용어의 사용은 [8][9]권장되지 않는다.
프로덕트 이온 스캔에서는, 제1 스테이지에서 전구체 이온을 선택해 프래그먼트화한 후, 그 결과 발생하는 모든 질량을 제2 질량 분석기로 스캔 해, 제2 질량 분석기 후에 위치하는 검출기로 검출한다.이 실험은 일반적으로 탠덤 MS에 의한 정량화에 사용되는 전이를 식별하기 위해 수행됩니다.
중성 손실 스캔에서는 첫 번째 질량 분석기가 모든 질량을 스캔합니다.두 번째 질량 분석기도 첫 번째 질량 [10]분석기에서 설정된 오프셋으로 스캔합니다.이 간격띄우기는 화합물 클래스에 대해 일반적으로 관측되는 중성 손실에 해당합니다.상시 중성 손실 스캔에서는 특정 공통 중성 손실을 겪는 모든 전구체가 모니터링된다.이 정보를 얻기 위해 두 질량 분석기가 동시에 스캔되지만 지정된 중성 질량과 상관되는 질량 오프셋을 사용합니다.전구체-이온 스캔과 마찬가지로 이 기술은 혼합물에서 밀접하게 관련된 화합물의 종류를 선택적으로 식별하는 데도 유용하다.
선택된 반응 모니터링에서는 두 질량 분석 장치가 선택된 질량으로 설정된다.이 모드는 MS 실험을 위해 선택된 이온 모니터링과 유사합니다.감도를 [11]높일 수 있는 선택적 분석 모드입니다.
단편화
기상 이온의 단편화는 탠덤 질량 분석에서 필수적이며 질량 분석의 다른 단계 사이에 발생합니다.이온을 파쇄하기 위해 사용되는 많은 방법들이 있으며, 이것들은 다양한 형태의 파쇄를 야기할 수 있고, 따라서 분자의 구조와 구성에 대한 다른 정보를 야기할 수 있다.
소스내 플래그멘테이션
종종 이온화 과정이 충분히 격렬하여 생성된 이온이 질량 분석계 내에서 파편화될 수 있는 충분한 내부 에너지를 남깁니다.생성물 이온이 자동 분해를 하기 전에 적당한 시간 동안 평형 상태가 아닌 상태를 유지하면 이 과정을 준안정 [12]파편화라고 합니다.노즐 스키머 단편화란 보통 일렉트로스프레이 기반의 계측기에서 노즐 스키머 전위를 증가시킴으로써 의도적인 소스 내 단편화 유도를 말한다.인소스 단편화는 단편화 분석을 가능케 하지만, 준안정 이온을 질량 분석 또는 선택하고, 그 결과 얻은 단편들에 대해 제2단계 분석을 하지 않는 한 기술적으로 탠덤 질량 분석법이 아니다.탠덤 질량 분석 [13]데이터와 직접 일치하는 플래그멘테이션을 생성하는 Enhanced In-Source Fragmentation Annotation(EISA) 기술을 사용하여 탠덤 질량 분석 대신 인 소스 플래그멘테이션을 사용할 수 있습니다.EISA에 의해 관측된 fragment는 탠덤 질량 분석계의 [14]충돌 셀에서 손실을 입는 기존 fragment보다 신호 강도가 높습니다.EISA는 비행시간 계측기와 단일 4극 계측기와 같은 MS1 질량 분석기에서 단편화 데이터 수집을 가능하게 합니다.인소스 플래그멘테이션은 종종 탠덤 질량 분석(포스트 소스 플래그멘테이션과 함께)과 함께 사용되어 의사 MS 유형의3 실험에서 [15]2단계 플래그멘테이션을 가능하게 한다.
충돌에 의한 해리
사후 선원 조각화는 종종 탠덤 질량 분석 실험에서 사용된다.에너지는 또한 중성 원자 또는 분자와 사후 소스 충돌, 방사선의 흡수 또는 다중 하전 이온에 의한 전자의 전달 또는 포획을 통해 이미 진동적으로 들뜬 이온에 추가될 수 있습니다.충돌 활성화 해리(CAD)라고도 불리는 충돌 유도 해리(CID)는 기체 단계에서 중성 원자 또는 분자와 이온의 충돌과 그에 따른 이온 [16][17]해리를 포함한다.예를 들어,
이온+ AB가 중성종 M과 충돌한 후 분해된다.이 과정의 세부 사항은 충돌 이론에 의해 설명됩니다.기기 구성이 다르기 때문에 (i) 빔형(전구체 이온이 비행 [18]중에 단편화되는)과 (ii) 이온 트랩형(전구체 이온이 먼저 포착되고 다음으로 단편화되는)[19][20]의 두 가지 주요 CID 유형이 가능합니다.
CID 단편화의 세 번째 및 최근 유형은 고에너지 충돌 해리(HCD)입니다.HCD는 이온 [21][22]트랩 외부에서 단편화가 발생하는 궤도 질량 분석기에 고유한 CID 기술입니다. HCD 세포에서 발생합니다('이온 라우팅 멀티폴'[23]이라는 이름의 일부 기구에서).HCD는 빔 타입의 [24][25]특성이 있는 것으로 판명된 트랩 타입의 플래그멘테이션입니다.자유롭게 이용할 수 있는 대규모 고해상도 탠덤 질량 분석 데이터베이스가 존재하며(예를 들어 각각 실험 CID MS/MS [26]데이터가 포함된 850,000개의 분자 표준을 가진 METLIN), 일반적으로 작은 분자 식별을 용이하게 하기 위해 사용된다.
전자 포획 및 전달 방법
전자가 다중 충전된 이온으로 이동하거나 포획될 때 방출되는 에너지는 분열을 유도할 수 있습니다.
전자 포획 해리
만약 전자가 다중 하전된 양이온에 추가된다면, 쿨롱 에너지는 해방된다.자유 전자를 추가하는 것은 전자 포획 해리(ECD)[27]라고 불리며, 다음과 같이 표현된다.
다중 양성자화 분자 M에 대한 것입니다.
전자 전달 해리
이온 이온 반응을 통해 전자를 추가하는 것을 전자 전달 해리(ETD)[28][29]라고 합니다.전자포획 해리와 유사하게 ETD는 전자를 양이온에 전달함으로써 양이온(예: 펩타이드 또는 단백질)의 파편을 유도한다.그것은 Donald F에 의해 발명되었다. 버지니아 [30]대학의 헌트, 조슈아 쿤, 존 E. P. 사이카, Jarrod Marto.
ETD는 자유 전자를 사용하지 않지만 이러한 목적을 위해 라디칼 음이온(안트라센 또는 아조벤젠)을 사용한다.
여기서 A는 [31]음이온입니다.
ETD는 펩타이드 골격(c 및 z 이온)을 따라 무작위로 갈라지는 반면, 측쇄 및 인산화와 같은 변형은 그대로 남아 있습니다.이 기술은 높은 전하 상태 이온(z>2)에 대해서만 잘 작동하지만, 충돌 유도 해리(CID)에 비해 ETD는 더 긴 펩타이드 또는 전체 단백질의 조각화에 유리하다.이것은 하향식 단백질학에서 이 기술을 중요하게 만든다.ETD는 ECD와 마찬가지로 인산화와 [32]같은 변형된 펩타이드에 효과적이다.
전자전달 및 고에너지 충돌해리(ETHcD)는 펩타이드 전구체가 처음에 c-와 z-이온을 생성하는 선형 이온트랩에서 불소 이온과 이온/[28][33]이온 반응을 일으키는 ETD와 HCD의 조합이다.두 번째 단계에서는 모든 ETD 유도 이온에 HCD 전 이온 단편화를 적용하여 궤도랩 [21]분석기에서 최종 분석 전에 b 이온과 y 이온을 생성한다.이 방법은 펩타이드 배열 및 PTM [34]국재화를 위해 이온과 데이터가 풍부한 MS/MS 스펙트럼을 생성하기 위해 이중 단편화를 사용한다.
음전자 전달 해리
또한 음전자가 전달 해리(NETD)[35]를 통해 전자가 탈양성자 종과 함께 발생할 수 있다.
이 전달 이벤트에 이어 전자 결핍 음이온은 내부 재배열과 파편을 겪는다.NETD는 전자분리해리(EDD)의 이온/이온 유사체이다.
NETD는 C-Cα 결합에서 골격을 따라 펩타이드 및 단백질과 호환된다.생성되는 조각은 보통• a형 및 x형 제품 이온입니다.
전자 이탈 해리
EDD(전자분리해리)는 [36]질량분석에서 음이온종을 단편화하는 방법이다.이것은 전자 포획 해리에 대한 음의 카운터 모드 역할을 합니다.음전하를 띤 이온은 적당한 운동 에너지의 전자 조사를 통해 활성화된다.그 결과 부모 이온 분자로부터 전자가 방출되어 재조합을 통해 해리를 일으킨다.
전하 전달 해리
양전하 펩타이드와 양이온성 [37]시약(CTD)[38] 간 반응은 최근 저전하 상태(1+ 또는 2+) 펩타이드에 대한 대체 고에너지 단편화 경로로 입증되었다.헬륨 양이온을 시약으로 사용하는 CTD의 제안된 메커니즘은 다음과 같습니다.
CTD가 펩타이드의 골격 C-Cα 결합 분열을 유발하고 a형• 및 x형 생성 이온을 제공한다는 최초 보고가 있다.
광분해
해리에 필요한 에너지는 광자 흡수에 의해 추가될 수 있으며, 결과적으로 이온 광분해와 다음과 같이 표현됩니다.
서 h { h는 이온에 흡수된 광자를 나타냅니다.자외선 레이저를 사용할 수 있지만, 생체 [39]분자의 과도한 분해를 초래할 수 있습니다.
적외선 다광자 해리
적외선 광자는 이온을 충분히 흡수하면 열을 가하고 해리를 일으킨다.이 과정은 적외선 다광자 해리(IRMPD)라고 불리며, 종종 이산화탄소 레이저와 [40]FTMS와 같은 이온 포획 질량 분석계로 이루어진다.
흑체 적외선 복사 해리
흑체 방사선은 흑체 적외선 방사 해리(BIRD)[41]로 알려진 기술로 광분해로 사용될 수 있습니다.BORD법은 질량분석계 진공챔버 전체를 가열하여 적외선을 발생시킨다.BORD는 이 방사선을 이용해 이온의 진동을 더욱 강하게 자극합니다. 결합이 깨져 [41][42]파편이 만들어질 때까지요.이것은 적외선 다중광자 해리와 유사하지만 다른 [17]광원으로부터의 적외선을 사용합니다.BORD는 푸리에 변환 이온 사이클로트론 공명 질량 분석과 함께 가장 자주 사용됩니다.
표면 유도 해리
표면 유도 해리(SID)를 사용하면 파편화는 고진공 [43][44]상태에서 이온과 표면이 충돌한 결과입니다.오늘날 SID는 광범위한 이온을 단편화하는 데 사용됩니다.수년 전에는 이온화 방법과 질량 분석기 기술이 높은 m/z의 이온을 적절하게 형성, 전송 또는 특성화할 수 있을 만큼 충분히 발전되지 않았기 때문에 낮은 질량의 단일 전하 종에만 SID를 사용하는 것이 일반적이었다.시간이 지남에 따라 금의 CF(CF2)10 CHCHS로22 구성된 자체3 조립 단층 표면(SAMs)은 탠덤 분광계에서 SID에 가장 많이 사용되는 충돌 표면이었다.SAMs는 유입 이온의 충돌에 대한 유효 질량이 크기 때문에 가장 바람직한 충돌 대상으로 작용했다.또한 이러한 표면은 단단한 불소 탄소 체인으로 구성되어 있어 발사체 이온의 에너지를 크게 감소시키지 않습니다.불소 탄소 사슬은 또한 금속 표면에서 들어오는 [45]이온으로 쉽게 전자가 전달되지 않기 때문에 유용합니다.SID가 안정적으로 유지되고 연결에 대한 귀중한 정보를 제공하는 하위 복합체를 생성하는 능력은 다른 어떤 분리 기술보다 뛰어납니다.SID에서 생성된 복합체는 안정적이고 단편에서 전하 분포를 유지하므로, 이는 복합체가 보다 좁은 m/z 분포를 중심으로 하는 고유한 스펙트럼을 생성한다.SID 제품과 SID 제품이 형성하는 에너지는 복합체의 강점과 토폴로지를 반영합니다.독특한 해리 패턴은 복합체의 4차 구조를 발견하는 데 도움이 됩니다.대칭 전하 분포와 해리 의존성은 SID에 고유하며 생성된 스펙트럼을 다른 해리 [45]기법과 구별되게 한다.
SID 기법은 이온 이동성 질량 분석(IM-MS)에도 적용할 수 있다.이 기술을 위한 세 가지 다른 방법에는 토폴로지의 특성 분석, 서브유닛 간 연결성 및 단백질 구조의 전개 정도가 포함됩니다.단백질 구조의 전개 분석은 SID 기술의 가장 일반적으로 사용되는 응용 프로그램입니다.이온이동성 질량분석(IM-MS)의 경우, SID는 C-반응성 단백질(CRP), 트랜스스티레틴(TTR) 및 콘카발린 A(Con A)의 세 가지 다른 단백질 복합체의 소스 활성 전구체의 해리에 사용된다.이 방법은 이러한 각 복합체의 전개 정도를 관찰하는 데 사용됩니다.이를 위해 SID는 표면과의 충돌 이전에 존재하는 전구체 이온의 구조를 보여주었다.IM-MS는 SID를 각 단백질의 [46]서브유닛에 대한 배열을 직접 측정하는 것으로 사용한다.
FTICR(Fourier-Transform Ion Cyclotron Resonance)은 질량을 측정하는 기기에 초고해상도 및 높은 질량 정확도를 제공할 수 있습니다.이러한 특징들은 FTICR 질량 분석기를 충돌 유도 해리(CID, 전자 전달 해리(ETD)[48] 등 여러 해리 실험과[47] 같은 다양한 응용 분야에 유용한 도구로 만듭니다.또한 기초적인 펩타이드 단편화 연구를 위해 표면 유도 해리가 본 기구에 의해 구현되었다.특히, SID는 ICR 기구 [49]내 에너지학 및 기상 단편화 역학의 연구에 적용되었다.이 접근방식은 양성자화된 펩타이드, 홀수전자 펩타이드 이온, 비공유성 리간드펩타이드 복합체 및 결합금속 클러스터의 기상 단편화를 이해하기 위해 사용되었다.
정량단백질학
정량적 단백질학은 [50][51][52]샘플에서 단백질의 상대적인 양 또는 절대적인 양을 결정하기 위해 사용된다.몇 가지 정량적 단백질학 방법은 탠덤 질량 분석법에 기초한다.MS/MS는 복잡한 생체 [53]분자의 구조를 설명하기 위한 벤치마크 절차가 되었다.
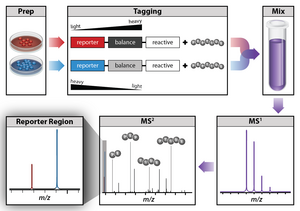
정량적 단백질학에서 일반적으로 사용되는 한 가지 방법은 이소바릭 태그 라벨링이다.이소바릭 태그 라벨링은 단일 분석에서 여러 샘플의 단백질을 동시에 식별하고 정량화할 수 있습니다.단백질을 정량화하기 위해 펩타이드는 구조 및 공칭 질량은 동일하지만 구조 내 무거운 동위원소 분포가 다른 화학 태그로 표시된다.일반적으로 탠덤 질량 태그라고 불리는 이러한 태그는 탠덤 질량 분석 중 고에너지 충돌 유도 해리(HCD) 시 특정 링커 영역에서 질량 태그를 절단하여 서로 다른 질량의 리포터 이온을 생성하도록 설계되었습니다.MS/MS 스펙트럼의 리포터 이온의 강도를 비교함으로써 단백질 정량화를 실시한다.시판되는 두 개의 이소바릭 태그는 iTRAQ 및 TMT 시약입니다.
상대 및 절대 정량(iTRAQ)을 위한 등압 태그
상대 절대 정량용 이소바릭 태그(iTRAQ)는 탠덤 질량 분석용 시약으로, 단일 [54][55][56]실험에서 서로 다른 소스로부터의 단백질 양을 결정하기 위해 사용됩니다.그것은 단백질의 N 말단 및 측쇄 아민과 공유 결합할 수 있는 안정적인 동위원소 라벨 분자를 사용한다.iTRAQ 시약은 액체 크로마토그래피 및 탠덤 질량 분석으로 풀링 및 분석되는 다양한 샘플의 펩타이드에 라벨을 붙이는 데 사용됩니다.부착된 태그의 조각은 저분자 질량 리포터 이온을 생성하며, 펩타이드와 펩타이드가 유래한 단백질을 상대적으로 정량화하는 데 사용될 수 있다.
탠덤 매스 태그(TMT)
탠덤 매스 태그(TMT)는 단백질 정량 및 식별에 사용되는 [57]이소바릭 매스 태그 화학 라벨입니다.태그에는 질량 리포터, 절단 가능한 링커, 질량 정규화 및 단백질 반응 그룹의 4가지 영역이 포함됩니다.TMT 시약을 사용하여 세포, 조직 또는 생물학적 액체에서 제조된 2 ~ 11개의 서로 다른 펩타이드 샘플을 동시에 분석할 수 있습니다.화학반응성이 다른 TMT 시약에는 (1) 1차 아민 라벨링을 위한 반응성 NHS 에스테르 관능기(TMTduplex, TMT6plex, TMT10plex + TMT11-131C), (2) 유리수프릴 라벨링을 위한 반응성 요도아세틸 관능기(Sulfhydydylsetylsetyls)의 세 종류가 있다.rbonyls(aminoxy TMT)
적용들
펩타이드
단백질 [58]배열 분석에는 탠덤 질량 분석법을 사용할 수 있다.온전한 단백질이 질량분석기에 도입될 때, 이것을 "하향식 단백질학"이라고 하며, 단백질이 더 작은 펩타이드로 소화되어 질량분석계에 도입될 때, 이것을 "하향식 단백질학"이라고 한다.샷건 프로테오믹스는 혼합물의 단백질이 분리 및 탠덤 질량 분석 전에 소화되는 보텀업 프로테오믹스의 변형이다.
탠덤 질량 분석법은 단백질 데이터베이스에서 [59][60][61]펩타이드를 식별하는 데 사용할 수 있는 펩타이드 배열 태그를 생성할 수 있다.탠덤 질량 [62]스펙트럼에서 발생하는 펩타이드 단편을 나타내는 표기가 개발되었다.펩타이드 단편 이온은 N 말단에 전하가 유지되면 a, b 또는 c, C 말단에 전하가 유지되면 x, y 또는 z로 나타낸다.첨자는 단편 내 아미노산 잔류물의 수를 나타낸다.간혹 위 첨자는 골격 조각 외에 중성 손실을 나타내기 위해 사용된다. * 암모니아 손실의 경우, *, 물 손실의 경우 °펩타이드 골격 분할은 배열 및 펩타이드 식별에 가장 유용하지만 다른 단편 이온은 높은 에너지 해리 조건에서 관찰될 수 있다.여기에는 측쇄 손실 이온 d, v, w 및 암모늄[63][64] 이온과 특정 아미노산 [65]잔류물과 관련된 추가적인 배열 특이적 단편 이온이 포함됩니다.
올리고당류
올리고당은 펩타이드 [66]배열과 유사한 방법으로 탠덤 질량분석법을 사용하여 배열할 수 있다.분열은 일반적으로 글리코시드 결합(b, c, y 및 z 이온)의 양쪽에서 발생하지만, 또한 교차 고리 분할(x 이온)의 당 고리 구조를 통해 보다 에너지적인 조건에서 발생합니다.끝 첨자는 체인을 따라 분할된 위치를 나타내기 위해 다시 사용됩니다.크로스링 분할 이온의 경우 크로스링 분할의 성질은 앞의 [67][68]윗첨자로 나타납니다.
올리고뉴클레오티드
탠덤 질량 분석법은 DNA와 RNA 염기서열 [69][70]분석에 적용되었다.올리고뉴클레오티드 이온의 기상 단편화를 위한 표기법이 [71]제안되었다.
신생아 검진
신생아 검진은 신생아의 치료 가능한 유전적, 내분비학적, 대사적,[72][73] 혈액학적 질병 여부를 검사하는 과정이다.1990년대 초 탠덤 질량 분석 검사의 개발로 유기산의 [74]혈중 수치에 영향을 미치는 잠재적으로 검출 가능한 선천성 대사 질환이 크게 확대되었다.
제한
탠덤 질량분석법은 단세포 분석에 적용할 수 없다. 왜냐하면 이렇게 적은 양의 세포를 분석하는 것은 둔감하기 때문이다.이러한 한계는 주로 [75]용제의 화학적 소음원으로 인한 기기 내 비효율적인 이온 생산과 이온 손실의 조합에 기인한다.
장래 전망
탠덤 질량 분석법은 단백질 특성 평가, 핵단백질 복합체 및 기타 생물학적 구조에 유용한 도구가 될 것이다.그러나 프로테옴의 특성을 양적, 질적으로 [76]분석하는 등 몇 가지 과제가 남았다.
「 」를 참조해 주세요.
레퍼런스
- ^ IUPAC, 화학 용어집, 제2판('골드북') (1997).온라인 수정판: (2006–) "탄뎀 질량 분석계". doi:10.1351/goldbook.T06250
- ^ Cody RB, Freiser BS (1982). "Collision-induced dissociation in a fourier-transform mass spectrometer". International Journal of Mass Spectrometry and Ion Physics. 41 (3): 199–204. Bibcode:1982IJMSI..41..199C. doi:10.1016/0020-7381(82)85035-3.
- ^ Cody RB, Burnier RC, Cassady CJ, Freiser BS (1 November 1982). "Consecutive collision-induced dissociations in Fourier transform mass spectrometry". Analytical Chemistry. 54 (13): 2225–2228. doi:10.1021/ac00250a021.
- ^ IUPAC, 화학 용어집, 제2판('골드북') (1997).온라인 수정판: (2006–) "프리저 이온" . doi : 10.1351 / goldbook . P04807
- ^ IUPAC, 화학 용어집, 제2판('골드북') (1997).온라인 수정판: (2006–) "부모 이온" . doi : 10.1351 / goldbook . P04406
- ^ IUPAC, 화학 용어집, 제2판('골드북') (1997).온라인 수정판: (2006–) "제품 이온" . doi : 10.1351 / goldbook . P04864
- ^ IUPAC, 화학 용어집, 제2판('골드북') (1997).온라인 수정판: (2006–) "딸 이온" . doi : 10.1351 / goldbook . D01524
- ^ Bursey, Maurice M. (1991). "Comment to readers: Style and the lack of it". Mass Spectrometry Reviews. 10 (1): 1–2. Bibcode:1991MSRv...10....1B. doi:10.1002/mas.1280100102.
- ^ Adams, J. (1992). "To the editor". Journal of the American Society for Mass Spectrometry. 3 (4): 473. doi:10.1016/1044-0305(92)87078-D.
- ^ Louris JN, Wright LG, Cooks RG, Schoen AE (1985). "New scan modes accessed with a hybrid mass spectrometer". Analytical Chemistry. 57 (14): 2918–2924. doi:10.1021/ac00291a039.
- ^ deHoffman E, Stroobant V (2003). Mass Spectrometry: Principles and Applications. Toronto: Wiley. p. 133. ISBN 978-0-471-48566-7.
- ^ IUPAC, 화학 용어집, 제2판('골드북') (1997).온라인 수정판: (2006–) "과도적(화학)종". doi:10.1351/goldbook.T06451
- ^ Domingo-Almenara, Xavier; Montenegro-Burke, J. Rafael; Guijas, Carlos; Majumder, Erica L.-W.; Benton, H. Paul; Siuzdak, Gary (5 March 2019). "Autonomous METLIN-Guided In-source Fragment Annotation for Untargeted Metabolomics". Analytical Chemistry. 91 (5): 3246–3253. doi:10.1021/acs.analchem.8b03126. PMC 6637741. PMID 30681830.
- ^ Xue, Jingchuan; Domingo-Almenara, Xavier; Guijas, Carlos; Palermo, Amelia; Rinschen, Markus M.; Isbell, John; Benton, H. Paul; Siuzdak, Gary (21 April 2020). "Enhanced in-Source Fragmentation Annotation Enables Novel Data Independent Acquisition and Autonomous METLIN Molecular Identification". Analytical Chemistry. 92 (8): 6051–6059. doi:10.1021/acs.analchem.0c00409. PMID 32242660.
- ^ Körner R, Wilm M, Morand K, Schubert M, Mann M (February 1996). "Nano electrospray combined with a quadrupole ion trap for the analysis of peptides and protein digests". Journal of the American Society for Mass Spectrometry. 7 (2): 150–6. doi:10.1016/1044-0305(95)00626-5. PMID 24203235.
- ^ Wells JM, McLuckey SA (2005). "Collision-Induced Dissociation (CID) of Peptides and Proteins". Collision-induced dissociation (CID) of peptides and proteins. Methods in Enzymology. Vol. 402. pp. 148–85. doi:10.1016/S0076-6879(05)02005-7. ISBN 9780121828073. PMID 16401509.
- ^ a b Sleno L, Volmer DA (October 2004). "Ion activation methods for tandem mass spectrometry". Journal of Mass Spectrometry. 39 (10): 1091–112. Bibcode:2004JMSp...39.1091S. doi:10.1002/jms.703. PMID 15481084.
- ^ Xia Y, Liang X, McLuckey SA (February 2006). "Ion trap versus low-energy beam-type collision-induced dissociation of protonated ubiquitin ions". Analytical Chemistry. 78 (4): 1218–27. doi:10.1021/ac051622b. PMID 16478115.
- ^ March RE (1 April 1997). "An Introduction to Quadrupole Ion Trap Mass Spectrometry". Journal of Mass Spectrometry. 32 (4): 351–369. Bibcode:1997JMSp...32..351M. doi:10.1002/(sici)1096-9888(199704)32:4<351::aid-jms512>3.0.co;2-y.
- ^ Bantscheff M, Boesche M, Eberhard D, Matthieson T, Sweetman G, Kuster B (September 2008). "Robust and sensitive iTRAQ quantification on an LTQ Orbitrap mass spectrometer". Molecular & Cellular Proteomics. 7 (9): 1702–13. doi:10.1074/mcp.M800029-MCP200. PMC 2556025. PMID 18511480.
- ^ a b Olsen JV, Macek B, Lange O, Makarov A, Horning S, Mann M (September 2007). "Higher-energy C-trap dissociation for peptide modification analysis". Nature Methods. 4 (9): 709–12. doi:10.1038/nmeth1060. PMID 17721543. S2CID 2538231.
- ^ Senko MW, Remes PM, Canterbury JD, Mathur R, Song Q, Eliuk SM, Mullen C, Earley L, Hardman M, Blethrow JD, Bui H, Specht A, Lange O, Denisov E, Makarov A, Horning S, Zabrouskov V (December 2013). "Novel parallelized quadrupole/linear ion trap/Orbitrap tribrid mass spectrometer improving proteome coverage and peptide identification rates". Analytical Chemistry. 85 (24): 11710–4. doi:10.1021/ac403115c. PMID 24251866.
- ^ Riley NM, Westphall MS, Coon JJ (July 2017). "Activated Ion-Electron Transfer Dissociation Enables Comprehensive Top-Down Protein Fragmentation". Journal of Proteome Research. 16 (7): 2653–2659. doi:10.1021/acs.jproteome.7b00249. PMC 5555583. PMID 28608681.
- ^ Nagaraj N, D'Souza RC, Cox J, Olsen JV, Mann M (December 2010). "Feasibility of large-scale phosphoproteomics with higher energy collisional dissociation fragmentation". Journal of Proteome Research. 9 (12): 6786–94. doi:10.1021/pr100637q. PMID 20873877.
- ^ Jora M, Burns AP, Ross RL, Lobue PA, Zhao R, Palumbo CM, Beal PA, Addepalli B, Limbach PA (August 2018). "Differentiating Positional Isomers of Nucleoside Modifications by Higher-Energy Collisional Dissociation Mass Spectrometry (HCD MS)". Journal of the American Society for Mass Spectrometry. 29 (8): 1745–1756. Bibcode:2018JASMS..29.1745J. doi:10.1007/s13361-018-1999-6. PMC 6062210. PMID 29949056.
- ^ "Article Metrics - METLIN MS 2 molecular standards database: a broad chemical and biological resource Nature Methods". ISSN 1548-7105.
{{cite journal}}:Cite 저널 요구 사항journal=(도움말) - ^ Cooper HJ, Håkansson K, Marshall AG (2005). "The role of electron capture dissociation in biomolecular analysis". Mass Spectrometry Reviews. 24 (2): 201–22. Bibcode:2005MSRv...24..201C. doi:10.1002/mas.20014. PMID 15389856.
- ^ a b Syka JE, Coon JJ, Schroeder MJ, Shabanowitz J, Hunt DF (June 2004). "Peptide and protein sequence analysis by electron transfer dissociation mass spectrometry". Proceedings of the National Academy of Sciences of the United States of America. 101 (26): 9528–33. Bibcode:2004PNAS..101.9528S. doi:10.1073/pnas.0402700101. PMC 470779. PMID 15210983.
- ^ Mikesh LM, Ueberheide B, Chi A, Coon JJ, Syka JE, Shabanowitz J, Hunt DF (December 2006). "The utility of ETD mass spectrometry in proteomic analysis". Biochimica et Biophysica Acta (BBA) - Proteins and Proteomics. 1764 (12): 1811–22. doi:10.1016/j.bbapap.2006.10.003. PMC 1853258. PMID 17118725.
- ^ 미국 특허 7534622, 도널드 F.헌트, 조슈아 J.쿤, 존 EP사이카, 재로드 A마르토, "생물고분자 배열 질량 분석용 전자 전달 해리", 2009-05-19 발행
- ^ McLuckey SA, Stephenson JL (1998). "Ion/ion chemistry of high-mass multiply charged ions". Mass Spectrometry Reviews. 17 (6): 369–407. Bibcode:1998MSRv...17..369M. doi:10.1002/(SICI)1098-2787(1998)17:6<369::AID-MAS1>3.0.CO;2-J. PMID 10360331.
- ^ Chi A, Huttenhower C, Geer LY, Coon JJ, Syka JE, Bai DL, Shabanowitz J, Burke DJ, Troyanskaya OG, Hunt DF (February 2007). "Analysis of phosphorylation sites on proteins from Saccharomyces cerevisiae by electron transfer dissociation (ETD) mass spectrometry". Proceedings of the National Academy of Sciences of the United States of America. 104 (7): 2193–8. Bibcode:2007PNAS..104.2193C. doi:10.1073/pnas.0607084104. PMC 1892997. PMID 17287358.
- ^ Frese CK, Altelaar AF, van den Toorn H, Nolting D, Griep-Raming J, Heck AJ, Mohammed S (November 2012). "Toward full peptide sequence coverage by dual fragmentation combining electron-transfer and higher-energy collision dissociation tandem mass spectrometry". Analytical Chemistry. 84 (22): 9668–73. doi:10.1021/ac3025366. PMID 23106539.
- ^ Frese CK, Zhou H, Taus T, Altelaar AF, Mechtler K, Heck AJ, Mohammed S (March 2013). "Unambiguous phosphosite localization using electron-transfer/higher-energy collision dissociation (EThcD)". Journal of Proteome Research. 12 (3): 1520–5. doi:10.1021/pr301130k. PMC 3588588. PMID 23347405.
- ^ Coon JJ, Shabanowitz J, Hunt DF, Syka JE (June 2005). "Electron transfer dissociation of peptide anions". Journal of the American Society for Mass Spectrometry. 16 (6): 880–2. doi:10.1016/j.jasms.2005.01.015. PMID 15907703.
- ^ Budnik BA, Haselmann KF, Zubarev RA (2001). "Electron detachment dissociation of peptide di-anions: an electron–hole recombination phenomenon". Chemical Physics Letters. 342 (3–4): 299–302. Bibcode:2001CPL...342..299B. doi:10.1016/S0009-2614(01)00501-2.
- ^ Chingin K, Makarov A, Denisov E, Rebrov O, Zubarev RA (January 2014). "Fragmentation of positively-charged biological ions activated with a beam of high-energy cations". Analytical Chemistry. 86 (1): 372–9. doi:10.1021/ac403193k. PMID 24236851.
- ^ Hoffmann WD, Jackson GP (November 2014). "Charge transfer dissociation (CTD) mass spectrometry of peptide cations using kiloelectronvolt helium cations". Journal of the American Society for Mass Spectrometry. 25 (11): 1939–43. Bibcode:2014JASMS..25.1939H. doi:10.1007/s13361-014-0989-6. PMID 25231159. S2CID 1400057.
- ^ Morgan JW, Hettick JM, Russell DH (2005). "Peptide Sequencing by MALDI 193-nm Photodissociation TOF MS". Peptide sequencing by MALDI 193-nm photodissociation TOF MS. Methods in Enzymology. Vol. 402. pp. 186–209. doi:10.1016/S0076-6879(05)02006-9. ISBN 9780121828073. PMID 16401510.
- ^ Little DP, Speir JP, Senko MW, O'Connor PB, McLafferty FW (September 1994). "Infrared multiphoton dissociation of large multiply charged ions for biomolecule sequencing". Analytical Chemistry. 66 (18): 2809–15. doi:10.1021/ac00090a004. PMID 7526742.
- ^ a b Schnier PD, Price WD, Jockusch RA, Williams ER (July 1996). "Blackbody infrared radiative dissociation of bradykinin and its analogues: energetics, dynamics, and evidence for salt-bridge structures in the gas phase". Journal of the American Chemical Society. 118 (30): 7178–89. doi:10.1021/ja9609157. PMC 1393282. PMID 16525512.
- ^ Dunbar RC (2004). "BIRD (blackbody infrared radiative dissociation): evolution, principles, and applications". Mass Spectrometry Reviews. 23 (2): 127–58. Bibcode:2004MSRv...23..127D. doi:10.1002/mas.10074. PMID 14732935.
- ^ Grill V, Shen J, Evans C, Cooks RG (2001). "Collisions of ions with surfaces at chemically relevant energies: Instrumentation and phenomena". Review of Scientific Instruments. 72 (8): 3149. Bibcode:2001RScI...72.3149G. doi:10.1063/1.1382641.
- ^ Mabud, M. (1985). "Surface-induced dissociation of molecular ions". International Journal of Mass Spectrometry and Ion Processes. 67 (3): 285–294. Bibcode:1985IJMSI..67..285M. doi:10.1016/0168-1176(85)83024-X.
- ^ a b Stiving, Alyssa; VanAernum, Zachary; Busch, Florian; Harvey, Sophie; Sarni, Samantha; Wysocki, Vicki (9 November 2018). "Surface-Induced Dissociation: An Effective Method for Characterization of Protein Quaternary Structure". Analytical Chemistry. 91 (1): 190–191. doi:10.1021/acs.analchem.8b05071. PMC 6571034. PMID 30412666.
- ^ Quintyn, Royston S.; Zhou, Mowei; Yan, Jing; Wysocki, Vicki H. (1 December 2015). "Surface-Induced Dissociation Mass Spectra as a Tool for Distinguishing Different Structural Forms of Gas-Phase Multimeric Protein Complexes". Analytical Chemistry. 87 (23): 11879–11886. doi:10.1021/acs.analchem.5b03441. ISSN 0003-2700. PMID 26499904.
- ^ Laskin, Julia; Futrell, Jean H. (2005). "Activation of large lons in FT-ICR mass spectrometry". Mass Spectrometry Reviews. 24 (2): 135–167. Bibcode:2005MSRv...24..135L. doi:10.1002/mas.20012. ISSN 0277-7037. PMID 15389858.
- ^ Kaplan, Desmond A.; Hartmer, Ralf; Speir, J. Paul; Stoermer, Carsten; Gumerov, Dmitry; Easterling, Michael L.; Brekenfeld, Andreas; Kim, Taeman; Laukien, Frank; Park, Melvin A. (2008). "Electron transfer dissociation in the hexapole collision cell of a hybrid quadrupole-hexapole Fourier transform ion cyclotron resonance mass spectrometer". Rapid Communications in Mass Spectrometry. 22 (3): 271–278. Bibcode:2008RCMS...22..271K. doi:10.1002/rcm.3356. ISSN 0951-4198. PMID 18181247.
- ^ Laskin, Julia (June 2015). "Surface-Induced Dissociation: A Unique Tool for Studying Energetics and Kinetics of the Gas-Phase Fragmentation of Large Ions". European Journal of Mass Spectrometry. 21 (3): 377–389. doi:10.1255/ejms.1358. ISSN 1469-0667. PMID 26307719. S2CID 19837927.
- ^ Ong SE, Mann M (October 2005). "Mass spectrometry-based proteomics turns quantitative". Nature Chemical Biology. 1 (5): 252–62. doi:10.1038/nchembio736. PMID 16408053.
- ^ Bantscheff M, Schirle M, Sweetman G, Rick J, Kuster B (October 2007). "Quantitative mass spectrometry in proteomics: a critical review". Analytical and Bioanalytical Chemistry. 389 (4): 1017–31. doi:10.1007/s00216-007-1486-6. PMID 17668192.
- ^ Nikolov M, Schmidt C, Urlaub H (2012). "Quantitative mass spectrometry-based proteomics: an overview". Quantitative Methods in Proteomics. Methods in Molecular Biology. Vol. 893. pp. 85–100. doi:10.1007/978-1-61779-885-6_7. hdl:11858/00-001M-0000-0029-1A75-8. ISBN 978-1-61779-884-9. PMID 22665296.
- ^ Maher S, Jjunju FP, Taylor S (2015). "100 years of mass spectrometry: Perspectives and future trends". Rev. Mod. Phys. 87 (1): 113–135. Bibcode:2015RvMP...87..113M. doi:10.1103/RevModPhys.87.113.
- ^ Ross PL, Huang YN, Marchese JN, Williamson B, Parker K, Hattan S, Khainovski N, Pillai S, Dey S, Daniels S, Purkayastha S, Juhasz P, Martin S, Bartlet-Jones M, He F, Jacobson A, Pappin DJ (December 2004). "Multiplexed protein quantitation in Saccharomyces cerevisiae using amine-reactive isobaric tagging reagents". Molecular & Cellular Proteomics. 3 (12): 1154–69. doi:10.1074/mcp.M400129-MCP200. PMID 15385600.
- ^ Zieske LR (2006). "A perspective on the use of iTRAQ reagent technology for protein complex and profiling studies". Journal of Experimental Botany. 57 (7): 1501–8. doi:10.1093/jxb/erj168. PMID 16574745.
- ^ Gafken PR, Lampe PD (2006). "Methodologies for characterizing phosphoproteins by mass spectrometry". Cell Communication & Adhesion. 13 (5–6): 249–62. doi:10.1080/15419060601077917. PMC 2185548. PMID 17162667.
- ^ Thompson A, Schäfer J, Kuhn K, Kienle S, Schwarz J, Schmidt G, Neumann T, Johnstone R, Mohammed AK, Hamon C (April 2003). "Tandem mass tags: a novel quantification strategy for comparative analysis of complex protein mixtures by MS/MS". Analytical Chemistry. 75 (8): 1895–904. doi:10.1021/ac0262560. PMID 12713048.
- ^ Angel TE, Aryal UK, Hengel SM, Baker ES, Kelly RT, Robinson EW, Smith RD (May 2012). "Mass spectrometry-based proteomics: existing capabilities and future directions". Chemical Society Reviews. 41 (10): 3912–28. doi:10.1039/c2cs15331a. PMC 3375054. PMID 22498958.
- ^ Hardouin J (2007). "Protein sequence information by matrix-assisted laser desorption/ionization in-source decay mass spectrometry". Mass Spectrometry Reviews. 26 (5): 672–82. Bibcode:2007MSRv...26..672H. doi:10.1002/mas.20142. PMID 17492750.
- ^ Shadforth I, Crowther D, Bessant C (November 2005). "Protein and peptide identification algorithms using MS for use in high-throughput, automated pipelines". Proteomics. 5 (16): 4082–95. doi:10.1002/pmic.200402091. PMID 16196103.
- ^ Mørtz E, O'Connor PB, Roepstorff P, Kelleher NL, Wood TD, McLafferty FW, Mann M (August 1996). "Sequence tag identification of intact proteins by matching tanden mass spectral data against sequence data bases". Proceedings of the National Academy of Sciences of the United States of America. 93 (16): 8264–7. Bibcode:1996PNAS...93.8264M. doi:10.1073/pnas.93.16.8264. PMC 38658. PMID 8710858.
- ^ Roepstorff P, Fohlman J (November 1984). "Proposal for a common nomenclature for sequence ions in mass spectra of peptides". Biomedical Mass Spectrometry. 11 (11): 601. doi:10.1002/bms.1200111109. PMID 6525415.
- ^ Johnson RS, Martin SA, Klaus Biemann (December 1988). "Collision-induced fragmentation of (M + H)+ ions of peptides. Side chain specific sequence ions". International Journal of Mass Spectrometry and Ion Processes. 86: 137–154. Bibcode:1988IJMSI..86..137J. doi:10.1016/0168-1176(88)80060-0.
- ^ Falick AM, Hines WM, Medzihradszky KF, Baldwin MA, Gibson BW (November 1993). "Low-mass ions produced from peptides by high-energy collision-induced dissociation in tandem mass spectrometry". Journal of the American Society for Mass Spectrometry. 4 (11): 882–93. doi:10.1016/1044-0305(93)87006-X. PMID 24227532.
- ^ Downard KM, Biemann K (January 1995). "Methionine specific sequence ions formed by the dissociation of protonated peptides at high collision energies". Journal of Mass Spectrometry. 30 (1): 25–32. Bibcode:1995JMSp...30...25D. doi:10.1002/jms.1190300106.
- ^ Zaia J (2004). "Mass spectrometry of oligosaccharides". Mass Spectrometry Reviews. 23 (3): 161–227. Bibcode:2004MSRv...23..161Z. doi:10.1002/mas.10073. PMID 14966796.
- ^ Bruno Domon; Catherine E Costello (1988). "A systematic nomenclature for carbohydrate fragmentations in FAB-MS/MS spectra of glycoconjugates". Glycoconj. J. 5 (4): 397–409. doi:10.1007/BF01049915.
- ^ Spina E, Cozzolino R, Ryan E, Garozzo D (August 2000). "Sequencing of oligosaccharides by collision-induced dissociation matrix-assisted laser desorption/ionization mass spectrometry". Journal of Mass Spectrometry. 35 (8): 1042–8. Bibcode:2000JMSp...35.1042S. doi:10.1002/1096-9888(200008)35:8<1042::AID-JMS33>3.0.CO;2-Y. PMID 10973004.
- ^ Banoub JH, Newton RP, Esmans E, Ewing DF, Mackenzie G (May 2005). "Recent developments in mass spectrometry for the characterization of nucleosides, nucleotides, oligonucleotides, and nucleic acids". Chemical Reviews. 105 (5): 1869–915. doi:10.1021/cr030040w. PMID 15884792.
- ^ Thomas B, Akoulitchev AV (March 2006). "Mass spectrometry of RNA". Trends in Biochemical Sciences. 31 (3): 173–81. doi:10.1016/j.tibs.2006.01.004. PMID 16483781.
- ^ Wu J, McLuckey SA (2004). "Gas-phase fragmentation of oligonucleotide ions". International Journal of Mass Spectrometry. 237 (2–3): 197–241. Bibcode:2004IJMSp.237..197W. doi:10.1016/j.ijms.2004.06.014.
- ^ Tarini BA (August 2007). "The current revolution in newborn screening: new technology, old controversies". Archives of Pediatrics & Adolescent Medicine. 161 (8): 767–72. doi:10.1001/archpedi.161.8.767. PMID 17679658.
- ^ Kayton A (2007). "Newborn screening: a literature review". Neonatal Network. 26 (2): 85–95. doi:10.1891/0730-0832.26.2.85. PMID 17402600.
- ^ Chace DH, Kalas TA, Naylor EW (November 2003). "Use of tandem mass spectrometry for multianalyte screening of dried blood specimens from newborns". Clinical Chemistry. 49 (11): 1797–817. doi:10.1373/clinchem.2003.022178. PMID 14578311.
- ^ Angel, Thomas E.; Aryal, Uma K.; Hengel, Shawna M.; Baker, Erin S.; Kelly, Ryan T.; Robinson, Errol W.; Smith, Richard D. (21 May 2012). "Mass spectrometry based proteomics: existing capabilities and future directions". Chemical Society Reviews. 41 (10): 3912–3928. doi:10.1039/c2cs15331a. ISSN 0306-0012. PMC 3375054. PMID 22498958.
- ^ Han, Xuemei; Aslanian, Aaron; Yates, John R. (October 2008). "Mass Spectrometry for Proteomics". Current Opinion in Chemical Biology. 12 (5): 483–490. doi:10.1016/j.cbpa.2008.07.024. ISSN 1367-5931. PMC 2642903. PMID 18718552.
참고 문헌
- McLuckey SA, Busch KL, Glish GL (1988). Mass spectrometry/mass spectrometry: techniques and applications of tandem mass spectrometry. New York, N.Y: VCH Publishers. ISBN 978-0-89573-275-0.
- McLuckey SA, Glish GL. Mass Spectrometry/Mass Spectrometry: Techniques and Applications of Tandem. Chichester: John Wiley & Sons. ISBN 978-0-471-18699-1.
- McLafferty FW (1983). Tandem mass spectrometry. New York: Wiley. ISBN 978-0-471-86597-1.
- Sherman NE, Kinter M (2000). Protein sequencing and identification using tandem mass spectrometry. New York: John Wiley. ISBN 978-0-471-32249-8.
외부 링크
- Alison E박사의 질량분석 입문. Ashcroft는 2020년 8월 8일 Wayback Machine에서 아카이브 완료



![{\displaystyle [{\ce {M}}+n{\ce {H}}]^{n+}+{\ce {e^{-}->}}\left[[{\ce {M}}+(n-1){\ce {H}}]^{(n-1)+}\right]^{*}{\ce {->fragments}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/80535d501352b7854827d81bd70a7811d203d639)
![{\displaystyle [{\ce {M}}+n{\ce {H}}]^{n+}+{\ce {A^{-}->}}\left[[{\ce {M}}+(n-1){\ce {H}}]^{(n-1)+}\right]^{*}+{\ce {A->fragments}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/a0f39c0e76fd3f9d3862b8b49cb144a43c1cd907)
![{\displaystyle [{\ce {M}}-n{\ce {H}}]^{n-}+{\ce {A+->}}\left[[{\ce {M}}-n{\ce {H}}]^{(n+1)-}\right]^{*}+{\ce {A->fragments}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/383244602d9ce4607f68c0696b8c55fb10951a3f)
![{\displaystyle {\ce {{[{M}+H]^{1}+}+He+->}}\left[{\ce {[{M}+H]^{2}+}}\right]^{*}+{\ce {He^{0}->fragments}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/a8ccd097f31299c546cd6e591efb6ed4f8556e2e)

 이온에 흡수된 광자를 나타냅니다.자외선 레이저를 사용할 수 있지만, 생체
이온에 흡수된 광자를 나타냅니다.자외선 레이저를 사용할 수 있지만, 생체