자가형질
Autapomorphy계통학에서 자가형질은 특정 분류군에 고유한 파생 특성으로 알려진 독특한 특성이다.즉, 하나의 분류군에서만 발견되지만, 다른 분류군이나 다른 분류군에서는 발견되지 않으며, 심지어 초점 분류군과 가장 밀접한 관련이 있는 분류군(종, 과 또는 일반적으로 어떤 [2]분류군일 수 있음)에서도 발견되지 않는다.따라서 단일 [3]분류군에 대한 아포몰피로 간주할 수 있다.독일 곤충학자 빌리 헤니그가 1950년에 처음 소개한 'autapomorphy'라는 단어는 그리스어 'α'[4]에서 유래했다.
논의
자동 형상은 단일 분류군에만 존재하기 때문에 관계에 대한 정보를 전달하지 않습니다.그러므로, 자기동형은 계통발생적 관계를 추론하는데 유용하지 않다.그러나 시너포몰피와 플레시오몰피와 같은 자가포몰피는 해당 분류군에 따라 상대적인 개념이다.소정의 레벨에서의 자가조식은 덜포함적인 [5]레벨에서의 시나포몰피일 수 있다.현생 뱀에서 자가형성의 한 예가 설명될 수 있다.뱀은 테트라포다를 특징짓는 두 쌍의 다리를 잃었고, 오피디아에 가장 가까운 분류군과 그들의 공통 조상들은 모두 두 쌍의 다리를 가지고 있다.그러므로 오피디아 분류군은 다리가 [3]없는 것에 대해 자가형성을 나타낸다.
자형종의 개념은 과학자들이 종을 정의하고 서로 구별하기 위해 사용할 수 있는 많은 방법 중 하나이다.이 정의는 기본적으로 자가 [6]형성의 수에 의해 측정되는 생식 비호환성과 관련된 발산량에 기초하여 종을 할당한다.이 그룹화 방법은 종종 "단일종 개념" 또는 "필로종" 개념으로 언급되며 D.E.에 의해 대중화되었습니다.1979년 로젠.이 정의에서, 종은 "적어도 하나의 자기질식에 의해 정의될 수 있는 가장 덜 포괄적인 단일통군"[7]으로 간주된다.이 사양화 모델은 비단일화 그룹화를 회피한다는 점에서 유용하지만 비판도 있다.예를 들어, N.I. Platnick은 자형종의 개념이 어미 개체군의 "종" 상태를 취소하면서 생식적인 고립과 분화의 가능성을 허용하기 때문에 부적절하다고 생각한다.즉, 말초 개체군이 분열하여 생식적으로 고립된다면, 다른 종으로 인식되기 위해서는 적어도 하나의 자기형질이 발달할 필요가 있을 것이다.만약 이것이 더 많은 어미 개체군이 새로운 자기 형질을 발달시키지 않고 일어날 수 있다면, 어미 개체군은 자기 형질 종 개념으로 남아있을 수 없다: 더 이상 딸 [8]종에 의해 공유되지 않는 동형성을 가질 수 없을 것이다.
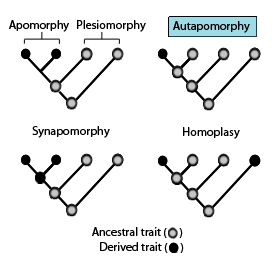
계통학적 유사성:이러한 계통발생학적 용어는 위의 도표에서 언급된 바와 같이 시나포메이션과 [1]관련하여 조상 및 파생된 특성 또는 특성 상태의 다른 패턴을 설명하기 위해 사용됩니다.
- 생물학적 체계학에서 동질체는 특성이 진화 과정에서 다른 계통에서 독립적으로 획득되거나 상실되는 것을 말한다.이 수렴된 진화는 종들이 독립적으로 그들의 공통 [9][10][11]조상에 존재했던 것으로 추측되는 특성과는 다른 특성을 공유하도록 이끈다.
- 아포몰피 – 파생된 특성.둘 이상의 분류군에 의해 공유되고 공통의 조상으로부터 유전되는 아포몰피는 시나포몰피이다.특정 분류군에 고유한 아포몰피는 자가형질이다.[14][15][16][17]
- Synapomorphy/Homology – 분지군의 일부 또는 모든 말단 그룹에서 발견되는 파생 특성으로, 공통 조상으로부터 유전되며, 그 조상으로부터는 자가형질(즉, 직계 조상에 존재하지 않음)이었다.
- 기초 시너포몰피 – 많은 시너포몰피에서 다시 상실된 시너포몰피.만약 하나를 제외하고 모두 사라진다면, 그것은 자가형성과 구별하기 어려울 수 있다.
- 자가형질 – 특정 분류군 또는 [3]그룹에 고유한 고유 파생 특성.
- Symplesiomorphy – 두 개 이상의 분류군이 공유하는 조상 특성.
- Plesiomorphy – 더 파생된 상태와 관련하여 논의된 심볼시오몰피.
- 가성질형 – 플리시오몰피 또는 [18]반전인 아포몰피로 식별될 수 없는 특성입니다.
- 반전 – 조상에 존재하는 파생 특성의 상실과 플라시모형 특성의 재정립이다.
- 컨버전스 – 2개 이상의 분류군에서 유사한 특성을 독립적으로 진화시킵니다.
- 헤미플라스시[19][20]
레퍼런스
- ^ a b Page RD, Holmes EC (14 July 2009). Molecular Evolution: A Phylogenetic Approach. John Wiley & Sons. ISBN 978-1-4443-1336-9. OCLC 609843839.
- ^ Futuyma DJ (1998). Evolutionary Biology (3rd ed.). Sinauer Associates, Inc. p. 95.
- ^ a b c Appel RD, Feytmans E (2009). "Chapter 3: Introduction of Phylogenetics and its Molecular Aspects". Bioinformatics: a Swiss Perspective (1st ed.). World Scientific Publishing Company.
- ^ Calow PP (2009). Encyclopedia of Ecology and Environmental Management. John Wiley & Sons. ISBN 978-1-4443-1324-6. OCLC 1039167559.
- ^ Forey PL (1997). History of the Coelacanth Fishes (1st ed.). Sprinter.
- ^ Howard DJ, Berlocher SH (1998). Endless Forms: Species and Speciation (1st ed.). USA: Oxford University Press. ISBN 978-0-19-510901-6. OCLC 60181901.
- ^ Bull AT (2004). Microbial Diversity and Bioprospecting. ASM Press.
- ^ Platnick NI (2001). "From Cladograms to Classifications: The Road to DePhylocode." (PDF). The Systematics Association.
- ^ Gauger A (April 17, 2012). "Similarity Happens! The Problem of Homoplasy". Evolution Today & Science News.
- ^ Sanderson MJ, Hufford L (21 October 1996). Homoplasy: The Recurrence of Similarity in Evolution. Elsevier. ISBN 978-0-08-053411-4. OCLC 173520205.
- ^ Brandley MC, Warren DL, Leaché AD, McGuire JA (April 2009). "Homoplasy and clade support". Systematic Biology. 58 (2): 184–98. doi:10.1093/sysbio/syp019. PMID 20525577.
- ^ Archie JW (September 1989). "Homoplasy Excess Ratios: New Indices for Measuring Levels of Homoplasy in Phylogenetic Systematics and a Critique of the Consistency Index". Systematic Zoology. 38 (3): 253–269. doi:10.2307/2992286. JSTOR 2992286.
- ^ Wake DB, Wake MH, Specht CD (February 2011). "Homoplasy: from detecting pattern to determining process and mechanism of evolution". Science. 331 (6020): 1032–5. Bibcode:2011Sci...331.1032W. doi:10.1126/science.1188545. PMID 21350170. S2CID 26845473.
- "Homoplasy: A good thread to pull to understand the evolutionary ball of yarn". ScienceDaily (Press release). February 25, 2011.
- ^ Simpson MG (9 August 2011). Plant Systematics. Elsevier. Amsterdam: Elsevier. ISBN 9780080514048.
- ^ Russell PJ, Hertz PE, McMillan B (2013). Biology: The Dynamic Science. Cengage Learning. ISBN 978-1-285-41534-5.
- ^ Lipscomb D (1998). "Basics of Cladistic Analysis" (PDF). Washington D.C.: George Washington University.
- ^ Choudhuri S (2014-05-09). Bioinformatics for Beginners: Genes, Genomes, Molecular Evolution, Databases and Analytical Tools (1st ed.). Academic Press. p. 51. ISBN 978-0-12-410471-6. OCLC 950546876.
- ^ Williams D, Schmitt M, Wheeler Q (2016-07-21). The Future of Phylogenetic Systematics: The Legacy of Willi Hennig. ISBN 978-1-107-11764-8. OCLC 951563305.
- ^ Avise JC, Robinson TJ (June 2008). "Hemiplasy: a new term in the lexicon of phylogenetics". Systematic Biology. 57 (3): 503–7. doi:10.1080/10635150802164587. PMID 18570042.
- ^ Copetti D, Búrquez A, Bustamante E, Charboneau JL, Childs KL, Eguiarte LE, et al. (November 2017). "Extensive gene tree discordance and hemiplasy shaped the genomes of North American columnar cacti". Proceedings of the National Academy of Sciences of the United States of America. 114 (45): 12003–12008. doi:10.1073/pnas.1706367114. PMC 5692538. PMID 29078296.