BLAT(생물정보학)
BLAT (bioinformatics)| 개발자 | Jim Kent, UCSC |
|---|---|
| 저장소 | |
| 유형 | 바이오 인포매틱스 툴 |
| 면허증. | 비상업적 사용, 상업적 사용, 소스 이용 가능 |
| 웹 사이트 | genome |
BLAT(BLAST-like alignment tool)는 2000년대 초 캘리포니아 대학 산타 크루즈(UCSC)의 Jim Kent가 인간 [1]게놈의 조립과 주석을 지원하기 위해 개발한 쌍별 배열 알고리즘이다.이것은 주로 인간 게놈 배열에 대해 수백만 개의 마우스 게놈 판독과 발현된 염기서열 태그를 맞추는 데 필요한 시간을 줄이기 위해 고안되었습니다.당시의 정렬 도구는 인간 게놈 조립체를 정기적으로 업데이트할 수 있는 방식으로 이러한 작업을 수행할 수 없었습니다.기존 툴에 비해 BLAT는 mRNA/DNA 정렬 수행 시 최대 500배, 단백질/[1]단백질 정렬 시 최대 50배 더 빨랐습니다.
개요
BLAT는 DNA, RNA 및 단백질과 같은 생물학적 배열의 분석과 비교를 위해 개발된 다중 알고리즘 중 하나이며, [2]게놈 배열의 생물학적 기능을 발견하기 위해 호몰로지를 추론하는 것이 주된 목표이다.기존의 니들맨-운쉬[3] 및 스미스-워터맨[4] 동적 프로그래밍 알고리즘처럼 두 시퀀스 간에 수학적으로 최적의 정렬을 찾는 것은 보장되지 않습니다. 대신, 먼저 동음이의 가능성이 더 높은 짧은 시퀀스를 빠르게 감지하고, 그 다음 동음이의 영역을 정렬하고 확장합니다.이것은 발견적[5][6] BLAST 계열의 알고리즘과 유사하지만, 각 툴은 서로 다른 알고리즘 [2][7]기술을 시도함으로써 생물학적 시퀀스를 시기적절하고 효율적으로 정렬하는 문제를 해결하려고 노력해왔다.
BLAT의 용도
BLAT는 단백질 및 번역된 뉴클레오티드(mRNA 또는 DNA) 염기서열뿐만 아니라 DNA 염기서열을 정렬하는 데 사용될 수 있다.유사성이 큰 시퀀스에서 가장 잘 작동하도록 설계되었습니다.DNA 검색은 영장류에게 가장 효과적이며 단백질 검색은 육지 [1][8]척추동물에게 효과적이다.또한 단백질 또는 번역된 배열 쿼리는 DNA 배열 [9]쿼리보다 원거리 일치 식별 및 이종 간 분석에 더 효과적입니다.BLAT의 일반적인 용도는 다음과 같습니다.
- 게놈 [10]좌표를 추론하기 위해 게놈 조립체에 대한 다중 mRNA 배열 정렬
- 호몰로지를 결정하기 위해 한 종의 단백질 또는 mRNA 배열을 다른 종의 배열 데이터베이스로 정렬하는 것.두 종이 너무 발산되지 않는 한, 이종 간 정렬은 일반적으로 BLAT에 효과적이다.이는 BLAT가 완벽한 일치를 요구하지 않고 오히려 [11]정렬의 불일치를 받아들이기 때문에 가능하다.
- BLAT는 두 단백질 배열의 정렬에 사용될 수 있다.그러나 이러한 유형의 정렬에는 이 도구를 사용할 수 없습니다.표준 단백질 BLAST 도구인 BLASTP는 단백질-단백질 [1]정렬에서 더 효율적입니다.
- 유전자의 [9][10]외부 및 내부 영역의 분포 결정
- 특정 유전자 [9][10]쿼리의 유전자 패밀리 구성원의 검출
- 특정 [9][10]유전자의 단백질 코드 배열 표시.
BLAT는 95% 이상의 뉴클레오티드 동일성 또는 80% 이상의 번역 단백질 [9][10]동일성을 공유하는 최소 40개의 염기서열 간의 일치를 찾도록 설계되었다.
과정
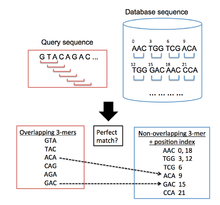
BLAT는 대상 게놈 데이터베이스에서 조사 중인 쿼리 시퀀스와 유사한 영역을 찾기 위해 사용됩니다.BLAT에 이은 일반적인 알고리즘 프로세스는 BLAST와 유사합니다.BLAST는 먼저 데이터베이스에서 짧은 세그먼트를 검색하고 일정 수의 일치하는 요소를 가진 쿼리 시퀀스를 검색합니다.이러한 정렬 시드는 높은 점수 [12]쌍을 형성하기 위해 시퀀스의 양방향으로 확장됩니다.그러나, BLAT는 BLAST와 다른 색인 접근법을 사용하며, 이를 통해 매우 큰 게놈 및 단백질 데이터베이스를 신속하게 스캔하여 쿼리 시퀀스와 유사성을 확인할 수 있다.이를 위해 타깃 데이터베이스의 인덱스리스트(해시 테이블)를 메모리에 저장함으로써 쿼리 시퀀스와 타깃 데이터베이스를 비교하는 데 필요한 시간을 대폭 단축합니다.이 인덱스는 매우 반복적인 k-mer를 제외하고 타깃 데이터베이스에서 겹치지 않는 모든 k-mer(k글자가 있는 단어)의 좌표를 취함으로써 구축됩니다.그런 다음 BLAT는 쿼리 시퀀스에서 중복되는 모든 k-mer 목록을 작성하고 대상 데이터베이스에서 이를 검색하여[1] 시퀀스 간에 일치하는 히트 목록을 구축한다(그림 1은 이 프로세스를 보여준다).
검색 단계
후보 호몰로지 영역을 검색하기 위해 사용되는 전략은 세 가지가 있습니다.
- 첫 번째 방법에서는 쿼리와 데이터베이스 시퀀스 간에 하나의 완벽한 일치가 필요합니다. 즉, 두 개의 k-mer 단어가 완전히 동일합니다.이 접근방식은 가장 실용적이라고 생각되지 않습니다.이는 높은 수준의 감도를 달성하려면 작은 k-mer 크기가 필요하지만, 이는 잘못된 양의 적중 횟수를 증가시켜 [1]알고리즘의 정렬 단계에서 소요되는 시간을 증가시키기 때문이다.
- 두 번째 방법은 두 개의 k-mer 단어 간에 적어도 하나의 불일치를 허용합니다.이는 잘못된 양의 양을 감소시켜 이전 방법에서 생성된 것보다 계산 비용이 덜 드는 더 큰 k-mer 크기를 처리할 수 있게 한다.이 방법은 작은 상동 [1]영역을 식별하는 데 매우 효과적입니다.
- 세 번째 방법에서는 서로 근접한 여러 개의 완벽한 매치가 필요합니다.Kent가 [1]보여주듯이, 이것은 상동 영역 내의 작은 삽입과 결실을 고려할 수 있는 매우 효과적인 기술이다.
뉴클레오티드를 정렬할 때, BLAT는 크기 11(11-mers)의 두 개의 완벽한 단어 일치를 요구하는 세 번째 방법을 사용합니다.단백질을 정렬할 때 BLAT 버전은 사용되는 검색 방법을 결정합니다. 클라이언트/서버 버전이 사용될 경우 BLAT 버전은 3개의 완벽한 4-mer를 검색하며, 독립 실행형 버전이 사용될 경우 BLAT 버전은 쿼리와 데이터베이스 [1]시퀀스 사이에서 하나의 완벽한 5-mer를 검색합니다.
블래트 vs. 블라스트
BLAT와 BLAST의 차이점 중 일부는 다음과 같다.
- BLAT는 게놈/단백질 데이터베이스를 인덱싱하고, 이 인덱스를 메모리에 유지한 다음 일치하는 쿼리 시퀀스를 스캔합니다.한편 BLAST는 쿼리 시퀀스의 인덱스를 작성하고 데이터베이스를 검색하여 [1]일치하는 항목을 찾습니다.MegaBLAST라는 BLAST 변형은 [9]정렬 속도를 높이기 위해 4 데이터베이스를 인덱싱합니다.
- BLAT는 여러 개의 완벽한 일치와 거의 완벽한 일치(기본값은 뉴클레오티드 검색을 위한 길이 11의 2개의 완벽한 일치와 단백질 검색을 위한 길이 4의 3개의 완벽한 일치)에서 확장될 수 있는 반면,[1][9] BLAST는 하나 또는 두 개의 일치하는 경우에만 확장됩니다.
- BLAT는 각 상동 영역을 별도의 국소 정렬로 반환하는 BLAST와 대조적으로 두 시퀀스 사이의 상동 영역을 하나의 큰 정렬로 연결합니다.BLAST의 결과는 각 정렬이 엑손의 끝을 지나 확장되는 엑손의 목록입니다.그러나 BLAT는 각 염기를 단 한 번만 사용하여 mRNA의 각 염기를 게놈에 정확하게 배치하고, 인트론-엑손 경계(즉, 스플라이스 부위)[1][13]를 식별하는 데 사용할 수 있다.
- BLAT는 [2]BLAST보다 덜 민감하다.
프로그램 사용방법
BLAT는 웹 기반 서버 클라이언트 프로그램 또는 독립 실행형 [9]프로그램으로 사용할 수 있습니다.
서버 클라이언트
BLAT의 웹 기반 애플리케이션은 UCSC Genome Biorinformatics [8]사이트에서 액세스할 수 있습니다.인덱스 작성은 비교적 느린 절차입니다.따라서 웹 기반 BLAT에 의해 사용되는 각 게놈 어셈블리는 BLAT 서버와 관련지어져 사전 계산된 지수를 정렬에 사용할 수 있다.이러한 웹 기반 BLAT 서버는 사용자가 쿼리 [11]시퀀스를 입력할 수 있도록 인덱스를 메모리에 보관합니다.
쿼리 시퀀스가 업로드/검색 필드에 붙여넣기되면 사용자는 타겟팅할 종의 게놈(현재 50종 이상이 사용 가능) 및 해당 게놈의 어셈블리 버전(예를 들어 인간 게놈은 선택할 수 있는 4개의 어셈블리가 있음), 쿼리 유형(즉, 시퀀스 관련 여부)과 같은 다양한 파라미터를 선택할 수 있습니다.DNA, 단백질 등) 및 출력 설정(즉, 출력을 정렬하고 시각화하는 방법)에 대해 설명합니다.사용자는 쿼리를 제출하거나 BLAT "I'm feeling lucky"[8] 검색을 사용하여 검색을 실행할 수 있습니다.
Bhagwat [9]등은 BLAT를 사용하여 다음을 수행하는 방법에 대한 단계별 프로토콜을 제공한다.
- mRNA/cDNA 염기서열을 게놈 염기서열에 매핑한다.
- 단백질 배열을 게놈에 매핑합니다.
- 호몰로지 검색을 수행합니다.
입력
BLAT는 긴 데이터베이스 시퀀스를 처리할 수 있지만 긴 쿼리 시퀀스보다 짧은 쿼리 시퀀스가 효과적입니다.Kent는[1] 최대 쿼리 길이를 200,000 base로 권장합니다.UCSC 브라우저는 DNA 검색의 경우 쿼리 시퀀스를 25,000자(뉴클레오티드) 미만으로 제한하고 단백질 및 번역된 시퀀스 [8]검색의 경우 10,000자(아미노산) 미만으로 제한한다.
UCSC 웹사이트에서 사용할 수 있는 BLAT 검색 게놈은 쿼리 시퀀스를 텍스트(쿼리 상자에 잘라 붙여넣기) 또는 텍스트 파일로 업로드할 수 있습니다.BLAT 검색 게놈은 한 번에 동일한 유형의 여러 시퀀스를 최대 25개까지 수용할 수 있습니다.다중 배열의 경우, 총 뉴클레오티드의 수는 DNA 검색을 위해 50,000개를 초과해서는 안 되며 단백질 또는 번역된 배열 검색을 위해 25,000개를 초과해서는 안 됩니다.DNA 쿼리 시퀀스를 사용하여 타깃 데이터베이스를 검색하는 예를 그림 2에 나타냅니다.
산출량
BLAT 검색은 점수를 기준으로 내림차순으로 정렬된 결과 목록을 반환합니다.다음 정보가 반환됩니다. 정렬 점수, 데이터베이스 시퀀스와 일치하는 쿼리 시퀀스의 영역, 쿼리 시퀀스의 크기, 정렬 비율로서의 정체성 수준 및 쿼리 시퀀스가 매핑되는 [9]염색체와 위치.Bhagwat [9]등은 BLAT의 "점수"와 "정체성" 측정이 어떻게 계산되는지를 설명한다.
각 검색 결과에 대해 UCSC Genome Browser(UCSC 게놈 브라우저)에 대한 링크가 제공되므로 사용자는 염색체 상의 정렬을 시각화할 수 있습니다.이것은 독립형 BLAT에 비해 웹 기반 BLAT의 주요 이점입니다.사용자는 쿼리가 [9]일치하는 유전자에 대한 정보 등 정렬과 관련된 생물학적 정보를 얻을 수 있습니다.사용자는 또한 게놈 어셈블리와 쿼리 시퀀스의 정렬을 볼 수 있는 링크도 제공됩니다.쿼리와 게놈 어셈블리의 일치는 파란색이며 정렬의 경계는 더 옅은 색입니다.이러한 엑손 경계는 스플라이스 [8][9]부위를 나타냅니다."[8]I'm feeling lucky" 검색 결과는 사용자가 선택한 출력 정렬 옵션에 따라 첫 번째 쿼리 시퀀스의 최고 점수 정렬을 반환합니다.
스탠드아론
독립형 BLAT는 웹 기반 BLAT보다 배치 실행에 더 적합하고 효율적입니다.지표를 [1][9]메모리에만 저장하는 웹 기반 애플리케이션과 달리 게놈을 메모리에 저장할 수 있어 효율적이다.
면허증.
BLAT의 소스 바이너리와 사전 컴파일 바이너리 모두 학술적 및 개인적 용도로 자유롭게 사용할 수 있다.독립형 BLAT의 상용 라이선스는 Kent Informatics, Inc.에 의해 배포됩니다.
「 」를 참조해 주세요.
- BLAST 기본 로컬 정렬 검색 도구
- 시퀀스 얼라인먼트 소프트웨어
레퍼런스
- ^ a b c d e f g h i j k l m n Kent, W James (2002). "BLAT--the BLAST-like alignment tool". Genome Research. 12 (4): 656–664. doi:10.1101/gr.229202. PMC 187518. PMID 11932250.
- ^ a b c Imelfort, Michael (2009). Edwards, D; Stajich, J; Hansen, D (eds.). Bioinformatics: Tools and Applications. New York: Springer. pp. 19–20. ISBN 978-0-387-92737-4.
- ^ Needleman, SB; Wunsch, CD (1970). "A general method applicable to the search for similarities in the amino acid sequence of two proteins". Journal of Molecular Biology. 48 (3): 443–53. doi:10.1016/0022-2836(70)90057-4. PMID 5420325.
- ^ Smith, TF; Waterman, MS (1981). "Identification of common molecular subsequences". Journal of Molecular Biology. 147 (1): 195–7. CiteSeerX 10.1.1.63.2897. doi:10.1016/0022-2836(81)90087-5. PMID 7265238.
- ^ Altschul, SF; Gish, W; Miller, W; Myers, EW; Lipman, DJ (1990). "Basic local alignment search tool". Journal of Molecular Biology. 215 (3): 403–10. doi:10.1016/S0022-2836(05)80360-2. PMID 2231712.
- ^ Altschul, SF; Madden, TL; Schäffer, AA; Zhang, J; Zhang, Z; Miller, W; Lipman, DJ (1997). "Gapped BLAST and PSI-BLAST: a new generation of protein database search programs". Nucleic Acids Research. 25 (17): 3389–402. doi:10.1093/nar/25.17.3389. PMC 146917. PMID 9254694.
- ^ Baxevanis, Andreas D.; Ouellette, B.F. Francis (2001). Bioinformatics: A Practical Guide to the Analysis of Genes and Proteins (2nd ed.). New York: Wiley-Interscience. pp. 187–214. ISBN 978-0-471-22392-4.
- ^ a b c d e f g UCSC 게놈 생물정보학 사이트
- ^ a b c d e f g h i j k l m n Bhagwat, Medha; Young, Lynn; Robison, Rex R (March 2012). Using BLAT to find sequence similarity in closely related genomes. Current Protocols in Bioinformatics. 10.8. Vol. 10. pp. Unit10.8. doi:10.1002/0471250953.bi1008s37. ISBN 978-0-471-25095-1. PMC 4101998. PMID 22389010.
- ^ a b c d e Ye, Shui Qing (2008). Bioinformatics: A Practical Approach. London: Chapman & Hall. pp. 11–12. ISBN 978-1-58488-810-9.
- ^ a b Kuhn, RM; Haussler, D; Kent, WJ (2013). "The UCSC genome browser and associated tools". Briefings in Bioinformatics. 14 (2): 144–61. doi:10.1093/bib/bbs038. PMC 3603215. PMID 22908213.
- ^ Lobo, Ingrid. "Basic Local Alignment Search Tool (BLAST)". Nature Education. Retrieved 15 October 2013.
- ^ Pevsner, J (2009). Bioinformatics and Functional Genomics. New Jersey: John Wiley & Sons, Inc. pp. 166–167. ISBN 978-0-470-08585-1.
- ^ "NCBI – GenBank: AACZ03015565.1". Retrieved 12 October 2013.