생물 몬테카를로 방법
Biology Monte Carlo method생물학 몬테카를로 방법(BioMOCA)은 일리노이 대학교 어바나 샴페인의 일리노이 대학에서 이온 채널이나 막에 내장된 나노 포자를 통한 전해질 환경에서의 이온 이동을 시뮬레이션하기 위해 개발되었다.[1]습식/생물학적 환경에서 이온 채널 시스템이나 이와 유사한 나노포어의 이온 전달 문제를 분석하고 연구하기 위한 3-D 입자 기반 몬테카를로 시뮬레이터다.시뮬레이션된 시스템은 이온 채널을 형성하는 단백질(또는 탄소 나노 튜브, CNT와 같은 인공 나노포어)과 양쪽에 두 개의 이온 욕조를 분리하는 막(즉, 지질 빌레이어)으로 구성된다.바이오모카는 볼츠만 운송수송 몬테카를로([2]BTMC)와 입자-입자-입자-입자-입자-입자-입자-입자-mesh(PM3) 등 두 가지 방법을 기반으로 하고 있으며,[3] 첫 번째 방법은 몬테카를로(Monte Carlo) 방식을 사용하여 볼츠만 방정식을 푸는 반면, 이후는 정전력을 단거리와 장거리 부품으로 분리한다.
배경
이온 채널의 완전 원자 분자 역학 시뮬레이션에서 계산비용의 대부분은 시스템 내 물 분자의 궤적을 따라가기 위한 것이다.그러나, 바이오모카에서 물은 연속 유전적 배경 매체로 취급된다.그 외에도, 이온 채널의 단백질 원자는 주어진 유전계수를 가진 유한 볼륨에 내장된 정적 점 전하를 모델로 한다.이온이 접근할 수 없는 정적 유전체 영역으로 취급되는 지질막도 마찬가지다.사실 이 시스템의 유일한 정전기 입자는 이온이다.그들의 움직임은 정전기적 상호작용과 쌍방향으로 레너드 존스의 전위를 통해 다른 이온들과 상호작용하면서 고전적인 것으로 가정된다.그들은 또한 물 배경 매체와 상호작용하는데, 이것은 산란 메커니즘을 사용하여 모델링된다.
시뮬레이션 영역에 있는 이온의 앙상블은 2차 정확한 도약구도를 사용하여 동작 방정식을 통합하여 시간과 3-D 공간에서 동시에 전파된다.이온 위치 r과 힘 F는 시간 단계 t와 t + dt에서 정의된다.이온 속도는 t – dt/2, t + dt/2에서 정의된다.운동의 지배적인 유한차 방정식은
여기서 F는 정전력과 쌍방향 이온 상호작용력의 합이다.
정전기장용액
정전기 전위는 포아송 방정식을 풀어서 일정한 시간 간격으로 계산한다.
여기서 , t) ) _{\은 각각 단백질에 대한 이온의 전하 밀도 및 영구 전하량이다.( ) 은 국부 유전체 상수 또는 허용률이고, ( r, ) 은 국부 정전기 전위이다.이 방정식을 해결하면 유전체 경계에서 유도된 영상 전하 효과와 적용된 바이어스를 포함할 수 있는 자기 일관적인 방법을 제공한다.
단백질 잔류물의 이온 및 부분 전하를 CIC(Cloud-in-cell) 방식을 사용하여 유한 직사각형 그리드에 할당한다.[3]그리드 상의 포아송 방정식의 해결은 PM3 체계의 미립자 메쉬 성분에 대한 카운트다.그러나 이러한 탈소화는 정전력의 단거리 구성요소가 불가피하게 잘리게 되는데, 단거리 충전-쿨롬빅 상호작용을 계산하여 교정할 수 있다.
유전계수
단백질, 멤브레인, 수용성 부위의 유전 허용성에 적절한 값을 할당하는 것은 매우 중요하다.유전체 계수는 서로 다른 허용률을 가진 두 영역 사이의 경계에 접근하는 이온에 대한 전하 입자 사이의 상호작용의 강도 및 유전체(DBF)의 강도를 결정한다.그러나 나노 단위에서 특정 허용률을 할당하는 작업은 문제가 있고 간단하지 않다.
단백질이나 멤브레인 환경은 여러 가지 다른 방법으로 외부 장에 반응할 수 있다.[1][4][5][6][7]현장 유도 쌍극자, 영구 쌍극자의 방향 전환, 단백질 잔류물의 양성 및 감응, 단백질 내부와 표면에 있는 이온화된 측면 체인과 물 분자의 대규모 재구성은 모두 허용성의 배정이 얼마나 복잡한지를 보여주는 예들이다.모든 전하, 쌍극자 및 현장 유도 원자 쌍극자를 명시적으로 취급하는 MD 시뮬레이션에서는 유전체 값 1이 적절하다고 제안한다.그러나 단백질, 막, 물이 연속 배경이고 암묵적으로 처리되는 우리 같은 환원 입자 이온 시뮬레이션 프로그램에서는 이온 운동이 그 존재에 대한 단백질의 반응과 같은 시간 척도로 일어나므로 유전계수를 할당하기가 매우 어렵다.실제로 유전계수를 변경하면 이온투과 선택성 등 채널특성을 쉽게 바꿀 수 있다.물에 대한 유전계수 할당도 또 다른 핵심 쟁점이다.이온 채널 내부의 물 분자는 고충전 잔류물 또는 물 분자와 단백질 사이의 수소 결합 형성이 종종 줄지어 있는 모공의 테이퍼형 크기 때문에 매우 질서 정연할 수 있었다.[8]결과적으로, 이온 채널 내부의 물의 유전 상수는 벌크 조건에서의 값과 상당히 다를 수 있다.문제를 더욱 복잡하게 만들기 위해 나노포어 내부의 물의 유전계수는 반드시 등방성 스칼라 값이 아니라 서로 다른 방향으로 다른 값을 갖는 등방성 텐서이다.
비등방성 허용률
시스템의 거시적 특성이 반드시 분자 길이 척도로 확장되는 것은 아니라는 것이 명백해졌다.그들은 일리노이 대학교의 Reza Toghrae, R. Jay Mashl, Eric Jakobsson이 최근 수행한 연구에서 직경 1 ~ 12 nm의 특징 없는 소수성 실린더의 물의 특성을 연구하기 위해 Molecular Dynamics 시뮬레이션을 이용했다.[4]이 연구는 관 지름이 변화함에 따라 물이 구조, 유전적 특성, 이동성에서 뚜렷한 변화를 겪는다는 것을 보여주었다.특히 그들은 1~10nm 범위의 유전체 성질이 벌크수와 상당히 다르며, 실제로 자연에서는 비등방성이라는 것을 발견했다.그러나 이러한 특징 없는 소수성 채널은 실제 이온 채널을 나타내지 않으며, 이온 채널에 그러한 데이터를 사용할 수 있기 전에 이 영역에서 더 많은 연구가 이루어져야 한다. 이온 채널이나 나노 포어 내부의 허용률과 같은 물 특성이 이전에 생각했던 것보다 훨씬 더 복잡할 수 있다는 것은 명백하다.높은 축방향 유전체 상수는 (채널을 따라) 축방향으로 이온의 정전기 전하를 보호하지만, 낮은 방사형 유전체 상수는 이동 이온과 부분 전하 사이의 상호작용을 증가시키거나 채널의 유전체 전하 이미지를 증가시켜 이온 채널에서 더 강한 선택성을 전달한다.
비등방성 허용성에 기반한 포아송 방정식을 해결하는 것은 아래에서 간략하게 설명한 박스 통합 [9]탈소화 방법을 이용하여 바이오 MOCA에 통합되었다.
계산
박스 통합 디스트리뷰트
D-차원 포아송 방정식의 분산을 위해 상자 통합을 사용하는 방법
D × D 텐서인 을(를) 사용하여 이 미분 방정식을 적분 방정식으로 재구성한다.D차원 영역 에대한 위의 방정식을 통합하고 Gauss 정리를 사용하여 적분 제형을 구한다.
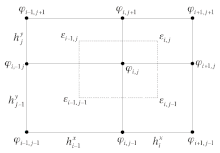
이 부록에서는 2차원 사례로 가정한다.가우스 정리가 1차원과 3차원에 대해서도 유효하기 때문에 3차원 시스템으로 업그레이드하는 것은 간단하고 합법적일 것이다. 사이의 직사각형 영역에 이(가) 주어지는 것으로 가정하고, 노드에는 이(가) 정의된다(오른쪽 그림 참조).
그런 다음 통합 영역 을(를) 노드를 중심으로 4개의 가장 가까운 인접 노드로 확장되는 사각형으로 선택한다.그라데이션 {\은(는) 통합 표면 Ω{\ 평균 {\의 경계에 대한 중심 차이를 사용하여 근사치를 구한다. 이 접근방법으로 우리는 대략적으로 알 수 있다.e 위의 포아송 방정식의 좌측은 다음과 같이 첫 번째 순서로 배열한다.
여기서 와 y 는 텐서 의 대각선의 두 성분이다 포아송 방정식의 오른쪽을 분해하는 것은 상당히 간단하다. 은(는) 에 대해 수행되었듯이 동일한 그리드 노드에서 디스코트된다.
이온 크기
이온의 유한 크기는 6–12 레나드-존스 전위로부터 파생된 쌍방향 반발력을 사용하여 바이오모카에서 설명된다.레너드 존스 전위성의 잘린 형태는 이온적인 코어 거부반응을 모방하기 위해 시뮬레이터에서 사용된다.혐오성분만을 보존하는 레나드 존스의 쌍방향 전위성의 변형된 형태는 다음과 같다.
Here, is the Lennard-Jones energy parameter and is the average of the individual Lennard-Jones distance parameters for particles i and j.잘린 형태의 전위를 사용하는 것은 계산적으로 효율적이며, 이온이 겹치거나 결합하는 것을 방지하는데, 이것은 분명히 물리적인 것이 아닐 것이다.
이온-단백질 상호작용
완전한 분자 구조의 고해상도 X선 결정학적 측정의 가용성은 단백질을 형성하는 모든 원자의 유형과 위치에 대한 정보를 제공한다.BioMOCA에서 단백질 원자는 이온에 접근할 수 없는 유한 볼륨에 내장된 정적 점 전하를 모델로 하며 사용자 정의 유전계수와 연관된다.게다가, 서로 다른 아미노산 그룹에 있는 원자의 전하와 반지름에 대한 정보를 제공하는 많은 힘-장 매개변수가 이용 가능하다.분자 구조와 힘 장의 결합은 단백질 채널에 있는 각 원자의 좌표, 반지름 및 전하를 제공한다.바이오모카(BioMOCA)는 이러한 정보를 표준 PQR(Position-Charge-Radius) 형식으로 사용하여 직사각형 그리드에 단백질 시스템을 매핑한다.
이상적으로는 수용성 매체에 있는 단백질 원자와 이온 사이의 강직 상호작용이 레너드 존스와 같은 역진 전위를 사용하여 이온이 단백질을 침투하는 것을 방지하는 것이다.이 접근방식은 계산량에 상당한 하중을 가할 수 있으므로 단백질 표면을 미리 결정된 단단한 벽 경계로 취급하는 간단한 접근법을 선택한다.최근의 많은 오픈소스 분자생물학 패키지에는 단백질 시스템에서 이온이 접근할 수 있는 부피를 결정하는 시설이 내장되어 있다.Adaptive Poisson Boltzmann Solver(APBS) 체계는[10] 접근 가능한 볼륨 영역을 얻기 위해 BioMOCA에 통합되었으며, 따라서 시뮬레이션 영역을 연속 영역으로 분할한다.
이온은 단백질과 지질 부위에 접근할 수 있는 것으로 간주되며, 이온 구의 유한 크기 내의 어떤 지점이 단백질이나 막 경계를 통과할 경우 충돌을 가정하고 이온을 확산적으로 반사한다.
이온-물 상호작용
감소된 입자 접근방식으로 바이오모카(BioMOCA)는 명시적 물 분자를 연속적 배경으로 대체하고 적절한 산란율을 선택해야 하는 BTMC 방법을 사용하여 이온-물 상호작용을 처리한다.즉, 이온 궤도는 물에서 이온의 확산 운동을 설명하는 산란 이벤트에 의해 무작위로 중단된다.[1]이러한 산란 현상 사이에 이온은 뉴턴의 힘을 따른다.자유 비행 시간 T는f 다음과 같이 총 산란 속도에서 통계적으로 생성된다.
여기서 r은 단위 간격에 균일하게 분포된 임의의 숫자다.모멘텀의 인은(는) 모든 충돌 메커니즘에 대한 총 산란율이다.각 자유 비행의 끝에서, 이온의 속도는 맥스웰리안 분포로부터 무작위로 재선택된다.비불크 전해액 용액에서 이온-수 상호작용에 대한 정확한 산란 메커니즘이 아직 개발되지 않았기 때문에, 국부적 확산성과 연결된 위치 의존적 산란율이 우리 모델에서 사용된다.이러한 위치에 대한 의존은 물 분자가 지역마다 다른 조직 질서를 가질 수 있다는 사실에서 비롯되며, 이는 산란율에 영향을 미칠 것이다.
위치 의존적 확산도
이온과 물 분자는 밀폐된 지역에서 대량과 같은 이동성 또는 확산성을 가지지 않는다는 것이 널리 받아들여지고 있다.[2][6]사실, 이온 채널에서 이온의 효과적인 이동성이 감소할 가능성이 더 높다.[5]채널수가 암묵적 연속체 배경으로 가정되는 감소된 입자 방법에서는 국소 정전력과 무작위 사건으로 인해 이온이 어떻게 확산될 수 있는지를 밝혀내기 위해 평균 이온 이동성이 필요하다.Transport Monte Carlo 시뮬레이션에서, 총 산란율( )은 이온-물 상호작용에 의해서만 발생하는 것으로 가정된다. 이는 표현과 이온 확산성과 관련이 있다.
여기서 m은 이온의 질량이고 D는 확산 상수다.방정식이 나타내듯이 채널의 루멘 내 이온의 확산 감소는 산란 현상의 발생률을 증가시킨다.
수화 껍질
물 분자는 극성 때문에 이온 이동에 있어 확산 효과가 있을 뿐만 아니라 개별 이온 주위에 수화 껍데기를 형성하기도 한다.수화 껍질은 다른 이온에서 이온에 대한 전하를 차단할 뿐만 아니라 이온 방사상 분포 기능을 조절하여 피크와 수조 형성을 유발한다.두 이온 사이의 평균 최소 거리는 두 이온이 너무 가까이 접근하는 것을 방지하는 물리적 억제제 역할을 하면서 두 이온 사이에 항상 적어도 한 층의 물 분자가 존재하기 때문에 증가하는데, 이는 레너드 존스 전위의 단거리 반발 성분과 유사한 방식이다.
수화 껍데기 이론은 물리 화학 문헌에서 잘 발달되어 있지만, 가능한 적은 연산 오버헤드로 본질적인 효과를 포착할 수 있는 간단한 모델이 필요하다.이러한 목적을 위해 임과 루크가[11] 논의한 동일한 쌍의 잠재력을 구현하여 수화 껍질의 효과를 포함시킨다.
계수 c는i 평형 몬테카를로 시뮬레이션에 대비하여 이온 방사형 분포 함수를 벤치마킹하기 위해 MD 시뮬레이션을 사용하여 1 M KCl 솔루션에 대해 경험적으로 결정되었다.수화 껍질의 효과는 전해질 욕조의 염분 농도가 더욱 높아짐에 따라 그 중에서도 포린인 많은 이온 채널의 전도성이 포화되는 것으로 관측되는 높은 염분 농도의 시뮬레이션에서 중요한 것으로 밝혀졌다.수화 쉘 모델을 포함하지 않은 초기 시뮬레이션은 전도도 포화 동작을 재현하지 않았다.이는 이온 혼잡을 방지하기 위해 작용하는 추가적인 반발성 전위를 시사하며, 따라서 높은 욕실 염분 농도에서도 모공 밀도의 이온 농도와 전류 밀도를 제한한다.반발 전위가 포함되었을 때 적당한 채널 전도성이 관찰되었다.
조건 및 방법
경계 조건
이온 채널의 전기적 및 생리학적 특성은 특정 농도의 용액을 포함하는 욕조 2개를 분리하는 지질 막에 채널을 삽입하여 실험적으로 측정한다.두 개의 욕조에 전극을 담가 채널 전체에 일정한 정전기 바이어스를 가한다.이러한 접촉 영역을 정확하게 나타내는 경계 조건을 형성하는 것은 엄청나게 큰 욕조 영역을 필요로 할 수 있으며 어려운 작업이다.막으로부터의 데비 길이 이상의 정전기 전위와 이온 밀도는 눈에 띄게 달라지지 않는다.이러한 가정은 앞에서 제시된 연속체 결과의 결과로 뒷받침되었다.[12]이온 채널 시뮬레이션에 사용되는 일반적인 염분 농도의 경우 데비예 길이는 10 å이다.이 가정을 이용하여 디리클레 경계 조건은 이 평면이 막에서 충분히 멀리 떨어져 있는지 주의하면서 채널에 횡방향인 두 개의 도메인 경계면에서 전위에 부과된다.
실험 조건을 복제하는 또 다른 문제는 두 욕조에서 고정된 전하 밀도를 유지하는 문제다.이 문제는 경계면에서 막 쪽으로 확장되는 두 개의 완충 영역에서 지정된 밀도를 유지함으로써 처리된다.두 개의 버퍼 영역에서 밀도를 유지하는 데 필요한 이온 수는 시뮬레이션 시작 시 계산된다.이러한 버퍼의 이온 수는 시뮬레이션 전체에 걸쳐 샘플링되며, 적자가 관찰될 때마다 이온을 주입한다.주입된 입자의 초기 속도는 Maxwellian 분포에 따라 결정된다.이온은 두 개의 디리클레 경계면을 통해서만 시스템을 떠날 수 있으며, 이온은 이러한 완충 지역에서 인위적으로 제거되지 않는다.노이만 경계면에서 반사된 부분은 탄성 반사로 처리된다.
다중그리드 및 그리드 포커싱 방법
이온 채널의 시뮬레이션에서 대부분의 방법에서 주요 계산 비용은 이온에 작용하는 정전력의 계산에서 나온다.예를 들어 명시적 이온보다는 이온 밀도가 존재하는 연속형 모델에서는 포아송 방정식을 풀어서 정전기 전위를 자가 정합성 있게 계산한다.반면에 MD 시뮬레이션에서는 입자에 작용하는 정전기력은 쿨롱 힘 용어의 명시적 평가에 의해 계산되며, 단거리 정전기력과 장거리 정전기력을 분할하여 다른 방법으로 계산할 수 있다.감소된 입자법과 같은 모델에서는 포아송 방정식을 풀고 단거리 구성요소로 얻은 힘을 증가시켜 장거리 정전력을 평가한다.포아송 방정식을 풀면 MD 시뮬레이션에서 다루기 어려운 문제인 반면, 시스템에 대한 편향에서 발생하는 힘을 자기 일관성 있게 포함할 수 있다.
현재 바이오MOCA에는 유한차분법에 근거한 2개의 포아송 솔버가 구현되어 있다.하나는 사전 조건화된 결합구배 방식(pCG)을 사용하며, 기본적으로 사용된다.후자는 V-멀티-그리드 체계를 사용하는 APBS 해결사로부터 차용된다.포아송 방정식을 풀기 위한 수치적 접근법 외에, 두 해결사 사이의 주요 차이는 그들이 시스템의 허용률을 어떻게 다루는지에 관한 것이다.첫 번째 솔버에서는 유전체 값이 그리드의 각 셀에 할당되며, APBS 솔버에서는 유전체 계수가 그리드 노드에 정의된다.앞에서 논의한 바와 같이, 포아송 방정식을 가장 정확한 방법으로 취급하는 PCG 솔버에는 박스 통합 방법이 사용된다.박스 적분법에 기초한 풀 멀티그리드 솔버가 개발되고 있지만, 이미 종료된 코드를 재사용하고 이온 채널 시스템을 처리할 수 있는 깔끔한 방법이 있다.
이온 채널 시뮬레이션은 선별의 정확한 처리를 위해 대형 욕조 부위가 있어야 한다.[1]그러한 욕조 영역의 존재는 포아송 방정식의 메쉬 영역을 크게 만들고 미세한 메시 분해능을 가진 많은 수의 격자점 또는 매우 거친 탈색성을 가진 소수의 격자점을 유도한다.대량 시뮬레이션에서 거친 망사는 PM3 방식을 사용하여 욕조를 설명하기에 충분하다.단, 채널 영역에서는 이러한 영역의 높은 충전 특성과 공간적으로 다양한 유전체 영역이 존재하기 때문에 미세한 분해능이 요구된다.궁극적인 관심은 이온 투과성, 선택성, 탕도, 밀도 등의 측면에서 채널 동작을 연구하는 것이다.즉, 채널 영역에 더 많은 계산 리소스를 배치하고, 대신 전체 계산 비용과 시뮬레이션 속도를 몇 주에서 며칠로 줄이기 위해 욕조에 최소량을 두는 것이 좋다.대규모 욕조 구역의 요건과 채널의 미세한 격자 분해능을 동시에 연산적으로 효과적인 방법으로 충족시킬 수 있는 그리드 포커싱 방법에 기초한 계획이 개발되었다.이 방법론은 다중 미세 메시 영역을 가질 수 있는데, 이는 동일한 욕조 영역을 공유하거나 니코틴 수용체 채널과 같이 이온 통로가 좁은 비교적 큰 채널에 대해 미세한 망사 내부에 미세한 메쉬를 포함시키는 데 필요할 수 있다.[13]
첫 번째 그리드는 욕조 영역과 채널 영역을 포함한 전체 문제 영역에 걸쳐 있는 굵은 메시이다.두 번째 그리드(다른 그리드에 대해서도, 3번째, 4번째 등)는 채널 모공과 같이 미세한 분해능이 요구되는 영역을 포함하는 시스템의 하위 도메인에 걸쳐 있는 상대적으로 훨씬 미세한 망사이다.포아송 방정식은 우선 적용된 편향을 고려하여 모든 디리클레와 노이만 경계 조건과 함께 굵은 그물망 위에서 해결된다.다음으로 2차 메시에 대한 경계 조건은 포아송 방정식의 첫 번째 또는 이전 해법에서 보간하여 얻는다.포아송 방정식은 새로운 경계 조건을 사용하여 보다 미세한 메시에 대해 다시 해결된다.이렇게 하면 지역별로 메쉬 디크리트화가 다른 정전기장을 생성할 수 있다.
EMF 및 DBF
전기력(EMF)은 이온과 같이 충전된 입자가 막에 내장된 이온 채널을 가로지르는 데 필요한 에너지를 측정하는 것이다.이 잠재적 에너지 장벽의 일부는 단백질 잔류물에 대한 교차 이온과 영구/부분 전하 사이의 상호작용에 기인한다.다른 부분은 단백질/메브레인 유전체 매개체에서 유도된 쌍극점으로부터 발생하며, 유전 경계 힘(DBF)이라고 한다.DBF만을 계산하기 위해 단백질 잔류물의 정적 전하를 모두 끄고 이온을 모공을 통해 끌고 가 에너지 장벽을 계산한다.
EMF나 DBF 측정은 질적 측정일 뿐인데, 이온은 반드시 루멘의 중심을 통해 직선으로 채널을 가로지르지 않으며, 다른 이온이 동일하거나 반대 방향으로 이동하는 경우가 많아 시스템의 역학을 극적으로 변화시키므로 유의해야 한다.더욱이, 이온이나 이온이 채널을 가로질러 튕겨 나가면서 단백질 잔류물이 동적으로 위치를 바꾸는 조향 MD 계산과는 달리, 우리의 EMF 또는 DBF 계산에서 단백질은 정적 연속체로 모델링되며, 이는 에너지 계산에 더 정량적인 방식으로 영향을 미친다.측정에 추가로 영향을 미치는 또 다른 문제는 이온과 차폐로 움직이는 수화 분자의 부재다.위에서 모두 말했듯이, 여전히 EMF나 DBF를 계산하는 것은 채널 선택성이나 게이트를 다루는데 가치가 있다.이 두 가지 에너지 장벽 중 하나를 계산하는 것은 바이오모카에서 옵션으로 이용할 수 있다.
VMD를 사용한 시각화
VMD에는[14] 바이오모카 구조 로딩 옵션이 탑재됐다.이것은 비교를 위해 BioMOCA에서 생성된 구조와 함께 단백질 구조(PDB 또는 PQR 파일)를 모두 로드할 수 있기 때문에 매우 유용한 기능이다.오른쪽 그림은 바이오모카가 어떻게 그람시딘 채널에 막을 감싼 구조물을 만들었는지를 보여준다.또한 바이오모카(BioMOCA)는 이온 궤적을 표준 형식으로 덤프하여 나중에 VMD와 같은 분자 시각화 도구로 로드하고 프레임별로 영화 형식으로 시청할 수 있도록 한다.
궤적을 2진수로 기록
채널을 가로지르는 이온의 수를 세는 것 외에 때로는 채널의 다른 영역에서 이들의 행동을 연구하는 것이 바람직하다.그러한 예로는 채널 또는 나노포어 내부의 평균 이온 점유율 또는 평균 이동속도가 될 수 있다.BioMOCA는 시뮬레이션의 모든 단계(또는 몇 단계)에서 모든 이온 위치, 평균 및 순간 속도, 전위 및 운동에너지, 평균 및 순간 변위 및 기타 정보를 ASCII 형식으로 덤핑하는 옵션을 갖추고 있으므로 그러한 궤적 정보는 나중에 추가 통계를 수집하기 위해 연구할 수 있다.그러나 기술적 관점에서 볼 때 수십 개의 이온에 대해 그러한 정보를 덤핑하는 것은 수백 개의 시간 단계마다 시뮬레이션을 느리게 하고 수십 기가바이트까지 엄청난 파일이 축적되는 결과를 초래할 수 있다.나중에 디스크 스토리지에서 이러한 파일을 로드하는 것도 시간이 많이 걸리고 계산적으로 비효율적인 절차다.그 이상 ASCII 형식으로 수치 정보를 재코딩하면 기계 정밀도가 유지되지 않고 정확도가 떨어진다.
이러한 문제를 해결하는 것은 사실 쉬운 일이며, 단순히 ASCII 형식을 사용하지 않고 바이너리 형식을 사용하는 것이다.기계 정확도를 유지할 뿐만 아니라 파일 시스템에 대한 쓰기 및 읽기 속도도 훨씬 빠르다.궤적을 버리기 위한 계산적 오버헤드는 무시해도 될 정도로 작아지고 궤적 파일은 크기가 약 2배 작아진다.단점은 데이터 프로그래밍과 디코딩이 매우 까다로워질 수 있다는 점일 수 있지만, 일단 정확하고 신중하게 수행되면 바이너리 포맷을 사용할 때의 이점은 추가적인 노력을 기울일 가치가 충분히 있다.바이오모카에는 이제 궤적 정보를 이진 형식으로 기록하는 툴이 탑재됐다.
참고 항목
참조
- ^ a b c d van der Straaten, T.A.; Kathawala, G.; Trellakis, A.; Eisenberg §, R.S.; Ravaioli, U. (2005-02-15). "BioMOCA—a Boltzmann transport Monte Carlo model for ion channel simulation". Molecular Simulation. Informa UK Limited. 31 (2–3): 151–171. doi:10.1080/08927020412331308700. ISSN 0892-7022. S2CID 96166501.
- ^ a b C. Jacoboni, P. Lugli, Springer Verlag, 뉴욕 (1989)
- ^ a b R. Hockney, J. Eastwood, McGraw-Hill, 뉴욕 (1981)
- ^ a b Toghraee, Reza; Mashl, R. Jay; Lee, Kyu Il; Jakobsson, Eric; Ravaioli, Umberto (2009). "Simulation of charge transport in ion channels and nanopores with anisotropic permittivity". Journal of Computational Electronics. Springer Science and Business Media LLC. 8 (2): 98–109. doi:10.1007/s10825-009-0272-4. ISSN 1569-8025. PMC 2863032. PMID 20445807.
- ^ a b Warshel, Arieh; Russell, Stephen T. (1984). "Calculations of electrostatic interactions in biological systems and in solutions". Quarterly Reviews of Biophysics. Cambridge University Press (CUP). 17 (3): 283–422. doi:10.1017/s0033583500005333. ISSN 0033-5835. PMID 6098916.
- ^ a b Schutz, Claudia N.; Warshel, Arieh (2001). "What are the dielectric "constants" of proteins and how to validate electrostatic models?". Proteins: Structure, Function, and Genetics. Wiley. 44 (4): 400–417. doi:10.1002/prot.1106. ISSN 0887-3585. PMID 11484218. S2CID 9912122.
- ^ Warshel, Arieh; Papazyan, Arno (1998). "Electrostatic effects in macromolecules: fundamental concepts and practical modeling". Current Opinion in Structural Biology. Elsevier BV. 8 (2): 211–217. doi:10.1016/s0959-440x(98)80041-9. ISSN 0959-440X. PMID 9631295.
- ^ Roux, Benoît; Allen, Toby; Bernèche, Simon; Im, Wonpil (2004). "Theoretical and computational models of biological ion channels" (PDF). Quarterly Reviews of Biophysics. Cambridge University Press (CUP). 37 (1): 15–103. Bibcode:2004APS..MAR.J7004R. doi:10.1017/s0033583504003968. ISSN 0033-5835. PMID 17390604. S2CID 6213437.
- ^ S. S. Selberher, Semiconductor Devices 분석 및 시뮬레이션, 뉴욕, Springer-Verlag Wien, (1984)ISBN 3-211-81800-6
- ^ Baker, N. A.; Sept, D.; Holst, M. J.; McCammon, J. A. (2001). "The adaptive multilevel finite element solution of the Poisson-Boltzmann equation on massively parallel computers". IBM Journal of Research and Development. IBM. 45 (3.4): 427–438. doi:10.1147/rd.453.0427. ISSN 0018-8646.
- ^ Im, Wonpil; Roux, Benoı̂t (2002). "Ion Permeation and Selectivity of OmpF Porin: A Theoretical Study Based on Molecular Dynamics, Brownian Dynamics, and Continuum Electrodiffusion Theory". Journal of Molecular Biology. Elsevier BV. 322 (4): 851–869. doi:10.1016/s0022-2836(02)00778-7. ISSN 0022-2836. PMID 12270719.
- ^ T. A. 반 데어 스트라텐, J. M. 탕, U. 라바이올리, R. S. 에이젠버그, N.알루루, 제이 컴프선거 2, 29 (2003)
- ^ Wang, Hai-Long; Toghraee, Reza; Papke, David; Cheng, Xiao-Lin; McCammon, J. Andrew; Ravaioli, Umberto; Sine, Steven M. (2009). "Single-Channel Current Through Nicotinic Receptor Produced by Closure of Binding Site C-Loop". Biophysical Journal. Elsevier BV. 96 (9): 3582–3590. Bibcode:2009BpJ....96.3582W. doi:10.1016/j.bpj.2009.02.020. ISSN 0006-3495. PMC 2711404. PMID 19413963.
- ^ "VMD - Visual Molecular Dynamics". www.ks.uiuc.edu.



 각각 단백질에 대한 이온의 전하 밀도 및 영구 전하량이다
각각 단백질에 대한 이온의 전하 밀도 및 영구 전하량이다
 국부 유전체
국부 유전체  국부 정전기 전위이다.이 방정식을 해결하면 유전체 경계에서 유도된 영상 전하 효과와 적용된 바이어스를 포함할 수 있는 자기 일관적인 방법을 제공한다.
국부 정전기 전위이다.이 방정식을 해결하면 유전체 경계에서 유도된 영상 전하 효과와 적용된 바이어스를 포함할 수 있는 자기 일관적인 방법을 제공한다.