DNA 부호화 화학 라이브러리
DNA-encoded chemical libraryDNA 인코딩 화학 라이브러리(DEL)는 소분자 화합물 수집의 전례 없는 규모로 합성 및 스크리닝을 위한 기술입니다.DEL은 조합 화학과 분자 생물학 분야를 연결하기 위해 의학 화학에서 사용됩니다.DEL 기술의 목적은 약물 발견 과정, 특히 표적 검증 및 표적 식별과 같은 초기 단계 발견 활동을 가속화하는 것이다.
DEL 기술은 식별 바코드로 기능하는 짧은 DNA 조각에 화학 화합물 또는 구성 블록을 결합하는 것을 포함하며, 경우에 따라서는 화학 합성을 지시하고 제어합니다.이 기술은 일반적으로 고정화된 단백질 표적에서 친화력 선택을 통해 라이브러리의 대량 생성 및 질문을 가능하게 한다.단일 튜브 접근방식으로 개별 리간드 타깃 복합체를 분리, 계수 및 식별하기 위해 기름 내 수분 에멀전 기술을 사용하는 DNA 인코딩 라이브러리를 선별하는 균질한 방법이 최근 개발되었습니다.높은 스루풋 스크리닝과 같은 기존 스크리닝 절차와 달리 생화학적 어세이드는 바인더 식별에 필요하지 않으며, 원칙적으로 기존 스크리닝 기술로는 역사적으로 다루기 어려운 광범위한 단백질에 대한 바인더를 분리할 수 있다.따라서 표적 특정 분자 화합물의 일반적인 발견과 더불어, 약리학적으로 중요하지만, 지금까지는 "파괴할 수 없는" 표적 단백질에 대한 결합제의 이용은 지금까지 치료될 수 없었던 질병을 위한 새로운 약을 개발할 수 있는 새로운 가능성을 열어준다.히트의 활성을 초기에 평가할 필요가 없어짐에 따라 식별된 많은 고친화성 바인더가 선택된 히트의 독립적인 분석에 활성화되어 고품질 히트와 제약 리드를 식별하는 효율적인 방법을 제공할 것으로 기대된다.
DNA 부호화 화학 라이브러리 및 디스플레이 기술
최근까지, 실험실에서 분자 진화의 적용은 생물학적 분자와 관련된 디스플레이 기술에 국한되어 있었으며, 여기서 작은 분자가 이러한 생물학적 접근법을 넘어서는 것으로 간주되었다.DEL은 소분자 등 비천연성 화합물을 포함하는 디스플레이 기술 분야를 개방하여 분자진화와 자연선택의 적용을 바람직한 활성과 기능을 가진 소분자 화합물 식별에까지 확대하였습니다.DNA 부호화 화학 라이브러리는 항체 파지 디스플레이 기술, 효모 디스플레이, mRNA 디스플레이 및 압타머 SELEX와 같은 생물학적 디스플레이 기술과 유사합니다.항체 파지 디스플레이에서, 항체는 부착된 항체를 위한 유전자 코드를 가진 파지 입자와 물리적으로 연결되어 있으며, 이것은 "페노타입"과 "유전자형"[1]의 물리적 연결과 동등합니다.파이지 표시 항체는 분자 진화를 모방함으로써 대형 항체 라이브러리에서 분리될 수 있습니다. 즉, (고정화 단백질 표적에 대한) 여러 차례의 선택을 통해, 증폭 및 [2]번역이 이루어집니다.DEL에서는 작은 분자와 식별자 DNA 코드의 연계에 의해 결합 분자의 쉬운 식별이 가능하다.DEL 라이브러리는 선택된 고정화 표적 단백질에 대해 친화력 선택 절차를 거친 후 세척 단계에 의해 비결합체를 제거하고, 그 후 중합효소 연쇄반응(PCR)에 의해 바인더를 증폭하여 DNA 코드(e.g.by DNA 배열 처리)에 의해 동정할 수 있다.진화 기반 DEL 기술(아래 참조)에서는 항체 파지 디스플레이와 같은 생물학적 디스플레이 시스템과 유사한 선택, PCR 증폭 및 번역을 여러 차례 수행함으로써 히트 수를 더욱 강화할 수 있습니다.이를 통해 훨씬 더 큰 라이브러리로 작업할 수 있습니다.
역사
"단일 공정에서 복합성분 혼합물을 합성하고 단일 공정에서도 선별합니다."이것은 교수에 의해 발명된 조합 화학의 원리이다.1982년 Furka A.(Eötvös Loránd University Budapast Hungary)는 같은 [3]해에 공증된 문서에 조합 라이브러리의 합성 방법과 디콘볼루션 전략의 합성 방법을 포함시켰다.이 발명에 이르게 된 동기는 [4]2002년에 발표되었다.DEL은 DNA 부호화 조합 라이브러리(DEC)이며, 조합 원리는 그 응용 분야에서 분명히 우세하다.
DNA 인코딩의 개념은 1992년 Brenner와 Lerner에 의해 이론적인 논문에서 처음 설명되었는데, 이 이론에서는 화학적으로 합성된 실체의 각 분자를 병렬로 구성된 특정 올리고뉴클레오티드 배열에 연결하고 활성 [5]화합물을 식별하고 농축하기 위해 이 인코딩 유전자 태그를 사용하는 것이 제안되었다.1993년에 S. Brenner와 K. Janda가 이 접근방식의 첫 번째 실질적인 구현을 제시했으며, M.A.[6][7] Gallop의 그룹도 이와 유사하게 제시하였다.브레너와 얀다는 헤테로폴리머 화합물과 적절한 올리고뉴클레오티드 배열을 "분할" 기반 방식으로 번갈아 병렬 합성함으로써 개별 부호화된 라이브러리 구성원을 생성할 것을 제안했다(아래 [6]참조).
비보호 DNA는 기존의 반응 조건의 좁은 창으로 제한되기 때문에, 1990년대 말까지 주로 불편한 고체상 DNA를 피하기 위해 많은 대체 부호화 전략(MS 기반 화합물 태그 부착, 펩타이드 부호화, 할로방향족 태그 부착, 2차 아민에 의한 부호화, 반도체 장치)이 예상되었다.높은 throughput [8]방식으로 쉽게 스크리닝할 수 있는 조합 라이브러리를 만듭니다.그러나 DNA의 선택적 증폭성은 라이브러리 스크리닝을 크게 촉진하고 전례 없는 크기의 유기화합물 라이브러리 부호화에 필수불가결하다.그 결과 2000년대 초에 DNA-공생화학이 부활했다.
밀레니엄이 시작되면서 DEL 기술에 몇 가지 독립적인 개발이 도입되었습니다.이러한 기술은 비진화 기반 DEL 기술과 분자 진화가 가능한 진화 기반 DEL 기술이라는 두 가지 일반적인 범주로 분류할 수 있습니다.첫 번째 범주는 기성 시약을 사용할 수 있는 능력에서 이익을 얻으며, 따라서 비교적 간단한 라이브러리 생성이 가능하다.히트는 DNA 염기서열 분석으로 식별될 수 있지만, DNA 번역과 분자 진화는 이러한 방법으로는 가능하지 않습니다.Praecis Pharmacuticals(현 GlaxoSmithKline 소유), Nuevolution(덴마크 코펜하겐) 및 ESAC 테크놀로지의 연구자가 개발한 분할 및 풀 접근법.Neri(스위스 취리히 약학 연구소)는 이 범주에 속합니다.ESAC 테크놀로지는, fragment 베이스의 히트 검출과 유사한 조합의 자기 조립 방식(그림 1 b)이라고 하는 특징을 가지고 있습니다.여기서 DNA 어닐링은 개별 구성 블록 조합을 샘플링할 수 있지만, 그들 사이에 화학 반응은 일어나지 않습니다.진화 기반의 DEL 테크놀로지의 예로는 D.R. Halpin 교수와 P.B. Harbury 교수(Stanford University, Stanford, CA)가 개발한 DNA 라우팅과 Professor가 개발한 DNA 템플릿 합성 등이 있습니다.D. Liu(Harvard University, Cambridge, MA) 및 YoctoReactor [9]기술에 의해 상용화되었습니다.Vipergen(덴마크 코펜하겐)에 의해 개발 및 상용화되었습니다.이러한 테크놀로지에 대해서는, 이하에 자세하게 설명합니다.DNA 템플릿 합성 및 YoctoReactor 기술은 라이브러리 조립 전에 DNA 올리고뉴클레오티드 태그에 대한 화학 구성 블록(BB)의 사전 결합을 필요로 하므로 라이브러리 조립 전에 더 많은 선행 작업이 필요합니다.또한 DNA 태그 부착 BB는 합성 화합물에 대한 유전자 코드 생성을 가능하게 하며 유전자 코드의 인위적인 번역이 가능하다.BB는 PCR 증폭된 유전자 코드에 의해 회수될 수 있고 라이브러리 화합물은 재생될 수 있다.이것은, 차례로, 다윈의 자연 선택과 진화의 원리를 생물학적 디스플레이 시스템과 직접적으로 유사하게; 선택, 증폭, 그리고 번역을 통해 작은 분자 선택에 적용할 수 있게 합니다.
비진화 기반 테크놀로지
조합 라이브러리
조합 라이브러리는 단일 단계적 프로세스에서 합성되는 특수 다성분 화합물 혼합물입니다.그것들은 개별 화합물의 집합뿐만 아니라 병렬 합성에 의해 만들어진 일련의 화합물들과도 다르다.조합 라이브러리에는 중요한 기능이 있습니다.
∙ 혼합물은 그 합성에 사용된다.혼합물을 사용하면 공정의 효율성이 매우 높아집니다.두 반응물은 혼합물이 될 수 있지만 실질적인 이유로 분할 혼합 절차가 사용됩니다. 즉, 한 혼합물은 BB와 [10][11]결합된 부분으로 나뉩니다.혼합물은 매우 중요하기 때문에 합성에 혼합물을 사용하지 않는 조합 라이브러리가 없으며 혼합물이 불가피하게 조합 라이브러리 형태로 사용되는 경우.
② 라이브러리 구성요소는 거의 동일한 몰 양으로 존재해야 한다.이를 위해 혼합물을 동일한 부분으로 나누고 풀링한 후 철저한 혼합이 필요합니다.
∙ 구성부품의 구조를 알 수 없기 때문에 선별 시 디콘볼루션 방법을 사용해야 한다.이러한 이유로 부호화 방법이 개발되었습니다.코드 분자는 결합된 BB와 그 시퀀스를 기록하는 고체 지지대의 비드에 부착됩니다.이들 방법 중 하나는 DNA 올리고머에 의한 부호화이다.
② 혼합물을 한 번에 모두 스크리닝할 수 있는 것은 조합 라이브러리의 특징입니다.
합성 및 스크리닝은 모두 매우 효율적인 절차이기 때문에 제약 연구에 조합 라이브러리를 사용하면 엄청난 비용 절감으로 이어집니다.
고체상 조합 합성에서는 각 비드에 단일 화합물만 형성됩니다.따라서 라이브러리의 컴포넌트 수는 솔리드 서포트의 비즈 수를 초과할 수 없습니다.이는 이러한 라이브러리의 구성 요소 수가 제한적이라는 것을 의미합니다.이 구속은 Harbury와 Halpin에 의해 완전히 제거되었다.이들의 DEL 합성에 있어서 고체 지지체는 생략되고 BB는 코드화 DNA 올리고머에 [12]직접 부착된다.이 새로운 접근방식은 DNA 부호화 조합 라이브러리(DEC)의 컴포넌트 수를 실질적으로 무제한으로 늘리는 데 도움이 됩니다.
스플릿&풀 DNA 부호화
DNA 부호화 화학 라이브러리의 합성에 조합 화학을 적용하기 위해, Split-&Pool 접근법을 [10][11]추구했다.처음에 특정 코드 배열을 포함한 고유 DNA-올리고뉴클레오티드(n) 세트를 대응하는 작은 유기 분자 세트와 화학적으로 결합한다.이것에 의해 올리고뉴클레오티드-공역화합물이 혼합('풀')되어 다수의 기(m)로 분할된다.적절한 조건에서 제2의 블록(m)을 제1의 블록(m)에 결합하고, 다시 혼합하기 전에 제2의 수정을 코드하는 올리고뉴클레오티드를 효소적으로 도입한다.이 "분할 및 풀" 단계는 각 라운드에서 (n x m)r 조합 방식으로 라이브러리 크기를 증가시키는 횟수(r)를 반복할 수 있습니다.또는 펩타이드 핵산은 "split-&pool"[13] 방법으로 제조된 라이브러리를 인코딩하기 위해 사용되어 왔다.PNA 인코딩의 장점은 표준 SPPS에 [14]의해 화학을 수행할 수 있다는 것입니다.
초기 유기 분자에 대한 코드화 DNA 조각의 단계적 결합

라이브러리 합성을 위한 "핵심 구조"로서 기능하는 올리고뉴클레오티드에 공유 결합되는 다기능 구성 블록을 사용함으로써 DNA 부호화 라이브러리 구축을 위한 유망한 전략이 제시된다.'풀 앤 스플릿' 방식으로 다기능 발판 세트는 일련의 적절한 반응성 파트너와 직교 반응을 거칩니다.각 반응 단계에 따라, 수정의 동일성은 원래 DNA "핵심 구조"[15][16]에 DNA 세그먼트를 효소적으로 추가함으로써 부호화된다.DNA 단편에 공유 결합되어 있는 N-보호 아미노산의 사용은 적절한 탈보호 단계 후에 일련의 카르본산 또는 알데하이드와의 환원 아미노화와의 추가적인 아미드 결합 형성을 가능하게 한다.마찬가지로 아미노변성 올리고뉴클레오티드의 5' 말단에서 라이브러리 구축용 발판으로 사용되는 디엔카르본산은 다양한 말레이미드 유도체와 디엘알데르 반응을 일으킬 수 있다.원하는 반응공정 완료 후 올리고뉴클레오티드에 첨가되는 화학부분의 동일성은 부분적으로 상보적인 올리고뉴클레오티드의 아닐화 및 후속 클레노우 필인 DNA 중합에 의해 결정되며 이중가닥 DNA 단편을 생성한다.위에서 설명한 합성 및 부호화 전략은 두 세트의4 "구성 블록"을 포함하는 최대 10개의 구성원 화합물 크기의 DNA 인코딩 라이브러리를 쉽게 구성할 수 있도록 합니다.그러나 매우 큰 DNA 부호화 라이브러리(최대6 10개의 화합물 포함)의 구축 및 부호화를 위해 3가지 기능 코어 구성 블록에 적어도 3개의 독립된 화학 부분 세트를 단계적으로 추가하는 것도 생각할 [15]수 있다.(그림2).
조합 자체 조립
인코딩된 자가 조립 화학 라이브러리

부호화된 자체 조립 화학(ESAC) 라이브러리는 일정한 상호 보완적 하이브리드화 도메인을 포함하는 x 멤버 크기의 두 개의 하위 라이브러리(예3: 10)가 x 균일하게 표현된 라이브러리 멤버의 복잡성2(예: 106)[17]을 가진 하이브리드화 후 조합 DNA 이중 라이브러리를 생성할 수 있다는 원리에 의존한다.각 서브라이브러리 부재는 일정한 DNA 배열로 양옆에 있는 가변 부호화 영역을 포함하는 올리고뉴클레오티드로 구성되며 올리고뉴클레오티드 [17]말단에서 적절한 화학적 변형을 담당한다.ESAC 서브 라이브러리는 적어도 4개의 다른 실시 [17]형태에서 사용할 수 있다.
- 서브라이브러리는 상보적 올리고뉴클레오티드와 쌍을 이루며 어피니티 기반 선택실험을 위한 단일 공유결합화합물을 표시하는 DNA 부호화 라이브러리로서 사용할 수 있다.
- 서브라이브러리는 타겟에 대한 기존의 바인더를 나타내는 올리고뉴클레오티드와 쌍을 이룰 수 있어 친화성 성숙 전략을 가능하게 한다.
- 2개의 개별 서브 라이브러리는 조합적으로 조립되어 바인덴테이트 결합 분자의 de novo 동정용으로 사용될 수 있다.
- 3개의 다른 서브 라이브러리를 조합하여 트리플플렉스 라이브러리를 형성할 수 있습니다.
어피니티 베이스 선택에서 분리된 우선결합제는 상보적인 올리고뉴클레오티드 마이크로어레이[18] 또는 코드 연결, 서브클로닝 및 [17]시퀀싱에 의해 PCR 증폭 및 디코딩될 수 있다.개별 구성 요소는 결국 적절한 링커를 사용하여 결합되어 약물 유사 고친화성 화합물을 생성할 수 있습니다.링커의 특성(예: 길이, 유연성, 기하학, 화학적 성질 및 용해성)은 결합 친화력과 결과 결합의 화학적 특성에 영향을 미칩니다.(그림3)
600명의 구성원으로 구성된 ESAC 라이브러리의 HSA에 대한 바이오 패닝 실험을 통해 4-(p-iodophenyl) 부탄성 부분을 분리할 수 있었다.이 화합물은 일련의 휴대용 알부민 결합 분자와 현재 임상평가 [19]중인 최근 개발된 플루오레세인 혈관 조영제인 Albufluor의 핵심 구조를 나타냅니다.
ESAC 기술은 소 트립신의 잠재적 억제제 분리 및 생리학적 및 병리학적 조직 리모델링 과정뿐만 아니라 관절염 및 [20]전이 같은 질병 과정에도 관여하는 매트릭스 메탈로프로테이나아제인 스트로멜리신-1(MMP-3)의 새로운 억제제 식별에 사용되어 왔다.
진화 기반의 테크놀로지
DNA 라우팅
2004년, D.R. Halpin과 P.B.Harbury는 DNA 인코딩 라이브러리 구축을 위한 새로운 흥미로운 방법을 제시했습니다.처음으로 DNA 결합 템플릿은 라이브러리 [21]컴포넌트의 "분할 및 풀" 합성의 인프라스트럭처를 인코딩 및 프로그래밍하는 역할을 했습니다.Halpin과 Harbury의 디자인은 파지 디스플레이 기술과 완전히 유사한 작은 유기 분자로 번갈아 선택, PCR 증폭 및 다양화를 가능하게 했습니다.DNA 라우팅 기계는 수지 결합 안티코돈(antiodon)을 가진 일련의 연결된 기둥으로 구성되어 있으며, 이는 DNA 템플릿의 집단을 하이브리드화에 [21]의해 공간적으로 구별되는 위치로 특별히 분리할 수 있다.이 스플릿 앤 풀 프로토콜에 따라 10명의 멤버로6 인코딩된 펩타이드 조합 라이브러리 DNA가 [22]생성되었다.
DNA템플릿합성

2001년에 David Liu와 동료들은 상보적인 DNA 올리고뉴클레오티드가 저농도의 [23][24]용액에서 효율적으로 일어나지 않는 특정 합성 반응을 돕는 데 사용될 수 있다는 것을 보여주었다.DNA-헤테로듀플렉스는 두 DNA 가닥의 끝에 나타나는 화학적 부분 간의 반응을 가속화하기 위해 사용되었다.또한, 쌍분자 반응을 가속화하는 "근접 효과"는 거리에 의존하지 않는 것으로 나타났다(최소한 30 뉴클레오티드의 [23][24]거리 이내).하나의 화학반응물 그룹을 운반하는 올리고뉴클레오티드가 다른 반응성 화학 그룹을 운반하는 상보적 올리고뉴클레오티드 유도체로 교배되었다.DNA 교잡에 의해 부여되는 근접성은 올리고뉴클레오티드에 부착된 반응 시약의 유효 몰 농도를 획기적으로 증가시켜 해당 기존 유기 리아에 필요한 농도보다 몇 배 낮은 수성 환경에서도 원하는 반응이 일어날 수 있게 한다.DNA [25]템플릿이 아닙니다. DNA 템플릿이 아닙니다.DNA 템플릿 설정 및 시퀀스 프로그래밍된 합성 기능을 사용하여 Liu와 동료들은 64명으로 구성된 복합 DNA 부호화 매크로 [26]사이클 라이브러리를 생성했습니다.
3차원 근접 기반 기술(YoctoReactor 기술)
YoctoReactor(yR)는 DNA 올리고뉴클레오티드의 자가조립 특성을 이용하여 접합부의 중심에 작은 분자 합성을 지시하는 3D 근접성 접근법이다.그림 5는 4방향 DNA 접합의 기본 개념을 보여줍니다.
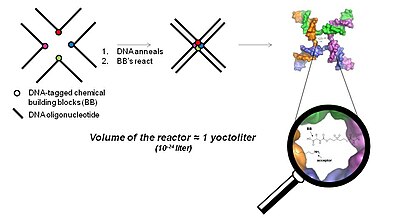
DNA 접합의 중심은 헥토리터 순서로 볼륨을 구성하므로 헥토리터라는 이름이 붙습니다.이 부피는 높은 mM 범위에서 반응 농도를 생성하는 단일 분자 반응을 포함한다.DNA에 의해 촉진된 유효 농도는 화학 반응을 크게 가속화하는데, 그렇지 않았다면 실제 농도는 몇 배 더 낮았을 것이다.
yR 라이브러리 구축
그림 6은 3방향 DNA 접합을 사용한 yR 라이브러리의 생성을 보여줍니다.
요약하면 화학빌딩블록(BB)은 분해성 또는 비분열성 링커를 통해 yR의 각 팔을 나타내는 3종류의 2특이적 DNA올리고뉴클레오티드(oligo-BBs)에 부착된다.조합적으로 합성을 용이하게 하기 위해 올리고-BB는 (a)올리고(색선)의 선단에 부착 BB의 코드를 포함하고 (b) 일정한 DNA 배열의 선단부(검은색선)에 DNA의 자기집합을 (BB와는 독립적으로) 가져오도록 설계되어 있다.혈중 반응단계적 절차를 통해 화학반응을 하고 각 단계 후 DNA를 결속시켜 폴리아크리아미드겔 전기영동으로 정제한다.분해성 링커(BB-DNA)는 DNA 코드에 대한 단일 공유 링크를 가진 작은 분자의 라이브러리를 생성하는 단일 위치를 제외한 모든 위치에 사용됩니다.표 1은 yR 기술을 사용하여 다양한 크기의 라이브러리를 생성하는 방법을 정리한 것입니다.
yR 설계 접근방식은 (a) 반응물 간 거리와 (b) 반응 부위를 둘러싼 배열 환경 둘 다와 관련하여 불변 반응 부위를 제공한다.또, 1개의 포토에 조합적으로 혼합된 올리고-BB상의 코드와 BB와의 밀접한 접속은, 라이브러리의 부호화에 높은 충실도를 부여한다.게다가 합성 제품의 코드는 프리셋이 아니고, 조합적으로 조립되어 선천적인 제품과 동기해 합성된다.
육반응기 라이브러리의 균질 스크리닝
개별 리간드 타깃 복합체를 분리하기 위해 유중수 에멀전 기술을 사용하는 균질한 육토액터 라이브러리(yR) 스크리닝 방법이 최근 개발되었습니다.바인더 트랩 농축(BTE)이라 불리는 단백질 표적에 대한 배위자는 해리 지배적 키네틱스 중에 에멀젼 방울에 결합 쌍(DNA 라벨이 부착된 단백질 표적 및 yR 배위자)을 포착함으로써 식별된다.일단 포착되면 표적 DNA와 배위자 DNA가 결찰에 의해 결합되어 결합 정보를 보존한다.
이후, 히트 식별은 본질적으로 계수 연습이다. 결합 이벤트에 대한 정보는 시퀀싱을 통해 해독되며, 결합 DNA 선택 바인더는 무작위 바인더보다 훨씬 높은 빈도로 계수된다.이는 표적과 배위자의 무작위 포획이 유제 내 많은 수의 물방울에 의해 "희박"되기 때문에 가능합니다.BTE의 낮은 노이즈 및 배경 신호 특성은 랜덤 신호의 "희석"과 표면 아티팩트의 부족, yR 라이브러리와 스크리닝 방법의 높은 충실도에 기인한다.스크리닝은 단일 튜브 방식으로 수행됩니다.생물학적 활성 적중률은 낮은 거짓 양성률로 특징지어지는 BTE의 한 라운드에서 식별된다.
BTE는 생체 내 리간드-타겟 상호작용의 비균형 특성을 모방하고 결합 복합체를 가두는 에멀젼이 동적 해리 단계에서 형성되기 때문에 리간드-타겟 체류 시간에 기초한 표적 특이 리간드에 대한 선별 가능성을 제공한다.
DNA 부호화 화학 라이브러리 디코딩
DNA 부호화 화학 라이브러리에서 선택한 후, 특정 결합 화합물의 빠르고 효율적인 식별을 위한 해독 전략은 DEL 기술의 추가 개발에 매우 중요하다.지금까지 생어 시퀀싱 기반 디코딩, 마이크로 어레이 기반 방법론 및 높은 스루풋 시퀀싱 기술은 DNA 인코딩 라이브러리 선택을 디코딩하는 주요 방법론이었다.
생거 시퀀싱 기반 디코딩
많은 저자들이 전통적인 Sanger 시퀀싱 기반 [6][7][17][22][26]디코딩을 암묵적으로 구상하고 있지만, 라이브러리의 복잡성에 따라 단순히 시퀀싱하는 코드 수는 전통적인 Sanger 시퀀싱 접근법에 있어 분명 비현실적인 작업이다.그럼에도 불구하고, DNA 인코딩 화학 라이브러리를 높은 처리량 방식으로 디코딩하기 위한 Sanger 시퀀싱의 구현이 최초로 설명되었다.[17]라이브러리 화합물의 DNA-태그 선택 및 PCR 증폭 후, 복수의 부호화 배열을 포함한 콘카타머가 생성되어 벡터에 결합되었다.결과 콜로니의 대표 번호를 Sanger로 시퀀싱한 후 선택 [17]전후에 DNA 인코딩 라이브러리 샘플에 존재하는 코드의 빈도가 밝혀졌다.
마이크로 어레이 기반 디코딩
DNA 마이크로어레이는 분자생물학 및 의학에서 널리 사용되는 높은 처리량 조사를 위한 장치입니다.특정 DNA 서열을 운반하는 올리고뉴클레오티드의 몇 개의 피코몰을 포함하는 일련의 현미경 점('특징' 또는 '위치')으로 구성됩니다.이것은 적절한 조건에서 DNA 또는 RNA 샘플을 교배시키기 위한 탐침으로 사용되는 유전자 또는 다른 DNA 요소의 짧은 단면일 수 있습니다.프로브-타깃 교배는 보통 불소포자 표적의 형광 기반 검출에 의해 검출되고 정량화되어 표적 핵산 배열의 상대적 풍부성을 결정한다.마이크로 어레이는 ESAC DNA 인코딩[17] 라이브러리 및 PNA 인코딩 라이브러리를 성공적으로 [27]디코딩하기 위해 사용되어 왔습니다.라이브러리의 개별 화학 화합물을 나타내는 부호화 올리고뉴클레오티드는 바이오칩 어레이로봇을 사용하여 마이크로어레이 슬라이드에 화학적으로 연결되어 있습니다.이어서 선택에서 분리된 결합화합물의 올리고뉴클레오티드 태그는 형광프라이머를 사용하여 PCR을 증폭하여 DNA 마이크로어레이 슬라이드상에 하이브리드화한다.그 후 레이저 스캔을 이용하여 마이크로 어레이를 분석하여 검출하고 정량화한다.선택 [17]전후의 DNA 마이크로어레이 슬라이드의 스폿 강도를 비교하여 선호 결합 화합물의 농도를 알 수 있다.
높은 스루풋 시퀀싱을 통한 디코딩
DNA 부호화 화학 라이브러리의 복잡성(일반적으로 103~106 멤버)에 따르면 염기서열을 위한 베이스당 비용이 높고 절차가 [28]번거롭기 때문에 기존의 생거 염기서열 기반 디코딩을 실제로 사용할 수 없을 것 같다.높은 처리량 시퀀싱 기술은 캐피럴리 전기영동 사용을 대체하고 동시에 수천 또는 수백만 개의 시퀀스를 생성하는 시퀀싱 프로세스를 병렬화하는 전략을 이용했습니다.2008년에는 4000개의 [15]화합물로 구성된 DNA 부호화 화학 라이브러리의 빠르고 효율적인 해독을 위해 게놈 염기서열 분석(즉, "454 기술")을 위해 개발된 높은 처리량 염기서열 분석 기술의 첫 구현이 설명되었다.이 연구는 스트렙타비딘에 대한 준미량 해리 상수를 가진 새로운 화학 화합물의 확인으로 이어졌고 수백만 개의 화학 [15]화합물을 포함하는 DNA 인코딩 라이브러리를 구축하고, 선택을 수행하고, 해독할 수 있는 가능성을 확실히 보여주었다.
「 」를 참조해 주세요.
레퍼런스
- ^ Smith GP (June 1985). "Filamentous fusion phage: novel expression vectors that display cloned antigens on the virion surface". Science. 228 (4705): 1315–7. Bibcode:1985Sci...228.1315S. doi:10.1126/science.4001944. PMID 4001944.
- ^ Hoogenboom HR (2002). "Overview of antibody phage-display technology and its applications". Antibody Phage Display. Methods in Molecular Biology. Vol. 178. pp. 1–37. doi:10.1385/1-59259-240-6:001. ISBN 978-1-59259-240-1. PMID 11968478.
- ^ 푸르카 A.타눌마니, 규야스자틸락 하즈노시 펩타이드크 지즈테마티쿠스 펠쿠타사냐크 레헤제릴.약학적으로 유용한 펩타이드를 체계적으로 검색할 가능성에 관한 연구) https://mersz.hu/mod/object.php?objazonosito=matud202006_f42772_i2
- ^ Furka AH (2002년)조합 화학 20년 전... 'Drug Discov오늘 7시~1시~4시~
- ^ Brenner S, Lerner RA (June 1992). "Encoded combinatorial chemistry". Proceedings of the National Academy of Sciences of the United States of America. 89 (12): 5381–3. Bibcode:1992PNAS...89.5381B. doi:10.1073/pnas.89.12.5381. PMC 49295. PMID 1608946.
- ^ a b c Nielsen J, Brenner S, Janda KD (1993). "Synthetic methods for the implementation of encoded combinatorial chemistry". Journal of the American Chemical Society. 115 (21): 9812–9813. doi:10.1021/ja00074a063.
- ^ a b Needels MC, Jones DG, Tate EH, Heinkel GL, Kochersperger LM, Dower WJ, Barrett RW, Gallop MA (November 1993). "Generation and screening of an oligonucleotide-encoded synthetic peptide library". Proceedings of the National Academy of Sciences of the United States of America. 90 (22): 10700–4. Bibcode:1993PNAS...9010700N. doi:10.1073/pnas.90.22.10700. PMC 47845. PMID 7504279.
- ^ Mukund S. Chorghade (2006). Drug discovery and development. New York: Wiley-Interscience. pp. 129–167. ISBN 978-0-471-39848-6.
- ^ Heitner TR, Hansen NJ (November 2009). "Streamlining hit discovery and optimization with a yoctoliter scale DNA reactor". Expert Opinion on Drug Discovery. 4 (11): 1201–13. doi:10.1517/17460440903206940. PMID 23480437. S2CID 20881306.
- ^ a b A. F. 세베스티엔, M. 아스게돔, G. 디보, 코누코피아에 의한 합성 펩타이드의 현대 생화학 하이라이트, 제14회 국제 생화학 콩그레스, VSP.위트레흐트, 네덜란드, 1988, 제5권, 페이지 47.
- ^ a b Furka AH, 세베스티엔 F, 아스게돔 M, 디보 G(1991) 다성분 펩타이드 혼합물의 신속한 합성을 위한 일반적인 방법.Int J 펩타이드 단백질 레스 37; 487-93.
- ^ Harbury DR, Halpin DR(2000) WO 00/23458.
- ^ Winssinger N, Damoiseaux R, Tully DC, Geierstanger BH, Burdick K, Harris JL (October 2004). "PNA-encoded protease substrate microarrays". Chemistry & Biology. 11 (10): 1351–60. doi:10.1016/j.chembiol.2004.07.015. PMID 15489162.
- ^ Zambaldo C, Barluenga S, Winssinger N (June 2015). "PNA-encoded chemical libraries". Current Opinion in Chemical Biology. 26: 8–15. doi:10.1016/j.cbpa.2015.01.005. PMID 25621730.
- ^ a b c d Mannocci L, Zhang Y, Scheuermann J, Leimbacher M, De Bellis G, Rizzi E, Dumelin C, Melkko S, Neri D (November 2008). "High-throughput sequencing allows the identification of binding molecules isolated from DNA-encoded chemical libraries". Proceedings of the National Academy of Sciences of the United States of America. 105 (46): 17670–5. Bibcode:2008PNAS..10517670M. doi:10.1073/pnas.0805130105. PMC 2584757. PMID 19001273.
- ^ Buller F, Mannocci L, Zhang Y, Dumelin CE, Scheuermann J, Neri D (November 2008). "Design and synthesis of a novel DNA-encoded chemical library using Diels-Alder cycloadditions". Bioorganic & Medicinal Chemistry Letters. 18 (22): 5926–31. doi:10.1016/j.bmcl.2008.07.038. PMID 18674904.
- ^ a b c d e f g h i Melkko S, Scheuermann J, Dumelin CE, Neri D (May 2004). "Encoded self-assembling chemical libraries". Nature Biotechnology. 22 (5): 568–74. doi:10.1038/nbt961. PMID 15097996. S2CID 24806778.
- ^ Lovrinovic M, Niemeyer CM (May 2005). "DNA microarrays as decoding tools in combinatorial chemistry and chemical biology". Angewandte Chemie. 44 (21): 3179–83. doi:10.1002/anie.200500645. PMID 15861437.
- ^ Dumelin CE, Trüssel S, Buller F, Trachsel E, Bootz F, Zhang Y, Mannocci L, Beck SC, Drumea-Mirancea M, Seeliger MW, Baltes C, Müggler T, Kranz F, Rudin M, Melkko S, Scheuermann J, Neri D (2008). "A portable albumin binder from a DNA-encoded chemical library". Angewandte Chemie. 47 (17): 3196–201. doi:10.1002/anie.200704936. PMID 18366035.
- ^ Melkko S, Zhang Y, Dumelin CE, Scheuermann J, Neri D (2007). "Isolation of high-affinity trypsin inhibitors from a DNA-encoded chemical library". Angewandte Chemie. 46 (25): 4671–4. doi:10.1002/anie.200700654. PMID 17497616.
- ^ a b Halpin DR, Harbury PB (July 2004). "DNA display I. Sequence-encoded routing of DNA populations". PLOS Biology. 2 (7): E173. doi:10.1371/journal.pbio.0020173. PMC 434148. PMID 15221027.

- ^ a b Halpin DR, Harbury PB (July 2004). "DNA display II. Genetic manipulation of combinatorial chemistry libraries for small-molecule evolution". PLOS Biology. 2 (7): E174. doi:10.1371/journal.pbio.0020174. PMC 434149. PMID 15221028.

- ^ a b Gartner ZJ, Liu DR (July 2001). "The generality of DNA-templated synthesis as a basis for evolving non-natural small molecules". Journal of the American Chemical Society. 123 (28): 6961–3. doi:10.1021/ja015873n. PMC 2820563. PMID 11448217.
- ^ a b Calderone CT, Puckett JW, Gartner ZJ, Liu DR (November 2002). "Directing otherwise incompatible reactions in a single solution by using DNA-templated organic synthesis". Angewandte Chemie. 41 (21): 4104–8. doi:10.1002/1521-3773(20021104)41:21<4104::AID-ANIE4104>3.0.CO;2-O. PMID 12412096.
- ^ Li X, Liu DR (September 2004). "DNA-templated organic synthesis: nature's strategy for controlling chemical reactivity applied to synthetic molecules". Angewandte Chemie. 43 (37): 4848–70. doi:10.1002/anie.200400656. PMID 15372570.
- ^ a b Gartner ZJ, Tse BN, Grubina R, Doyon JB, Snyder TM, Liu DR (September 2004). "DNA-templated organic synthesis and selection of a library of macrocycles". Science. 305 (5690): 1601–5. Bibcode:2004Sci...305.1601G. doi:10.1126/science.1102629. PMC 2814051. PMID 15319493.
- ^ Harris J, Mason DE, Li J, Burdick KW, Backes BJ, Chen T, Shipway A, Van Heeke G, Gough L, Ghaemmaghami A, Shakib F, Debaene F, Winssinger N (October 2004). "Activity profile of dust mite allergen extract using substrate libraries and functional proteomic microarrays". Chemistry & Biology. 11 (10): 1361–72. doi:10.1016/j.chembiol.2004.08.008. PMID 15489163.
- ^ Sanger F, Nicklen S, Coulson AR (December 1977). "DNA sequencing with chain-terminating inhibitors". Proceedings of the National Academy of Sciences of the United States of America. 74 (12): 5463–7. Bibcode:1977PNAS...74.5463S. doi:10.1073/pnas.74.12.5463. PMC 431765. PMID 271968.




