단백질역학
Protein dynamics단백질은 일반적으로 아미노산 염기서열에 의해 결정되는 독특한 구조를 채택하는 것으로 생각된다. 그러나 단백질은 엄격히 정적인 물체가 아니라 (때로는 비슷한) 순응의 앙상블을 채우는 것이다. 이러한 상태 사이의 전환은 다양한 길이 척도(ELT의 10분의 1 ~ nm)와 시간 척도(ns ~ s)에서 발생하며, 알로스테릭 신호[1] 전달 및 효소 카탈루션과 같은 기능적으로 관련되는 현상과 연계되어 왔다.[2]
단백질 역학 연구는 이러한 상태들 사이의 전환과 가장 직접적으로 관련이 있지만, 상태 자체의 자연과 평형 인구도 포함할 수 있다. 이 두 가지 관점(각각 키네틱스와 열역학)은 "에너지 풍경" 패러다임에서 개념적으로 합성될 수 있다.[3] 즉, 인구밀도가 높은 상태와 이들 사이의 전환의 운동학은 각각 에너지 우물 깊이와 에너지 장벽 높이에 의해 설명될 수 있다.
국부 유연성: 원자와 잔류물
단백질 구조의 일부는 종종 평형 상태에서 벗어난다. 화학적 결합과 결합 각도의 확률적 변동과 같이 그러한 편차는 조화롭다. 다른 것들은 별개의 분리된 에너지 미니마 또는 로타머 사이에서 점프하는 사이드체인과 같은 조화로운 것이다.[4]
국부적 유연성에 대한 증거는 종종 NMR 분광학에서 얻는다. 단백질의 유연하고 잠재적으로 흐트러진 부위는 무작위 코일 지수를 사용하여 검출할 수 있다. 접힌 단백질의 유연성은 단백질 내 개별 원자의 스핀 이완을 분석하여 확인할 수 있다. 또한 X선 결정학에 의해 생산된 매우 고해상도 전자 밀도 지도에서도 유연성이 관찰될 수 있으며,[5] 특히 기존의 극저온(일반적으로 100K 가까이) 대신 상온에서 회절 데이터를 수집할 때 더욱 그러하다.[6] 국소 단백질 유연성의 주파수 분포 및 역학 정보는 테라헤르츠 주파수 영역에서 라만과 광학 커 효과 분광법을 사용하여 얻을 수 있다.[7]
지역적 유연성: 도메인 내 멀티 레시듀 커플링
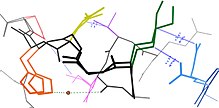
단백질 구조에서 많은 잔류물이 공간적으로 근접해 있다. 이는 일차 순서에 따라 연속되는 대부분의 잔류물에도 해당하지만, 순서에 따라 원위치가 있는 많은 잔류물도 최종 접힌 구조물에 접촉한다. 이러한 근접성 때문에 이러한 잔류물의 에너지 경관은 수소 결합, 이온 결합, 판 데르 발스 상호작용과 같은 다양한 생물물리학적 현상을 기반으로 결합된다(그림 참조). 따라서 그러한 잔류물 집합에 대한 상태 간의 전환은 상관 관계가 된다.[8]
이것은 종종 다른 결정 구조에서 다른 순응을 채택하기 위해 집합적으로 이동하는 표면 노출 루프에 대해 가장 명백할 수 있다(그림 참조). 그러나 결합형 순응 이질성은 이차 구조에서도 뚜렷하게 나타난다.[9] 예를 들어 1차 순서에서 4로 상쇄되는 연속 잔류물과 잔류물은 종종 α 나선형으로 상호작용한다. 또한 1차 시퀀스에서 2로 오프셋되는 잔류물은 옆면 체인을 β 시트의 동일한 면으로 향하며, 동일한 β 시트의 인접 가닥에 잔류물이 있는 것처럼 강직하게 상호작용할 수 있을 정도로 충분히 가깝다. 이러한 순응적 변화들 중 일부는 인산화나 메틸화 같은 단백질 구조에서 변환 후 수정으로 유도된다.[9][10]
이러한 결합 잔류물이 기능적으로 중요한 단백질 부분을 연결하는 경로를 형성할 때, 그것들은 알로스테릭 신호에 참여할 수 있다. 예를 들어, 산소 분자가 헤모글로빈 테트라머의 한 서브유닛에 결합할 때, 그 정보는 다른 세 서브유닛에 알로스테릭하게 전파되어 산소에 대한 친화력을 강화한다. 이 경우 헤모글로빈의 결합 유연성은 협조적인 산소 결합을 가능하게 하는데, 폐조직에서는 산소 부하가 빨라지고 산소가 부족한 조직(예: 근육)에서는 산소 하역 속도가 빨라지기 때문에 생리학적으로 유용하다.
글로벌 유연성: 여러 도메인
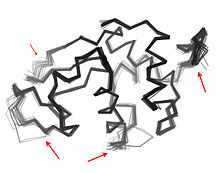
단백질에 여러 도메인이 존재하면 유연성과 이동성이 크게 나타나 단백질 영역 역학으로 이어진다.[1] 영역 운동은 (분자 운동 데이터베이스에서와 같이) 단백질의 서로 다른 구조를 비교하여 유추할 수 있으며, 중성자 스핀 에코 분광법에 의해 측정된[11][12] 스펙트럼을 사용하여 직접 관찰할 수 있다. 또한 광범위한 분자역학 궤적과[13] 주요 성분 분석에서 표본을 추출하여 제안할 수 있다.[14] 도메인 동작은 다음을 위해 중요하다.
- ABC 수송기 [15]
- 강직성[16]
- 세포 운동과 운동 단백질[17]
- 단백질 복합체[18] 형성
- 이온 통로[19]
- 기계 수용체와 기계 전도[20]
- 규제 활동
- 세포막을[citation needed] 가로지르는 대사물 수송
가장 크게 관찰된 도메인 운동 중 하나는 피루베이트 인산염 디키나아제의 '스위블링' 메커니즘이다. 인산염 영역은 뉴클레오티드 결합 영역의 활성 부위에서 인산염 그룹을 인산염 피루베이트/피루베이트 도메인으로 가져오기 위해 두 상태 사이를 회전한다.[22] 인산염 그룹은 단일 잔류물 주위로 약 100도의 도메인 운동을 포함하여 45 å의 거리로 이동한다. 효소에서는 한 도메인을 다른 영역으로 폐쇄하는 것이 유도 적합에 의해 기질을 포착하여 반응이 통제된 방식으로 일어날 수 있게 한다. 게르슈타인의 상세한 분석은 두 가지 기본적인 도메인 운동 유형인 힌지와 전단 구분을 이끌어냈다.[19] 체인에서 비교적 작은 부분, 즉 도메인 간 연결기와 사이드 체인만이 도메인 재배치에 따라 상당한 순응적 변화를 겪는다.[23]
2차 구조물에 의한 힌지
헤이워드의[24] 연구는 α-헬리케스와 β-시트의 종단부가 많은 경우에 힌지를 형성한다는 것을 발견했다. 많은 경첩이 도어의 경첩처럼 작용하는 2차 구조 요소를 포함하고 있어 개폐 동작이 일어날 수 있는 것으로 밝혀졌다. 이것은 한 영역에 위치한 β-시트 내의 두 개의 인접한 가닥이 다른 영역에 결합하면서 분리될 때 발생할 수 있다. 그 결과 두 개의 종단부는 두 도메인 사이의 벤딩 영역을 형성한다. 휘었을 때 수소 본딩 네트워크를 보존하는 ααes가 기계적 힌지 역할을 하는 것으로 밝혀져 기질을 빠르게 포착하기 위해 도메인의 폐쇄를 구동하는 '촉매 에너지'를 저장한다.[24]
연장 순응에 나선형
영역 경계 현장에서 나선형 및 확장형 순응의 상호 변환은 드물지 않다. Calmodulin에서는 α-헬릭스(α-helix)를 연결하는 영역의 중간에 있는 5개의 잔류물에 대해 토션 각도가 변화한다. 나선은 거의 수직인 두 개의 작은 나선형으로 나뉘며, 확장된 가닥의 네 개의 잔여물로 분리된다.[25][26]
전단 운동
전단 모션은 인터페이스 내의 아미노산 사이드 체인에 의해 제어되는 도메인 인터페이스의 작은 슬라이딩 이동을 포함한다. 전단 운동을 나타내는 단백질은 종종 2차 구조의 쌓기라는 계층 구조를 가지고 있다. 도메인 간 링커는 도메인을 근접하게 유지하는 역할만 할 뿐이다.[citation needed]
효소의 도메인 운동 및 기능적 역학
구조적으로 다르지만 기능적으로 유사한 효소의 내부 역학 분석은 활성 부위의 위치설정과 두 개의 주요 단백질 하위 영역 사이의 공통 관계를 강조하였다. 실제로 수산화물 슈퍼 패밀리의 여러 구성원에 대해 촉매 현장은 두 개의 주요 준강체 도메인을 분리하는 인터페이스 가까이에 위치한다.[13] 이러한 위치는 두 하위 도메인의 상대적인 움직임으로 인한 측면 부위의 기능 지향적 변조를 허용하는 동시에 능동 부위의 정확한 형상을 유지하는 데 중요한 것으로 보인다.
고분자 진화에 대한 시사점
증거는 단백질 역학이 DHFR의 효소 카탈루션과 같은 기능에 중요하지만, 그것들은 또한 분자 진화에 의한 새로운 기능의 획득을 용이하게 하기 위해 존재한다는 것을 암시한다.[27] 이 주장은 단백질이 안정적이고 대부분 독특한 접힌 구조를 갖도록 진화해왔다는 것을 암시하지만, 피할 수 없는 잔류 유연성은 어느 정도 기능적 난잡함으로 이어지며, 이는 이후의 돌연변이에 의해 증폭/해상/분해될 수 있다.
그러나 진핵 게놈에는 본질적으로 구조화되지 않은 단백질이 상당히 널리 퍼져 있다는 인식이 확산되고 있어 [28]안핀센의 독단어인 '시퀀스가 구조를 결정한다(가수)'의 가장 간단한 해석에 더욱 의구심을 제기하고 있다. 사실상, 새로운 패러다임은 "시퀀스와 세포 환경이 구조 앙상블을 결정한다"라는 두 가지 주의사항이 추가된 것이 특징이다.
참조
- ^ a b Bu Z, Callaway DJ (2011). "Proteins move! Protein dynamics and long-range allostery in cell signaling". Protein Structure and Diseases. Advances in Protein Chemistry and Structural Biology. 83. pp. 163–221. doi:10.1016/B978-0-12-381262-9.00005-7. ISBN 9780123812629. PMID 21570668.
- ^ Fraser JS, Clarkson MW, Degnan SC, Erion R, Kern D, Alber T (Dec 2009). "Hidden alternative structures of proline isomerase essential for catalysis". Nature. 462 (7273): 669–673. Bibcode:2009Natur.462..669F. doi:10.1038/nature08615. PMC 2805857. PMID 19956261.
- ^ Frauenfelder H, Sligar SG, Wolynes PG (Dec 1991). "The energy landscapes and motions of proteins". Science. 254 (5038): 1598–1603. Bibcode:1991Sci...254.1598F. doi:10.1126/science.1749933. PMID 1749933.
- ^ Dunbrack, Roland L (August 2002). "Rotamer Libraries in the 21st Century". Current Opinion in Structural Biology. 12 (4): 431–440. doi:10.1016/s0959-440x(02)00344-5. PMID 12163064.
- ^ Davis IW, Arendall WB, Richardson DC, Richardson JS (Feb 2006). "The backrub motion: how protein backbone shrugs when a sidechain dances". Structure. 14 (2): 265–274. doi:10.1016/j.str.2005.10.007. PMID 16472746.
- ^ Fraser JS, van den Bedem H, Samelson AJ, Lang PT, Holton JM, Echols N, Alber T (Sep 2011). "Accessing protein conformational ensembles using room-temperature X-ray crystallography". Proceedings of the National Academy of Sciences of the United States of America. 108 (39): 16247–16252. Bibcode:2011PNAS..10816247F. doi:10.1073/pnas.1111325108. PMC 3182744. PMID 21918110.
- ^ Turton DA, Senn HM, Harwood T, Lapthorn AJ, Ellis EM, Wynne K (June 2014). "Terahertz underdamped vibrational motion governs protein-ligand binding in solution". Nature Communications. 5: 3999. Bibcode:2014NatCo...5.3999T. doi:10.1038/ncomms4999. PMID 24893252.
- ^ Bu Z, Cook J, Callaway DJ (Sep 2001). "Dynamic regimes and correlated structural dynamics in native and denatured alpha-lactalbumin". Journal of Molecular Biology. 312 (4): 865–873. doi:10.1006/jmbi.2001.5006. PMID 11575938.
- ^ a b Costa CH, Oliveira AR, Dos Santos AM, da Costa KS, Lima AH, Alves CN, Lameira J (October 2019). "Computational study of conformational changes in human 3-hydroxy-3-methylglutaryl coenzyme reductase induced by substrate binding". Journal of Biomolecular Structure & Dynamics. 37 (16): 4374–4383. doi:10.1080/07391102.2018.1549508. PMID 30470158. S2CID 53717806.
- ^ Groban ES, Narayanan A, Jacobson MP (April 2006). Shakhnovich E (ed.). "Conformational changes in protein loops and helices induced by post-translational phosphorylation". PLOS Computational Biology. 2 (4): e32. Bibcode:2006PLSCB...2...32G. doi:10.1371/journal.pcbi.0020032. PMC 1440919. PMID 16628247.
- ^ Farago B, Li J, Cornilescu G, Callaway DJ, Bu Z (Nov 2010). "Activation of nanoscale allosteric protein domain motion revealed by neutron spin echo spectroscopy". Biophysical Journal. 99 (10): 3473–3482. Bibcode:2010BpJ....99.3473F. doi:10.1016/j.bpj.2010.09.058. PMC 2980739. PMID 21081097.
- ^ Bu Z, Biehl R, Monkenbusch M, Richter D, Callaway DJ (Dec 2005). "Coupled protein domain motion in Taq polymerase revealed by neutron spin-echo spectroscopy" (PDF). Proceedings of the National Academy of Sciences of the United States of America. 102 (49): 17646–17651. Bibcode:2005PNAS..10217646B. doi:10.1073/pnas.0503388102. PMC 1345721. PMID 16306270.
- ^ a b Potestio R, Pontiggia F, Micheletti C (Jun 2009). "Coarse-grained description of protein internal dynamics: an optimal strategy for decomposing proteins in rigid subunits". Biophysical Journal. 96 (12): 4993–5002. Bibcode:2009BpJ....96.4993P. doi:10.1016/j.bpj.2009.03.051. PMC 2712024. PMID 19527659.
- ^ Baron R, Vellore NA (Jul 2012). "LSD1/CoREST is an allosteric nanoscale clamp regulated by H3-histone-tail molecular recognition". Proceedings of the National Academy of Sciences of the United States of America. 109 (31): 12509–14. Bibcode:2012PNAS..10912509B. doi:10.1073/pnas.1207892109. PMC 3411975. PMID 22802671.
- ^ Ponte-Sucre A, ed. (2009). ABC Transporters in Microorganisms. Caister Academic. ISBN 978-1-904455-49-3.
- ^ Kamerlin SC, Warshel A (May 2010). "At the dawn of the 21st century: Is dynamics the missing link for understanding enzyme catalysis?". Proteins. 78 (6): 1339–75. doi:10.1002/prot.22654. PMC 2841229. PMID 20099310.
- ^ Howard J (2001). Mechanics of motor proteins and the cytoskeleton (1st ed.). Sunderland,MA: Sinauer Associates. ISBN 9780878933334.
- ^ Callaway DJ, Matsui T, Weiss T, Stingaciu LR, Stanley CB, Heller WT, Bu Z (Apr 2017). "Controllable Activation of Nanoscale Dynamics in a Disordered Protein Alters Binding Kinetics". Journal of Molecular Biology. 429 (7): 987–998. doi:10.1016/j.jmb.2017.03.003. PMC 5399307. PMID 28285124.
- ^ a b Gerstein M, Lesk AM, Chothia C (Jun 1994). "Structural mechanisms for domain movements in proteins". Biochemistry. 33 (22): 6739–49. doi:10.1021/bi00188a001. PMID 8204609.
- ^ Nicholl ID, Matsui T, Weiss TM, Stanley CB, Heller WT, Martel A, Farago B, Callaway DJ, Bu Z (Aug 21, 2018). "Alpha-catenin structure and nanoscale dynamics in solution and in complex with F-actin". Biophysical Journal. 115 (4): 642–654. Bibcode:2018BpJ...115..642N. doi:10.1016/j.bpj.2018.07.005. hdl:2436/621755. PMC 6104293. PMID 30037495.
- ^ Voet D (2011). Biochemistry. Voet, Judith G. (4th ed.). Hoboken, NJ: John Wiley & Sons. ISBN 9780470570951. OCLC 690489261.
- ^ Herzberg O, Chen CC, Kapadia G, McGuire M, Carroll LJ, Noh SJ, Dunaway-Mariano D (Apr 1996). "Swiveling-domain mechanism for enzymatic phosphotransfer between remote reaction sites". Proceedings of the National Academy of Sciences of the United States of America. 93 (7): 2652–7. Bibcode:1996PNAS...93.2652H. doi:10.1073/pnas.93.7.2652. PMC 39685. PMID 8610096.
- ^ Janin J, Wodak SJ (1983). "Structural domains in proteins and their role in the dynamics of protein function". Progress in Biophysics and Molecular Biology. 42 (1): 21–78. doi:10.1016/0079-6107(83)90003-2. PMID 6353481.
- ^ a b Hayward S (Sep 1999). "Structural principles governing domain motions in proteins". Proteins. 36 (4): 425–35. doi:10.1002/(SICI)1097-0134(19990901)36:4<425::AID-PROT6>3.0.CO;2-S. PMID 10450084.
- ^ Meador WE, Means AR, Quiocho FA (Aug 1992). "Target enzyme recognition by calmodulin: 2.4 A structure of a calmodulin-peptide complex". Science. 257 (5074): 1251–1255. Bibcode:1992Sci...257.1251M. doi:10.1126/science.1519061. PMID 1519061.
- ^ Ikura M, Clore GM, Gronenborn AM, Zhu G, Klee CB, Bax A (May 1992). "Solution structure of a calmodulin-target peptide complex by multidimensional NMR". Science. 256 (5057): 632–638. Bibcode:1992Sci...256..632I. doi:10.1126/science.1585175. PMID 1585175.
- ^ Tokuriki N, Tawfik DS (Apr 2009). "Protein dynamism and evolvability". Science. 324 (5924): 203–207. Bibcode:2009Sci...324..203T. doi:10.1126/science.1169375. PMID 19359577. S2CID 28576496.
- ^ Dyson HJ, Wright PE (Mar 2005). "Intrinsically unstructured proteins and their functions". Nature Reviews Molecular Cell Biology. 6 (3): 197–208. doi:10.1038/nrm1589. PMID 15738986. S2CID 18068406.