중성자 스핀 에코
Neutron spin echo| 중성자를 이용한 과학 |
|---|
 |
| 기초 |
| 중성자 산란 |
| 기타 응용 프로그램 |
| 사회 기반 시설 |
| 중성자 시설 |
중성자 스핀 에코 분광법은 1970년대 Ferenc Mezei에 의해 발명되어 John Hayter와 공동으로 개발된 비탄성 중성자 산란 기술이다.[1]그의 업적과 다른 분야에서, Mezei는 1999년에 제1회 Walter Haelg 상을 받았습니다.
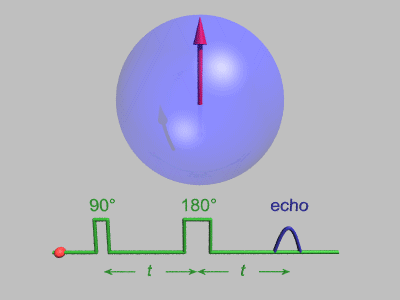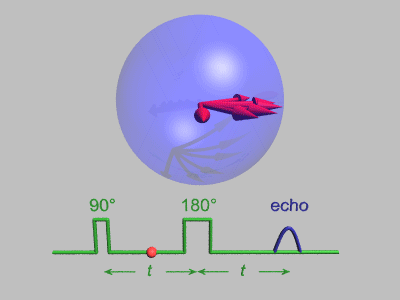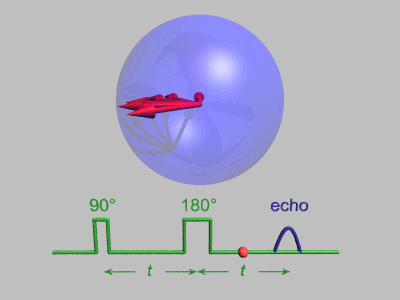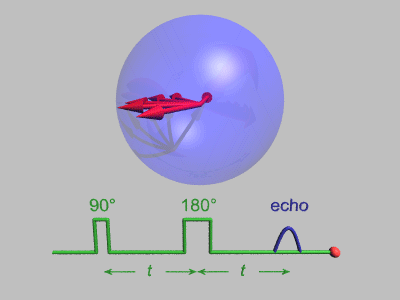
자기 공진에서 스핀 에코(spin echo)는 공명 전자기 방사 펄스에 의한 스핀 자화의 재초점화이다.스핀 에코 분광계는 매우 높은 에너지 분해능을 가지고 있습니다(대략 100,000분의 1).또한 운동량 전달 Q 및 시간의 함수로서 밀도-밀도 상관(또는 중간 산란 함수) F(Q,t)를 측정한다.다른 중성자 산란 기술은 실제로 어려울 수 있는 푸리에 변환에 의해 F(Q,t)로 변환될 수 있는 동적 구조 인자 S(Q,θ)를 측정한다.약한 비탄성 특징의 경우 S(Q,θ)가 더 적합하지만, (느린) 이완의 경우 자연 표현은 F(Q,t)로 주어진다.NSE는 다른 중성자 산란 기술에 비해 매우 높은 유효 에너지 분해능으로 인해 폴리머 블렌드, 알칸 체인 또는 마이크로 에멀션과 같은 재료에서 과잉 감쇠된 내부 동적 모드(완화) 및 기타 확산 과정을 관찰하는 데 이상적인[2] 방법이다.NSE 분광법의[3] 비범한 힘은 최근 NHERF1과 Taq 중합효소 단백질의 결합 내부 단백질 역학과 아데렌스 [6]접합부의 직접 관찰에 의해[4][5] 더욱 입증되었으며, 이는 단백질 나노 기계의 움직임을 직접 시각화할 수 있게 했다.기법에 대한 몇 가지 기본적인 검토가 존재한다.[7][8][9][10][11]
구조
중성자 스핀 에코(spin echo)는 비행시간 기술이다.중성자 스핀에 관해서는 NMR 분야에서 잘 알려진 이른바 Hahn [12]echo와 매우 유사하다. 두 경우 모두 스핀의 소멸에 의한 편광 손실(자화)은 효과적인 시간 반전 연산에 의해 복원되어 편광의 회복(반복)으로 이어진다.NMR에서 디페이징은 핵 위치에서의 국소장 변화로 인해 발생하며, NSE에서 디페이징은 들어오는 중성자 빔의 다른 중성자 속도로 인해 발생한다.샘플이 빔에서 중성자의 개별 속도를 세차 각도로 인코딩하기 전에 자기장이 있는 준비 구역에서 중성자 스핀의 라모르 세차.샘플에 가까운 시간 반전은 이른바 플리퍼에 의해 영향을 받습니다.대칭 복호화 구역은 준비 구역에서 축적된 세차 운동 각도가 정확히 보정되도록 한다(샘플이 중성자 속도를 바꾸지 않은 경우, 즉 탄성 산란), 모든 스핀이 "spin-echo"를 형성하기 위해 위상을 바꾼다.이상적으로는 완전한 편광은 복원됩니다.이 영향은 들어오는 중성자의 속도/에너지/파장에 의존하지 않는다.표본의 산란이 탄성이 없지만 중성자 속도가 변화하면, 재위즈가 불완전해지고 최종 편파 결과가 손실된다. 이는 중성자가 대칭적인 첫 번째(부호화) 및 두 번째(복호화) 세차 영역을 통과하는 데 필요한 시간 차이의 분포에 따라 달라진다.시료의 비탄성 산란으로 얻은 속도 변화로 인해 시간 차이가 발생한다.이러한 시간 차이의 분포는 (준탄성 고해상도 분광법에 적합한 선형화 근사에서) 산란 함수 S(Q,θ)의 스펙트럼 부분에 비례한다.측정된 빔 편광에 대한 영향은 스펙트럼 함수인 중간 산란 함수 F(Q,t)의 cos-Fourier 변환에 비례합니다.시간 매개변수는 중성자 파장과 세차각과 (반복) 속도를 연결하는 인자에 따라 달라지며, 예를 들어 준비 및 디코딩 구역에서 특정 자기장을 설정하여 제어할 수 있다.그런 다음 자기장을 변화시켜 t 스캔을 수행할 수 있습니다.
주의할 점은 모든 스핀 조작은 측정된 강도의 스펙트럼 함수의 푸리에 변환 측면에서 기술적인 이유로 영향을 미치는 중성자의 속도 변화를 감지하는 수단일 뿐이라는 점이다.중성자의 속도 변화는 NSE를 사용하여 얻을 수 있는 물리적 정보를 전달한다.
(Q ,t ) ( Q ) + " " ") ( ," ) \ style ( , t ) + \\ cos ( \ 서 ( , \ ) \ 。
B는 세차 운동장 강도, δ (평균) 중성자 파장 및 δV 시료 산란 시 중성자 속도 변화를 나타낸다.
NSE를 사용하는 주된 이유는 위의 방법으로 최대 100ns의 푸리에 시간에 도달할 수 있으며, 이는 neV 범위의 에너지 분해능에 해당하기 때문입니다.분광 중성자 계측기 유형, 즉 후방 산란 분광계(BSS)에 의한 이 분해능에 가장 가까운 접근법은 0.5~1μeV 범위이다.스핀 에코 트릭은 파장 분포가 10% 이상인 강력한 중성자 빔을 사용하는 동시에 10 미만의−4 범위에서의 속도 변화에 민감하게 반응할 수 있다.
주의: 위의 설명은 Institut Laue-Langevin(IL)의 IN11 계측기에서 최초로 사용된 일반적인 NSE 구성을 가정한 것입니다.공진 스핀 에코, DC 필드가 집중된 NRSE 및 준비 및 디코딩 구역의 마지막에 리플리퍼에 RF 필드가 포함된 다른 접근법도 가능하며, 이 접근법은 자기장(제로 필드)이 없습니다.원칙적으로 이러한 접근법은 최종 강도 신호와 중간 산란 기능의 연결에 대해 동등하다.지금까지 기술적인 문제로 인해 일반적인(IN11) NSE [citation needed]유형과 동일한 수준의 성능을 달성하지 못했습니다.
측정할 수 있는 것
연질 물질 연구에서 고분자 물체의 구조는 종종 소각 중성자 산란(SANS)에 의해 조사된다.몇몇 분자에서 수소와 중수소의 교환은 심지어 동일한 화학종들 사이에 산란 대비를 만들어낸다.SANS 회절 패턴(실제 공간에서 해석되는 경우)은 분자 배열의 스냅샷 사진에 해당합니다.중성자 스핀 에코 기기는 SAN 강도의 비탄성적 확대를 분석하여 고분자 [13]물체의 움직임을 분석할 수 있다.대략적인 예로 스냅샷과 같은 SAN이 아닌 특정 오픈 시간을 가진 사진을 들 수 있습니다.그래서 우리는 분자의 배열의 변화를 시간의 함수로 분석할 수 있습니다.개방 시간은 NSE 분광계의 설정에 따라 달라지는 푸리에 시간에 해당하며, 자기장(적분) 및 중성자 파장의 세 번째 거듭제곱에 비례합니다.최대 수백 나노초의 값을 사용할 수 있습니다.산란 실험의 공간 분해능은 나노미터 범위에 있으며, 이는 예를 들어 100ns의 시간 범위가 1nm/100ns = 1cm/s의 유효 분자 운동 속도에 해당함을 의미한다.이는 전형적인 중성자 속도인 200과 비교될 수 있다.이러한 유형의 실험에 사용된 1000m/s.
NSE 및 스핀-인코히런트 산란(양자로부터의)
이 섹션은 확장해야 합니다.추가해서 도와주시면 됩니다. (2022년 1월) |
정상 비행 시간(TOF) 또는 후방 산란 분광계를 사용하는 많은 비탄성 연구는 양성자의 거대한 일관성 중성자 산란 단면에 의존한다.산란 신호는 양성자의 (평균) 자기 상관 함수(시간)를 나타내는 해당 기여에 의해 지배된다.
NSE 스핀의 경우 비간섭 산란은 2/3 확률로 산란 중에 중성자 스핀을 뒤집는 단점이 있다.따라서 산란 강도의 2/3을 "편광되지 않은" 배경으로 변환하고 일관성이 없는 강도와 관련된 cos-Fourier 적분 기여 앞에 -1/3의 계수를 둡니다.이 신호는 코히런트 에코 신호에서 감산됩니다.그 결과 NSE만 사용할 경우 분해할 수 없는 복잡한 조합이 발생할 수 있습니다.그러나 순수한 경우, 즉 양성자로 인한 압도적인 강도 기여가 있는 경우 NSE를 사용하여 일관성이 없는 스펙트럼을 측정할 수 있다.
예를 들어 연질 물질 샘플에 대한 NSE의 강도 상황은 소각 중성자 산란(SANs)과 동일하다.저운동량 전달(Q)에서 간섭성 산란 대비가 있는 분자 물체는 간섭성 배경 산란보다 상당히 높은 강도로 간섭성 산란을 나타낸다.이 효과는 Q가 커질수록 약해집니다.수소를 포함하는 시스템의 경우 대비는 일부 양성자의 존재를 필요로 하며, 이는 산란 강도에 일정량의 일관되지 않은 기여를 반드시 추가한다.게다가 듀테론조차 약한 스핀 불간섭 산란 강도를 더한다.SANS에서는 Q에 의존하지 않는 이러한 강도가 일반적으로 배경으로 간주되고 감산됩니다.NSE 실험에서는 이러한 혼합물이 존재하며 Q가 증가할수록 더 유의한 혼합물이 될 수 있습니다.
완전 양성자화된 샘플은 SAN 백그라운드 [14]레벨의 강도로 일관성이 없는 측정을 가능하게 한다.참고: NSE 기술의 스핀 조작에 대한 이러한 간섭은 스핀 비간섭 산란에서만 발생합니다.동위원소 비간섭 산란은 "정상" NSE 신호를 생성합니다.
기존 분광계
IN11(IL, 그르노블, 프랑스)
IN15(IL, 그르노블, 프랑스)
NL2a J-NSE "PHOENIX" (독일 JCNS, Juelich, FRM II 뮌헨 주최, 독일 뮌헨 주최)
NL5-S RESEDA (FRM II 뮌헨, 독일 뮌헨)
V5/SPAN(독일 베를린 한마이트너 연구소)
C2-3-1 iNSE(일본 도카이 JRR-3)
BL06 VIN-ROSE(MLF, J-PARC, 도카이, 일본)
BL-15 NSE(미국 Oak Ridge, ORNL, SNS)
NG5-NSE(NCNSR, NIST, Gaithersburg, 미국)
「 」를 참조해 주세요.
레퍼런스
- ^ Mezei, F., ed. (1980). Neutron Spin Echo. Lecture Notes in Physics Vol. 128. Berlin, Heidelberg, New York: Springer.
- ^ B. Farago (2006). "Neutron spin echo study of well organized soft matter systems". Physica B. 385–386: 688–691. Bibcode:2006PhyB..385..688F. doi:10.1016/j.physb.2006.05.292.
- ^ Callaway, D. J.; Farago, B; Bu, Z (2013). "Nanoscale protein dynamics: A new frontier for neutron spin echo spectroscopy". The European Physical Journal E. 36 (7): 76. doi:10.1140/epje/i2013-13076-1. PMID 23884624. S2CID 10246098.
- ^ B. Farago, Li J, Cornilescu G, Callaway DJE, Bu Z (November 2010). "Activation of Nanoscale Allosteric Protein Domain Motion Revealed by Neutron Spin Echo Spectroscopy". Biophysical Journal. 99 (10): 3473–3482. Bibcode:2010BpJ....99.3473F. doi:10.1016/j.bpj.2010.09.058. PMC 2980739. PMID 21081097.
{{cite journal}}: CS1 maint: 여러 이름: 작성자 목록(링크) - ^ Bu Z, Biehl R, Monkenbusch M, Richter D, Callaway DJE (2005). "Coupled protein domain motion in Taq polymerase revealed by neutron spin-echo spectroscopy". Proc Natl Acad Sci USA. 102 (49): 17646–17651. Bibcode:2005PNAS..10217646B. doi:10.1073/pnas.0503388102. PMC 1345721. PMID 16306270.
{{cite journal}}: CS1 maint: 여러 이름: 작성자 목록(링크) - ^ Farago B, Nicholl ID, Wang S, Cheng X, Callaway DJ, Bu Z (March 30, 2021). "Activated nanoscale actin-binding domain motion in the catenin-cadherin complex revealed by neutron spin echo spectroscopy". Proc Natl Acad Sci USA. 118 (13): e2025012118. Bibcode:2021PNAS..11825012F. doi:10.1073/pnas.2025012118. PMC 8020631. PMID 33753508.
- ^ L. Kay Nicholson (1981). "The neutron spin-echo spectrometer: A new high resolution technique in neutron scattering". Contemp. Phys. 22 (4): 451–475. Bibcode:1981ConPh..22..451N. doi:10.1080/00107518108231544.
- ^ Higgins JS, Benoit HC (1997). Polymers and neutron scattering. Oxford Series on Neutron Scattering in Condensed Matter (Book 8). Clarendon Press. ISBN 978-0198500636.
- ^ Callaway DJ, Bu Z (2017). "Visualizing the nanoscale: protein internal dynamics and neutron spin echo spectroscopy". Curr. Opin. Struct. Biol. 42: 1–5. doi:10.1016/j.sbi.2016.10.001. PMC 5374024. PMID 27756047.
- ^ Richter D (2006). "Neutron spin echo for the exploration of large scale macromolecular dynamics". J. Phys. Soc. Jpn. 75 (11): 110041–11004112. Bibcode:2006JPSJ...75k1004R. doi:10.1143/JPSJ.75.111004.
- ^ Jacrot, B (1976). "The study of biological structures by neutron scattering from solution". Reports on Progress in Physics. 39 (10): 911–53. Bibcode:1976RPPh...39..911J. doi:10.1088/0034-4885/39/10/001.
- ^ E.L. Hahn (1950). "Spin Echoes". Physical Review. 80 (4): 580. Bibcode:1950PhRv...80..580H. doi:10.1103/PhysRev.80.580.
- ^ M. Monkenbusch & D. Richter (2007). "High resolution neutron spectroscopy - a tool for the investigation of dynamics of polymers and soft matter". Comptes Rendus Physique. 8 (7–8): 845–864. Bibcode:2007CRPhy...8..845M. doi:10.1016/j.crhy.2007.10.001.
- ^ A. Wischnewski and M. Monkenbusch and L. Willner and D. Richter and G. Kali (2003). "Direct observation of the transition from free to constrained single-segment motion in entangled polymer melts". Physical Review Letters. 90 (5): 058302. Bibcode:2003PhRvL..90e8302W. doi:10.1103/PhysRevLett.90.058302. PMID 12633402.



