파이로시퀀싱
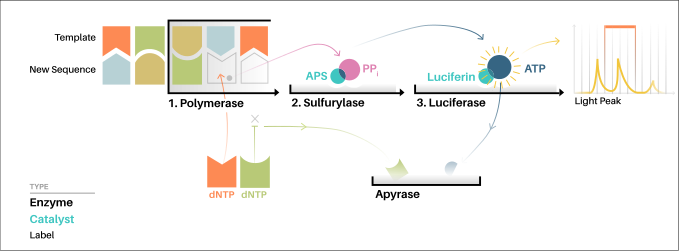
Pyrosequencing파이로시퀀싱은 '합성에 의한 염기서열결정' 원리에 기초한 DNA 염기서열결정법(DNA 내 뉴클레오티드의 순서를 결정하는 방법)으로, DNA 중합효소에 의해 함유된 뉴클레오티드를 검출함으로써 염기서열이 이루어진다.파이로시퀀싱은 피로인산염이 방출될 때 연쇄 반응에 기초한 빛 검출에 의존합니다.따라서 파이로시퀀싱이라는 이름이 붙습니다.
pyrosequencing의 원칙 먼저 1993[1]에 Bertil Pettersson, 마티아스 Uhlen과 Pål Nyren이 고상 재조합 DNA중합 효소는 반딧불이luciferase enzym을 사용하여 3´5´exonuclease 활동(proof-reading)과 발광 검출이 streptavidin를 막론하고 코팅 자기 구슬을 사용하여 method[2]의 염기 서열을 결합시켜 묘사되었다.e.[3]3가지 효소(DNA 중합효소, ATP 술푸릴라아제 및 반딧불 루시페라아제)와 뉴클레오티드(dNTP)를 혼합하여 배열하고, 이어서 뉴클레오티드의 혼입에 의해 방출되는 빛을 측정한다.빛의 세기는 0, 1 이상의 뉴클레오티드가 포함되었는지 여부를 결정하여 템플릿 가닥에 얼마나 많은 상보적인 뉴클레오티드가 존재하는지 보여준다.뉴클레오티드 혼합물은 다음 뉴클레오티드 혼합물이 첨가되기 전에 제거된다.이 과정은 단일 가닥 템플릿의 DNA 배열이 결정될 때까지 4개의 뉴클레오티드 각각에 대해 반복된다.
파이로시퀀싱을 위한 두 번째 솔루션 기반 방법은 1998년[4] Mostafa Ronaghi, Mathias Uhlen 및 Pöl Nyren에 의해 설명되었습니다.본 대체방법에서는 DNA 중합효소에 포함되지 않은 뉴클레오티드를 제거하기 위해 추가적인 효소 아피라아제가 도입된다.이를 통해 DNA 중합효소, 루시퍼라아제 및 아피라아제를 포함한 효소 혼합물이 처음에 첨가되고 시술 내내 유지되므로 자동화에 적합한 간단한 설정이 제공되었습니다.이 원리에 기초한 자동 계측기는 이듬해 파이로시퀀싱에 의해 시장에 출시되었습니다.
파이로시퀀싱 방법의 세 번째 미세 유체 변형은 2005년[5] Jonathan Rothberg와 454 Life Sciences의 동료들에 의해 설명되었습니다.파이로시퀀싱을 위한 이 대체 접근법은 DNA를 고체 지지대에 부착하는 원리에 기초하고 있었으며, 그들은 미세 가공된 마이크로어레이를 사용하여 매우 병렬적으로 시퀀싱을 수행할 수 있음을 보여주었다.이를 통해 높은 처리량 DNA 염기서열 분석을 가능하게 했고 자동화된 기기가 시장에 출시되었습니다.이는 게놈 연구의 새로운 시대를 여는 최초의 차세대 염기서열 분석 기구가 되었으며, DNA 염기서열 분석의 가격이 급격히 하락하면서 저렴한 가격에 전체 게놈 염기서열을 분석할 수 있게 되었다.
절차.
"합성에 의한 배열"은 배열될 DNA의 단일 가닥을 가져다가 그 상보적인 가닥을 효소적으로 합성하는 것을 포함한다.파이로시퀀싱 방법은 DNA 중합효소(DNA 합성효소)와 다른 화학발광효소의 활성을 검출하는 것에 기초하고 있다.본질적으로 본 방법은 상보적인 가닥을 따라 한 번에 한 쌍의 염기쌍을 합성하고 각 단계에서 실제로 첨가된 염기를 검출함으로써 DNA의 단일 가닥을 배열할 수 있다.템플릿 DNA는 움직이지 않으며, A, C, G, T 뉴클레오티드의 용액이 순차적으로 첨가되고 반응에서 제거된다.빛은 뉴클레오티드 용액이 템플릿의 첫 번째 비쌍 염기를 보완할 때만 발생한다.화학 발광 신호를 생성하는 용액 시퀀스에 따라 템플릿 [6]시퀀스를 결정할 수 있습니다.
단사슬 DNA(ssDNA) 템플릿은 배열 프라이머로 하이브리드화되어 DNA 중합효소, ATP 술푸릴라아제, 루시페라아제 및 아피라아제 및 기질 아데노신5'포스포황산(APS) 및 루시페인과 함께 배양된다.
- 4개의 디옥시뉴클레오티드 트리포스페이트(dNTPs) 중 하나를 첨가하면 두 번째 단계가 시작된다(dATP 대신 dATP αS가 잡음을 피하기 위해 루시페라아제 기질이 아니다).DNA 중합효소는 정확한 상보적 dNTP를 템플릿에 통합합니다.이 혼합물은 피로인산염(PPI)을 방출한다.
- ATP 술푸릴라아제는 아데노신 5' 포스포황산염의 존재 하에서 PPi를 ATP로 변환한다.이 ATP는 루시페린에서 옥시루시페린으로의 루시페라아제 매개 변환의 기질로 작용하며, 그 양에 비례하는 양의 가시광을 생성합니다.루시페라아제 촉매 반응에서 생성된 빛을 카메라로 검출하여 프로그램에서 분석합니다.
- 비혼합 뉴클레오티드와 ATP는 아피라아제에 의해 분해되며, 반응은 다른 뉴클레오티드와 함께 다시 시작될 수 있다.
프로세스는 다음 방정식으로 나타낼 수 있습니다.
- PPI + APS → ATP + 황산염(ATP-술푸릴라아제에 의해 촉매됨)
- ATP + 루시페린 + O2 → AMP + PPi + 옥시루시페린 + CO2 + hv( 루시페라아제에 의해 촉매됨)
여기서:
- PPi는 피로인산염이다
- APS는 아데노신 5-인황산염이다.
- ATP는 아데노신 삼인산이다.
- O2는 산소 분자이다.
- AMP는 아데노신 일인산염이다.
- CO2는 이산화탄소이다.
- hv는 가볍다.
제한 사항
현재 이 방법의 한계는 DNA 배열의 개별 판독 길이가 300~500뉴클레오티드 부근에 있으며, 이는 체인 종단 방법으로 얻을 수 있는 800~1000보다 짧다는 것이다(예: 생거 배열).이것은 게놈 조립 과정을 더욱 어렵게 만들 수 있으며, 특히 많은 양의 반복적인 DNA를 포함하는 배열의 경우 더욱 그렇습니다.교정 활동이 부족하면 이 방법의 정확도가 제한됩니다.
상용화
스웨덴 웁살라에 있는 Pyrosequencing AB사는 파이로시퀀싱 기술을 사용하여 짧은 길이의 DNA를 염기서열 분석하기 위한 기계와 시약을 상용화하기 위해 HealthCap이 제공한 벤처 캐피털로 설립되었습니다.파이로시퀀싱 AB는 1999년 스톡홀름 증권거래소에 상장되었다.2003년에 Biotage로 이름을 바꿨다.파이로시퀀싱 비즈니스 라인은 2008년에 키아겐에 인수되었습니다.파이로시퀀싱 기술은 454 Life Sciences에 한층 더 라이센스 되었습니다.454는 어레이 기반의 파이로시퀀싱 기술을 개발하여 게놈 염기서열 분석과 메타게노믹스를 포함한 대규모 DNA 염기서열의 플랫폼으로 부상했습니다.
Roche는 2013년에 [7]454 시퀀싱 플랫폼의 단종을 발표했습니다.
레퍼런스
- ^ Nyren, Pettersson 및 Uhlen(1993) "효소 발광계 무기 피로인산염 검출 어세이(Luminometric 무기물 검출 어세이)" 분석 생화학 208(1), 171-175, https://doi.org/10.1006/abio.1993.1024
- ^ Uhlen (표준) "DNA의 자기 분리" Nature 340 : 733-4, https://doi.org/10.1038/340733a0
- ^ Nyren 및 Lundin(1985) "무기 피로인산염 합성의 지속적인 모니터링을 위한 효소적 방법" 분석 생화학 151 (2): 504-509.https://doi.org/10.1016/0003-2697(85)90211-8
- ^ Ronaghi, Mostafa; Uhlén, Mathias; Nyrén, Pål (1998-07-17). "A Sequencing Method Based on Real-Time Pyrophosphate". Science. 281 (5375): 363–365. doi:10.1126/science.281.5375.363. PMID 9705713. S2CID 26331871.
- ^ Marguiles et al(2005) "미세조립 고밀도 피콜리트로의 유전자 배열" Nature 437, 376-380.https://doi.org/doi:10.1038/nature03959;
- ^ QIAGEN. "Pyrosequencing Technology and Platform Overview". Retrieved 4 August 2017.
- ^ Hollmer, Mark (October 17, 2013). "Roche to close 454 Life Sciences as it reduces gene sequencing focus". Fierce Biotech.
추가 정보
- Metzker M. (2005). "Emerging Technologies in DNA Sequencing". Genome Research. 15 (12): 1767–76. doi:10.1101/gr.3770505. PMID 16339375.