분자 복제
Molecular cloning분자 복제는 분자 생물학에서 재조합 DNA 분자를 조립하고 숙주 [1]유기체 내에서 그들의 복제를 지시하기 위해 사용되는 일련의 실험 방법입니다.복제라는 단어의 사용은 동일한 DNA 분자를 가진 세포 집단을 만들기 위해 한 분자의 복제를 수반한다는 사실을 의미한다.분자 복제는 일반적으로 두 개의 다른 유기체의 DNA 서열을 사용한다: 복제될 DNA의 원천인 종과 재조합 DNA의 복제를 위한 살아있는 숙주 역할을 할 종.분자 복제 방법은 현대 생물학과 [2]의학의 많은 현대 영역의 중심이다.
종래의 분자 복제 실험에서는, 대상 생물로부터 복제하는 DNA를 취득해, 시험관내에서 효소로 처리해, 보다 작은 DNA 단편을 생성한다.이어서 이들 단편은 벡터 DNA와 결합되어 재조합 DNA 분자를 생성한다.그런 다음 재조합 DNA는 숙주 유기체에 도입됩니다(일반적으로 대장균의 배양하기 쉽고 양성인 실험실 변종).이것은 숙주 DNA와 함께 재조합 DNA 분자가 복제되는 유기체 집단을 만들어 낼 것이다.이것들은 외래 DNA 조각들을 포함하고 있기 때문에 트랜스제닉 또는 유전자 변형 미생물이다.[3]이 과정은 단일 박테리아 세포가 단일 재조합 DNA 분자를 흡수하고 복제하도록 유도될 수 있다는 사실을 이용한다.그리고 나서 이 단일 세포는 기하급수적으로 확장되어 많은 양의 박테리아를 생성할 수 있으며, 각각의 박테리아는 원래의 재조합 분자의 복사본을 포함합니다.따라서, 결과 박테리아 집단과 재조합 DNA 분자는 일반적으로 "클론"이라고 불린다.엄밀히 말하면, 재조합 DNA는 DNA 분자를 지칭하는 반면, 분자 복제는 그것들을 조립하기 위해 사용되는 실험적인 방법을 말한다.다른 DNA 서열이 플라스미드에 삽입될 수 있고 이 외부 서열이 박테리아로 옮겨져 플라스미드의 일부로 소화된다는 생각이 떠올랐다.즉,[4] 이 플라스미드는 유전자를 운반하는 복제 벡터 역할을 할 수 있다.
사실상 모든 DNA 염기서열이 복제되고 증폭될 수 있지만, 이 과정의 성공을 제한할 수 있는 몇 가지 요소들이 있다.복제하기 어려운 DNA 배열의 예로는 역반복, 복제의 기원, 동원체 및 텔로미어가 있다.또한 큰 크기의 DNA 배열을 삽입할 때 성공할 확률이 낮습니다.10kbp보다 큰 삽입은 성공이 매우 제한적이지만, 박테리오파지 can 등의 박테리오파지를 수정하여 최대 40kbp까지 [5]시퀀스를 삽입할 수 있습니다.
역사
1970년대 이전에는 유전학과 분자생물학의 이해는 복잡한 유기체로부터 개별 유전자를 분리하여 연구할 수 없었기 때문에 심각하게 방해를 받았다.이것은 분자 복제 방법의 출현과 함께 극적으로 변화했다.미생물학자들은 박테리아가 박테리오파지의 성장을 제한하는 분자 메커니즘을 이해하려고 노력하며, 분리된 제한 핵산 분해효소, 특정한 DNA 배열이 [6]있을 때만 DNA 분자를 쪼갤 수 있는 효소들을 이해하려고 한다.그들은 제한 효소가 특정 위치에서 염색체 길이의 DNA 분자를 쪼개고, 더 큰 분자의 특정 부분이 크기 분화에 의해 정제될 수 있다는 것을 보여주었다.두 번째 효소인 DNA 연결효소를 사용하여, 제한 효소에 의해 생성된 조각들은 재조합 DNA라고 불리는 새로운 조합에 결합될 수 있다.박테리오파지나 플라스미드와 같이 박테리아 내부에서 자연적으로 복제되는 벡터 DNA와 관심 DNA 세그먼트를 재조합함으로써, 정제된 재조합 DNA 분자가 박테리아 배양에서 대량으로 생산될 수 있었다.최초의 재조합 DNA 분자는 [7][8]1972년에 생성되어 연구되었다.
개요
분자 복제는 DNA의 화학적 구조가 모든 생물에서 근본적으로 같다는 사실을 이용한다.따라서, 어떤 유기체로부터의 DNA의 어떤 부분이 DNA 복제에 필요한 분자 배열을 포함하는 DNA 세그먼트에 삽입되고, 그 결과 생성된 재조합 DNA가 복제 배열을 얻은 유기체에 도입된다면, 외부 DNA는 트랜지스터에서 숙주 세포의 DNA와 함께 복제될 것이다.에닉 유기체
분자 복제는 DNA 서열의 복제를 허용한다는 점에서 중합효소 연쇄 반응과 유사하다.두 방법의 근본적인 차이점은 분자 복제는 살아있는 미생물의 DNA 복제를 수반하는 반면 PCR은 살아있는 세포 없이 체외 용액에서 DNA를 복제한다는 것이다.
실리코 복제 및 시뮬레이션에서
실제 복제 실험이 실험실에서 이루어지기 전에, 대부분의 복제 실험은 특수 소프트웨어를 사용하여 컴퓨터에서 계획된다.클로닝의 자세한 계획은 모든 텍스트 편집기에서 수행할 수 있지만 PCR 프라이머 설계와 같은 온라인 유틸리티와 함께 목적을 위한 전용 소프트웨어가 존재합니다.이를 위한 소프트웨어에는 ApE [1](오픈 소스), DNAStrider [2](오픈 소스), 시리얼 클로너 [3](무료), 콜라진 [4](오픈 소스), SnapGene(상용) 등이 있습니다.이러한 프로그램을 통해 PCR 반응, 제한 다이제스트, 연결 등을 시뮬레이션할 수 있습니다. 즉, 아래에 설명된 모든 단계를 시뮬레이션할 수 있습니다.
순서
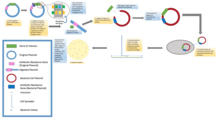
표준 분자 복제 실험에서 DNA 조각의 복제는 기본적으로 7단계를 포함한다. (1) 숙주 생물과 복제 벡터의 선택 (2) 벡터 DNA의 준비 (3) 복제할 DNA의 준비 (4) 숙주 생물로의 재조합 DNA의 생성 (5) 재조합 DNA의 도입 (6) 유기체의 선택.g재조합 DNA, (7) 원하는 DNA 삽입 및 생물학적 특성을 가진 클론 선별.
특히 DNA 합성 플랫폼의 용량과 충실도가 높아짐에 따라 분자 공학에서 점점 더 복잡한 설계가 가능해졌습니다.이러한 프로젝트에는 매우 긴 가닥의 새로운 DNA 염기서열 및/또는 전체 라이브러리를 개별 염기서열이 아닌 동시에 테스트할 수 있다.이러한 변화는 설계가 평평한 뉴클레오티드 기반 표현에서 벗어나 더 높은 수준의 추상화로 이동해야 하는 복잡성을 야기합니다.이러한 도구의 예로는 GenoCAD, Teselagen [5](학계 무료) 또는 GeneticConstructor [6](학계 무료)가 있습니다.
숙주 유기체 및 클로닝 벡터 선택
비록 매우 많은 숙주 유기체와 분자 복제 벡터가 사용되고 있지만, 분자 복제 실험의 대부분은 박테리아 대장균의 실험실 변종과 플라스미드 복제 벡터로 시작합니다.대장균과 플라스미드 벡터는 기술적으로 정교하고, 다용도적이며, 널리 이용 가능하며,[3] 최소한의 장비로 재조합 생물의 빠른 성장을 제공하기 때문에 일반적으로 사용됩니다.복제할 DNA가 예외적으로 클 경우(수십만에서 수백만 개의 염기쌍), 세균성 인공염색체[10] 또는 효모 인공염색체 벡터가 선택된다.
특수한 애플리케이션에서는, 특수한 호스트 벡터 시스템이 필요하게 되는 경우가 있습니다.예를 들어, 실험자가 재조합 생물로부터 특정 단백질을 수확하고 싶다면, 원하는 숙주 생물에서 전사 및 번역을 위한 적절한 신호를 포함하는 발현 벡터가 선택된다.또는 다른 종에서 DNA의 복제가 바람직한 경우(예를 들어 박테리아에서 식물로의 DNA 이동), 다중 숙주 범위 벡터(셔틀 벡터라고도 함)를 선택할 수 있다.하지만 실제로, 전문화된 분자 복제 실험은 보통 박테리아 플라스미드로 복제하는 것으로 시작하고, 그 후에 전문화된 벡터로 하위 복제한다.
호스트와 벡터의 어떤 조합이 사용되든 간에 벡터는 거의 항상 기능 및 실험 [3]효용에 매우 중요한 4개의 DNA 세그먼트를 포함합니다.
- DNA 복제의 기원은 벡터(및 그 연결된 재조합 배열)가 숙주 생물 내에서 복제하기 위해 필요하다.
- 외래 DNA가 도입될 수 있는 부위로서 기능하는 하나 이상의 고유 제한 핵산가수분해효소 인식 부위
- 벡터 배열을 차지한 세포의 생존을 가능하게 하는 데 사용할 수 있는 선택 가능한 유전자 표지자 유전자
- 외래 DNA를 포함한 세포를 선별하는 데 사용할 수 있는 태그 유전자
벡터 DNA의 작성
클로닝 벡터는 외부 DNA가 삽입되는 부위에서 DNA를 절단하기 위해 제한적 핵산가수분해효소로 처리된다.제한 효소는 외부 DNA의 끝과 호환되는 분할 부위에서 구성을 생성하기 위해 선택됩니다(DNA 끝 참조).일반적으로, 이것은 벡터 DNA와 외부 DNA를 동일한 제한 효소(예: EcoRI)로 분해함으로써 이루어집니다.대부분의 현대 벡터는 벡터 분자 내에서 독특하고(그래서 벡터는 한 곳에서만 절단할 수 있다) 다양한 편리한 절단 부위를 포함하고 있으며, 그 과정의 후반 단계에서 불활성화 생물과 재조합을 구별하기 위해 사용될 수 있는 유전자(흔히 베타 갈락토시다아제) 내에 위치한다.재조합체와 비재조합체의 비율을 개선하기 위해 분해된 벡터는 벡터 말단을 탈인산화효소(알칼린 포스파타아제)로 처리해도 된다.탈인산화 말단을 가진 벡터 분자는 복제할 수 없으며, 외부 DNA가 절단 [11]부위에 통합되어야만 복제가 복원될 수 있다.
복제할 DNA 준비
게놈 DNA 복제를 위해 대상 생물로부터 복제 대상 DNA를 추출한다.DNA가 광범위하게 분해되지 않는 한 사실상 모든 조직 소스(멸종된 [12]동물의 조직도)를 사용할 수 있다.그런 다음 DNA는 오염 단백질(페놀 추출), RNA(리보핵산가수분해효소) 및 더 작은 분자(침전 및/또는 크로마토그래피)를 제거하기 위한 간단한 방법을 사용하여 정제된다.중합효소 연쇄반응(PCR) 방법은 종종 분자 복제 전에 특정 DNA 또는 RNA(RT-PCR) 시퀀스의 증폭에 사용된다.
복제 실험을 위한 DNA는 역전사효소(상보적 DNA 또는 cDNA 복제)를 사용하거나 합성 DNA(인공 유전자 합성)의 형태로 RNA로부터 얻을 수도 있다.cDNA 복제는 일반적으로 관심 세포의 mRNA 모집단을 대표하는 클론을 얻기 위해 사용되는 반면 합성 DNA는 설계자가 정의한 정확한 염기서열을 얻기 위해 사용됩니다.그러한 설계된 배열은 유전자 코드를 가로질러 유전자를 이동할 때(예를 들어, 마이토크론드리아에서 [13]핵으로) 또는 단순히 코돈 [14]최적화를 통해 발현을 증가시키기 위해 필요할 수 있다.
정제된 DNA는 제한 효소로 처리되어 벡터의 끝부분과 연결될 수 있는 단편을 생성한다.필요에 따라 원하는 제한 부위를 포함하는 DNA(링커)의 짧은 이중가닥 세그먼트를 추가하여 [3][11]벡터와 호환되는 끝 구조를 만들 수 있다.
DNA 연결효소에 의한 재조합 DNA 생성
재조합 DNA의 생성은 많은 면에서 분자 복제 과정의 가장 간단한 단계이다.벡터와 외부 소스로부터 제조된 DNA는 단순히 적절한 농도로 함께 섞이고 양끝을 공유 결합하는 효소(DNA 연결효소)에 노출된다.이 결합 반응은 종종 결찰이라고 불립니다.무작위로 결합된 끝을 포함하는 결과 DNA 혼합물은 숙주 유기체에 도입될 준비가 되어 있다.
DNA 연결효소는 선형 DNA 분자의 끝부분만을 인식하고 작용하며, 보통 무작위로 결합된 끝부분을 가진 복잡한 DNA 분자의 혼합을 야기합니다.원하는 제품(외부 DNA와 공유로 연결된 벡터 DNA)이 존재하지만, 다른 배열(예: 자신과 연결된 외부 DNA, 자신과 연결된 벡터 DNA 및 벡터 및 외부 DNA의 고차 조합)도 존재한다.이 복잡한 혼합물은 DNA 혼합물이 [3][11]세포에 도입된 후 복제 과정의 후속 단계에서 분류된다.
숙주 생물에 재조합 DNA 도입
이전에 체외에서 조작되었던 DNA 혼합물은 숙주 유기체라고 불리는 살아있는 세포로 다시 이동됩니다.DNA를 세포에 넣는 방법은 다양하며, 분자 복제 과정의 이 단계에 적용되는 이름은 종종 선택된 실험 방법(예: 변형, 변환, 트랜스펙션, 일렉트로포메이션)[3][11]에 따라 달라집니다.
미생물이 현지 환경에서 DNA를 채취해 복제할 수 있는 것을 변형이라고 하며, 생리학적으로 DNA를 채취할 수 있는 세포는 [15]유능하다고 한다.포유류의 세포 배양에서 DNA를 세포에 도입하는 유사한 과정을 흔히 트랜스펙션이라고 한다.형질전환과 전염은 일반적으로 사용되는 특정 종과 세포 유형에 따라 달라지는 특별한 성장 과정과 화학적 처리 과정을 통해 세포를 준비해야 한다.
일렉트로포레이션은 고전압의 전기 펄스를 사용하여 세포막(및 [16]존재하는 경우 세포벽)을 통해 DNA를 변환합니다.이와는 대조적으로, 변환은 바이러스 감염과 유사한 과정을 통해 캡슐화된 DNA를 세포에 도입하기 위해 DNA를 바이러스 유래 입자로 패키징하고 이러한 바이러스 유사 입자를 사용하는 것을 포함한다.비록 전기 탐사와 변환이 매우 전문적인 방법이지만, 그것들은 DNA를 세포로 이동시키는 가장 효율적인 방법일 수 있다.
벡터 배열을 포함하는 생물 선택
어떤 방법을 사용하든, 선택된 숙주 유기체에 재조합 DNA를 도입하는 것은 보통 낮은 효율의 과정이다; 즉, 세포의 극히 일부만이 실제로 DNA를 차지할 것이다.실험 과학자들은 DNA를 흡수하지 않은 세포를 선택적으로 죽이는 인위적 유전자 선택 단계를 통해 이 문제에 대처하고 벡터에 의해 암호화된 선택 가능한 표시 유전자를 가진 DNA를 능동적으로 복제할 수 있는 세포만이 살아남을 [3][11]수 있다.
박테리아 세포가 숙주 유기체로 사용될 때, 선택 가능한 마커는 보통 세포를 죽일 항생제에 내성을 부여하는 유전자이다. 전형적으로 암피실린이다.플라스미드를 품고 있는 세포는 항생제에 노출되면 생존할 수 있지만 플라스미드 염기서열을 흡수하지 못한 세포는 죽게 된다.포유류 세포(예: 인간 또는 마우스 세포)를 사용할 때, 유사한 전략이 사용된다. 단, 마커 유전자(이 경우 일반적으로 kanMX 카세트의 일부로 인코딩됨)가 항생제 Geneticin에 대한 내성을 갖는다는 점을 제외한다.
원하는 DNA 삽입물과 생물학적 특성을 가진 클론 선별
현대의 박테리아 클로닝 벡터(예: pUC19 및 pGEM 벡터를 포함한 이후의 유도체)는 부모 벡터(재조합 배열을 삽입하지 않은 벡터 DNA)를 포함하는 것과 트랜스제닉 세포의 콜로니(클론)를 구별하기 위해 청백색 스크리닝 시스템을 사용한다.이들 벡터에서는 외부 DNA가 베타갈락토시드가수분해효소의 필수부분을 코드하는 배열에 삽입된다.β갈락토시드가수분해효소는 활성으로 인해 본 작업에 사용되는 배지에 푸른색 콜로니를 형성한다.외부 DNA를 베타갈락토시다아제 코드 배열에 삽입하면 변환된 DNA를 포함한 집락이 무색(흰색)으로 유지되도록 효소의 기능이 비활성화된다.따라서, 실험자들은 유전자 변형 DNA를 포함하지 않는 복제 박테리아를 무시하고 유전자 변형 박테리아 복제에 대한 추가 연구를 쉽게 식별하고 수행할 수 있다.
분자 복제 실험에서 얻은 개인 복제의 총 집단은 종종 DNA 라이브러리라고 불립니다.라이브러리는 매우 복잡하거나 비교적 단순할 수 있다(기존에 복제된 DNA 조각을 다른 플라스미드로 이동시킬 때처럼). 그러나 원하는 DNA 구조를 확실히 얻기 위해 거의 항상 많은 다른 클론을 검사할 필요가 있다.이는 핵산 교배, 항체 프로브, 중합효소 연쇄 반응, 제한 단편 분석 및/또는 DNA 염기서열 분석을 [3][11]포함한 매우 광범위한 실험 방법을 통해 달성될 수 있다.
적용들
분자 복제는 과학자들에게 어떤 게놈에서 유래한 어떤 개별 DNA 세그먼트의 본질적으로 무한한 양을 제공한다.이 재료는 기초 및 응용 생물 과학 모두를 포함한 다양한 용도로 사용될 수 있습니다.여기에서는, 보다 중요한 애플리케이션의 일부를 정리하고 있습니다.
게놈 구성 및 유전자 발현
분자 복제는 직접적으로 많은 종의 게놈의 완전한 DNA 배열의 해명과 개별 종들 내의 유전적 다양성의 탐구로 이어졌습니다, 대부분의 작업은 게놈의 무작위로 복제된 조각들의 많은 수의 DNA 배열을 결정하고, 그리고 오버레이를 조립함으로써 이루어졌습니다.적용 시퀀스.
개별 유전자의 수준에서, 분자 복제는 유전자가 어떻게 발현되는지를 검사하기 위해 사용되는 탐침을 생성하기 위해 사용된다. 그리고 그 발현이 대사 환경, 세포 외 신호, 발달, 학습, 노화 그리고 세포 사멸을 포함한 생물학에서의 다른 과정과 어떻게 관련이 있는지.복제 유전자는 또한 연구자들이 유전자를 비활성화하거나 국소 돌연변이 유발이나 부위 지향 돌연변이 유발을 사용하여 더 미묘한 돌연변이를 만들 수 있도록 함으로써 개별 유전자의 생물학적 기능과 중요성을 검사하는 도구를 제공할 수 있다.기능성 복제를 위해 발현 벡터로 복제된 유전자는 발현된 단백질의 기능을 바탕으로 유전자를 선별하는 수단을 제공한다.
재조합단백질 생산
유전자의 분자 클론을 얻는 것은 재조합 단백질이라고 불리는 복제된 유전자의 단백질 제품을 생산하는 유기체의 개발을 이끌 수 있다.실제로, 유전자를 복제하는 것보다 바람직한 양으로 재조합 단백질의 활성 형태를 생산하는 유기체를 개발하는 것이 종종 더 어렵다.이것은 유전자 발현을 위한 분자 신호가 복잡하고 다양하기 때문이고, 단백질 접힘, 안정성 그리고 수송이 매우 어려울 수 있기 때문이다.
많은 유용한 단백질들이 현재 재조합 제품으로 이용 가능하다.이 뿐만 아니라 행정부는 미흡하거나 표현된 유전자(예를 들어 재조합 제8인자, hemophilia,[17]과 재조합형 인슐린의 종류, diabetes[18]몇가지 종류의 치료에 사용되는에서blood-clotting 요인이 부족해), 생명이 위험한을 돕기 위해 통치 될 수 있(2)단백질 결함 고칠 수 있는 의료적으로 유용한 단백질(1)을 포함한다.드러나다Ncy(예를 들어 조직 플라스미노겐 활성 인자를 치료하는 데 사용 strokes[19]), 순수 단백질 전염병 환자들을 면역시키는, 감염원 자체(예를 들어 B형 간염 vaccine[20])에 노출되지 않게 하지 않고, 진단 laborato을 위한 표준 재료로 재조합형 단백질(4)사용할 수 있(3)가 재조합형 소단위 백신.ry 시험.
트랜스제닉 생물
일단 적절한 발현을 위한 신호를 제공하기 위해 특징지어지고 조작되면, 복제 유전자는 유기체에 삽입되어 유전자 변형 유기체(GMO)라고도 불리는 트랜스제닉 유기체를 생성할 수 있다.대부분의 GMO는 기본적인 생물학적 연구 목적으로 생성되지만(예: 유전자 변환 마우스 참조), 의약품이나 다른 화합물(약초제)을 생산하는 동식물, 제초제 내성 작물 식물 및 가정용 형광 열대어(글로피시)에 이르기까지 많은 GMO가 상업적 사용을 위해 개발되었다.아이네이션[1]
유전자 치료
유전자 치료는 유전 질환이나 후천성 질환을 교정하기 위해 기능이 부족한 세포에 기능성 유전자를 공급하는 것을 포함한다.유전자 치료는 크게 두 가지로 나눌 수 있다.첫 번째는 생식세포의 변화, 즉 정자나 난자의 변화입니다. 이것은 전체 유기체와 그 다음 세대에게 영구적인 유전자 변화를 가져옵니다.이 "독선 유전자 치료"는 많은 사람들에게 [21]비윤리적인 것으로 여겨진다.유전자 치료의 두 번째 유형인 "체세포 유전자 치료"는 장기 이식과 유사하다.이 경우 1개 이상의 특이조직을 직접 치료 또는 조직의 제거, 연구실 내 치료유전자 또는 유전자의 첨가 및 치료세포의 환자에 대한 복귀에 의해 표적화한다.체세포 유전자 치료의 임상시험은 1990년대 말에 시작되었으며, 주로 암과 혈액, 간,[22] 폐 질환의 치료를 위한 것이었다.
엄청난 홍보와 약속에도 불구하고, 인간 유전자 치료의 역사는 상대적으로 제한된 [22]성공으로 특징지어져 왔다.세포에 유전자를 도입하는 효과는 종종 치료 중인 질병의 증상으로부터 부분적 및/또는 일시적인 완화만을 촉진한다.일부 유전자 치료 실험 환자들은 사망을 포함한 치료 자체의 부작용에 시달리고 있다.어떤 경우, 부작용은 삽입 불활성화에 의한 환자의 게놈 내 필수 유전자의 교란에서 비롯된다.유전자 치료에 사용되는 바이러스 벡터가 감염성 바이러스에 오염된 사례도 있다.그럼에도 불구하고, 유전자 치료는 여전히 의학의 유망한 미래 분야로 여겨지고 있으며 상당한 수준의 연구 개발 활동이 있는 분야이다.
레퍼런스
- ^ a b Watson JD (2007). Recombinant DNA: genes and genomes: a short course. San Francisco: W.H. Freeman. ISBN 978-0-7167-2866-5.
- ^ Patten CL, Glick BR, Pasternak J (2009). Molecular Biotechnology: Principles and Applications of Recombinant DNA. Washington, D.C: ASM Press. ISBN 978-1-55581-498-4.
- ^ a b c d e f g h Brown T (2006). Gene cloning and DNA analysis: an introduction. Cambridge, MA: Blackwell Pub. ISBN 978-1-4051-1121-8.
- ^ M., Grisham, Charles (2013-01-01). Biochemistry. Brooks/Cole, Cengage Learning. ISBN 978-1133106296. OCLC 777722371.
- ^ Garret, Grisham (2010). Biochemistry. Belmont, CA, Brooks/Cole: Cengage Learning. p. 380.
- ^ Nathans D, Smith HO (1975). "Restriction endonucleases in the analysis and restructuring of dna molecules". Annual Review of Biochemistry. 44: 273–93. doi:10.1146/annurev.bi.44.070175.001421. PMID 166604.
- ^ Cohen SN, Chang AC, Boyer HW, Helling RB (Nov 1973). "Construction of biologically functional bacterial plasmids in vitro". Proceedings of the National Academy of Sciences of the United States of America. 70 (11): 3240–4. Bibcode:1973PNAS...70.3240C. doi:10.1073/pnas.70.11.3240. PMC 427208. PMID 4594039.
- ^ Jackson DA, Symons RH, Berg P (Oct 1972). "Biochemical method for inserting new genetic information into DNA of Simian Virus 40: circular SV40 DNA molecules containing lambda phage genes and the galactose operon of Escherichia coli". Proceedings of the National Academy of Sciences of the United States of America. 69 (10): 2904–9. Bibcode:1972PNAS...69.2904J. doi:10.1073/pnas.69.10.2904. PMC 389671. PMID 4342968.
- ^ "plasmid / plasmids Learn Science at Scitable". www.nature.com. Retrieved 2017-12-06.
- ^ Shizuya H, Birren B, Kim UJ, Mancino V, Slepak T, Tachiiri Y, Simon M (Sep 1992). "Cloning and stable maintenance of 300-kilobase-pair fragments of human DNA in Escherichia coli using an F-factor-based vector". Proceedings of the National Academy of Sciences of the United States of America. 89 (18): 8794–7. Bibcode:1992PNAS...89.8794S. doi:10.1073/pnas.89.18.8794. PMC 50007. PMID 1528894.
- ^ a b c d e f Russell DW, Sambrook J (2001). Molecular cloning: a laboratory manual. Cold Spring Harbor, N.Y: Cold Spring Harbor Laboratory. ISBN 978-0-87969-576-7.
- ^ Higuchi R, Bowman B, Freiberger M, Ryder OA, Wilson AC (1984). "DNA sequences from the quagga, an extinct member of the horse family". Nature. 312 (5991): 282–4. Bibcode:1984Natur.312..282H. doi:10.1038/312282a0. PMID 6504142. S2CID 4313241.
- ^ Boominathan, A; Vanhoozer, S; Basisty, N; Powers, K; Crampton, AL; Wang, X; Friedricks, N; Schilling, B; Brand, MD; O'Connor, MS (2 November 2016). "Stable nuclear expression of ATP8 and ATP6 genes rescues a mtDNA Complex V null mutant". Nucleic Acids Research. 44 (19): 9342–9357. doi:10.1093/nar/gkw756. PMC 5100594. PMID 27596602.
- ^ Plotkin, J. B.; Kudla, G (2011). "Synonymous but not the same: The causes and consequences of codon bias". Nature Reviews Genetics. 12 (1): 32–42. doi:10.1038/nrg2899. PMC 3074964. PMID 21102527.
- ^ Lederberg J (Feb 1994). "The transformation of genetics by DNA: an anniversary celebration of Avery, MacLeod and McCarty (1944)". Genetics. 136 (2): 423–6. doi:10.1093/genetics/136.2.423. PMC 1205797. PMID 8150273.
- ^ Wirth R, Friesenegger A, Fiedler S (Mar 1989). "Transformation of various species of gram-negative bacteria belonging to 11 different genera by electroporation". Molecular & General Genetics. 216 (1): 175–7. doi:10.1007/BF00332248. PMID 2659971. S2CID 25214157.
- ^ Oldenburg J, Dolan G, Lemm G (Jan 2009). "Haemophilia care then, now and in the future". Haemophilia. 15 Suppl 1: 2–7. doi:10.1111/j.1365-2516.2008.01946.x. PMID 19125934. S2CID 29118026.
- ^ The MJ (Nov 1989). "Human insulin: DNA technology's first drug". American Journal of Hospital Pharmacy. 46 (11 Suppl 2): S9-11. PMID 2690608.
- ^ Lewandowski C, Barsan W (Feb 2001). "Treatment of acute ischemic stroke". Annals of Emergency Medicine. 37 (2): 202–16. doi:10.1067/mem.2001.111573. PMID 11174240.
- ^ Chang MH, Chen CJ, Lai MS, Hsu HM, Wu TC, Kong MS, Liang DC, Shau WY, Chen DS (Jun 1997). "Universal hepatitis B vaccination in Taiwan and the incidence of hepatocellular carcinoma in children. Taiwan Childhood Hepatoma Study Group". The New England Journal of Medicine. 336 (26): 1855–9. doi:10.1056/NEJM199706263362602. PMID 9197213.
- ^ August JT (1997). Gene Therapy. Vol. 40. Academic Press. p. 508. ISBN 978-0-08-058132-3.
- ^ a b Pfeifer A, Verma IM (2001). "Gene therapy: promises and problems". Annual Review of Genomics and Human Genetics. 2: 177–211. doi:10.1146/annurev.genom.2.1.177. PMID 11701648.
추가 정보
- Matsumura, Ichiro (September 2015). "Why Johnny can't clone: Common pitfalls and not so common solutions". BioTechniques. 53 (3): IV–XIII. doi:10.2144/000114324. PMID 26345511. Archived from the original on 2015-09-16. Retrieved 2 February 2016.
외부 링크
| 라이브러리 리소스 정보 분자 복제 |