잔류 이극 커플링
Residual dipolar coupling분자 내 두 개의 스핀들 사이의 잔여 2극 결합은 용액 내 분자가 부분 정렬을 보여 공간적으로 비등방성 2극 커플링의 불완전한 평균을 얻을 경우 발생한다.
부분 분자 정렬은 자기 이중극-디폴 상호작용(이중극 커플링이라고도 함), 화학적 이동 음이소트로피 또는 전기적 4극 상호작용과 같은 비등방성 자기 상호작용의 불완전한 평균화로 이어진다.이에 따른 소위 잔류 비등방성 자기 상호작용은 생체분자 NMR 분광학에서 점점 더 중요해지고 있다.[1]
역사와 선구자적 작품
부분 지향적 매체에서의 NMR 분광학은 1963년에 처음 발견되었으며,[2] 매우 기초적인 논문에서 알프레드 사우페도 겨우 1년 후에 관측 가능한 현상을 기술하고 이해할 수 있는 필수 이론을 제시할 수 있었다.[3]이 개시 후에 다양한 액체 결정 단계에서 NMR 스펙트럼의 홍수가 보고되었다(예: 참조).
최소 음이소트로피에 의해 제한되지 않는 부분 정렬의 두 번째 기법은 델로체와 사물스키의 선구적인 작업을 바탕으로 한 젤(SAG)에서의 변형 유도 정렬이다.[8]이 기술은 고해상도 중수소 NMR을 이용하여 폴리머젤의 성질을 연구하기 위해 광범위하게 사용되었지만,[9] 최근에 와서야 젤 정렬을 사용하여 젤에 용해된 분자의 RDC를 유도하였다.[10][11]SAG는 광범위한 범위의 무제한 정렬 스케일링을 허용하며, 사용되는 폴리머에 따라 수성 용매뿐만 아니라 유기 용매에도 사용할 수 있다.유기용제의 첫 번째 예로서 CDCl에서3 부풀어 오른 폴리스티렌(PS)겔의 RDC 측정은 유망한 정렬 방법으로 보고되었다.[12]
1995년, 제임스 H. Prestegard와 동료들은 매우 높은 현장에서 채취한 특정 단백질(이 경우 매우 높은 비등방성 파라자성 감수성을 갖는 시안오메트미오글로빈)의 NMR 스펙트럼이 3차 접힘 결정에서 NOE를 유용하게 보완할 수 있는 데이터를 포함할 수 있다는 것을 입증했다.[13]
1996년과 1997년에 Adriaan Bax와 동료들은 RDC를 직경 단백질(유비퀴틴)로 측정했다.그 결과는 수정 구조와 잘 일치했다.[14][15]
물리학
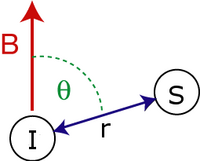
개의 스핀인 I {\ 과S, {\ S의 세속적 2극 커플링 해밀턴안은 다음을 통해 주어진다.
어디에
- 은(는) 축소된 플랑크 상수임.
- 와 S _{는 각각 스핀 과 스핀S {\의 자석 비율이다.
- 는 스핀간 거리.
- }은(는) 쌍방향 벡터와 외부 자기장 사이의 각도다.
- → → 는 스핀 연산자의 벡터다.
위의 방정식은 다음과 같은 형태로 다시 쓸 수 있다.
어디에
등방성 용액에서 분자 텀블링은 S 의 평균값을 감소시킨다.0.따라서 우리는 2극 결합을 관찰하지 않는다.용액이 등방성이 아닌 경우 의 평균값은 0과 다를 수 있으며, 잔류 커플링을 관찰할 수 있다.
이 잔류 이극 결합은 표본 추출되는 각도의 범위에 따라 양 또는 음일 수 있다는 점에 유의하십시오.[16]
정적 거리와 각도 정보 외에도, RDC는 분자의 내부 움직임에 대한 정보를 포함할 수 있다.분자의 각 원자에 대하여 다음과 같은 관계에 따라 RDC에서 계산될 수 있는 모션 텐서 B를 연결할 수 있다.[17]
여기서 A는 분자 정렬 텐서 입니다.B의 행은 각 원자에 대한 모션 텐더를 포함한다.모션 텐서에도 5도의 자유도가 있다.각 모션 텐서로부터 관심 매개변수 5개를 계산할 수 있다.변수 Si2, ηi, αi, βi 및 γ은i 원자 i에 대해 이 5개의 파라미터를 나타내기 위해 사용된다.S는i2 원자 i 운동의 크기, η은i 원자 i 운동의 음이소트로피의 척도, α와i β는i 초기 임의의 기준 프레임(즉 PDB 프레임)에 표현된 본드 벡터의 극좌표와 관련이 있다.원자의 움직임이 비등방성(즉, ηi = 0)인 경우, 최종 매개변수 γ은i 운동의 주요 방향을 측정한다.
RDC에서 파생된 모션 파라미터는 로컬 측정이라는 점에 유의하십시오.
측정
용액 내 모든 RDC 측정은 분자와 NMR 연구를 정렬하는 두 단계로 구성된다.
분자 정렬 방법
중간 정도의 자기장 강도에 있는 직경 분자의 경우 분자는 방향 선호도가 거의 없고, 텀블링 표본은 거의 등방성 분포에 가깝고, 평균 2극 커플링은 0이 된다.사실, 대부분의 분자는 자기장이 있는 곳에서 방향을 선호한다. 왜냐하면 대부분의 분자는 비등방성 자기 감수성 텐서인 χ을 가지고 있기 때문이다.[13]
이 방법은 자기 감수성 텐서 값이 큰 시스템에 가장 적합하다.여기에는 다음이 포함된다.단백질-핵산 복합체, 핵산, 방향제 잔류물이 많은 단백질, 단백질과 금속 결합 단백질(금속은 란타니드로 대체될 수 있다).
완전 지향성 분자의 경우, H-N15 아미드 그룹의 2극 결합은 20 kHz 이상일 것이며, 5 by으로 분리된 양성자 쌍은 최대 1 kHz의 결합을 가질 것이다.그러나 자기장을 적용함으로써 달성되는 정렬 정도는 매우 낮아서 가장 큰 H-N15 또는 H-C13 2극 커플링은 <5 Hz이다.[18]따라서 다음과 같은 여러 가지 정렬 매체가 설계되었다.
- Lipid Bicelles (큰 자석 감수성 포함): 측정된 RDC는 수백 Hz의 순서였다.[19]
- 액체 결정체 바이셀: 측정된 RDC는 -40에서 +20Hz 사이였습니다.[20]
- 필라멘트성 박테리오파지(대형 비등방성 자기 감수성)를 포함한 로드형 바이러스.[18][21]
- DNA 나노튜브(막 단백질을 용해하는 데 사용되는 세제와 호환)[22]
NMR 실험
핵 사이의 결합 상수를 정확하게 측정하기 위해 고안된 수많은 방법이 있다.[23]주파수 영역에서 피크 중심 분리(분할)를 측정하는 주파수 기반 방법과 분할 대신 공명 강도에서 커플링을 추출하는 강도 기반 방법 등 두 그룹으로 분류됐다.두 가지 방법은 각각 다른 종류의 계통 오류가 발생하기 때문에 서로 보완한다.다음은 두 그룹 각각에 속하는 NMR 실험의 원형 예시들이다.
- 강도 방법: 정량적 J-변조 실험 및 위상변조 방법
- 주파수 분해 방법: SCE-HSQC, E. COSY 및 스핀 상태 선택 실험
구조생물학
RDC 측정은 단백질 또는 단백질 복합체의 전지구적 접힘에 대한 정보를 제공한다.기존의 NOE 기반 NMR 구조 결정과는 반대로, RDC는 장거리 구조 정보를 제공한다.그것은 또한 나노초보다 느린 시간 척도에서의 분자의 역학에 대한 정보를 제공한다.
생체분자구조에 관한 연구
단백질 구조에 대한 대부분의 NMR 연구는 단백질 내의 서로 다른 양성자 사이의 핵 과부하 효과 NOE의 분석에 기초한다.NOE는 핵, r−6, NOE 사이의 거리의 반전된 6번째 힘에 의존하기 때문에, NOE는 분자역학형 구조 계산에 사용할 수 있는 거리 구속장치로 변환될 수 있다.RDC는 거리 구속장치보다는 방향 구속장치를 제공하며 NOE에 비해 몇 가지 장점이 있다.
- RDC는 외부 자기장에 상대적인 각도에 대한 정보를 제공하며, 이는 구조에서 멀리 떨어져 있는 분자 부분의 상대적 방향에 대한 정보를 제공할 수 있다는 것을 의미한다.
- 큰 분자(>25kDa)에서는 스핀 확산으로 인해 NOE를 기록하기 어려운 경우가 많다.이것은 RDC의 문제가 아니다.
- 많은 수의 NOE를 분석하는 것은 매우 시간 소모적일 수 있다.
매우 완전한 RDC 세트를 이용할 수 있다면, NOE 구속장치에 의지하지 않고 이러한 비등방성 상호작용에 기반하여 분자구조를 독점적으로 계산할 수 있다는 것이 여러 모델 시스템에 대해 입증되었다.단, 실무에서는 이를 달성할 수 없으며 주로 NOE 데이터와 J-커플링에 의해 결정되는 구조물을 다듬는 데 RDC를 사용한다.구조 결정에서 2극 커플링을 사용할 때의 한 가지 문제는 2극 커플링이 내부 핵 벡터 방향을 고유하게 설명하지 않는다는 것이다.게다가, 극소량의 2극 커플링을 이용할 수 있다면, 정교함이 원래의 커플링보다 더 나쁜 구조로 이어질 수 있다.N개의 아미노산이 있는 단백질의 경우, 백본에 대한 2N RDC 구속조건은 정확한 정교화에 필요한 최소값이다.[24]
특정 결합 벡터(단백분자 내 특정 백본 NH 결합 등)에 대한 개별 RDC 측정의 정보 내용은 관측된 RDC 값과 모델에서 계산된 값 사이의 완벽한 합치 방향을 추적하는 목표 곡선을 보여줌으로써 이해할 수 있다.그러한 곡선(그림 참조)은 자기장 방향을 따라 극축이 있는 구체에 놓여 있는 두 개의 대칭적인 가지를 가지고 있다.구 적도에서 나온 이들의 높이는 RDC 값의 크기에 따라 달라지며, 이들의 모양은 분자 정렬 텐서의 "횡위성"(비대칭성)에 따라 달라진다.분자 정렬이 자기장 방향을 중심으로 완전히 대칭이 되었다면, 대상 곡선은 특정 결합 벡터가 적용된 자기장에 만드는 각도 와 동일한 각도에서 두 개의 원만으로 구성될 것이다.[24]
국소 비틀림 정보와 단거리만으로는 구조를 구속할 수 없는 RNA와 같은 연장된 분자의 경우, RDC 측정은 단일 좌표 프레임에 관하여 핵산 전체에 걸친 특정 화학 결합의 방향에 대한 정보를 제공할 수 있다.특히 RNA 분자는 양성자가 빈약하고 리보스 공진의 중복으로 J커플링과 NOE 데이터를 사용하여 구조를 결정하는 것이 매우 어렵다.또한 5-6 å 이상의 거리를 가진 핵 사이의 RDC도 검출할 수 있다.이 거리는 NOE 신호 생성에 너무 과하다.이는 RDC가 r에−3 비례하는 반면 NOE는 r에−6 비례하기 때문이다.
또한 RDC 측정은 단백질에서 알려진 구조 단위의 상대적 방향을 신속하게 결정하는 데 매우 유용한 것으로 입증되었다.[25][26]원칙적으로, 나선형 회전만큼 작거나 전체 영역만큼 클 수 있는 구조 서브 유닛의 방향은 서브 유닛당 최대 5개의 RDC에서 설정될 수 있다.[24]
단백질역학
RDC는 외부 자기장과 분자 내 결합 벡터 사이의 각도에 대한 공간적 및 일시적 평균 정보를 제공하므로 단백질에서 느린 속도(>10초−9)의 역학에 대한 풍부한 기하학적 정보를 제공할 수 있다.특히, 방사상 의존성 때문에 RDC는 특히 대형 암페어 각도 프로세스에 민감하다 톨만 외 연구원의 초기 사례는 이전에 발표된 미오글로빈 구조물이 측정된 RDC 데이터를 설명하기에 불충분하다는 것을 발견했으며, 이를 개선하기 위해 저속 역학의 간단한 모델을 고안했다.[28]그러나 본질적으로 질서 정연한 단백질을 포함한 많은 종류의 단백질에 대해서는 정렬 프레임을 정의하는 것이 사소한 것이 아니기 때문에 RDC의 분석이 더욱 관여하게 된다.[29]최근 두 개의 논문은 정렬 프레임을 명시적으로 정의할 필요성을 회피함으로써 문제의 유망한 해결책에 대해 설명한다.[29][30]
참고 항목
- 자기 쌍극-디폴 상호작용
- 잔류 화학적 이동 음이소트로피(rCSA)
- 고체 핵자기공명(ssNMR)
참조
- ^ Brunner, E. (2001). "Residual dipolar couplings in protein NMR". Concepts in Magnetic Resonance. 13 (4): 238–259. doi:10.1002/cmr.1012.
- ^ Saupe, A.; Englert, G. (1963). "High-Resolution Nuclear Magnetic Resonance Spectra of Orientated Molecules". Physical Review Letters. 11 (10): 462–464. Bibcode:1963PhRvL..11..462S. doi:10.1103/PhysRevLett.11.462.
- ^ 소프, A. Z. 나튀르포르슈. 19a, 161-171. (1964)
- ^ Snyder, L. C. (1965). "Analysis of Nuclear Magnetic Resonance Spectra of Molecules in Liquid-Crystal Solvents". The Journal of Chemical Physics. 43 (11): 4041–4050. Bibcode:1965JChPh..43.4041S. doi:10.1063/1.1696638.
- ^ Sackmann, E.; Meiboom, S.; Snyder, L. C. (1967). "Relation of nematic to cholesteric mesophases". Journal of the American Chemical Society. 89 (23): 5981–5982. doi:10.1021/ja00999a062.
- ^ Yannoni, C. S.; Ceasar, G. P.; Dailey, B. P. (1967). "Nuclear magnetic resonance spectrum of oriented (cyclobutadiene)iron tricarbonyl". Journal of the American Chemical Society. 89 (12): 2833–2836. doi:10.1021/ja00988a006.
- ^ Luckhurst, G. R. (1968). "Liquid crystals as solvents in nuclear magnetic resonance". Quarterly Reviews, Chemical Society. 22 (2): 179–4621. doi:10.1039/qr9682200179.
- ^ Deloche, B.; Samulski, E. T. (1981). "Short-range nematic-like orientational order in strained elastomers: A deuterium magnetic resonance study". Macromolecules. 14 (3): 575–581. Bibcode:1981MaMol..14..575D. doi:10.1021/ma50004a024.
- ^ Samulski, E. T. (1985). "Investigations of polymer chains in oriented fluid phases with deuterium nuclear magnetic resonance". Polymer. 26 (2): 177–189. doi:10.1016/0032-3861(85)90027-8.
- ^ Sass, H. J. R.; Musco, G.; Stahl, S. J.; Wingfield, P. T.; Grzesiek, S. (2000). "Solution NMR of proteins within polyacrylamide gels: Diffusional properties and residual alignment by mechanical stress or embedding of oriented purple membranes". Journal of Biomolecular NMR. 18 (4): 303–309. doi:10.1023/A:1026703605147. PMID 11200524. S2CID 30050084.
- ^ Tycko, R.; Blanco, F. J.; Ishii, Y. (2000). "Alignment of Biopolymers in Strained Gels: A New Way to Create Detectable Dipole−Dipole Couplings in High-Resolution Biomolecular NMR". Journal of the American Chemical Society. 122 (38): 9340–9341. doi:10.1021/ja002133q.
- ^ Luy, B.; Kobzar, K.; Kessler, H. (2004). "An Easy and Scalable Method for the Partial Alignment of Organic Molecules for Measuring Residual Dipolar Couplings". Angewandte Chemie International Edition. 43 (9): 1092–1094. doi:10.1002/anie.200352860. PMID 14983442.
- ^ a b Tolman, J. R.; Flanagan, J. M.; Kennedy, M. A.; Prestegard, J. H. (1995). "Nuclear magnetic dipole interactions in field-oriented proteins: Information for structure determination in solution". Proceedings of the National Academy of Sciences. 92 (20): 9279–9283. Bibcode:1995PNAS...92.9279T. doi:10.1073/pnas.92.20.9279. PMC 40968. PMID 7568117.
- ^ Tjandra, N.; Szabo, A.; Bax, A. (1996). "Protein Backbone Dynamics and15N Chemical Shift Anisotropy from Quantitative Measurement of Relaxation Interference Effects". Journal of the American Chemical Society. 118 (29): 6986–6991. doi:10.1021/ja960510m.
- ^ Tjandra, N.; Bax, A. (1997). "Measurement of Dipolar Contributions to 1JCH Splittings from Magnetic-Field Dependence of J Modulation in Two-Dimensional NMR Spectra". Journal of Magnetic Resonance. 124 (2): 512–515. Bibcode:1997JMagR.124..512T. doi:10.1006/jmre.1996.1088. PMID 9169226.
- ^ Sanders, C. R.; Hare, B. J.; Howard, K. P.; Prestegard, J. H. (1994). "Magnetically-oriented phospholipid micelles as a tool for the study of membrane-associated molecules". Progress in Nuclear Magnetic Resonance Spectroscopy. 26: 421–444. doi:10.1016/0079-6565(94)80012-X.
- ^ Tolman, J. R. (2002). "A Novel Approach to the Retrieval of Structural and Dynamic Information from Residual Dipolar Couplings Using Several Oriented Media in Biomolecular NMR Spectroscopy". Journal of the American Chemical Society. 124 (40): 12020–12030. doi:10.1021/ja0261123. PMID 12358549.
- ^ a b Hansen, M. R.; Mueller, L.; Pardi, A. (1998). "Tunable alignment of macromolecules by filamentous phage yields dipolar coupling interactions". Nature Structural Biology. 5 (12): 1065–1074. doi:10.1038/4176. PMID 9846877. S2CID 29222802.
- ^ Metz, G.; Howard, K. P.; Van Liemt, W. B. S.; Prestegard, J. H.; Lugtenburg, J.; Smith, S. O. (1995). "NMR Studies of Ubiquinone Location in Oriented Model Membranes: Evidence for a Single Motionally-Averaged Population". Journal of the American Chemical Society. 117: 564–565. doi:10.1021/ja00106a078.
- ^ Tjandra, N.; Bax, A. (1997). "Direct Measurement of Distances and Angles in Biomolecules by NMR in a Dilute Liquid Crystalline Medium". Science. 278 (5340): 1111–1114. Bibcode:1997Sci...278.1111T. doi:10.1126/science.278.5340.1111. PMID 9353189.
- ^ Clore GM, Starich MR, Gronenborn AM (1998). "measurement of residual dipolar couplings of macromolecules aligned in the nematic phase of a colloidal suspension of rod shaped viruses". Journal of the American Chemical Society. 120 (40): 10571–10572. doi:10.1021/ja982592f.
{{cite journal}}: CS1 maint: 작성자 매개변수 사용(링크) - ^ Douglas, S. M.; Chou, J. J.; Shih, W. M. (2007). "DNA-nanotube-induced alignment of membrane proteins for NMR structure determination". Proceedings of the National Academy of Sciences. 104 (16): 6644–6648. Bibcode:2007PNAS..104.6644D. doi:10.1073/pnas.0700930104. PMC 1871839. PMID 17404217.
- ^ Prestegard, J. H.; Al-Hashimi, H. M.; Tolman, J. R. (2000). "NMR structures of biomolecules using field oriented media and residual dipolar couplings". Quarterly Reviews of Biophysics. 33 (4): 371–424. doi:10.1017/S0033583500003656. PMID 11233409. S2CID 46687434.
- ^ a b c Bax, A.; Grishaev, A. (2005). "Weak alignment NMR: A hawk-eyed view of biomolecular structure". Current Opinion in Structural Biology. 15 (5): 563–570. doi:10.1016/j.sbi.2005.08.006. PMID 16140525.
- ^ Clore G.M. (2000). "Accurate and rapid docking of protein-protein complexes on the basis of intermolecular nuclear Overhauser enhancement data and dipolar couplings by rigid body minimization". Proceedings of the National Academy of Sciences of the United States of America. 97 (16): 9021–9025. Bibcode:2000PNAS...97.9021C. doi:10.1073/pnas.97.16.9021. PMC 16814. PMID 10922057.
- ^ Tang, C.; Williams Jr, D. C.; Ghirlando, R.; Clore, G. M. (2005). "Solution Structure of Enzyme IIAChitobiose from the N,N'-Diacetylchitobiose Branch of the Escherichia coli Phosphotransferase System". Journal of Biological Chemistry. 280 (12): 11770–11780. doi:10.1074/jbc.M414300200. PMID 15654077.
- ^ Bouvignies, G.; Bernadó, P.; Blackledge, M. (2005). "Protein backbone dynamics from N–HN dipolar couplings in partially aligned systems: A comparison of motional models in the presence of structural noise". Journal of Magnetic Resonance. 173 (2): 328–338. Bibcode:2005JMagR.173..328B. doi:10.1016/j.jmr.2005.01.001. PMID 15780926.
- ^ Tolman, J. R.; Flanagan, J. M.; Kennedy, M. A.; Prestegard, J. H. (1997). "NMR evidence for slow collective motions in cyanometmyoglobin". Nature Structural Biology. 4 (4): 292–297. doi:10.1038/nsb0497-292. PMID 9095197. S2CID 29605996.
- ^ a b Olsson, Simon; Ekonomiuk, Dariusz; Sgrignani, Jacopo; Cavalli, Andrea (2015). "Molecular Dynamics of Biomolecules through Direct Analysis of Dipolar Couplings". Journal of the American Chemical Society. 137 (19): 6270–8. doi:10.1021/jacs.5b01289. PMID 25895902.
- ^ Camilloni, Carlo; Vendruscolo, Michele (2015). "A Tensor-Free Method for the Structural and Dynamical Refinement of Proteins using Residual Dipolar Couplings". The Journal of Physical Chemistry B. 119 (3): 653–61. doi:10.1021/jp5021824. PMID 24824082.
추가 읽기
책:
- 액정 용제를 사용한 Lindon, J. C. NMR Spectroscopy; Pergamon Press: 영국 옥스포드, 1975.
검토 문서:
- Ad Bax and Alexander Grishaev, Current Office in Structural Biology, 15:563–570 (2005)
- 레베카 S.립시츠와 니코 탄드라, 안누. 바이오피스 목사님 바이오몰. 구조적인.33:387–413 (2004)
일반 논문:
- Saupe, A.; Englert, G. (1963). "High-Resolution Nuclear Magnetic Resonance Spectra of Orientated Molecules". Physical Review Letters. 11 (10): 462–464. Bibcode:1963PhRvL..11..462S. doi:10.1103/physrevlett.11.462.
- Saupe, A (1964). "Kernresonanzen in kristallinen Flüssigkeiten und in kristallinflüssigen Lösungen. Teil I". Zeitschrift für Naturforschung. 19a (2): 161–171. Bibcode:1964ZNatA..19..161S. doi:10.1515/zna-1964-0201. S2CID 94943333.
- Deloche, B.; Samulski, E. T. (1981). "Short-range nematic-like orientational order in strained elastomers: a deuterium magnetic resonance study". Macromolecules. 14 (3): 575–581. Bibcode:1981MaMol..14..575D. doi:10.1021/ma50004a024.
- Tjandra, Nico; Bax, Ad (1997). "Direct measurement of distances and angles in biomolecules by NMR in a dilute liquid crystalline medium". Science. 278 (5340): 1111–1114. Bibcode:1997Sci...278.1111T. doi:10.1126/science.278.5340.1111. PMID 9353189.
- Tjandra, N.; Omichinski, J.G; Gronenborn, A.M.; Clore, G.M.; Bax, A. (1997). "Use of dipolar 1H–15N and 1H–13C couplings in the structure determination of magnetically oriented macromolecules in solution". Nature Structural Biology. 4 (9): 732–738. doi:10.1038/nsb0997-732. PMID 9303001. S2CID 22357937.
- Tjandra, N.; Bax, A. (1997). "Measurement of dipolar contributions to 1JCH splittings from magnetic-field dependence of J modulation in two-dimensional NMR spectra". J. Magn. Reson. 124 (2): 512–515. Bibcode:1997JMagR.124..512T. doi:10.1006/jmre.1996.1088. PMID 9169226.
- Tolman, J. R.; et al. (1997). "NMR evidence for slow collective motions in cyanometmyoglobin". Nature Structural Biology. 4 (4): 292–297. doi:10.1038/nsb0497-292. PMID 9095197. S2CID 29605996.
- Tolman, J.R.; Prestegard, J.H. (1996). "A QuantitativeJ-Correlation Experiment for the Accurate Measurement of One-Bond Amide15N–1H Couplings in Proteins". J. Magn. Reson. B. 112 (3): 245–252. Bibcode:1996JMRB..112..245T. doi:10.1006/jmrb.1996.0138. PMID 8921605.
- Tolman, J.R.; Flanagan, J.M.; Kennedy, M.A.; Prestegard, J.H. (1995). "Nuclear magnetic dipole interactions in field-oriented proteins: information for structure determination in solution". Proc. Natl. Acad. Sci. U.S.A. 92 (20): 9279–9283. Bibcode:1995PNAS...92.9279T. doi:10.1073/pnas.92.20.9279. PMC 40968. PMID 7568117.
- Clore, G.M. (2000). "Accurate and rapid docking of protein-protein complexes on the basis of intermolecular nuclear Overhauser enhancement data and dipolar couplings by rigid body minimization". Proc. Natl. Acad. Sci. U.S.A. 97 (16): 9021–9025. Bibcode:2000PNAS...97.9021C. doi:10.1073/pnas.97.16.9021. PMC 16814. PMID 10922057.
- Sanders, C.R.; Hare, B.J.; Howard, K.P.; Prestegard, J.H. (1994). "Magnetically-oriented phospholipid micelles as a tool for the study of membrane-associated molecules". Prog. Nucl. Magn. Reson. Spectrosc. 26: 421–444. doi:10.1016/0079-6565(94)80012-x.
- Bastiaan, E. W.; Maclean, C.; Van Zijl, P. C. M.; Bothner-By, A. A. (1987). "High-Resolution NMR of Liquids and Gases: Effects of Magnetic-Field-Induced Molecular Alignment". Annu. Rep. NMR Spectrosc. Annual Reports on NMR Spectroscopy. 19: 35–77. doi:10.1016/s0066-4103(08)60245-8. ISBN 9780125053198.



}](https://wikimedia.org/api/rest_v1/media/math/render/svg/64cb9a1de4ac67a4d920322838be224b4330f0b2)
 (는) 축소된
(는) 축소된 

 .
. 스핀간 거리.
스핀간 거리.


![H_{{\mathrm {D}}}=D_{{IS}}(\theta )[2I_{z}S_{z}-(I_{x}S_{x}+I_{y}S_{y})]\!](https://wikimedia.org/api/rest_v1/media/math/render/svg/f1ef53aaf415fafa421f740336087b2fe905859d)
![{\displaystyle D_{IS}(\theta )={\frac {\hbar ^{2}\gamma _{I}\gamma _{S}}{4\pi r_{IS}^{3}}}[1-3\cos ^{2}\theta ].\!}](https://wikimedia.org/api/rest_v1/media/math/render/svg/5304d624d47f35b40cf0fcb7e7cb645a5339c377)