리보솜 프레임시프트
Ribosomal frameshift리보솜 프레임 쉬핑(Ribosomal frameshifting)은 번역 중에 발생하는 생물학적 현상으로, 단일 mRNA에서 복수의 고유 단백질이 생성된다.[1]이 과정은 mRNA의 뉴클레오티드 순서에 의해 프로그래밍될 수 있으며 때때로 2차원의 3차원 mRNA 구조에 의해 영향을 받는다.[2]주로 바이러스(특히 레트로바이러스), 레트로트랜스포존, 박테리아 삽입원소 등에 설명되어 왔으며, 일부 세포 유전자에서도 설명되어 왔다.[3]
프로세스 개요
단백질은 시작(초기) 코돈으로 아미노산 메티오닌을 시작으로 mRNA의 한쪽 끝에서 다른 쪽 끝(5'에서 3'' 끝)으로 코돈이라고도 하는 mRNA 가닥의 트라이 뉴클레오티드(tri-nucleotide)를 읽음으로써 번역된다.각각의 코돈은 단일 아미노산으로 번역된다.코드 자체는 퇴행성으로 간주되는데, 이는 특정 아미노산이 둘 이상의 코돈에 의해 지정될 수 있다는 것을 의미한다.그러나 판독 틀에서 3으로 분할되지 않는 뉴클레오티드의 개수를 변경하면 이후의 코돈은 다르게 읽히게 된다.[4]이것은 효과적으로 리보솜 판독 프레임을 변화시킨다.
문장 예
이 예에서 세 글자로 된 다음 문장은 처음부터 읽으면 일리가 있다.
'고양이를 시작하면 남자는 뚱뚱해진다'123 123 123 123 123 123 시작...
단, 판독 프레임이 첫 번째 단어의 T와 H 사이에 한 글자씩 이동되는 경우(0의 위치를 T의 초기 위치로 고려할 때 효과적으로 +1 프레임시프트)
T Start HEC ATA NDT HEM ANA REF AT... - 123 123 123 123 123 123 123 123 123 123...
문장이 다르게 읽혀지니까 말이 안 돼
DNA의 예
이 예에서 다음 순서는 MT-ATP8과 MT-ATP6의 두 유전자를 겹치는 인간 미토콘드리아 게놈의 영역이다.처음부터 읽었을 때, 이 코돈들은 리보솜에 이치에 맞고 척추동물 미토콘드리아 코드에 따라 아미노산(AA)으로 번역될 수 있다.
AAC GAA AAT CTG TTC GCT TCA 시작...123 123 123 123 123 123 시작...AA N E N L F A S ...
단, 하나의 뉴클레오티드 다운스트림(A의 초기 위치로 0 위치를 고려할 때 효과적으로 "+1 프레임시프트")을 시작하여 판독 프레임을 변경하자.
시작 ACG AAA ATC TGT TCG CTT CA... - 시작 123 123 123 123 123 123 123 123 123...AA T K I C S L ...
이제, 이 +1 프레임 쉬핑 때문에, DNA 염기서열은 다르게 읽힌다.따라서 다른 코돈 판독 프레임은 다른 아미노산을 산출한다.
번역 리보솜의 경우 프레임시프트는 프레임시프트 후 헛소리(조기 정지 코돈)가 생기거나, 프레임시프트 후 완전히 새로운 단백질이 생성될 수 있다.프레임시프트가 헛소리를 일으키는 경우, NMD(nonsense-mediated mRNA 붕괴) 경로가 mRNA 대본을 파괴할 수 있으므로, 프레임시프트는 관련 유전자의 발현 수준을 조절하는 방법이 될 것이다.[5]
함수
바이러스에서는 이 현상 특정 사이트에 일어나는 것을 하고 바이러스 같은 mRNA로부터 단백질의 여러 형식을 인코딩할 수 있습니다. 주목할 만한 예 HIV-1(인체 면역 결핍 바이러스)[6]원격 제어 감시 장비(라우스 육종 바이러스)[7]고 모든 frameshifting에0-frame(n.의 적절한 비율을 만들기 위해 의존하고 있는 인플루엔자 바이러스(독감)[8]을 포함한다 프로그램될 것ormal translation) 및 "trans-frame"(프레임쉬프트 시퀀스에 의해 결정됨) 단백질.바이러스에 사용하는 것은 주로 더 많은 유전 정보를 더 짧은 양의 유전 물질로 압축하는 것이다.
eukaryotes에서 그것은 조기 정지를 발생시키고 비기능적인 대본을 생성함으로써 유전자 발현 수준을 조절하는 역할을 하는 것으로 보인다.[3][9]
프레임 쉬핑 유형
프레임 쉬핑의 가장 일반적인 유형은 -1 프레임 쉬핑 또는 프로그래밍된 -1 리보솜 프레임 쉬핑(-1 PRF)이다.기타 희귀한 유형의 프레임 쉬핑에는 +1 및 -2 프레임 쉬핑이 포함된다.[2]-1 및 +1 프레임 시프팅은 아래에서 설명하는 서로 다른 메커니즘에 의해 제어되는 것으로 생각된다.두 메커니즘은 모두 운동적으로 움직인다.
프로그래밍된 -1 리보솜 프레임 쉬프팅
-1 프레임 쉬프팅에서 리보솜은 하나의 뉴클레오티드를 뒤로 미끄러뜨리고 -1 프레임에서 번역을 계속한다.일반적으로 -1 프레임시프트 신호로 구성되는 세 가지 요소가 있는데, 미끄러운 시퀀스, 스페이서 영역, RNA 이차 구조.미끄러운 순서는 X_XXY_에 맞는다.YYH motif, where XXX is any three identical nucleotides (though some exceptions occur), YYY typically represents UUU or AAA, and H is A, C or U. Because the structure of this motif contains 2 adjacent 3-nucleotide repeats it is believed that −1 frameshifting is described by a tandem slippage model, in which the ribosomal P-site tRNA anticodon re-paXXY에서 XXX로 irs 및 A-site 안티코돈은 YYH에서 YYY로 동시에 재생된다.이러한 새 쌍은 세 번째 위치를 제외하고 0-프레임 쌍과 동일하다.이 차이는 흔들림 위치로 알려진 코돈의 세 번째 뉴클레오티드가 첫 번째 뉴클레오티드와 두 번째 뉴클레오티드에 비해 더 약한 tRNA 안티코돈 결합 특수성을 가지기 때문에 크게 탐탁하지 않다.[2][10]이 모델에서 모티브 구조는 안티코돈의 첫 번째와 두 번째 위치가 0과 -1 프레임 모두에서 완벽하게 쌍을 이룰 수 있어야 한다는 사실에 의해 설명된다.따라서 뉴클레오티드 2와 1은 동일해야 하며, 뉴클레오티드 3과 2도 동일해야 하며, 미끄러지는 각 tRNA에 대해 필요한 3개의 동일한 뉴클레오티드의 순서가 있어야 한다.[11]
+1 리보솜 프레임 쉬프팅

+1 프레임시프트 신호의 미끄러운 시퀀스는 동일한 모티브를 가지지 않으며, 대신 희귀 아미노산을 인코딩하는 시퀀스에서 리보솜을 일시 중지하여 기능하는 것으로 보인다.[12]리보솜은 순서에 상관없이 단백질을 일정한 속도로 번역하지 않는다.어떤 코돈들은 번역하는데 더 오랜 시간이 걸린다. 왜냐하면 사이토솔에는 그 코돈의 같은 양의 tRNA가 없기 때문이다.[13]이러한 지연으로 인해 리보솜 프레임 쉬핑 속도를 제어하는 코돈 시퀀스의 작은 섹션에 존재한다.구체적으로는 희귀한 tRNA의 도착을 기다리기 위해 리보솜을 잠시 멈춰야 하며, 이로 인해 리보솜의 운동적 호감도가 높아져 관련 tRNA가 새로운 프레임으로 미끄러져 들어간다.[12][14]이 모델에서, 판독 프레임의 변화는 2개가 아닌 1개의 tRNA 슬립에 의해 발생한다.
제어 메커니즘
리보솜 프레임 쉬핑은 mRNA 시퀀스(cis-action)에서 발견된 메커니즘에 의해 제어될 수 있다.이것은 일반적으로 미끄러운 염기서열, RNA 이차구조, 또는 둘 다를 가리킨다.-1 프레임시프트 신호는 일반적으로 5–9 뉴클레오티드 길이 스페이서 영역으로 분리된 두 원소로 구성된다.[2]프레임 쉬핑은 리보솜이나 mRNA와 상호작용하는 다른 분자에 의해서도 유도될 수 있다.
프레임시프트 신호 요소

미끄러운 순서
미끄러운 시퀀스는 잠재적으로 판독 리보솜을 "슬립"하게 만들고 다수의 뉴클레오티드(보통 1개만)를 건너뛰고 이후 완전히 다른 프레임을 읽을 수 있다.프로그래밍된 -1 리보솜 프레임 쉬프팅에서 미끄러운 시퀀스는 X_XXY_에 적합함YYH motif, where XXX is any three identical nucleotides (though some exceptions occur), YYY typically represents UUU or AAA, and H is A, C or U. In the case of +1 frameshifting, the slippery sequence contains codons for which the corresponding tRNA is more rare, and the frameshift is favored because the codon in the new frame has a more common associved [12]tRNA미끄러운 염기서열의 한 예는 mRNA의 폴리A로, 다른 원소가 없어도 리보솜의 미끄러짐을 유발하는 것으로 알려져 있다.[15]
RNA 이차 구조
효율적인 리보솜 프레임 쉬핑은 일반적으로 미끄러운 시퀀스의 효과를 높이기 위해 RNA 2차 구조의 존재를 요구한다.[11]RNA 구조(줄기-루프 또는 가성질일 수 있음)는 번역 중에 미끄러운 부위의 리보솜을 정지시켜, 리보솜을 재배치하고 -1 위치에서 복제를 계속할 수밖에 없는 것으로 생각된다.구조물이 리보솜 mRNA 터널에 박혀 리보솜의 움직임을 물리적으로 차단하기 때문에 이런 현상이 발생하는 것으로 생각된다.[2]이 모델은 가성비의 강도가 관련 mRNA에 대한 프레임 쉬핑 수준과 양적으로 상관관계가 있다는 사실에 의해 뒷받침된다.[3][16]
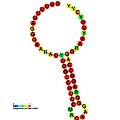
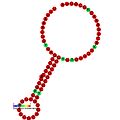
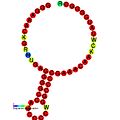
아래는 다양한 유기체에서 프레임 쉬핑을 자극하는 것으로 보이는 프레임시프트 요소에 대한 예측된 이차 구조의 예들이다.표시된 구조물의 대부분은 줄기-루프(ALIL (apical loop-internal 루프) 가성비 구조를 제외하고 있다.이러한 이미지에서 mRNA의 더 크고 불완전한 원은 선형 영역을 나타낸다.같은 가닥의 다른 영역과 mRNA 베이스 페어링 영역에 의해 "스템"이 형성되는 2차 "스템 루프" 구조는 선형 DNA에서 돌출된 것으로 나타난다.HIV 리보솜 프레임시프트 신호의 선형 영역에는 매우 보존된 UUU UUU UUU A 미끄러운 시퀀스가 포함되어 있다. 다른 많은 예측 구조물에는 미끄러운 시퀀스에 대한 후보들도 포함되어 있다.
영상의 mRNA 시퀀스는 일련의 지침에 따라 판독할 수 있다.A, T, C, G는 특정 위치에서 특정 뉴클레오티드를 나타내는 반면, 둘 이상의 뉴클레오티드가 그 위치에서 발생할 수 있을 때 사용되는 모호성을 나타내는 문자도 있다.국제순응화학연맹(IUPAC)의 규칙은 다음과 같다.[17]
| 기호[17] | 설명 | 표시된 기준 | 보완 | ||||
|---|---|---|---|---|---|---|---|
| A | 아데닌 | A | 1 | T | |||
| C | 시토신 | C | G | ||||
| G | 구아닌 | G | C | ||||
| T | 티민 | T | A | ||||
| U | 우라실 | U | A | ||||
| W | 약한 | A | T | 2 | W | ||
| S | 강하다 | C | G | S | |||
| M | a미노 | A | C | K | |||
| K | 케토 | G | T | M | |||
| R | 푸린 | A | G | Y | |||
| Y | 피리미딘 | C | T | R | |||
| B | A가 아니라 (B는 A 다음에 온다) | C | G | T | 3 | V | |
| D | C가 아니라(D는 C 다음에 온다) | A | G | T | H | ||
| H | G가 아님(H는 G 다음에 온다) | A | C | T | D | ||
| V | T가 아님(V는 T와 U 다음에 온다) | A | C | G | B | ||
| N | 모든 뉴클레오티드(공극이 아님) | A | C | G | T | 4 | N |
| Z | 제로 | 0 | Z | ||||
이러한 기호들은 T(지뢰)를 대체하는 U(우라실)를 제외하고는 RNA에도 유효하다.[17]
| 유형 | 분배 | 참조 |
|---|---|---|
| ALIL 유사점 | 박테리아 | [18] |
| Antijyme RNA 프레임시프팅 자극요소 | 무척추동물 | [19] |
| 코로나바이러스 프레임시프팅자극요소 | 코로나바이러스 | [20] |
| DnaX 리보솜 프레임 쉬프팅 원소 | 에우카리오타, 박테리아 | [21] |
| HIV 리보솜 프레임시프트 신호 | 바이러스 | |
| 삽입 시퀀스 IS1222 리보솜 프레임 쉬프팅 요소 | 에우카리오타, 박테리아 | |
| 리보솜 프레임시프트 | 바이러스 |
트랜스액션 요소
작은 분자, 단백질, 핵산이 프레임 쉬핑의 수준을 자극하는 것으로 밝혀졌다.예를 들어, 폴리아민 합성 경로에서 음성 피드백 루프의 메커니즘은 +1 프레임 전환의 증가를 자극하는 폴리아민 수치에 기초하여 억제 효소가 생성된다.코돈 인식에 필요하거나 mRNA 순서에 직접 바인딩되는 특정 단백질도 프레임시프화 수준을 조절하는 것으로 나타났다.마이크로RNA(miRNA) 분자는 RNA 이차 구조로 혼합되어 그 강도에 영향을 미칠 수 있다.[5]
참고 항목
- Antijyme RNA 프레임시프팅 자극요소
- 코로나바이러스 프레임시프팅자극요소
- DnaX 리보솜 프레임 쉬프팅 원소
- 프레임시프트 돌연변이
- HIV 리보솜 프레임시프트 신호
- 삽입 시퀀스 IS1222 리보솜 프레임 쉬프팅 요소
- 데이터베이스 재코딩
- 리보솜 일시 중지
- 미끄러운 순서
참조
- ^ Atkins JF, Loughran G, Bhatt PR, Firth AE, Baranov PV (September 2016). "Ribosomal frameshifting and transcriptional slippage: From genetic steganography and cryptography to adventitious use". Nucleic Acids Research. 44 (15): 7007–7078. doi:10.1093/nar/gkw530. PMC 5009743. PMID 27436286.
- ^ a b c d e Napthine S, Ling R, Finch LK, Jones JD, Bell S, Brierley I, Firth AE (June 2017). "Protein-directed ribosomal frameshifting temporally regulates gene expression". Nature Communications. 8: 15582. Bibcode:2017NatCo...815582N. doi:10.1038/ncomms15582. PMC 5472766. PMID 28593994.
- ^ a b c Ketteler R (2012). "On programmed ribosomal frameshifting: the alternative proteomes". Frontiers in Genetics. 3: 242. doi:10.3389/fgene.2012.00242. PMC 3500957. PMID 23181069.
- ^ Ivanov IP, Atkins JF (2007). "Ribosomal frameshifting in decoding antizyme mRNAs from yeast and protists to humans: close to 300 cases reveal remarkable diversity despite underlying conservation". Nucleic Acids Research. 35 (6): 1842–1858. doi:10.1093/nar/gkm035. PMC 1874602. PMID 17332016.
- ^ a b Dever TE, Dinman JD, Green R (August 2018). "Translation Elongation and Recoding in Eukaryotes". Cold Spring Harbor Perspectives in Biology. 10 (8): a032649. doi:10.1101/cshperspect.a032649. PMC 6071482. PMID 29610120.
- ^ a b Jacks T, Power MD, Masiarz FR, Luciw PA, Barr PJ, Varmus HE (January 1988). "Characterization of ribosomal frameshifting in HIV-1 gag-pol expression". Nature. 331 (6153): 280–283. Bibcode:1988Natur.331..280J. doi:10.1038/331280a0. PMID 2447506. S2CID 4242582.
- ^ a b Jacks T, Madhani HD, Masiarz FR, Varmus HE (November 1988). "Signals for ribosomal frameshifting in the Rous sarcoma virus gag-pol region". Cell. 55 (3): 447–458. doi:10.1016/0092-8674(88)90031-1. PMC 7133365. PMID 2846182.
- ^ Jagger BW, Wise HM, Kash JC, Walters KA, Wills NM, Xiao YL, Dunfee RL, Schwartzman LM, Ozinsky A, Bell GL, Dalton RM, Lo A, Efstathiou S, Atkins JF, Firth AE, Taubenberger JK, Digard P (July 2012). "An overlapping protein-coding region in influenza A virus segment 3 modulates the host response". Science. 337 (6091): 199–204. Bibcode:2012Sci...337..199J. doi:10.1126/science.1222213. PMC 3552242. PMID 22745253.
- ^ Advani VM, Dinman JD (January 2016). "Reprogramming the genetic code: The emerging role of ribosomal frameshifting in regulating cellular gene expression". BioEssays. 38 (1): 21–26. doi:10.1002/bies.201500131. PMC 4749135. PMID 26661048.
- ^ Crick FH (August 1966). "Codon—anticodon pairing: the wobble hypothesis". Journal of Molecular Biology. 19 (2): 548–555. doi:10.1016/S0022-2836(66)80022-0. PMID 5969078.
- ^ a b Brierley I (August 1995). "Ribosomal frameshifting viral RNAs". The Journal of General Virology. 76 (Pt 8) (8): 1885–1892. doi:10.1099/0022-1317-76-8-1885. PMID 7636469.
- ^ a b c d Harger JW, Meskauskas A, Dinman JD (September 2002). "An "integrated model" of programmed ribosomal frameshifting". Trends in Biochemical Sciences. 27 (9): 448–454. doi:10.1016/S0968-0004(02)02149-7. PMID 12217519.
- ^ Gurvich OL, Baranov PV, Gesteland RF, Atkins JF (June 2005). "Expression levels influence ribosomal frameshifting at the tandem rare arginine codons AGG_AGG and AGA_AGA in Escherichia coli". Journal of Bacteriology. 187 (12): 4023–4032. doi:10.1128/JB.187.12.4023-4032.2005. PMC 1151738. PMID 15937165.
- ^ Caliskan N, Katunin VI, Belardinelli R, Peske F, Rodnina MV (June 2014). "Programmed −1 frameshifting by kinetic partitioning during impeded translocation". Cell. 157 (7): 1619–1631. doi:10.1016/j.cell.2014.04.041. PMC 7112342. PMID 24949973.
- ^ Arthur L, Pavlovic-Djuranovic S, Smith-Koutmou K, Green R, Szczesny P, Djuranovic S (July 2015). "Translational control by lysine-encoding A-rich sequences". Science Advances. 1 (6): e1500154. Bibcode:2015SciA....1E0154A. doi:10.1126/sciadv.1500154. PMC 4552401. PMID 26322332.
- ^ Hansen TM, Reihani SN, Oddershede LB, Sørensen MA (April 2007). "Correlation between mechanical strength of messenger RNA pseudoknots and ribosomal frameshifting". Proceedings of the National Academy of Sciences of the United States of America. 104 (14): 5830–5835. Bibcode:2007PNAS..104.5830H. doi:10.1073/pnas.0608668104. PMC 1838403. PMID 17389398.
- ^ a b c Nomenclature Committee of the International Union of Biochemistry (NC-IUB) (1984). "Nomenclature for Incompletely Specified Bases in Nucleic Acid Sequences". Retrieved 4 February 2008.
- ^ Mazauric MH, Licznar P, Prère MF, Canal I, Fayet O (July 2008). "Apical loop-internal loop RNA pseudoknots: a new type of stimulator of −1 translational frameshifting in bacteria". The Journal of Biological Chemistry. 283 (29): 20421–20432. doi:10.1074/jbc.M802829200. PMID 18474594.
- ^ Ivanov IP, Anderson CB, Gesteland RF, Atkins JF (June 2004). "Identification of a new antizyme mRNA +1 frameshifting stimulatory pseudoknot in a subset of diverse invertebrates and its apparent absence in intermediate species". Journal of Molecular Biology. 339 (3): 495–504. doi:10.1016/j.jmb.2004.03.082. PMC 7125782. PMID 15147837.
- ^ Baranov PV, Henderson CM, Anderson CB, Gesteland RF, Atkins JF, Howard MT (February 2005). "Programmed ribosomal frameshifting in decoding the SARS-CoV genome". Virology. 332 (2): 498–510. doi:10.1016/j.virol.2004.11.038. PMC 7111862. PMID 15680415.
- ^ Larsen B, Gesteland RF, Atkins JF (August 1997). "Structural probing and mutagenic analysis of the stem-loop required for Escherichia coli dnaX ribosomal frameshifting: programmed efficiency of 50%". Journal of Molecular Biology. 271 (1): 47–60. doi:10.1006/jmbi.1997.1162. PMC 7126992. PMID 9300054.