반전반복
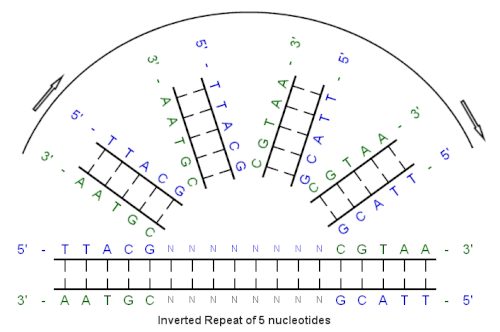
Inverted repeat역반복(또는 IR)은 핵물질의 역보충에 의해 하류로 이어지는 단일 좌초된 시퀀스다.[1] 초기 시퀀스와 역보완 사이에 뉴클레오티드의 간섭 시퀀스는 0을 포함한 어떤 길이도 될 수 있다. 예를 들어 5'-TTACGnnnnnnCGTAA---3'은 반전 반복 시퀀스다. 간섭 길이가 0일 때 복합 시퀀스는 팔린드로믹 시퀀스가 된다.[2]
반전반복과 직접반복 모두 반복적으로 발생하는 뉴클레오티드 시퀀스의 유형을 구성한다. 이러한 반복된 DNA 시퀀스는 종종 뉴클레오티드 한 쌍에서 전체 유전자에 이르기까지 다양하며, 반면에 반복 시퀀스의 근접성은 널리 분산된 탠덤 배열과 단순한 탠덤 배열 사이에서 다양하다.[3] 짧은 탠덤 반복 시퀀스는 대부분의 진핵생물의 게놈 전체에 흩어져 있는 수천 개의 복사본에 대해 작은 영역에 몇 개의 복사본으로 존재할 수 있다.[4] 기본 쌍이 약 10–100인 반복 시퀀스를 미니 위성이라고 하며, 기본 쌍이 대부분 2–4인 짧은 반복 시퀀스를 마이크로 위성이라고 한다.[5] 가장 흔한 반복실험으로는 디뉴클레오티드 반복실험이 있는데, 디뉴클레오티드 반복실험은 하나의 DNA 가닥에 기초 AC가 있고, 보완 가닥에 GT가 있다.[3] 독특한 염기서열을 가진 게놈의 일부 요소들은 엑손, 인트론, 그리고 규제 DNA로 기능한다.[6] 반복 시퀀스 중 가장 친숙한 곳은 센트롬과 텔로미어지만,[6] 게놈의 반복 시퀀스 중 많은 부분이 비코딩 DNA에서 발견된다.[5]
역반복은 많은 중요한 생물학적 기능을 가지고 있다. 그들은 트랜스포존의 경계를 정의하고 자기 보완적 베이스 페어링이 가능한 지역(서로 베이스 페어링할 수 있는 단일 시퀀스 내의 지역)을 나타낸다. 이러한 성질은 게놈 불안정에[7] 중요한 역할을 하며 세포 진화와 유전적 다양성뿐만[8] 아니라 돌연변이와 질병에도 기여한다.[9] 이러한 효과를 상세하게 연구하기 위해, 다양한 게놈에서 역반복의 발견과 주석을 지원하기 위해 다수의 프로그램과 데이터베이스가 개발되었다.
| 관련 항목 |
|---|
반전 반복측정 이해
반전 반복 예제
이 초기 시퀀스부터 시작:
5'-TTACG-3'
베이스 페어링에 의해 생성된 보완 요소는 다음과 같다.
3'-AATGC-5'
역보완은 다음과 같다.
5'-CGTAA-3'
그리고 역반복 순서는 다음과 같다.
5'-TTACGnnnnnnnnnCGTAA---3'
"nnnnn"은 간섭하는 뉴클레오티드의 수를 나타낸다.
Vs 직접 반복
다운스트림에서 동일한 패턴으로 시퀀스를 반복할 때 직접 반복이 발생한다.[1] 직접 반복과 관련된 반전 및 역보완은 없다. 굵은 글씨로 쓰여진 뉴클레오티드 순서는 반복된 순서를 나타낸다. 그것은 뉴클레오티드를 개입시킬 수도 있고 그렇지 않을 수도 있다.
- 5" TTACGnnnnnnnnnnnn.TTACG 3.
- 3"AATGCnnnnnnnnnnnn.AATGC 5.
언어학적으로, 전형적인 직접적인 반복은 "time on a dime"에서와 같이 라임과 비슷하다.
대 탠덤 반복
초기 수열과 그것의 다운스트림 복사 사이에 방해 뉴클레오티드가 없는 직접 반복은 탠덤 반복이다. 굵은 글씨로 쓰여진 뉴클레오티드 순서는 반복된 순서를 나타낸다.
- 5" TTACGTTACG 3"
- 3"AATGCAATGC 5"
언어학적으로 전형적인 탠덤 반복은 "안녕"에서처럼 말을 더듬거나 의도적으로 반복되는 단어와 비슷하다.
대 팔린드롬
초기 시퀀스와 그 다운스트림 역보완 사이에 뉴클레오티드가 개입되지 않은 역반복 시퀀스는 구문(palindrome이다.[1]
예:
1단계: 역반복으로 시작: 5' TTACGnnnnnCTAA 3'
2단계: 간섭 뉴클레오티드 제거: 5' TTACGTAA 3'
이 결과적인 순서는 그 자체의 역보완이기 때문에 팔린드로믹하다.[1]
- 5' TTACGCGTAA 3' 테스트 시퀀스(중간 뉴클레오티드가 제거된 2단계부터)
- 3' AATGCCATT 5' 시험 시퀀스 보완
- 5' TTACGCGTAA 3의 역보완 이것은 위의 시험 순서와 같으므로 팔목이다.
생물학적 특징 및 기능
합성을 선호하는 조건
다양한 게놈 폭의 반복은 전이 가능한 요소로부터 파생되는데, 이 요소들은 현재 다른 게놈 위치에 대해 원래의 복사본을 전송하지 않고 "점프"로 이해되고 있다.[10] 동일한 시퀀스를 수많은 세대에 걸쳐 후속적으로 폐쇄함으로써 게놈 전체에서 그 다중성을 보장한다.[10] 보수적인 현장 고유 재조합(CSSR)이라고 알려진 두 개의 구별되는 시퀀스 요소 사이의 시퀀스들을 제한적으로 재조합하면 기증자 DNA와 수령자 DNA의 재조합 인식 시퀀스의 배열에 기초하여 DNA 세그먼트를 역전시킬 수 있다.[10] 다시 말하지만, 교차 영역이라고 알려진 DNA 갈라짐 시퀀스의 비대칭성에 상대적인 기증자 DNA 분자 내의 재결합 사이트 두 곳의 방향은 반전 반복 또는 직접 반복의 형성에 중추적인 역할을 한다.[10] 따라서 반전된 사이트 쌍에서 발생하는 재결합은 두 사이트 사이의 DNA 순서를 역전시킬 것이다.[10] 매우 안정된 염색체는 직접 반복보다 역반복 횟수가 상대적으로 적은 것으로 관찰되어 염색체의 안정성과 반복 횟수의 관계를 시사하고 있다.[11]
입회 의무가 있는 지역
말단 역반복은 여러 가지 진핵 전이체의 DNA에서 그 근원을 알 수 없음에도 불구하고 관찰되어 왔다.[12] 역반복은 주로 페이지 플라스미드, 미토콘드리아, 진핵 바이러스로부터 포유류 세포에 이르는 세포 유기체와 유기체의 복제에서 발견된다.[13] 페이지 G4 및 기타 관련 페이지의 복제 기원은 복제 프라이밍에 필수적인 세 개의 반전 반복실험을 포함하는 거의 139개의 뉴클레오티드 베이스로 구성된다.[13]
게놈에서
대체로, 뉴클레오티드 반복의 일부는 희귀한 DNA 조합의 일부로 꽤 자주 관찰된다.[14] 특히 DNA 구성에서 주로 발견되는 세 가지 주요 반복측정에는 정밀도가 높은 호모푸린-호모피리미딘 역반복 반복측정(H palindromes라고도 함)이 포함되며, 이는 TAT 또는 CGC 뉴클레오티드 3중으로 구성될 수 있는 삼중 나선 H 순응에서 흔히 발생한다. 다른 것들은 긴 역반복으로 설명될 수 있는데, 머리핀과 십자가형을 생산하는 경향이 있고, 마침내 직접 탠덤 반복으로 설명될 수 있는데, 이것은 일반적으로 슬립 루프, 십자가형, 왼손잡이 Z-DNA로 묘사되는 구조물에 존재한다.[14]
다른 유기체에서 공통됨
과거의 연구들은 반복이 원핵생물이나 고고학과는 달리 진핵생물의 공통적인 특징임을 시사한다.[14] 다른 보고서들은 원핵 게놈의 반복 요소들의 비교 부족과 무관하게, 그럼에도 불구하고 그것들은 수백 혹은 수천 개의 큰 반복적인 요소들을 포함하고 있다고 말한다.[15] 현재의 게놈 분석은 진핵 게놈에 비해 많은 원핵 게놈에서 완전 역반복 반복의 큰 과잉이 존재함을 시사하는 것으로 보인다.[16]
여러 종(예: 고고학) 사이의 역반복 반복측정 정량화 및 비교는 다음을 참조하십시오.
반전 반복측정(가명)
가성비는 RNA에서 흔히 볼 수 있는 구조 모티브다. 그것들은 두 개의 내포된 줄기줄기에 의해 형성되어 한 구조물의 줄기가 다른 구조물의 고리로부터 형성된다. 가성 사이에 여러 개의 접이식 위상이 있고 루프 길이의 차이가 커서 구조적으로 다양한 집단이 된다.[18]
역반복은 인간의 텔로머레이즈 RNA 성분에서 발견되는 자연발생 유사불명의 예에서 볼 수 있는 유사불명의 핵심 성분이다.[19] 이 구조에는 네 가지 서로 다른 반전된 반복측정기가 포함되어 있다. 세트 1과 세트 2는 스템 루프 A의 스템이며 스템 루프 B의 루프에 속한다. 마찬가지로, 세트 3과 세트 4는 스템 루프 B의 스템이며 스템 루프 A의 일부분이다.
가성비는 생물학에서 많은 다른 역할을 한다. 그림의 텔로머레이즈 유사점은 그 효소의 활동에 매우 중요하다.[19] 델타형 간염 바이러스(HDV)의 리보핵은 이중 의사체 구조로 접혀 원형 게놈을 스스로 다듬어 단일 유전체 길이의 RNA를 만들어 낸다.가성체 또한 일부 바이러스에서 발견되고 복고 바이러스 복제에 필요한 프로그램된 리보솜 프레임 쉬프화에도 역할을 한다.[18]
리보스워치에서
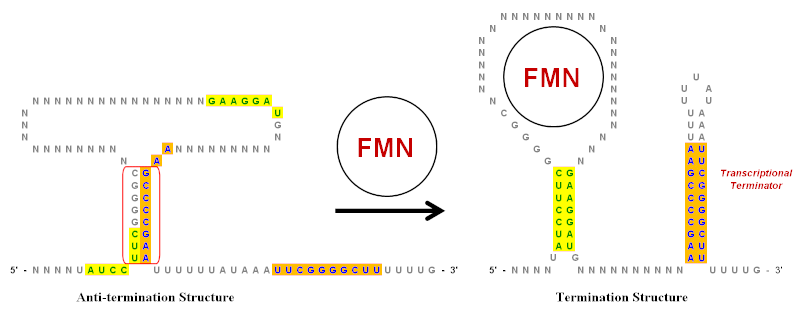
역반복은 리보스랑크에서 중요한 역할을 하는데, 리보스랑크는 그들이 속한 mRNA를 생산하는 유전자의 발현을 조절하는 RNA 규제 요소다.[10] 그림에는 플라빈 단핵화(FMN) 리보스위치의 간단한 예가 나와 있다. 이 리보스위치는 mRNA 대본에 존재하며 코딩 영역에서 업스트림하는 여러 개의 스템 루프 구조를 가지고 있다. 그러나 그림에는 키 스템 루프만 나와 있어 반전된 반복의 역할을 보여주는 데 도움이 되도록 크게 단순화되었다. 이 리보스위치에는 녹색(노란색 배경)과 파란색(주황색 배경)으로 표시된 여러 개의 반전 반복이 있다.
FMN이 없는 경우, 결정방지 구조는 mRNA 성적서에 선호되는 순응이다. 빨간색으로 동그라미를 친 역반복 부위의 베이스 페어링에 의해 생성된다. FMN이 존재할 경우, 그것은 루프에 결합되어 종단방지 구조의 형성을 막을 수 있다. 이를 통해 두 가지 다른 세트의 반전 반복이 베이스 페어에 허용되고 종료 구조를 형성한다.[20] 3의 끝에 있는 줄기-루프는 바로 이어지는 순서가 우라실(U)의 끈이기 때문에 전사적 터미네이터다. RNA 중합효소 복합체에서 성장하는 RNA 가닥이 출현함에 따라 이 스템 루프가 형성(FMN의 존재로 인해)되면 RNA 가닥이 분리되어 전사가 종료될 정도로 충분한 구조적 장력을 생성하게 된다. RNA에 있는 U와 템플릿 가닥에 있는 A 사이의 베이스 페어링이 모든 베이스 페어링 중 가장 약하기 때문에 쉽게 분리된다.[10] 따라서 높은 농도 수준에서 FMN은 종료 구조의 형성을 증가시킴으로써 자체 전사를 하향 조절한다.
돌연변이와 질병
역반복은 진핵 및 원핵 유전체 불안정성의 "핫스팟"으로 설명된다.[7] 긴 역반복은 다양한 생물의 게놈의 안정성에 큰 영향을 미치는 것으로 여겨진다.[21] 이것은 대장균에서 예시되는데, 긴 역반복 반복을 가진 게놈 염기서열은 거의 복제되지 않고 오히려 빠른 속도로 삭제된다.[21] 다시 말하지만, 효모에서 관찰된 긴 반전 반복은 동일하고 인접한 염색체 내의 재조합을 크게 선호하여 똑같이 매우 높은 삭제율을 초래한다.[21] 마지막으로, 포유류 염색체 영역에서도 반복반복과 함께 매우 높은 삭제 및 재조합 비율이 관찰되었다.[21] 상호 관련 유기체의 게놈 안정성 차이는 항상 역반복의 차이를 나타낸다.[11] 불안정성은 반전된 반복의 경향에서 비롯된다. 머리핀이나 십자가 모양의 DNA 구조로 접히는 것이다. 이러한 특별한 구조는 DNA 복제와 다른 유전학적 활동을 방해하거나 혼동시킬 수 있다.[7] 따라서 반전반복은 결국 돌연변이와 질병을 유발할 수 있는 RNA와 DNA 둘 다의 특별한 구성으로 이어진다.[9]
그림에는 십자가형 압출이 발생하는 역반복 반복이 표시된다. 반전된 영역의 DNA는 바람을 풀고 나서 다시 결합하여 두 개의 줄기-루프 구조로 4방향 접합부를 형성한다. 십자가형 구조는 역반복된 반복이 그들 자신의 가닥에서 서로에 대한 자기 페어링을 시퀀스하기 때문에 발생한다.[22]
돌출된 십자가형은 DNA 염기서열이 반전되어 양쪽의 직접 반복 영역과 결합되어 팔목의 형태로 반복될 때 프레임 변형으로 이어질 수 있다. 전사하는 동안 템플릿 스트랜드에서 폴리머아제 미끄러짐과 부분적인 분리는 삭제와 삽입 돌연변이를 유발할 수 있다.[9] 삭제는 풀리지 않은 템플릿 스트랜드의 일부가 전사 기계에 의해 "스키핑"되는 스템 루프를 형성할 때 발생한다. 삽입은 스템 루프가 초기(신규 합성) 스트랜드의 분리된 부분에 형성되어 템플릿 스트랜드의 일부가 두 번 복사될 때 발생한다.[9]
포인트 돌연변이로 인한 안티트롬빈 결핍증
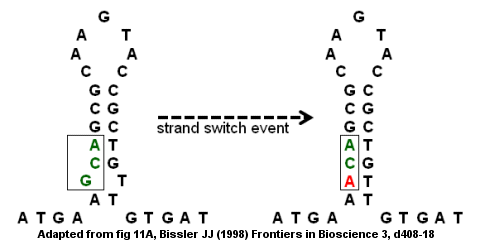
불완전한 역반복은 주내 및 주내 전환을 통해 돌연변이를 유발할 수 있다.[9] 안티트롬빈 III 유전자의 코딩 부위는 오른쪽 그림과 같이 불완전한 역반복의 예다. 줄기-루프 구조는 G와 T가 짝을 이루지 않기 때문에 하단에 혹이 생겨난다. 스트랜드 스위치 이벤트로 인해 G(범프 내)가 A로 교체될 수 있으며, 이 경우 반전된 반복에서 "불완전"이 제거되고 더 강한 스템 루프 구조를 제공할 수 있다. 그러나, 그 교체는 또한 CCA 코돈을 ACA로 변환하는 점 돌연변이를 생성한다. 만약 스트랜드 스위치 사건이 2차 DNA 복제에 따른다면, 돌연변이는 게놈에 고정되어 질병으로 이어질 수 있다. 구체적으로, 오감 돌연변이는 결함이 있는 유전자와 항혈전증(정맥 내의 혈전)의 발달을 초래할 수 있는 항혈전자의 결핍으로 이어질 수 있다.[9]
골격변형으로 인한 불완전성
콜라겐 유전자의 돌연변이는 뼈가 부서지기 쉬운 것이 특징인 Osteogenesis 불완전성 질환으로 이어질 수 있다.[9] 그림에서 불완전한 역반복으로 형성된 스템루프는 근거리 또는 근거리 내 스위치의 결과로 티민(T) 뉴클레오티드 삽입으로 변이된다. T를 추가하면 기존에 스템 왼쪽에 '범프'였던 아데닌(A)과 베이스 페어링 '매칭 업'이 생성된다. 이 추가는 줄기를 더 강하게 하고 반전된 반복을 완벽하게 하는 반면, 뉴클레오티드 시퀀스에 프레임-변형을 만들어내서 판독 프레임을 변경하고 유전자의 잘못된 발현을 초래하게 된다.[9]
프로그램 및 데이터베이스
다음 목록은 반전 반복을 위한 다양한 프로그램 및 데이터베이스에 대한 정보와 외부 링크를 제공한다.
- 비-B DB 통합 주석 및 비-B DNA 형성 모티브 분석을 위한 데이터베이스.[23] 이 데이터베이스는 당시 FNLCR(Frederick National Laboratory for Cancer Research)에 있는 ABCC(Advanced Biomedical Computing Center)에서 제공한다. 그것은 오른손잡이 왓슨 크릭 더블 헥스의 더 일반적인 B-DNA 형태가 아니기 때문에 "비-B DNA"로 알려진 A-DNA와 Z-DNA 순응을 다룬다. 이러한 "B가 아닌 DNA"에는 왼손잡이 Z-DNA, 십자가형, 트리플렉스, 테트라플렉스, 머리핀 구조가 포함된다.[23] 검색은 다양한 "반복 유형"(역반복 반복 포함)과 여러 종에 대해 수행할 수 있다.
- 반전 반복 데이터베이스 보스턴 대학교. 이 데이터베이스는 PULITY DATABASE 프로젝트에서 보유 중인 반복실험에 대한 질의와 분석이 가능한 웹 애플리케이션이다. 과학자들은 또한 반전 반복 검출 알고리즘으로 그들 자신의 시퀀스를 분석할 수 있다.[24]
- P-MITE: 플랜트 MITE 데이터베이스 — MITE(Minimal Invert-repeat Transposable Elements)를 위한 이 데이터베이스는 플랜트 게놈의 시퀀스를 포함한다. 시퀀스는 데이터베이스에서 검색하거나 다운로드할 수 있다.[25]
- EMBOSS는 유닉스(UNIX)와 유닉스(UNIX) 유사 운영 체제에서 실행되는 '유럽 분자생물 오픈 소프트웨어 세트'이다.[26] 문서와 프로그램 원본 파일은 EMBOSS 웹사이트에서 이용할 수 있다. 역반복과 관련된 애플리케이션은 다음과 같다.
- EMBOSS einverted: 뉴클레오티드 시퀀스에서 반전 반복측정 찾기 임계값은 검색 범위를 제한하도록 설정할 수 있다.[26]
- 엠보스 팔레드롬: 뉴클레오티드 시퀀스에서 스템 루프 영역과 같은 팔레드롬을 찾는다. 이 프로그램은 줄기 루프에서 돌출에 해당할 수 있는 불일치 부분과 간격 부분을 포함하는 시퀀스를 찾을 것이다.[26]
참조
- ^ a b c d Ussery, David W.; Wassenaar, Trudy; Borini, Stefano (2008-12-22). "Word Frequencies, Repeats, and Repeat-related Structures in Bacterial Genomes". Computing for Comparative Microbial Genomics: Bioinformatics for Microbiologists. Computational Biology. Vol. 8 (1 ed.). Springer. pp. 133–144. ISBN 978-1-84800-254-8.
- ^ Ye, Congting; Ji, Guoli; Liang, Chun (2014). "detectIR: A Novel Program for Detecting Perfect and Imperfect Inverted Repeats Using Complex Numbers and Vector Calculation". PLoS ONE. 9 (11): e113349. doi:10.1371/journal.pone.0113349. PMID 25409465.
- ^ a b Richards, GR; Richards, RI (Apr 25, 1995). "Simple tandem DNA repeats and human genetic disease". Proceedings of the National Academy of Sciences of the United States of America. 92 (9): 3636–41. Bibcode:1995PNAS...92.3636S. doi:10.1073/pnas.92.9.3636. PMC 42017. PMID 7731957.
- ^ van Belkum, A; Scherer, S; van Alphen, L; Verbrugh, H (June 1998). "Short-sequence DNA repeats in prokaryotic genomes". Microbiology and Molecular Biology Reviews. 62 (2): 275–93. doi:10.1128/MMBR.62.2.275-293.1998. PMC 98915. PMID 9618442.
- ^ a b Ramel, C (June 1997). "Mini- and microsatellites". Environmental Health Perspectives. 105 Suppl 4 (Suppl 4): 781–9. doi:10.2307/3433284. JSTOR 3433284. PMC 1470042. PMID 9255562.
- ^ a b Eichler, EE (August 1998). "Masquerading repeats: paralogous pitfalls of the human genome". Genome Research. 8 (8): 758–62. doi:10.1101/gr.8.8.758. PMID 9724321.
- ^ a b c Mirkin, I; Narayanan, V; Lobachev, KS; Mirkin, SM (Jul 22, 2008). "Replication stalling at unstable inverted repeats: interplay between DNA hairpins and fork stabilizing proteins". Proceedings of the National Academy of Sciences of the United States of America. 105 (29): 9936–41. Bibcode:2008PNAS..105.9936V. doi:10.1073/pnas.0804510105. PMC 2481305. PMID 18632578.
- ^ Lin, CT; Lin, WH; Lyu, YL; Whang-Peng, J (Sep 1, 2001). "Inverted repeats as genetic elements for promoting DNA inverted duplication: implications in gene amplification". Nucleic Acids Research. 29 (17): 3529–38. doi:10.1093/nar/29.17.3529. PMC 55881. PMID 11522822.
- ^ a b c d e f g h Bissler, JJ (Mar 27, 1998). "DNA inverted repeats and human disease" (PDF). Frontiers in Bioscience. 3 (4): d408–18. doi:10.2741/a284. PMID 9516381. S2CID 12982. Archived from the original (PDF) on March 3, 2019.
- ^ a b c d e f g School, James D. Watson, Cold Spring Harbor Laboratory, Tania A. Baker, Massachusetts Institute of Technology, Stephen P. Bell, Massachusetts Institute of Technology, Alexander Gann, Cold Spring Harbor Laboratory, Michael Levine, University of California, Berkeley, Richard Losik, Harvard University ; with Stephen C. Harrison, Harvard Medical (2014). Molecular biology of the gene (Seventh ed.). Boston: Benjamin-Cummings Publishing Company. ISBN 9780321762436.
- ^ a b Achaz, G; Coissac, E; Netter, P; Rocha, EP (August 2003). "Associations between inverted repeats and the structural evolution of bacterial genomes". Genetics. 164 (4): 1279–89. doi:10.1093/genetics/164.4.1279. PMC 1462642. PMID 12930739.
- ^ Zhang, HH; Xu, HE; Shen, YH; Han, MJ; Zhang, Z (January 2013). "The Origin and Evolution of Six Miniature Inverted-Repeat Transposable Elements in Bombyx mori and Rhodnius prolixus". Genome Biology and Evolution. 5 (11): 2020–31. doi:10.1093/gbe/evt153. PMC 3845634. PMID 24115603.
- ^ a b Pearson, CE; Zorbas, H; Price, GB; Zannis-Hadjopoulos, M (October 1996). "Inverted repeats, stem-loops, and cruciforms: significance for initiation of DNA replication". Journal of Cellular Biochemistry. 63 (1): 1–22. doi:10.1002/(SICI)1097-4644(199610)63:1<1::AID-JCB1>3.0.CO;2-3. PMID 8891900. S2CID 22204780.
- ^ a b c Heringa, J (June 1998). "Detection of internal repeats: how common are they?". Current Opinion in Structural Biology. 8 (3): 338–45. doi:10.1016/S0959-440X(98)80068-7. PMID 9666330.
- ^ Treangen, TJ; Abraham, AL; Touchon, M; Rocha, EP (May 2009). "Genesis, effects and fates of repeats in prokaryotic genomes" (PDF). FEMS Microbiology Reviews. 33 (3): 539–71. doi:10.1111/j.1574-6976.2009.00169.x. PMID 19396957.
- ^ Ladoukakis, ED; Eyre-Walker, A (September 2008). "The excess of small inverted repeats in prokaryotes" (PDF). Journal of Molecular Evolution. 67 (3): 291–300. Bibcode:2008JMolE..67..291L. CiteSeerX 10.1.1.578.7466. doi:10.1007/s00239-008-9151-z. PMID 18696026. S2CID 29953202.
- ^ Hosseini, M; Pratas, D; Pinho, AJ (2017). "On the Role of Inverted Repeats in DNA Sequence Similarity". 11th International Conference on Practical Applications of Computational Biology & Bioinformatics. Advances in Intelligent Systems and Computing. Vol. 616. Springer. pp. 228–236. doi:10.1007/978-3-319-60816-7_28. ISBN 978-3-319-60815-0.
- ^ a b Staple, DW; Butcher, SE (June 2005). "Pseudoknots: RNA structures with diverse functions". PLOS Biology. 3 (6): e213. doi:10.1371/journal.pbio.0030213. PMC 1149493. PMID 15941360.

- ^ a b Chen, JL; Greider, CW (Jun 7, 2005). "Functional analysis of the pseudoknot structure in human telomerase RNA". Proceedings of the National Academy of Sciences of the United States of America. 102 (23): 8080–5, discussion 8077–9. Bibcode:2005PNAS..102.8080C. doi:10.1073/pnas.0502259102. PMC 1149427. PMID 15849264.
- ^ Winkler, WC; Cohen-Chalamish, S; Breaker, RR (Dec 10, 2002). "An mRNA structure that controls gene expression by binding FMN". Proceedings of the National Academy of Sciences of the United States of America. 99 (25): 15908–13. Bibcode:2002PNAS...9915908W. doi:10.1073/pnas.212628899. PMC 138538. PMID 12456892.
- ^ a b c d Stormo, G; Chang, KY; Varley, K; Stormo, GD (Feb 28, 2007). Hall, Neil (ed.). "Evidence for active maintenance of inverted repeat structures identified by a comparative genomic approach". PLOS ONE. 2 (2): e262. Bibcode:2007PLoSO...2..262Z. doi:10.1371/journal.pone.0000262. PMC 1803023. PMID 17327921.

- ^ Ramreddy, T; Sachidanandam, R; Strick, TR (May 2011). "Real-time detection of cruciform extrusion by single-molecule DNA nanomanipulation". Nucleic Acids Research. 39 (10): 4275–83. doi:10.1093/nar/gkr008. PMC 3105387. PMID 21266478.
- ^ a b Cer, RZ; Donohue, DE; Mudunuri, US; Temiz, NA; Loss, MA; Starner, NJ; Halusa, GN; Volfovsky, N; Yi, M; Luke, BT; Bacolla, A; Collins, JR; Stephens, RM (January 2013). "Non-B DB v2.0: a database of predicted non-B DNA-forming motifs and its associated tools". Nucleic Acids Research. 41 (Database issue): D94–D100. doi:10.1093/nar/gks955. PMC 3531222. PMID 23125372.
- ^ Gelfand, Y; Rodriguez, A; Benson, G (January 2007). "TRDB--the Tandem Repeats Database". Nucleic Acids Research. 35 (Database issue): D80–7. doi:10.1093/nar/gkl1013. PMC 1781109. PMID 17175540.
- ^ Chen, J; Hu, Q; Zhang, Y; Lu, C; Kuang, H (Oct 29, 2013). "P-MITE: a database for plant miniature inverted-repeat transposable elements". Nucleic Acids Research. 42 (1): D1176–81. doi:10.1093/nar/gkt1000. PMC 3964958. PMID 24174541.
- ^ a b c Rice, P; Longden, I; Bleasby, A (June 2000). "EMBOSS: the European Molecular Biology Open Software Suite". Trends in Genetics. 16 (6): 276–7. doi:10.1016/S0168-9525(00)02024-2. PMID 10827456.