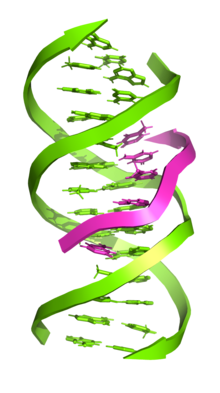
삼가닥 DNA
Triple-stranded DNA3가닥 DNA(H-DNA 또는 Triplex-DNA라고도 함)는 3개의 올리고뉴클레오티드가 서로 감겨 삼중나선을 형성하는 DNA 구조이다.3가닥 DNA에서, 세 번째 가닥은 후그스틴 염기쌍을 형성하거나 역후그스틴 수소 결합을 형성함으로써 (왓슨-크릭 염기쌍을 통해) B형 DNA에 결합한다.
구조.
세 가닥 DNA의 예는 예를 [1]들어 위성 DNA와 같이 필요한 구조적 요소를 가진 자연 소스에서 발견되었습니다.
Hoogsteen 염기쌍
티민(T) 핵염기는 후그스틴 수소 결합을 형성함으로써 T-A의 왓슨-크릭 염기쌍에 결합할 수 있다.티민 수소는 원래의 이중 가닥 DNA의 아데노신(A)과 결합하여 T-A*T 염기-트리플렛을 [2]생성한다.
분자간 및 분자내 상호작용
트리플렉스 DNA에는 분자간 형성과 분자내 형성의 두 가지 클래스가 있습니다.분자간 삼중배열은 이중배열과 다른 (세 번째) DNA 가닥 사이의 삼중배열 형성을 말한다.세 번째 가닥은 인접한 염색체 또는 올리고뉴클레오티드(TFO)를 형성하는 삼중 염색체일 수 있다.미러 반복 대칭을 가진 호모푸린 및 호모피리미딘 가닥을 가진 이중체로부터 분자 내 트리플렉스 DNA를 형성한다.[4]DNA의 초코일의 정도는 일어나는 [5]분자 내 삼중배 형성 양에 영향을 미친다.분자 내 삼중 DNA에는 H-DNA와 H*-DNA의 두 가지 다른 유형이 있습니다.H-DNA의 형성은 산성 조건 및 Mg 등의 2가의2+ 양이온 존재 하에서 안정화된다.이 형태에서 이중화 중의 호모피리미딘 가닥은 푸린 가닥에 병렬로 결합하기 위해 뒤로 구부러진다.이 구성을 안정화하기 위해 사용되는 베이스 트라이어드는 T-A*T와 C-G*A입니다+.이러한 분자 내 삼중 나선을 형성하기 위해서는 이 염기 삼중의 시토신이 양성자화되어야 하며, 이것이 산성 [6]조건 하에서 이러한 배치가 안정화되는 이유이다.H*-DNA는 중성 pH 및 2가 양이온 [5]존재 하에서 양호한 형성 조건을 가진다.이 분자 내 배치는 이중체의 호모푸린과 푸린 가닥이 반평행적으로 결합함으로써 형성된다.T-A*A 및 C-G*G 염기 [4][6]세쌍둥이에 의해 안정화된다.
기능.
올리고뉴클레오티드(TFO)를 형성하는 트리플렉스
TFO는 2가닥 DNA의 주요 홈에 결합하여 분자 내 3중 DNA 구조를 형성하는 짧은 핵산 가닥이다.또한 그들이 생체 내에서 유전자 활성을 조절할 수 있다는 몇 가지 증거가 있다.펩타이드핵산(PNA)은 DNA의 당인산 골격을 단백질상의 골격으로 치환한다.PNA는 이중 DNA와 상호작용하는 동안 P-루프를 형성하고, 다른 DNA를 치환하는 동안 한 가닥의 DNA로 3중고를 형성한다.상동 재조합 과정에서 RecA 단백질 아래에서 매우 특이한 재조합 [7]또는 병렬 삼중수소(R-DNA)가 형성되는 것으로 가정되어 왔다.
TFO는 특이적으로 호모푸린-호모피리미딘 영역에 결합하며, 호모푸린-호모피리미딘 영역은 종종 유전자의 프로모터 및 인트론 배열에서 흔하며 세포 신호 [8]전달에 영향을 미친다.TFO는 DNA 나선에 대한 높은 특이성으로 결합함으로써 전사를 억제할 수 있으며, 따라서 특정 배열에 대한 전사 인자의 결합과 기능을 차단할 수 있다.TFO를 세포에 도입함으로써(트랜스펙션 또는 다른 수단을 통해) 특정 유전자의 발현을 [9]제어할 수 있다.이 애플리케이션은 부위 특이적 돌연변이 유발과 유전자 치료에 새로운 의미를 갖는다.인간 전립선암세포에서는 전사인자 Ets2가 과잉 발현되어 세포의 성장과 생존을 촉진하는 것으로 생각된다.Carbone 등은 Ets2 프로모터 배열에 대한 배열 특이적 TFO를 설계하여 유전자 발현을 억제하고 세포 성장 속도를 늦추고 [10]세포 사멸을 초래했다.창셴 등는 또한 [11]아포토시스를 억제하는 유전자인 bcl-2의 프로모터 배열을 대상으로 하는 TFO를 제시했다.
관찰된 전사의 금지는 또한 프리드라이히의 [12]아탁시아에 대한 열성, 상염색체 유전자에 대한 역할과 같이 건강에 부정적인 영향을 미칠 수 있습니다.프레드릭의 아탁시아에서는 3중 DNA 형성은 FXN 유전자의 인트론 1의 발현을 손상시킨다.이것은 신경계와 척수의 퇴화를 초래하여 [13]팔다리의 움직임을 손상시킨다.이 3중 불안정성과 싸우기 위해, 뉴클레오티드 절제 복구 단백질은 이전에 억제되고 불안정한 유전자의 완전한 가용성을 회복하면서 [14]3중 사슬 DNA 구조를 인식하고 복구하는 것으로 나타났다.

펩타이드핵산(PNA)
펩타이드 핵산은 단백질 분해효소 분해에 저항하는 합성 올리고뉴클레오티드이며 DNA 게놈 부위의 부위 특이적 트리플렉스 형성 영역에서 복구를 유도하는 데 사용됩니다.PNA는 Watson-Crick 염기쌍 결합을 통해 상보적인 DNA 배열에 높은 친화력과 배열 특이성으로 결합할 수 있으며, DNA 가닥의 [15]5' 말단에 면한 PNA와 평행 배향 Hoogsteen 결합을 통해 3중 나선형을 형성할 수 있다.PNA는 중성적으로 충전된 의사펩타이드 골격으로 구성되어 이중 가닥 DNA(dsDNA) [16]배열에 결합하기 때문에 PNA-DNA 삼중배열은 안정적입니다.TFO의 호모피리미딘과 유사하게, PNA의 호모피리미딘은 dsDNA의 표적 배열에서 상보적인 호모푸린과 결합을 형성할 수 있다.이러한 DNA 유사체는 주변 DNA 조건과 다양한 인식 예측 모드를 이용하여 dsDNA와 결합할 수 있다.이는 dsDNA의 [15]주요 그루브 인식을 통해 바인드되는 TFO와는 다릅니다.
인식에 사용되는 인식의 예측 모드 중 하나는 이중 [17][16]침입을 통한 것이다.혼합 A-T/G-C dsDNA 배열은 각각 [17]아데닌과 티오우라실을 대체하는 디아미노푸린(D)과 티오우라실(Us)의 동시 형성을 통해 이중 침투를 통해 dsDNA에 결합할 수 있는 의사 상보적(pc) PNA 쌍에 의해 표적화된다.pc PNA 쌍은 각각의 상보적인 DNA 가닥과 함께 D-T와s U-A와 G-C 또는 C-G 왓슨-Crick 쌍으로 이루어진 PNA-DNA 나선을 형성한다.T–C [18]시퀀스가 혼재된dsDNA에서는 타깃시퀀스로 인식된 다른 형태의 듀플렉스 침해가 발생할 수 있습니다.이러한 이중 침입 형태는 호모푸린 PNA 올리고머의 상보적 염기서열을 통해 달성된다.이 삼중배열은 상보적 DNA 배열과 반병렬 결합하고 치환된 비상보적 DNA [16]가닥을 생성하는 PNA-DNA 하이브리드로부터 형성된다.
또한 PNA는 대상 부위에서 [17]"클램프" 트리플플렉스 구조를 형성하도록 수정할 수 있다.형성되는 '클램프'의 한 종류는 2개의 PNA 분자가 8-아미노-3,6-다이옥사옥탄산(O)[19]과 같은 가요성 링커에 의해 결합되는 비스-PNA 구조이다.bis-PNA 구조는 표적 부위에서 PNA-DNA-PNA 삼중배열을 형성하며, 여기서 한 가닥은 역평행 방향의 DNA와 왓슨-크릭 염기쌍을 형성하고, 다른 한 가닥은 DNA-PNA [18]이중체의 호모푸린 DNA 가닥과 후그스틴 염기쌍을 형성한다.테일 클램프 PNA(tcPNA)도 형성할 수 있는 트리플렉스 클램프의 또 다른 형태이다.TcPNA에는 PNA-DNA-PNA "clamp" 외에 PNA/DNA 듀플렉스를 형성하는 확장 5-10 bp 꼬리가 포함되어 있습니다.이를 통해 호모피리미디/[16]피리딘 스트레칭 없이 보다 특정 PNA 결합을 수행할 수 있습니다.이러한 클램프 구조는 높은 친화력과 특이성을 가진 것으로 나타났다.PNA의 한쪽 또는 양쪽 끝에 리신 잔기를 첨가하는 것은 세포 흡수 [17]및 결합을 증가시키는데 사용될 수 있다.
유전자 조절
3가닥 DNA는 여러 유전자의 조절에 관여해 왔다.예를 들어, c-myc 유전자는 유전자 조절에서 선형 염기서열이 아닌 삼중 DNA가 수행하는 역할을 조사하기 위해 광범위하게 변이되었다.핵산가수분해효소 감수성 원소 또는 NSE라고 불리는 c-myc 프로모터 원소는 H-DNA형의 분자 내 탠덤 3중시스를 형성할 수 있으며 반복 배열 모티브(ACCTCC)4를 가진다.돌연변이 NSE는 전사 활성과 분자 내 및 분자 간 삼중 형성 능력을 검사했다.돌연변이 NSE의 전사 활성은 반복 횟수, 위치 또는 돌연변이 염기쌍의 수가 아닌 H-DNA를 형성하는 원소의 능력에 의해 예측될 수 있습니다.따라서 DNA는 c-myc [20]유전자의 전사에 동적으로 참여할 수 있다.
유전자 발현
몇몇 발표된 기사에 따르면, H-DNA는 근접한 위치와 배열과 같은 요인에 따라 유전자 발현을 조절하는 능력을 가지고 있다.원핵 게놈의 유전자 간 영역은 자연적으로 발생하는 H-DNA 또는 3중 모티브의 낮은 흔적을 보였지만, H-DNA 구조는 진핵 게놈에서 더 널리 퍼져 있는 것으로 나타났다.H-DNA는 인간을 포함한 포유동물 세포에 특히 풍부한 것으로 나타났다(5만 [15]bp당 1개).유전자 조절에 관여하는 유전자 배열은 전형적으로 진핵생물 [15]게놈의 프로모터 영역에서 발견된다.
그 결과 프로모터 영역은 보다 높은 [15]빈도로 H-DNA를 형성할 수 있는 능력을 보였다.S. cerevisiae 게놈의 생물정보학적 분석은 인트론, 엑손, 프로모터 영역 및 기타 영역의 네 가지 조직 영역에서 H-DNA와 다른 3판 DNA 모티브의 발생을 관찰했다.생물정보학자는 총 148개의 H-DNA 또는 트리플렛 DNA 가능한 구조를 보였다.프로모터 영역은 71개의 삼판구조로 더 높은 빈도를 차지했으며 엑손은 57개의 삼판구조, 인트론 및 기타 구조는 2와 18개의 [21]구조를 차지했다.
진핵생물 게놈 발현에 대한 체외 및 체내 연구는 상승 조절, 하강 조절 또는 H-DNA [15]모티브의 존재에 변화가 없는 세 가지 결과 중 하나를 얻었다.가토 등은 B-락타마아제 프로모터에 [22][15]H-DNA가 도입되었을 때 lacZ의 상향 조절 발현을 보고했다.한편, 유사한 연구(Brachmachari 등)에서는 포유동물 COS [15]세포의 게놈에 H-DNA를 삽입했을 때 lacZ 리포터 유전자의 통계적으로 유의한 억제가 없었다고 보고했다.H-DNA의 조절을 시사하는 연구도 있지만, 그 메커니즘은 아직 연구 중이다.Potaman et al.은 유전자 조절 메커니즘을 Na, K-ATPase의 프로모터 영역에서 발견된 H-DNA와 TATA 상자 사이의 상호작용과 연관시킨다.TATA 박스에 인접한 H-DNA 형성은 H-DNA 구조가 전사에 필수적인 T-A 결합을 불안정하게 한다.TATA 상자와의 간섭은 유전자 [15][23]발현을 방해하는 전사 기계와 전사 시작을 억제합니다.H-DNA의 존재 하에서 유전자 배열의 게놈 발현과 관련된 다른 메커니즘은 TFO를 포함한다.체외 연구는 포유동물 [24]세포에서 TFO가 존재할 때 유전자 발현 감소가 두드러졌다.발렌티나 등에 의해 제시된 또 다른 가능한 메커니즘은 13-mer AG 모티브 올리고뉴클레오티드 트리플렉스 복합체(TFO 복합체)가 경쟁 [25]억제를 통해 mRNA의 전사를 하향 조절한다는 것을 시사한다.H-DNA로부터의 유전자 발현을 직접적으로 억제하는 것은 돌연변이 유발, 복제 억제, 그리고 [15]게놈의 DNA 재조합에 핵심이다.
재결합
H-DNA 모티브는 다른 메커니즘으로 상동 재조합을 자극하는 것으로 나타났다.재조합에서 H-DNA의 역할에 대한 최초의 의미는 1990년대 초 삼중나선 DNA로 구성된 박테리아 DNA 재조합 단백질인 RecA를 관찰했을 때 나타났다.RecA는 [7][26]재조합에 필수적인 효소 활성을 나타낸다.H-DNA 모티브를 포함한 상동 재조합도 진핵생물에서 발견되었다.RecA와 상동 단백질인 RadA는 RecA와 [27]동일한 재조합 효소 활성을 갖는 것으로 나타났다.단백질은 평행한 3중 가닥나선을 [28][29]통해 상동 가닥을 촉진하고 교환하는 능력을 가지고 있다.단일 가닥 DNA(ssDNA)와 보완적인 이중 가닥 DNA(dsDNA)는 D-루프 [30][15]구조를 형성합니다.RecA의 또 다른 가능한 메커니즘은 왓슨-크릭 염기쌍을 형성하기 위해 두 개의 분리된 H-DNA 구조에서 ssDNA를 포함한다.새로운 구조는 홀리데이 접합으로 알려져 있으며, 상동 [15]재조합의 중간체입니다.H-DNA는 다른 형태의 재조합에서도 발견된다.포유류 세포에서 H-DNA 염기서열은 높은 재조합 빈도를 보였다.예를 들어 생쥐 골수종 세포주를 대상으로 한 연구에서 자매 염색체 [15]교환에 관여하는 C22a와 C22b에서 H-DNA 구조가 발견되었다.
생물학적 영향
유전적 불안정성
주요 중단점 영역(Mbr) 및 특정 유전자의 이중 가닥 차단점에 H-DNA의 존재와 관련된 생물학적 의미에 상당한 연구가 쏟아졌다.최근의 연구는 비 B-DNA 구조의 존재를 유전적 불안정 [31]사례와 연결시켰다.
폴리퓨린 미러 반복 H-DNA 형성 서열이 c-MYC 유전자의 P1 프로모터 근처에서 발견되었으며, 이 영역의 주요 중단점 핫스팟과 관련이 있다.이전에 불안정성이 [32]보고되지 않았던 게놈에 Z-DNA 염기서열과 쌍을 이룬 인간 H-DNA 생성 염기서열을 통합한 후 유전자 변형 생쥐의 F1 자손에서도 유전자 불안정 사례가 관찰되었다.또한 BCL-2 유전자의 MBr에서 R.R.Y. H-DNA 배치가 형성되는 것이 관찰되었다.이러한 구조의 형성은 많은 암과 대부분의 모낭 림프종에서 관찰되는 t(14;18) 전위를 유발하는 것으로 추정되었다.이러한 관찰을 통해 이 영역의 염기서열을 [32][33]약간 변경하여 H-DNA 형성을 차단한 후 전위 이벤트의 상당한 감소를 관찰할 수 있다는 연구 결과가 나왔다.GAA의 긴 계약TTC는 또한 매우 안정적인 H-DNA 구조를 형성하는 것으로 관찰되었다.스틱 DNA라고 불리는 이 두 H-DNA 구조 사이의 상호작용은 X25, 즉 프라탁신 유전자의 전사를 방해하는 것으로 나타났다.단백질 프라탁신의 감소가 프리드라이히의 운동실조증과 관련이 있기 때문에, 이러한 불안정성의 형성은 이 유전병의 [34][35]기초가 될 것으로 제안되어 왔다.위성 DNA에서 큰 2.1kb 위성 DNA 반복 단위 내의 대칭 Z-DNA 구조와 함께 마이크로 위성 복사 번호가 매우 가변적인 지역에서 3가닥 DNA가 관찰되었다. [36][37]
또한, H-DNA는 DNA 복제와 [38]전사 같은 중요한 세포 과정과 관련된 돌연변이를 일으키는 것으로 나타났다.생존을 위한 이러한 과정의 중요성은 세포가 DNA 손상을 인식하고 고칠 수 있도록 하는 복잡한 DNA 복구 메커니즘의 개발을 이끌었다.비표준 DNA 구조는 세포에 의한 손상으로 인식될 수 있으며, 최근의 연구는 비 B-DNA 형성 [38]염기서열 근처에서 돌연변이의 유병률을 증가시켰다.이러한 돌연변이의 일부는 H-DNA와 DNA 복제와 전사에 관여하는 효소 사이의 상호작용에 기인하며, H-DNA는 이러한 과정을 방해하고 다양한 DNA 복구 메커니즘을 유발한다.이것은 유전적 불안정성을 유발할 수 있고 H-DNA가 암 [38]형성에 관여할 수 있다.
DNA복제
DNA 복제는 다양한 DNA 복구 효소의 기능에 영향을 미치는 것으로 나타났다.H-DNA 형성은 핵산 [38]분해 효소에 의해 공격받기 쉬운 단일 가닥 DNA(ssDNA)의 형성을 포함한다.다양한 핵산가수분해효소는 복제의존적 또는 복제의존적 방식으로 [38]H-DNA와 상호작용하는 것으로 나타났다.
인간 세포를 이용한 연구는 뉴클레오티드 절제 수리(NER)가 ERCC1-XPF와 ERCC1-XPG 유도 유전 [39]불안정성을 일으키는 것을 발견했다.이들 효소는 각각 두 개의 Hoogsteen 수소 결합 가닥에 의해 형성된 루프와 다른 Watson-Crick 수소 결합 가닥의 5' 말단에서 H-DNA를 분해한다.[39]이 분열은 유전자 [38][39]불안정을 초래할 수 있는 DNA의 이중 가닥 절단(DSB)을 일으키는 대규모 결실을 유도하는 것으로 나타났다.ERCC1-XPF 및 ERCC1-XPG가 결핍된 세포에서는 이러한 결손이 H-DNA 형성 근처에서 덜 흔했다.[39]또한 DNA 복제가 없는 상태에서 ERCC1-XPF 및 ERCC1-XPG 결핍 세포에서 더 많은 돌연변이가 발견되었으며, 이는 이들이 복제에 의존하지 않는 방식으로 [39]H-DNA를 처리함을 시사한다.
또는 DNA 복제 복구 [39]핵산가수분해효소 FEN1은 유전적 불안정성을 억제하는 것으로 밝혀졌다.ERCC1-XPG와 마찬가지로 FEN1은 Hoogsteen 수소 [39]결합에 관여하지 않는 가닥의 5' 말단에서 H-DNA를 절단한다.FEN1이 결핍된 Hela 세포는 H-DNA 형성 염기서열 부근에서 결실 유병률이 높았으나, H-DNA 유도 돌연변이 유발은 DNA [39]복제 존재 하에서 FEN1 결핍 세포에서 더 두드러졌다.이는 FEN1이 복제 의존적인 방식으로 [39]H-DNA 유도 돌연변이 유발을 억제한다는 것을 시사한다.
H-DNA는 암 [39]게놈에서 H-DNA를 형성하는 배열의 확산으로 인해 인간 암 병인과 관련이 있다.H-DNA에 의한 복제 매개 핵산가수분해효소 활성은 H-DNA 유도 돌연변이 유발을 강조하고 암 성장을 이끈다.
문자 변환
H-DNA의 염기서열 형성 또한 [38]전사를 조기에 방해하고 중단함으로써 유전적 불안정성을 야기할 수 있다.전사에 관여하는 DNA의 풀림은 손상을 받기 쉽게 만든다.전사결합수리(TCR)에서 DNA 템플릿 스트랜드상의 병변은 RNA 중합효소의 기능을 정지하고 [40]TCR 인자에 신호를 보내 손상을 제거함으로써 해결한다.H-DNA는 이러한 병변 중 하나로 인식될 수 있습니다.
안정적인 H-DNA 형성 배열 아날로그에서 T7 RNA 중합효소에 의한 전사를 관찰한 연구에서 이중-트리플렉스 접합부에서 전사 차단을 발견했다.여기서 템플릿 가닥은 H-DNA의 중심 가닥으로 왓슨-크릭 및 후그스틴 수소 결합을 파괴하기 어려웠기 때문에 전사가 [41]진행되지 않았다.
c-MYC 유전자의 P0 프로모터에서 T7에 의한 전사가 관찰되었을 때, 발견된 짧은 전사 생성물은 프로모터의 하류 H-DNA 형성 배열에 근접하여 전사가 정지되었음을 나타낸다.이 영역에 H-DNA가 형성되면 T7이 원인이 되는 입체 장애로 인해 템플릿 스트랜드를 따라 이동하는 것을 방지합니다.이것은 TCR 인자가 H-DNA를 분해하게 되는 전사와 신호를 멈추는데, 이것은 유전자 [40]불안을 야기할 수 있는 DNA 절제를 초래한다.c-MYC 유전자의 거울 대칭과 구아닌 잔기의 유병률은 비표준 DNA 구조 [42]형성에 대한 높은 경향을 제공한다.이는 전사 중 TCR 인자의 활성과 결합되어 Burkitt 림프종과 [40][42]백혈병의 발생에 역할을 하면서 높은 돌연변이 유발성을 만든다.
적용들
삼중가닥 DNA 영역은 트리플렉스 형성 올리고뉴클레오티드(TFO)와 펩타이드 핵산(PNA)의 결합을 통해 생성될 수 있다.역사적으로 TFO 결합은 DNA에 [17]대한 전사, 복제 및 단백질 결합을 억제하는 것으로 나타났다.또한 돌연변이에 얽매인 TFO는 DNA 손상을 촉진하고 돌연변이 유발을 [15]유도하는 것으로 나타났다.TFO는 DNA의 전사와 복제를 방해하는 것으로 알려져 있지만, 최근의 연구는 TFO가 체외와 [17]체내 모두에서 부위 고유의 유전자 변형을 중재하는 데 사용될 수 있다는 것을 보여주고 있다.또 다른 최근의 연구는 또한 TFO가 암세포의 성장을 감소시키기 위해 종양유전자와 원생유전자의 억제에 사용될 수 있다는 것을 보여주었다.예를 들어, 최근의 연구는 MET의 발현 감소를 통해 간종 세포의 세포사를 줄이기 위해 TFO를 사용했다.
PNA TFO는 유전자 재조합 빈도를 높일 수 있는 능력을 가지고 있어 표적화된 특정 유전자 편집으로 이어집니다.PNA-DNA-PNA 삼중나선은 세포 자체의 DNA 복구 메커니즘에 의해 인식될 수 있으며, 이는 상동 재조합을 위해 주변 DNA를 감작시킨다.부위 특이적 PNA 구조가 DNA 배열 내에서 재조합을 매개하기 위해 Bis-PNA 구조는 표적 유전자 상의 인접 영역에 [18]상동하는 40nt DNA 단편과 결합할 수 있다.기증자 DNA 가닥에 대한 TFO의 결합은 표적 유전자와 인접한 유전자 표적 [43]영역의 재조합을 유도하는 것으로 나타났다.이러한 형태의 재조합 및 복구 메커니즘은 삼중구조를 인식하고 [18][17]복구하는 역할을 하는 뉴클레오티드 절제 복구(NER) 경로와 연결되어 있다.복수의 조사에 따르면 NER인 건피색소염기 A(XPA)와 복제단백질 A(RPA)는 가교 3중 구조의 복합체로서 특이적으로 결합할 수 있다.이 메커니즘은 다른 메커니즘과 함께 트리플플렉스 구조를 인식하고 수리하는 역할을 하는 것으로 알려져 있다.
TFO의 생체 내 전달은 유전자 [44]수정을 위해 TFO를 사용하는 데 있어 주요 장벽이었다.조혈줄기세포의 생체내 표적에 관한 한 연구는 CCR5 [43]유전자의 6bp 변형을 가능하게 하기 위해 폴리(유산-코글리콜산)(PLGA) 나노입자와 함께 PNA 분자를 세포투과펩타이드(CPP)와 결합시키는 새로운 기술을 제안했다.CCR5 유전자의 편집은 HIV-1 [45]저항성과 관련이 있다.CPP는 작은 단백질이나 분자와 같은 "카고"를 세포로 성공적으로 운반할 수 있는 단백질이다.PGLA는 부위별 게놈 [43]수정을 위해 PNA 분자를 나노 입자로 캡슐화하는 생분해성 물질입니다.연구결과 PNA-DNA PGLA 나노입자는 독성이 낮고 바이러스가 없는 조혈모세포를 효과적으로 편집할 수 있었으며 CPP와의 결합은 줄기세포에서 부위 특이적 돌연변이 유발을 위한 유전자를 직접 겨냥했다.
낭포성 섬유증(CF) 유전자 치료에 대한 새로운 연구에서 기증자 DNA 분자와 함께 3개의 테일 클램프 펩타이드 핵산(PNA)이 나노입자에 의해 전달되도록 설계되어 인간 기관지 상피세포의 낭포성 섬유증 경막 전도도 조절기(CFTR)의 F508 델 돌연변이를 [46]교정했다.F508 del 돌연변이는 사람에게 [47]CF가 생기게 하는 가장 흔한 돌연변이입니다.F508 돌연변이는 순환 아데노신 일인산(cAMP)에 의해 조절되는 혈장막 염화물 채널인 CFTR의 기능 상실로 이어진다.본 연구에서, 그들은 인간 기관지 상피(HBE) 세포와 CF 마우스 모델에서 CFTR 의존성 [46]염화물의 출현을 초래한 생체 내 F508 del CFTR 돌연변이를 교정하기 위해 나노 입자를 사용하여 CF에 대한 새로운 치료 접근법을 만들 수 있었다.
역사
세 가닥의 DNA 구조는 과학자들이 DNA의 진짜 구조 형태를 발견하기 위해 고군분투하던 1950년대에 흔한 가설이었다.왓슨과 크릭(나중에 이중나선 모형으로 노벨상 수상자)은 원래 삼중나선 모형으로 여겨졌으며,[48][49] 1953년 그들의 삼중나선 모형 제안을 발표한 폴링과 코리, 그리고 동료 과학자 [50]프레이저도 마찬가지였다.그러나 Watson과 Crick은 곧 이러한 모델에서 몇 가지 문제를 발견했습니다.
- 축 근처의 음전하를 띤 인산염은 서로 밀어내고, 어떻게 3쇄 구조가 함께 유지되는지에 대한 의문을 남긴다.
- 삼중나선 모델(특히 폴링과 코리의 모델)에서는 일부 반데르발스 거리가 너무 작은 것으로 보입니다.
프레이저의 모델은 폴링과 코리의 모델과 달랐습니다. 그의 모델에서 인산염은 외부에 있고 염기는 내부에 있으며 수소 결합에 의해 서로 연결되어 있습니다.하지만 왓슨과 크릭은 프레이저의 모델이 너무 불명확해서 그것의 부적절함에 대해 구체적으로 언급할 수 없다는 것을 발견했다.
대체 3가닥 DNA 구조가 [51]1957년에 설명되었습니다.펠센펠트, 데이비스, 리치 박사는 만약 한 가닥이 푸린체만을 포함하고 다른 한 가닥이 푸린체만을 포함하고 있다면, 그 가닥은 3중 가닥 DNA 나선을 형성하기 위해 구조 변화를 겪을 것이라고 예측했다.삼중사슬 DNA(H-DNA)는 1개의 폴리퓨린과 2개의 폴리피리미딘 [7][51]가닥으로 구성될 것으로 예측되었다.이는 대장균 재조합 효소 RecA의 [51]작용 중 중간 생성물로서 체내 생물학적 과정에서만 발생하는 것으로 생각되었다.1960년대 초기 모델들은 폴리세틸산과 구아닌 올리고뉴클레오티드 사이의 복합체의 형성을 예측했다.이 모델들은 주요 [7]홈에 위치한 Hoogsten 쌍(Watson-Crick이 아닌 상호작용)으로 알려진 상호작용을 제안했습니다.얼마 후, 1개의 피리미딘과 2개의 퓨린 가닥으로 구성된 3중 나선형이 [7]예측되었다.H-DNA에서 발견된 초코일 플라스미드는 살아있는 [52]세포에서 삼중구조의 잠재적 기능에 대한 현대의 관심을 최고조에 달했습니다.또한 곧 호모피리미딘과 일부 퓨린이 풍부한 올리고뉴클레오티드가 DNA [53]이중체 상에서 호모푸린-호모피리미딘 결합 배열 특이적 구조와 함께 안정적인 H-DNA 구조를 형성할 수 있다는 것이 밝혀졌다.
레퍼런스
- ^ [Fowler, R. F.; 스키너, D. M. (1986-07-05)]"유럽 DNA는 길고 복잡한 피리미딘: 변경된 구조를 채택할 수 있는 퓨린 트랙에서 분리된다."생물 화학 저널.261(19): 8994~9001.PMID 3013872 ]
- ^ Rhee S, Han Z, Liu K, Miles HT, Davies DR (December 1999). "Structure of a triple helical DNA with a triplex-duplex junction". Biochemistry. 38 (51): 16810–5. doi:10.1021/bi991811m. PMID 10606513.
- ^ Mergny JL, Sun JS, Rougée M, Montenay-Garestier T, Barcelo F, Chomilier J, Hélène C (October 1991). "Sequence specificity in triple-helix formation: experimental and theoretical studies of the effect of mismatches on triplex stability". Biochemistry. 30 (40): 9791–8. doi:10.1021/bi00104a031. PMID 1911764.
- ^ a b Ussery DW, Sinden RR (June 1993). "Environmental influences on the in vivo level of intramolecular triplex DNA in Escherichia coli". Biochemistry. 32 (24): 6206–13. doi:10.1021/bi00075a013. PMID 8512930.
- ^ a b Dayn A, Samadashwily GM, Mirkin SM (December 1992). "Intramolecular DNA triplexes: unusual sequence requirements and influence on DNA polymerization". Proceedings of the National Academy of Sciences of the United States of America. 89 (23): 11406–10. Bibcode:1992PNAS...8911406D. doi:10.1073/pnas.89.23.11406. PMC 50559. PMID 1454828.
- ^ a b Lyamichev VI, Mirkin SM, Frank-Kamenetskii MD (February 1986). "Structures of homopurine-homopyrimidine tract in superhelical DNA". Journal of Biomolecular Structure & Dynamics. 3 (4): 667–9. doi:10.1080/07391102.1986.10508454. PMID 3271043.
- ^ a b c d e Frank-Kamenetskii MD, Mirkin SM (1995-01-01). "Triplex DNA structures". Annual Review of Biochemistry. 64: 65–95. doi:10.1146/annurev.bi.64.070195.000433. PMID 7574496. S2CID 21426188.
- ^ Brázdová M, Tichý V, Helma R, Bažantová P, Polášková A, Krejčí A, et al. (2016). "p53 Specifically Binds Triplex DNA In Vitro and in Cells". PLOS ONE. 11 (12): e0167439. Bibcode:2016PLoSO..1167439B. doi:10.1371/journal.pone.0167439. PMC 5131957. PMID 27907175.
- ^ Graham MK, Brown TR, Miller PS (April 2015). "Targeting the human androgen receptor gene with platinated triplex-forming oligonucleotides". Biochemistry. 54 (13): 2270–82. doi:10.1021/bi501565n. PMID 25768916.
- ^ Carbone GM, Napoli S, Valentini A, Cavalli F, Watson DK, Catapano CV (2004-08-03). "Triplex DNA-mediated downregulation of Ets2 expression results in growth inhibition and apoptosis in human prostate cancer cells". Nucleic Acids Research. 32 (14): 4358–67. doi:10.1093/nar/gkh744. PMC 514370. PMID 15314206.
- ^ Shen C, Rattat D, Buck A, Mehrke G, Polat B, Ribbert H, et al. (February 2003). "Targeting bcl-2 by triplex-forming oligonucleotide--a promising carrier for gene-radiotherapy". Cancer Biotherapy & Radiopharmaceuticals. 18 (1): 17–26. doi:10.1089/108497803321269296. PMID 12667305.
- ^ Sakamoto N, Chastain PD, Parniewski P, Ohshima K, Pandolfo M, Griffith JD, Wells RD (April 1999). "Sticky DNA: self-association properties of long GAA.TTC repeats in R.R.Y triplex structures from Friedreich's ataxia". Molecular Cell. 3 (4): 465–75. doi:10.1016/s1097-2765(00)80474-8. PMID 10230399.
- ^ Bacolla A, Wells RD (April 2009). "Non-B DNA conformations as determinants of mutagenesis and human disease". Molecular Carcinogenesis. 48 (4): 273–85. doi:10.1002/mc.20507. PMID 19306308. S2CID 5493647.
- ^ Kaushik Tiwari M, Adaku N, Peart N, Rogers FA (September 2016). "Triplex structures induce DNA double strand breaks via replication fork collapse in NER deficient cells". Nucleic Acids Research. 44 (16): 7742–54. doi:10.1093/nar/gkw515. PMC 5027492. PMID 27298253.
- ^ a b c d e f g h i j k l m n Jain A, Wang G, Vasquez KM (August 2008). "DNA triple helices: biological consequences and therapeutic potential". Biochimie. 90 (8): 1117–30. doi:10.1016/j.biochi.2008.02.011. PMC 2586808. PMID 18331847.
- ^ a b c d Hansen ME, Bentin T, Nielsen PE (July 2009). "High-affinity triplex targeting of double stranded DNA using chemically modified peptide nucleic acid oligomers". Nucleic Acids Research. 37 (13): 4498–507. doi:10.1093/nar/gkp437. PMC 2715256. PMID 19474349.
- ^ a b c d e f g Ricciardi AS, McNeer NA, Anandalingam KK, Saltzman WM, Glazer PM (2014). "Targeted genome modification via triple helix formation". In Wajapeyee N (ed.). Cancer Genomics and Proteomics. Methods in Molecular Biology. Vol. 1176. New York, NY: Springer New York. pp. 89–106. doi:10.1007/978-1-4939-0992-6_8. ISBN 978-1-4939-0991-9. PMC 5111905. PMID 25030921.
- ^ a b c d Rogers FA, Vasquez KM, Egholm M, Glazer PM (December 2002). "Site-directed recombination via bifunctional PNA-DNA conjugates". Proceedings of the National Academy of Sciences of the United States of America. 99 (26): 16695–700. Bibcode:2002PNAS...9916695R. doi:10.1073/pnas.262556899. PMC 139206. PMID 12461167.
- ^ Montazersaheb S, Hejazi MS, Nozad Charoudeh H (November 2018). "Potential of Peptide Nucleic Acids in Future Therapeutic Applications". Advanced Pharmaceutical Bulletin. 8 (4): 551–563. doi:10.15171/apb.2018.064. PMC 6311635. PMID 30607328.
- ^ Firulli AB, Maibenco DC, Kinniburgh AJ (April 1994). "Triplex forming ability of a c-myc promoter element predicts promoter strength". Archives of Biochemistry and Biophysics. 310 (1): 236–42. doi:10.1006/abbi.1994.1162. PMID 8161210.
- ^ Zain R, Sun JS (May 2003). "Do natural DNA triple-helical structures occur and function in vivo?". Cellular and Molecular Life Sciences. 60 (5): 862–70. doi:10.1007/s00018-003-3046-3. PMID 12827276. S2CID 6227195.
- ^ Kato M, Shimizu N (October 1992). "Effect of the potential triplex DNA region on the in vitro expression of bacterial beta-lactamase gene in superhelical recombinant plasmids". Journal of Biochemistry. 112 (4): 492–4. doi:10.1093/oxfordjournals.jbchem.a123927. PMID 1491004.
- ^ Potaman VN, Ussery DW, Sinden RR (June 1996). "Formation of a combined H-DNA/open TATA box structure in the promoter sequence of the human Na,K-ATPase alpha2 gene". The Journal of Biological Chemistry. 271 (23): 13441–7. doi:10.1074/jbc.271.23.13441. PMID 8662935. S2CID 46550975.
- ^ Seidman MM, Glazer PM (August 2003). "The potential for gene repair via triple helix formation". The Journal of Clinical Investigation. 112 (4): 487–94. doi:10.1172/JCI19552. PMC 171401. PMID 12925687.
- ^ Rapozzi V, Cogoi S, Spessotto P, Risso A, Bonora GM, Quadrifoglio F, Xodo LE (January 2002). "Antigene effect in K562 cells of a PEG-conjugated triplex-forming oligonucleotide targeted to the bcr/abl oncogene". Biochemistry. 41 (2): 502–10. doi:10.1021/bi011314h. PMID 11781088.
- ^ Bertucat G, Lavery R, Prévost C (December 1998). "A model for parallel triple helix formation by RecA: single-single association with a homologous duplex via the minor groove". Journal of Biomolecular Structure & Dynamics. 16 (3): 535–46. doi:10.1080/07391102.1998.10508268. PMID 10052612.
- ^ Chen J, Tang Q, Guo S, Lu C, Le S, Yan J (September 2017). "Parallel triplex structure formed between stretched single-stranded DNA and homologous duplex DNA". Nucleic Acids Research. 45 (17): 10032–10041. doi:10.1093/nar/gkx628. PMC 5622322. PMID 28973442.
- ^ Camerini-Otero RD, Hsieh P (April 1993). "Parallel DNA triplexes, homologous recombination, and other homology-dependent DNA interactions". Cell. 73 (2): 217–23. doi:10.1016/0092-8674(93)90224-e. PMID 8477443. S2CID 34585948.
- ^ Bertucat G, Lavery R, Prévost C (September 1999). "A molecular model for RecA-promoted strand exchange via parallel triple-stranded helices". Biophysical Journal. 77 (3): 1562–76. Bibcode:1999BpJ....77.1562B. doi:10.1016/S0006-3495(99)77004-9. PMC 1300444. PMID 10465767.
- ^ Yang H, Zhou C, Dhar A, Pavletich NP (October 2020). "Mechanism of strand exchange from RecA-DNA synaptic and D-loop structures". Nature. 586 (7831): 801–806. Bibcode:2020Natur.586..801Y. doi:10.1038/s41586-020-2820-9. PMC 8366275. PMID 33057191. S2CID 222349650.
- ^ McKinney JA, Wang G, Mukherjee A, Christensen L, Subramanian SH, Zhao J, Vasquez KM (January 2020). "Distinct DNA repair pathways cause genomic instability at alternative DNA structures". Nature Communications. 11 (1): 236. Bibcode:2020NatCo..11..236M. doi:10.1038/s41467-019-13878-9. PMC 6957503. PMID 31932649.
- ^ a b Wang G, Vasquez KM (July 2014). "Impact of alternative DNA structures on DNA damage, DNA repair, and genetic instability". DNA Repair. 19: 143–51. doi:10.1016/j.dnarep.2014.03.017. PMC 4216180. PMID 24767258.
- ^ Raghavan SC, Chastain P, Lee JS, Hegde BG, Houston S, Langen R, et al. (June 2005). "Evidence for a triplex DNA conformation at the bcl-2 major breakpoint region of the t(14;18) translocation". The Journal of Biological Chemistry. 280 (24): 22749–60. doi:10.1074/jbc.M502952200. PMID 15840562.
- ^ Wang G, Vasquez KM (September 2004). "Naturally occurring H-DNA-forming sequences are mutagenic in mammalian cells". Proceedings of the National Academy of Sciences of the United States of America. 101 (37): 13448–53. Bibcode:2004PNAS..10113448W. doi:10.1073/pnas.0405116101. PMC 518777. PMID 15342911.
- ^ Vetcher AA, Napierala M, Iyer RR, Chastain PD, Griffith JD, Wells RD (October 2002). "Sticky DNA, a long GAA.GAA.TTC triplex that is formed intramolecularly, in the sequence of intron 1 of the frataxin gene". The Journal of Biological Chemistry. 277 (42): 39217–27. doi:10.1074/jbc.M205209200. PMID 12161437.
- ^ last1 = Fowler first1 = R. F. last2 = Skinner first2 = D. M. title = 진핵 DNA는 길고 복잡한 피리미딘: 변경된 배치 저널을 채택할 수 있는 퓨린로 = Journal of Biological Chemistry date = 1986-07-05 pmid = 301372}
- ^ {{분개저널 볼륨 = 38호 = 1~3페이지 = 145~145호
- ^ a b c d e f g Wang G, Vasquez KM (January 2017). "Effects of Replication and Transcription on DNA Structure-Related Genetic Instability". Genes. 8 (1): 17. doi:10.3390/genes8010017. PMC 5295012. PMID 28067787.
- ^ a b c d e f g h i j Zhao J, Wang G, Del Mundo IM, McKinney JA, Lu X, Bacolla A, et al. (January 2018). "Distinct Mechanisms of Nuclease-Directed DNA-Structure-Induced Genetic Instability in Cancer Genomes". Cell Reports. 22 (5): 1200–1210. doi:10.1016/j.celrep.2018.01.014. PMC 6011834. PMID 29386108.
- ^ a b c Belotserkovskii BP, De Silva E, Tornaletti S, Wang G, Vasquez KM, Hanawalt PC (November 2007). "A triplex-forming sequence from the human c-MYC promoter interferes with DNA transcription". The Journal of Biological Chemistry. 282 (44): 32433–41. doi:10.1074/jbc.M704618200. PMID 17785457. S2CID 24211097.
- ^ Pandey S, Ogloblina AM, Belotserkovskii BP, Dolinnaya NG, Yakubovskaya MG, Mirkin SM, Hanawalt PC (August 2015). "Transcription blockage by stable H-DNA analogs in vitro". Nucleic Acids Research. 43 (14): 6994–7004. doi:10.1093/nar/gkv622. PMC 4538819. PMID 26101261.
- ^ a b Del Mundo IM, Zewail-Foote M, Kerwin SM, Vasquez KM (May 2017). "Alternative DNA structure formation in the mutagenic human c-MYC promoter". Nucleic Acids Research. 45 (8): 4929–4943. doi:10.1093/nar/gkx100. PMC 5416782. PMID 28334873.
- ^ a b c McNeer NA, Schleifman EB, Cuthbert A, Brehm M, Jackson A, Cheng C, et al. (June 2013). "Systemic delivery of triplex-forming PNA and donor DNA by nanoparticles mediates site-specific genome editing of human hematopoietic cells in vivo". Gene Therapy. 20 (6): 658–69. doi:10.1038/gt.2012.82. PMC 3713483. PMID 23076379.
- ^ Hnedzko D, Cheruiyot SK, Rozners E (September 2014). "Using triple-helix-forming Peptide nucleic acids for sequence-selective recognition of double-stranded RNA". Current Protocols in Nucleic Acid Chemistry. 58: 4.60.1–23. doi:10.1002/0471142700.nc0460s58. ISSN 1934-9270. PMC 4174339. PMID 25199637.
- ^ Schleifman EB, Bindra R, Leif J, del Campo J, Rogers FA, Uchil P, et al. (September 2011). "Targeted disruption of the CCR5 gene in human hematopoietic stem cells stimulated by peptide nucleic acids". Chemistry & Biology. 18 (9): 1189–98. doi:10.1016/j.chembiol.2011.07.010. PMC 3183429. PMID 21944757.
- ^ a b McNeer NA, Anandalingam K, Fields RJ, Caputo C, Kopic S, Gupta A, et al. (April 2015). "Nanoparticles that deliver triplex-forming peptide nucleic acid molecules correct F508del CFTR in airway epithelium". Nature Communications. 6 (1): 6952. Bibcode:2015NatCo...6.6952M. doi:10.1038/ncomms7952. PMC 4480796. PMID 25914116.
- ^ Lukacs GL, Verkman AS (February 2012). "CFTR: folding, misfolding and correcting the ΔF508 conformational defect". Trends in Molecular Medicine. 18 (2): 81–91. doi:10.1016/j.molmed.2011.10.003. PMC 3643519. PMID 22138491.
- ^ Pauling L, Corey RB (February 1953). "A Proposed Structure For The Nucleic Acids". Proceedings of the National Academy of Sciences of the United States of America. 39 (2): 84–97. Bibcode:1953PNAS...39...84P. doi:10.1073/pnas.39.2.84. PMC 1063734. PMID 16578429.
- ^ Pauling L, Corey RB (February 1953). "Structure of the nucleic acids". Nature. 171 (4347): 346. Bibcode:1953Natur.171..346P. doi:10.1038/171346a0. PMID 13036888. S2CID 4151877.
- ^ Fraser RD (March 2004). "The structure of deoxyribose nucleic acid". Journal of Structural Biology. 145 (3): 184–5. doi:10.1016/j.jsb.2004.01.001. PMID 14997898.
- ^ a b c Felsenfeld G, Davies DR, Rich A (April 1957). "Formation of a three-stranded polynucleotide molecule". Journal of the American Chemical Society. 79 (8): 2023–4. doi:10.1021/ja01565a074.
- ^ Hanvey JC, Shimizu M, Wells RD (September 1988). "Intramolecular DNA triplexes in supercoiled plasmids". Proceedings of the National Academy of Sciences of the United States of America. 85 (17): 6292–6. Bibcode:1988PNAS...85.6292H. doi:10.1073/pnas.85.17.6292. PMC 281955. PMID 3413097.
- ^ Mirkin SM, Lyamichev VI, Drushlyak KN, Dobrynin VN, Filippov SA, Frank-Kamenetskii MD (December 1987). "DNA H form requires a homopurine-homopyrimidine mirror repeat". Nature. 330 (6147): 495–7. Bibcode:1987Natur.330..495M. doi:10.1038/330495a0. PMID 2825028. S2CID 4360764.
추가 정보
- Rich A (December 1993). "DNA comes in many forms". Gene. 135 (1–2): 99–109. doi:10.1016/0378-1119(93)90054-7. PMID 8276285.
- Soyfer VN, Potaman VN (1995). Triple-Helical Nucleic Acids. New York: Springer. ISBN 978-0-387-94495-1.
- Mills M, Arimondo PB, Lacroix L, Garestier T, Hélène C, Klump H, Mergny JL (September 1999). "Energetics of strand-displacement reactions in triple helices: a spectroscopic study". Journal of Molecular Biology. 291 (5): 1035–54. doi:10.1006/jmbi.1999.3014. PMID 10518941.
- Watson JD, Crick FH (April 1953). "Molecular structure of nucleic acids; a structure for deoxyribose nucleic acid". Nature. 171 (4356): 737–8. Bibcode:1953Natur.171..737W. doi:10.1038/171737a0. PMID 13054692. S2CID 4253007.