N-메틸아데노신6
N6-Methyladenosine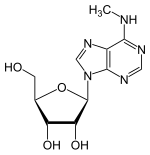 | |
| 이름 | |
|---|---|
| IUPAC 이름 N-메틸아데노신 | |
| 선호 IUPAC 이름 (2R,3S,4R,5R)-2-(히드록시메틸)-5-[6-(메틸아미노)-9H-푸린-9-yl]옥솔레인-2,3-다이올 | |
| 기타 이름 m6A | |
| 식별자 | |
3D 모델(JSmol) | |
| 체비 | |
| 켐스파이더 | |
펍켐 CID | |
| 유니 | |
CompTox 대시보드 (EPA) | |
| |
| |
| 특성. | |
| C11H15N5O4 | |
| 어금질량 | 281.272 g·190−1 |
달리 명시된 경우를 제외하고, 표준 상태(25°C [77°F], 100 kPa)의 재료에 대한 데이터가 제공된다. | |
| Infobox 참조 자료 | |
N-메틸아데노신6(mA6)은 원래 1970년대에 식별되어 부분적으로 특징지어졌으며,[1][2][3][4] mRNA와 DNA의 풍부한 변형이다.[5]그것은 일부 바이러스에서 발견되며 [4][3]포유류,[2][1][6][7] 곤충,[8] 식물[9][10][11], 효모를 포함한 대부분의 진핵생물에서 발견된다.[12][13]또한 Xist와 같은 몇 개의 긴 비코딩 RNA뿐만 아니라 tRNA, rRNA, snRNA에서도 발견된다.[14][15]
아데노신의 메틸화는 METTL3를 SAM-binding 서브유닛으로 함유한 대형6 mA 메틸트랜스퍼레이제 콤플렉스에 의해 지시된다.[16]시험관으로, 이 메틸기 전달 효소 복잡한 우선적으로 RNA올리고 핵산염 GGACU[17]고 비슷한 선호도 포함하는 vivo에 라우스 육종 바이러스 게놈 RNA[18]에 매핑 된 m6A 사이트와 소 프롤락틴에서 파악된 mRNA.[19]최근 연구 결과에 따르m의m6A 메틸기 전달 효소 단지의 다른 핵심 부품 생산해 와methylatesammals, inclu딩 METTL14,[20][21] Wilms 종양 1 관련 단백질(WTAP),[20][22] KIAA1429 및 METTL5.[24]2010년 mRNA에서 mA가6 동적이고 가역적이라는 추측에 이어 2011년[26] 첫 mA6 데메틸아제, 지방질량, 비만 관련 단백질(FTO)이 발견되면서 이 가설을 확인시켜 mA6 연구에 대한 관심이 활성화됐다.[25]두 번째 mA6 데메틸아제 알크B 호몰로지 5(ALKBH5)도 나중에 발견되었다.[27]
mA의6 생물학적 기능은 RNA의 메틸화 아데노신을 구체적으로 인식하는 RNA 결합 단백질 그룹을 통해 매개된다.이 결합 단백질은 mA6 리더라고 불린다.YT521-B 호몰로지(YTH) 단백질 영역 제품군(YTDF1, YTHDF2, YTHDF3, YTHDC1)은 직접 mA6 판독기로 특징지어졌으며, 보존된 mA-binding6 포켓을 가지고 있다.[15][28][29][30][31]인슐린과 같은 성장 인자-2 mRNA 결합 단백질 1, 2, 3(IGF2BP1–3)은 m6A 독자의 새로운 등급으로 보고된다.[32]IGF2BP는 K 호몰로지(KH) 도메인을 사용하여 m6A 함유 RNA를 선택적으로 인식하고 이들의 번역과 안정성을 촉진한다.[32]이러한 mA6 리더는 mA6 메틸트랜스퍼레이즈(작성자) 및 데메틸아제(지우기자)와 함께 RNA에 대한6 mA의 분포를 결정하는 mA6 규제의 복잡한 메커니즘을 확립하는 한편, 독자는 mA에6 의존하는 기능을 조정한다.mA는6 mA6 스위치라는 구조적 스위치도 중재하는 것으로 나타났다.[33]
종 분포
효모
출아 효모(Sacharomyces 세레비시아)에서, METTL3, IME4의 동족체 질소와 발효성의 탄소원 기아에 대응하여 이배체 세포에만 있으며 mRNA메틸화고 정확한 감수 분열과 포자 형성의 개시에 필요합니다 유도된다.IME1과 IME2의[12][13]mRNAs, 감수 분열의 주요 초 규제 당국, 필로폰 환각 상태에 공격 목표가 된다고 알려져 있다.Ilation, IME4 자체의 성적증명서와 같이.[13]
식물
식물에서, mA의6 대부분은 폴리(A) 꼬리가 시작되기 전에 150개의 뉴클레오티드 내에서 발견된다.[34]
MTA의 돌연변이, METTL3의 아라비도피스 탈리아나 호몰로뉴는 구상 단계에서 배아를 체포하게 된다.성숙한 식물에서 mA6 수치가 90% 이상 감소하면 성장 패턴이 급격히 변화하고 꽃무늬가 있는 가정형편이 이상해진다.[34]
포유류
인간과 생쥐 RNA에서의 mA6 매핑은 [G/A/U][G]m의6 일치된 순서를 가진 7,000개 이상의 인간 유전자의 대본에서 18,000개 이상의 mA 사이트를 확인했다6.AC[U]A/C][14][15][35]는 이전에 식별된 모티브와 일치한다.많은 mRNAs에서 개별 m6A 사이트의 국산화 고도로 mouse,[14][15]과transcriptome-wide 인간의 분석 사이에 비슷한 것은 m6A 높은 진화의 보존 및 지역에서 발견되는 것을 나타낸다.[14]m6A 긴 내부 구조 부위 안에 우선적으로 3'UTRs, 정지 codons에 안에 첨가되어 있다. 3'UTRs 또한 관련된 기지 내에 m6A 발견된다.h 마이크로RNA 바인딩 사이트의 존재; 3' UTR 내에 mA6 사이트를 포함하는 mRNA의 약 2/3은 최소 1개의 마이크로RNA 바인딩 사이트를 가지고 있다.[14]모든 mA6 염기서열 데이터를 통합함으로써 RMBase라는 새로운 데이터베이스가 RNA의 N6-메틸아데노신(mA6)에 해당하는 인간과 마우스 게놈에서 약 20만 개의 부지를 식별해 제공했다.[35]
m6A-CLIP/IP(간편 m6A-CLIP)에 의한 정확한 m6A 매핑을 통해 대부분의 m6A가 생쥐와 인간의 여러 조직/배양 세포에서 mRNA의 마지막 exon에 위치하고 있으며,[36] 정지 코돈 주변의 m6A 농축은 m6A가 진정으로 농축된 마지막 exon의 시작 부분에 많은 정지 코돈들이 위치한 우연의 일치라고 밝혔다.[36]last exon(>=70%)에 m6A의 주요 존재는 3'의 가능성을 허용한다.대체 폴리아데닐화를 포함한 UTR 규정.[36]m6A-CLIP와 엄격한 세포분열 생화학이 결합된 연구는 m6A mRNA 수정은 초기 mRNA에 축적되며 스플라이싱에 필요하지 않지만 세포질 전환은 명시해야 한다는 것을 보여준다.[37][38]
mA는6 발달 과정 전반과 세포 자극에 반응하여 동적 규제에 취약하다.생쥐 뇌 RNA에서 mA를6 분석한 결과 배아 발달 시 mA6 수치가 낮고 성인기에 따라 급격히 증가하는 것으로 나타났다.[14]또한 mA6 메틸전달효소를 침묵시키면 유전자 발현과 대체 RNA 스플라이싱 패턴에 유의하게 영향을 미쳐 p53(TP53) 신호 경로 및 사멸의 변조를 초래한다.[15]
mA는6 또한 RNA의 안정성 조절에 관여하는 인간 세포의 R-루프의 RNA 성분에서도 발견된다.DNA 하이브리드.[39]
생리적 과정에 대한 mA6 메틸화의 중요성이 최근에 입증되었다.세포 메틸화의 약리학적 억제를 통한 mA6 메틸화 억제 또는 mA6 메틸화효소 메틸화효소 메틸화3의 siRNA 매개 음소거에 의한 mA 메틸화 억제는 순환기간의 연장으로 이어졌다.이와는 대조적으로, 메틀3의 과도한 압박은 더 짧은 기간으로 이어졌다.약 24시간 동안 진동하도록 엄격하게 조절된 전사 피드백 루프로 구성된 포유류 순환 시계는 따라서 클럭 유전자 대본 내에 mA6 부위가 존재하기 때문에 mA 의존적6 RNA 처리의 섭동에 매우 민감하다.[40][41]전지구적 메틸화 억제가 쥐세포에서 순환기에 미치는 영향은 박테리아 메틸대사의 효소의 엑토픽 발현으로 막을 수 있다.이 박테리아성 단백질을 발현하는 생쥐 세포는 메틸 대사의 약리학적 억제에 내성이 있어 mRNA mA6 메틸화나 단백질 메틸화의 감소를 보이지 않았다.[42]
임상적 유의성
따라서 다양한 생리학적 과정에서 mA의6 다용도 기능을 고려할 때 mA와6 수많은 인간 질병 사이의 연관성을 찾는 것은 놀라운 일이 아니다. mA의6 동일인자의 돌연변이 또는 단일 뉴클레오티드 다형성(SNPs)에서 유래된 것이 많다.mA와6 수많은 암 유형의 연관성은 위암, 전립선암, 유방암, 췌장암, 신장암, 중피종, 육종, 백혈병을 포함하는 보고서에 나타나 있다.[43][44][45][46][47][48][49][50][51][52][53][54]mA가6 암세포 증식에 미치는 영향은 더 많은 데이터가 등장함에 따라 훨씬 더 심오할 수 있다.METTL3의 고갈과는 동안 ALKBH5 저산소증으로의 활성화 줄기 세포 농축 암을 유발하는 것으로 나타났다 암 cells,[55][56]의 침입성을 줄이암 세포의 세포를 유발한다고 알려져 있다.에너지 신진대사와 비만으로 해외 훈련 장교는 키 조절 유전자[57]m6A은 에너지 항상성과 비만의 규정에, 나타내고 있다.FTO의 SNPs는 인간의 체질량지수와 비만과 당뇨병의 발생과 관련이 있는 것으로 나타났다.[58][59][60][61][62]FTO가 아디포시 전 분화에 미치는 영향이 제시됐다.[63][64][65]mA와6 신경장애의 연관성도 연구됐다.예를 들어, 신경퇴행성 질환은 FTO에 의존하고 키 신호 대본에서 정확한 mA6 메틸레이션에 의존하는 것으로 보여져6 mA에 의해 영향을 받을 수 있다.[66]mA의6 잠재적 독자인 HNRNPA2B1의 돌연변이는 신경분열을 일으키는 것으로 알려져 왔다.[67]mA6 리더의 새로운 클래스인 IGF2BP1–3은 인공적인 기능을 가지고 있다.IGF2BP1–3 녹다운 또는 녹아웃은 인간 암세포 라인에서 MYC 단백질 표현, 세포 증식 및 군집 형성을 감소시켰다.[32]m6A 메틸전달효소 복합체의 일원인 ZC3H13은 쓰러졌을 때 대장암 세포의 성장을 현저하게 억제했다.[68]
게다가, mA는6 바이러스 감염에 영향을 미치는 것으로 보고되었다.SimianVirus40시 미안 바이러스 40, 아데노 바이러스, herpes 바이러스, 라우스 육종 바이러스, 인플루엔자 바이러스를 포함한 많은 RNA바이러스 바이러스 게놈 RNA.[69]이 넘어서더 최근의 연구에 내부m6A 메틸화 반응을 포함한다고 알려져 해 온m6A 감독 당국 감염의 효율성과 같은 RNA바이러스의 복제 지배하는 인체 면역 결핍 바이러스(HIV), h. 밝혔다epatitis C 바이러스(HCV), 지카 바이러스([70][71][72][73][74]ZIKV).이러한 결과는 mA와6 그 인지적 요인이 바이러스 수명주기 및 호스트-바이러스 상호작용을 규제하는 데 중요한 역할을 한다는 것을 시사한다.
참조
- ^ a b Adams JM, Cory S (May 1975). "Modified nucleosides and bizarre 5'-termini in mouse myeloma mRNA". Nature. 255 (5503): 28–33. Bibcode:1975Natur.255...28A. doi:10.1038/255028a0. PMID 1128665. S2CID 4199864.
- ^ a b Desrosiers R, Friderici K, Rottman F (October 1974). "Identification of methylated nucleosides in messenger RNA from Novikoff hepatoma cells". Proceedings of the National Academy of Sciences of the United States of America. 71 (10): 3971–5. Bibcode:1974PNAS...71.3971D. doi:10.1073/pnas.71.10.3971. PMC 434308. PMID 4372599.
- ^ a b Aloni Y, Dhar R, Khoury G (October 1979). "Methylation of nuclear simian virus 40 RNAs". Journal of Virology. 32 (1): 52–60. doi:10.1128/JVI.32.1.52-60.1979. PMC 353526. PMID 232187.
- ^ a b Beemon K, Keith J (June 1977). "Localization of N6-methyladenosine in the Rous sarcoma virus genome". Journal of Molecular Biology. 113 (1): 165–79. doi:10.1016/0022-2836(77)90047-X. PMID 196091.
- ^ Ji, Pengfei; Wang, Xia; Xie, Nina; Li, Yujing (2018). "N6-Methyladenosine in RNA and DNA: An Epitranscriptomic and Epigenetic Player Implicated in Determination of Stem Cell Fate". Stem Cells International. 2018: 3256524. doi:10.1155/2018/3256524. PMC 6199872. PMID 30405719.
- ^ Wei CM, Gershowitz A, Moss B (January 1976). "5'-Terminal and internal methylated nucleotide sequences in HeLa cell mRNA". Biochemistry. 15 (2): 397–401. doi:10.1021/bi00647a024. PMID 174715.
- ^ Perry RP, Kelley DE, Friderici K, Rottman F (April 1975). "The methylated constituents of L cell messenger RNA: evidence for an unusual cluster at the 5' terminus". Cell. 4 (4): 387–94. doi:10.1016/0092-8674(75)90159-2. PMID 1168101.
- ^ Levis R, Penman S (April 1978). "5'-terminal structures of poly(A)+ cytoplasmic messenger RNA and of poly(A)+ and poly(A)- heterogeneous nuclear RNA of cells of the dipteran Drosophila melanogaster". Journal of Molecular Biology. 120 (4): 487–515. doi:10.1016/0022-2836(78)90350-9. PMID 418182.
- ^ Nichols JL (1979). "In maize poly(A)-containing RNA". Plant Science Letters. 15 (4): 357–361. doi:10.1016/0304-4211(79)90141-X.
- ^ Kennedy TD, Lane BG (June 1979). "Wheat embryo ribonucleates. XIII. Methyl-substituted nucleoside constituents and 5'-terminal dinucleotide sequences in bulk poly(AR)-rich RNA from imbibing wheat embryos". Canadian Journal of Biochemistry. 57 (6): 927–31. doi:10.1139/o79-112. PMID 476526.
- ^ Zhong S, Li H, Bodi Z, Button J, Vespa L, Herzog M, Fray RG (May 2008). "MTA is an Arabidopsis messenger RNA adenosine methylase and interacts with a homolog of a sex-specific splicing factor". The Plant Cell. 20 (5): 1278–88. doi:10.1105/tpc.108.058883. PMC 2438467. PMID 18505803.
- ^ a b Clancy MJ, Shambaugh ME, Timpte CS, Bokar JA (October 2002). "Induction of sporulation in Saccharomyces cerevisiae leads to the formation of N6-methyladenosine in mRNA: a potential mechanism for the activity of the IME4 gene". Nucleic Acids Research. 30 (20): 4509–18. doi:10.1093/nar/gkf573. PMC 137137. PMID 12384598.
- ^ a b c Bodi Z, Button JD, Grierson D, Fray RG (September 2010). "Yeast targets for mRNA methylation". Nucleic Acids Research. 38 (16): 5327–35. doi:10.1093/nar/gkq266. PMC 2938207. PMID 20421205.
- ^ a b c d e f Meyer KD, Saletore Y, Zumbo P, Elemento O, Mason CE, Jaffrey SR (June 2012). "Comprehensive analysis of mRNA methylation reveals enrichment in 3' UTRs and near stop codons". Cell. 149 (7): 1635–46. doi:10.1016/j.cell.2012.05.003. PMC 3383396. PMID 22608085.
- ^ a b c d e Dominissini D, Moshitch-Moshkovitz S, Schwartz S, Salmon-Divon M, Ungar L, Osenberg S, Cesarkas K, Jacob-Hirsch J, Amariglio N, Kupiec M, Sorek R, Rechavi G (April 2012). "Topology of the human and mouse m6A RNA methylomes revealed by m6A-seq". Nature. 485 (7397): 201–6. Bibcode:2012Natur.485..201D. doi:10.1038/nature11112. PMID 22575960. S2CID 3517716.
- ^ Bokar JA, Shambaugh ME, Polayes D, Matera AG, Rottman FM (November 1997). "Purification and cDNA cloning of the AdoMet-binding subunit of the human mRNA (N6-adenosine)-methyltransferase". RNA. 3 (11): 1233–47. PMC 1369564. PMID 9409616.
- ^ Harper JE, Miceli SM, Roberts RJ, Manley JL (October 1990). "Sequence specificity of the human mRNA N6-adenosine methylase in vitro". Nucleic Acids Research. 18 (19): 5735–41. doi:10.1093/nar/18.19.5735. PMC 332308. PMID 2216767.
- ^ Kane SE, Beemon K (September 1985). "Precise localization of m6A in Rous sarcoma virus RNA reveals clustering of methylation sites: implications for RNA processing". Molecular and Cellular Biology. 5 (9): 2298–306. doi:10.1128/mcb.5.9.2298. PMC 366956. PMID 3016525.
- ^ Horowitz S, Horowitz A, Nilsen TW, Munns TW, Rottman FM (September 1984). "Mapping of N6-methyladenosine residues in bovine prolactin mRNA". Proceedings of the National Academy of Sciences of the United States of America. 81 (18): 5667–71. Bibcode:1984PNAS...81.5667H. doi:10.1073/pnas.81.18.5667. PMC 391771. PMID 6592581.
- ^ a b Liu J, Yue Y, Han D, Wang X, Fu Y, Zhang L, Jia G, Yu M, Lu Z, Deng X, Dai Q, Chen W, He C (February 2014). "A METTL3-METTL14 complex mediates mammalian nuclear RNA N6-adenosine methylation". Nature Chemical Biology. 10 (2): 93–5. doi:10.1038/nchembio.1432. PMC 3911877. PMID 24316715.
- ^ Wang Y, Li Y, Toth JI, Petroski MD, Zhang Z, Zhao JC (February 2014). "N6-methyladenosine modification destabilizes developmental regulators in embryonic stem cells". Nature Cell Biology. 16 (2): 191–8. doi:10.1038/ncb2902. PMC 4640932. PMID 24394384.
- ^ Ping XL, Sun BF, Wang L, Xiao W, Yang X, Wang WJ, Adhikari S, Shi Y, Lv Y, Chen YS, Zhao X, Li A, Yang Y, Dahal U, Lou XM, Liu X, Huang J, Yuan WP, Zhu XF, Cheng T, Zhao YL, Wang X, Rendtlew Danielsen JM, Liu F, Yang YG (February 2014). "Mammalian WTAP is a regulatory subunit of the RNA N6-methyladenosine methyltransferase". Cell Research. 24 (2): 177–89. doi:10.1038/cr.2014.3. PMC 3915904. PMID 24407421.
- ^ Schwartz S, Mumbach MR, Jovanovic M, Wang T, Maciag K, Bushkin GG, Mertins P, Ter-Ovanesyan D, Habib N, Cacchiarelli D, Sanjana NE, Freinkman E, Pacold ME, Satija R, Mikkelsen TS, Hacohen N, Zhang F, Carr SA, Lander ES, Regev A (July 2014). "Perturbation of m6A writers reveals two distinct classes of mRNA methylation at internal and 5' sites". Cell Reports. 8 (1): 284–96. doi:10.1016/j.celrep.2014.05.048. PMC 4142486. PMID 24981863.
- ^ Lafontaine, Denis L. J.; Graille, Marc; Jaffrey, Samie R.; Bohnsack, Markus T.; Bohnsack, Katherine E.; Hackert, Philipp; Ulryck, Nathalie; Zorbas, Christiane; Hawley, Ben R. (2019). "The human 18S rRNA m6A methyltransferase METTL5 is stabilized by TRMT112". Nucleic Acids Research. 47 (15): 7719–7733. doi:10.1093/nar/gkz619. PMC 6735865. PMID 31328227.
- ^ He C (December 2010). "Grand challenge commentary: RNA epigenetics?". Nature Chemical Biology. 6 (12): 863–5. doi:10.1038/nchembio.482. PMID 21079590.
- ^ Jia G, Fu Y, Zhao X, Dai Q, Zheng G, Yang Y, Yi C, Lindahl T, Pan T, Yang YG, He C (October 2011). "N6-methyladenosine in nuclear RNA is a major substrate of the obesity-associated FTO". Nature Chemical Biology. 7 (12): 885–7. doi:10.1038/nchembio.687. PMC 3218240. PMID 22002720.
- ^ Zheng G, Dahl JA, Niu Y, Fedorcsak P, Huang CM, Li CJ, Vågbø CB, Shi Y, Wang WL, Song SH, Lu Z, Bosmans RP, Dai Q, Hao YJ, Yang X, Zhao WM, Tong WM, Wang XJ, Bogdan F, Furu K, Fu Y, Jia G, Zhao X, Liu J, Krokan HE, Klungland A, Yang YG, He C (January 2013). "ALKBH5 is a mammalian RNA demethylase that impacts RNA metabolism and mouse fertility". Molecular Cell. 49 (1): 18–29. doi:10.1016/j.molcel.2012.10.015. PMC 3646334. PMID 23177736.
- ^ Wang X, Lu Z, Gomez A, Hon GC, Yue Y, Han D, Fu Y, Parisien M, Dai Q, Jia G, Ren B, Pan T, He C (January 2014). "N6-methyladenosine-dependent regulation of messenger RNA stability". Nature. 505 (7481): 117–20. Bibcode:2014Natur.505..117W. doi:10.1038/nature12730. PMC 3877715. PMID 24284625.
- ^ Wang X, Zhao BS, Roundtree IA, Lu Z, Han D, Ma H, Weng X, Chen K, Shi H, He C (June 2015). "N(6)-methyladenosine Modulates Messenger RNA Translation Efficiency". Cell. 161 (6): 1388–99. doi:10.1016/j.cell.2015.05.014. PMC 4825696. PMID 26046440.
- ^ Xu C, Wang X, Liu K, Roundtree IA, Tempel W, Li Y, Lu Z, He C, Min J (November 2014). "Structural basis for selective binding of m6A RNA by the YTHDC1 YTH domain". Nature Chemical Biology. 10 (11): 927–9. doi:10.1038/nchembio.1654. PMID 25242552.
- ^ Xiao W, Adhikari S, Dahal U, Chen YS, Hao YJ, Sun BF, et al. (February 2016). "Nuclear m(6)A Reader YTHDC1 Regulates mRNA Splicing". Molecular Cell. 61 (4): 507–519. doi:10.1016/j.molcel.2016.01.012. PMID 26876937.
- ^ a b c Huang H, Weng H, Sun W, Qin X, Shi H, Wu H, et al. (March 2018). "6-methyladenosine by IGF2BP proteins enhances mRNA stability and translation". Nature Cell Biology. 20 (3): 285–295. doi:10.1038/s41556-018-0045-z. PMC 5826585. PMID 29476152.
- ^ Liu N, Dai Q, Zheng G, He C, Parisien M, Pan T (February 2015). "N(6)-methyladenosine-dependent RNA structural switches regulate RNA-protein interactions". Nature. 518 (7540): 560–4. Bibcode:2015Natur.518..560L. doi:10.1038/nature14234. PMC 4355918. PMID 25719671.
- ^ a b Bodi Z, Zhong S, Mehra S, Song J, Graham N, Li H, May S, Fray RG (2012). "Adenosine Methylation in Arabidopsis mRNA is Associated with the 3' End and Reduced Levels Cause Developmental Defects". Frontiers in Plant Science. 3: 48. doi:10.3389/fpls.2012.00048. PMC 3355605. PMID 22639649.
- ^ a b Sun WJ, Li JH, Liu S, Wu J, Zhou H, Qu LH, Yang JH (January 2016). "RMBase: a resource for decoding the landscape of RNA modifications from high-throughput sequencing data". Nucleic Acids Research. 44 (D1): D259–65. doi:10.1093/nar/gkv1036. PMC 4702777. PMID 26464443.
- ^ a b c d Ke S, Alemu EA, Mertens C, Gantman EC, Fak JJ, Mele A, Haripal B, Zucker-Scharff I, Moore MJ, Park CY, Vågbø CB, Kusśnierczyk A, Klungland A, Darnell JE, Darnell RB (October 2015). "A majority of m6A residues are in the last exons, allowing the potential for 3' UTR regulation". Genes & Development. 29 (19): 2037–53. doi:10.1101/gad.269415.115. PMC 4604345. PMID 26404942.
- ^ Ke S, Pandya-Jones A, Saito Y, Fak JJ, Vågbø CB, Geula S, Hanna JH, Black DL, Darnell JE, Darnell RB (May 2017). "6A mRNA modifications are deposited in nascent pre-mRNA and are not required for splicing but do specify cytoplasmic turnover". Genes & Development. 31 (10): 990–1006. doi:10.1101/gad.301036.117. PMC 5495127. PMID 28637692.
- ^ Rosa-Mercado NA, Withers JB, Steitz JA (May 2017). "6A debate: methylation of mature mRNA is not dynamic but accelerates turnover". Genes & Development. 31 (10): 957–958. doi:10.1101/gad.302695.117. PMC 5495124. PMID 28637691.
- ^ Abakir, Abdulkadir; Giles, Tom C.; Cristini, Agnese; Foster, Jeremy M.; Dai, Nan; Starczak, Marta; Rubio-Roldan, Alejandro; Li, Miaomiao; Eleftheriou, Maria; Crutchley, James; Flatt, Luke; Young, Lorraine; Gaffney, Daniel J.; Denning, Chris; Dalhus, Bjørn; Emes, Richard D.; Gackowski, Daniel; Corrêa, Ivan R.; Garcia-Perez, Jose L.; Klungland, Arne; Gromak, Natalia; Ruzov, Alexey (January 2020). "N 6 -methyladenosine regulates the stability of RNA:DNA hybrids in human cells". Nature Genetics. 52 (1): 48–55. doi:10.1038/s41588-019-0549-x. PMC 6974403. PMID 31844323.
- ^ Fustin JM, Doi M, Yamaguchi Y, Hida H, Nishimura S, Yoshida M, Isagawa T, Morioka MS, Kakeya H, Manabe I, Okamura H (November 2013). "RNA-methylation-dependent RNA processing controls the speed of the circadian clock". Cell. 155 (4): 793–806. doi:10.1016/j.cell.2013.10.026. PMID 24209618.
- ^ Hastings MH (November 2013). "m(6)A mRNA methylation: a new circadian pacesetter". Cell. 155 (4): 740–1. doi:10.1016/j.cell.2013.10.028. PMID 24209613.
- ^ Fustin, J.M.; Ye, S., Rakers, C.; Kaneko, K.; Fukumoto, K.; Yamano, M.; Versteven, M.; Grünewald, E.; Cargill, S.J.; Tamai, T.K.; Xu, Y.; Jabbur, M.L.; Kojima, R.; Lamberti, M.L.; Yoshioka-Kobayashi, K.; Whitmore, D.; Tammam, S.; Howell, P.L.; Kageyama, R.; Matsuo, T.; Stanewsky, R.; Golombek, D.A.; Johnson, C.H.; Kakeya, H.; van Ooijen, G.; Okamura, H. (2020). "Methylation deficiency disrupts biological rhythms from bacteria to humans". Communications Biology. 3 (211): 211. doi:10.1038/s42003-020-0942-0. PMC 7203018. PMID 32376902.
{{cite journal}}: CS1 maint: 작성자 매개변수 사용(링크) - ^ Akilzhanova A, Nurkina Z, Momynaliev K, Ramanculov E, Zhumadilov Z, Zhumadilov Z, Rakhypbekov T, Hayashida N, Nakashima M, Takamura N (September 2013). "Genetic profile and determinants of homocysteine levels in Kazakhstan patients with breast cancer". Anticancer Research. 33 (9): 4049–59. PMID 24023349.
- ^ Reddy SM, Sadim M, Li J, Yi N, Agarwal S, Mantzoros CS, Kaklamani VG (August 2013). "Clinical and genetic predictors of weight gain in patients diagnosed with breast cancer" (PDF). British Journal of Cancer. 109 (4): 872–81. doi:10.1038/bjc.2013.441. PMC 3749587. PMID 23922112.
- ^ Heiliger KJ, Hess J, Vitagliano D, Salerno P, Braselmann H, Salvatore G, Ugolini C, Summerer I, Bogdanova T, Unger K, Thomas G, Santoro M, Zitzelsberger H (June 2012). "Novel candidate genes of thyroid tumourigenesis identified in Trk-T1 transgenic mice". Endocrine-Related Cancer. 19 (3): 409–21. doi:10.1530/ERC-11-0387. PMID 22454401.
- ^ Ortega A, Niksic M, Bachi A, Wilm M, Sánchez L, Hastie N, Valcárcel J (January 2003). "Biochemical function of female-lethal (2)D/Wilms' tumor suppressor-1-associated proteins in alternative pre-mRNA splicing". The Journal of Biological Chemistry. 278 (5): 3040–7. doi:10.1074/jbc.M210737200. PMID 12444081.
- ^ Jin DI, Lee SW, Han ME, Kim HJ, Seo SA, Hur GY, Jung S, Kim BS, Oh SO (December 2012). "Expression and roles of Wilms' tumor 1-associating protein in glioblastoma". Cancer Science. 103 (12): 2102–9. doi:10.1111/cas.12022. PMC 7659328. PMID 22957919.
- ^ Lin Y, Ueda J, Yagyu K, Ishii H, Ueno M, Egawa N, Nakao H, Mori M, Matsuo K, Kikuchi S (July 2013). "Association between variations in the fat mass and obesity-associated gene and pancreatic cancer risk: a case-control study in Japan". BMC Cancer. 13: 337. doi:10.1186/1471-2407-13-337. PMC 3716552. PMID 23835106.
- ^ Casalegno-Garduño R, Schmitt A, Wang X, Xu X, Schmitt M (October 2010). "Wilms' tumor 1 as a novel target for immunotherapy of leukemia". Transplantation Proceedings. 42 (8): 3309–11. doi:10.1016/j.transproceed.2010.07.034. PMID 20970678.
- ^ Linnebacher M, Wienck A, Boeck I, Klar E (2010-03-18). "Identification of an MSI-H tumor-specific cytotoxic T cell epitope generated by the (-1) frame of U79260(FTO)". Journal of Biomedicine & Biotechnology. 2010: 841451. doi:10.1155/2010/841451. PMC 2842904. PMID 20339516.
- ^ Machiela MJ, Lindström S, Allen NE, Haiman CA, Albanes D, Barricarte A, Berndt SI, Bueno-de-Mesquita HB, Chanock S, Gaziano JM, Gapstur SM, Giovannucci E, Henderson BE, Jacobs EJ, Kolonel LN, Krogh V, Ma J, Stampfer MJ, Stevens VL, Stram DO, Tjønneland A, Travis R, Willett WC, Hunter DJ, Le Marchand L, Kraft P (December 2012). "Association of type 2 diabetes susceptibility variants with advanced prostate cancer risk in the Breast and Prostate Cancer Cohort Consortium". American Journal of Epidemiology. 176 (12): 1121–9. doi:10.1093/aje/kws191. PMC 3571230. PMID 23193118.
- ^ Long J, Zhang B, Signorello LB, Cai Q, Deming-Halverson S, Shrubsole MJ, Sanderson M, Dennis J, Michailidou K, Michailiou K, Easton DF, Shu XO, Blot WJ, Zheng W (2013-04-08). "Evaluating genome-wide association study-identified breast cancer risk variants in African-American women". PLOS ONE. 8 (4): e58350. Bibcode:2013PLoSO...858350L. doi:10.1371/journal.pone.0058350. PMC 3620157. PMID 23593120.
- ^ Kaklamani V, Yi N, Sadim M, Siziopikou K, Zhang K, Xu Y, Tofilon S, Agarwal S, Pasche B, Mantzoros C (April 2011). "The role of the fat mass and obesity associated gene (FTO) in breast cancer risk". BMC Medical Genetics. 12: 52. doi:10.1186/1471-2350-12-52. PMC 3089782. PMID 21489227.
- ^ Pierce BL, Austin MA, Ahsan H (June 2011). "Association study of type 2 diabetes genetic susceptibility variants and risk of pancreatic cancer: an analysis of PanScan-I data". Cancer Causes & Control. 22 (6): 877–83. doi:10.1007/s10552-011-9760-5. PMC 7043136. PMID 21445555.
- ^ Bokar JA (2005-01-01). "The biosynthesis and functional roles of methylated nucleosides in eukaryotic mRNA". In Grosjean H (ed.). Fine-Tuning of RNA Functions by Modification and Editing. Topics in Current Genetics. Vol. 12. Springer Berlin Heidelberg. pp. 141–177. doi:10.1007/b106365. ISBN 9783540244950.
- ^ Lin S, Choe J, Du P, Triboulet R, Gregory RI (May 2016). "The m(6)A Methyltransferase METTL3 Promotes Translation in Human Cancer Cells". Molecular Cell. 62 (3): 335–345. doi:10.1016/j.molcel.2016.03.021. PMC 4860043. PMID 27117702.
- ^ Zhang C, Samanta D, Lu H, Bullen JW, Zhang H, Chen I, He X, Semenza GL (April 2016). "Hypoxia induces the breast cancer stem cell phenotype by HIF-dependent and ALKBH5-mediated m6A-demethylation of NANOG mRNA". Proceedings of the National Academy of Sciences of the United States of America. 113 (14): E2047–56. Bibcode:2016PNAS..113E2047Z. doi:10.1073/pnas.1602883113. PMC 4833258. PMID 27001847.
- ^ Loos RJ, Yeo GS (January 2014). "The bigger picture of FTO: the first GWAS-identified obesity gene". Nature Reviews. Endocrinology. 10 (1): 51–61. doi:10.1038/nrendo.2013.227. PMC 4188449. PMID 24247219.
- ^ Frayling TM, Timpson NJ, Weedon MN, Zeggini E, Freathy RM, Lindgren CM, Perry JR, Elliott KS, Lango H, Rayner NW, Shields B, Harries LW, Barrett JC, Ellard S, Groves CJ, Knight B, Patch AM, Ness AR, Ebrahim S, Lawlor DA, Ring SM, Ben-Shlomo Y, Jarvelin MR, Sovio U, Bennett AJ, Melzer D, Ferrucci L, Loos RJ, Barroso I, Wareham NJ, Karpe F, Owen KR, Cardon LR, Walker M, Hitman GA, Palmer CN, Doney AS, Morris AD, Smith GD, Hattersley AT, McCarthy MI (May 2007). "A common variant in the FTO gene is associated with body mass index and predisposes to childhood and adult obesity". Science. 316 (5826): 889–94. Bibcode:2007Sci...316..889F. doi:10.1126/science.1141634. PMC 2646098. PMID 17434869.
- ^ Wang L, Yu Q, Xiong Y, Liu L, Zhang X, Zhang Z, Wu J, Wang B (2013). "Variant rs1421085 in the FTO gene contribute childhood obesity in Chinese children aged 3-6 years". Obesity Research & Clinical Practice. 7 (1): e14–22. doi:10.1016/j.orcp.2011.12.007. PMID 24331679.
- ^ Kalnina I, Zaharenko L, Vaivade I, Rovite V, Nikitina-Zake L, Peculis R, Fridmanis D, Geldnere K, Jacobsson JA, Almen MS, Pirags V, Schiöth HB, Klovins J (September 2013). "Polymorphisms in FTO and near TMEM18 associate with type 2 diabetes and predispose to younger age at diagnosis of diabetes". Gene. 527 (2): 462–8. doi:10.1016/j.gene.2013.06.079. PMID 23860325.
- ^ Karra E, O'Daly OG, Choudhury AI, Yousseif A, Millership S, Neary MT, Scott WR, Chandarana K, Manning S, Hess ME, Iwakura H, Akamizu T, Millet Q, Gelegen C, Drew ME, Rahman S, Emmanuel JJ, Williams SC, Rüther UU, Brüning JC, Withers DJ, Zelaya FO, Batterham RL (August 2013). "A link between FTO, ghrelin, and impaired brain food-cue responsivity". The Journal of Clinical Investigation. 123 (8): 3539–51. doi:10.1172/jci44403. PMC 3726147. PMID 23867619.
- ^ Zhao X, Yang Y, Sun BF, Shi Y, Yang X, Xiao W, Hao YJ, Ping XL, Chen YS, Wang WJ, Jin KX, Wang X, Huang CM, Fu Y, Ge XM, Song SH, Jeong HS, Yanagisawa H, Niu Y, Jia GF, Wu W, Tong WM, Okamoto A, He C, Rendtlew Danielsen JM, Wang XJ, Yang YG (December 2014). "FTO-dependent demethylation of N6-methyladenosine regulates mRNA splicing and is required for adipogenesis". Cell Research. 24 (12): 1403–19. doi:10.1038/cr.2014.151. PMC 4260349. PMID 25412662.
- ^ Merkestein M, Laber S, McMurray F, Andrew D, Sachse G, Sanderson J, Li M, Usher S, Sellayah D, Ashcroft FM, Cox RD (April 2015). "FTO influences adipogenesis by regulating mitotic clonal expansion". Nature Communications. 6: 6792. Bibcode:2015NatCo...6.6792M. doi:10.1038/ncomms7792. PMC 4410642. PMID 25881961.
- ^ Zhang M, Zhang Y, Ma J, Guo F, Cao Q, Zhang Y, Zhou B, Chai J, Zhao W, Zhao R (2015-07-28). "The Demethylase Activity of FTO (Fat Mass and Obesity Associated Protein) Is Required for Preadipocyte Differentiation". PLOS ONE. 10 (7): e0133788. Bibcode:2015PLoSO..1033788Z. doi:10.1371/journal.pone.0133788. PMC 4517749. PMID 26218273.
- ^ Hess ME, Hess S, Meyer KD, Verhagen LA, Koch L, Brönneke HS, Dietrich MO, Jordan SD, Saletore Y, Elemento O, Belgardt BF, Franz T, Horvath TL, Rüther U, Jaffrey SR, Kloppenburg P, Brüning JC (August 2013). "The fat mass and obesity associated gene (Fto) regulates activity of the dopaminergic midbrain circuitry". Nature Neuroscience. 16 (8): 1042–8. doi:10.1038/nn.3449. PMID 23817550. S2CID 11452560.
- ^ Kim HJ, Kim NC, Wang YD, Scarborough EA, Moore J, Diaz Z, MacLea KS, Freibaum B, Li S, Molliex A, Kanagaraj AP, Carter R, Boylan KB, Wojtas AM, Rademakers R, Pinkus JL, Greenberg SA, Trojanowski JQ, Traynor BJ, Smith BN, Topp S, Gkazi AS, Miller J, Shaw CE, Kottlors M, Kirschner J, Pestronk A, Li YR, Ford AF, Gitler AD, Benatar M, King OD, Kimonis VE, Ross ED, Weihl CC, Shorter J, Taylor JP (March 2013). "Mutations in prion-like domains in hnRNPA2B1 and hnRNPA1 cause multisystem proteinopathy and ALS". Nature. 495 (7442): 467–73. Bibcode:2013Natur.495..467K. doi:10.1038/nature11922. PMC 3756911. PMID 23455423.
- ^ Wang, ZL; Li, B; Luo, YX; Lin, Q; Liu, SR; Zhang, XQ; Zhou, H; Yang, JH; Qu, LH (2 January 2018). "Comprehensive Genomic Characterization of RNA-Binding Proteins across Human Cancers". Cell Reports. 22 (1): 286–298. doi:10.1016/j.celrep.2017.12.035. PMID 29298429.
- ^ Narayan P, Rottman FM (1992-01-01). Nord FF (ed.). Advances in Enzymology and Related Areas of Molecular Biology. Vol. 65. John Wiley & Sons, Inc. pp. 255–285. doi:10.1002/9780470123119.ch7. ISBN 9780470123119. PMID 1315118.
- ^ Kennedy EM, Bogerd HP, Kornepati AV, Kang D, Ghoshal D, Marshall JB, Poling BC, Tsai K, Gokhale NS, Horner SM, Cullen BR (May 2016). "Posttranscriptional m(6)A Editing of HIV-1 mRNAs Enhances Viral Gene Expression". Cell Host & Microbe. 19 (5): 675–85. doi:10.1016/j.chom.2016.04.002. PMC 4867121. PMID 27117054.
- ^ Tirumuru N, Zhao BS, Lu W, Lu Z, He C, Wu L (July 2016). "N(6)-methyladenosine of HIV-1 RNA regulates viral infection and HIV-1 Gag protein expression". eLife. 5. doi:10.7554/eLife.15528. PMC 4961459. PMID 27371828.
- ^ Lichinchi G, Gao S, Saletore Y, Gonzalez GM, Bansal V, Wang Y, Mason CE, Rana TM (February 2016). "Dynamics of the human and viral m(6)A RNA methylomes during HIV-1 infection of T cells". Nature Microbiology. 1 (4): 16011. doi:10.1038/nmicrobiol.2016.11. PMC 6053355. PMID 27572442.
- ^ Lichinchi G, Zhao BS, Wu Y, Lu Z, Qin Y, He C, Rana TM (November 2016). "Dynamics of Human and Viral RNA Methylation during Zika Virus Infection". Cell Host & Microbe. 20 (5): 666–673. doi:10.1016/j.chom.2016.10.002. PMC 5155635. PMID 27773536.
- ^ Gokhale NS, McIntyre AB, McFadden MJ, Roder AE, Kennedy EM, Gandara JA, et al. (November 2016). "N6-Methyladenosine in Flaviviridae Viral RNA Genomes Regulates Infection". Cell Host & Microbe. 20 (5): 654–665. doi:10.1016/j.chom.2016.09.015. PMC 5123813. PMID 27773535.


