원산지인식복합체
Origin recognition complex| 원산지인식복합소단위2 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| 식별자 | |||||||||
| 기호 | ORC2 | ||||||||
| Pfam | PF04084 | ||||||||
| 인터프로 | IPR007220 | ||||||||
| |||||||||
| 원산지 인식 복합체(ORC) 단위 3 N단자 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| 식별자 | |||||||||
| 기호 | ORC3_N | ||||||||
| Pfam | PF07034 | ||||||||
| 인터프로 | IPR010748 | ||||||||
| |||||||||
| 원산지인식복합소단위6(ORC6) | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| 식별자 | |||||||||
| 기호 | ORC6 | ||||||||
| Pfam | PF05460 | ||||||||
| 인터프로 | IPR008721 | ||||||||
| |||||||||
분자생물학에서 원산지 인식 콤플렉스(ORC)는 복제의 기원에 ATP 의존적인 방식으로 모든 진핵생물 및 고고학에서 결합하는 다중단위 DNA 결합 콤플렉스(6개 서브유닛)이다.이 단지의 서브유닛은 ORC1, ORC2, ORC3, ORC4, ORC5, ORC6 유전자에 의해 암호화된다.[1][2][3]ORC는 진핵 DNA 복제를 위한 중심 성분이며, 세포 주기 내내 복제 기원에서 염색질에 결합되어 있다.[4]
ORC는 게놈 전체에 걸쳐 DNA 복제를 지시하며 게놈의 개시를 위해 필요하다.[5][6][7]복제 기원에 바인딩된 ORC와 Noc3p는 Cdc6, Tah11(a.k.a)을 포함하는 사전 복제 복합체(pre-RC)의 조립 기반 역할을 한다.Cdt1) 및 Mcm2-Mcm7 복합체.[8][9][10][11]S상 중 DNA 합성 전 염색체 복제허가를 위해서는 G1 중 사전RC 조립이 필요하다.[12][13][14]사이클린 의존성 단백질 키나아제 Cdc28에 의한 Orc2, Orc6, Cdc6, MCM의 세포주기 조절 인산화 작용은 G2/M상에서의 재진화를 차단하는 등 DNA 복제의 시작을 조절한다.[4][15][16][17]
ORC는 복제 기원에 바인딩된 세포 주기에 걸쳐 존재하지만 늦은 유사분열과 초기 G1에서만 활성화된다.
효모에서는 ORC가 짝짓기형 로키 히든 MAT Left(HML)와 HMR(Hidden MAT Right)에서도 음소거 설정의 역할을 하며,[5][6][7] ORC는 HML과 HMR에서 Siri1 소음 단백질을 채용하여 전사적으로 무성 색소체의 조립에 참여한다.[7][18][19]
Orc1만이 ATPase 활동을 가지고 있지만, Orc1과 Orc5는 모두 ATP를 바인딩한다.[20]Orc1에 의한 ATP의 결합은 DNA에 대한 ORC 결합에 필요하며 세포 생존에 필수적이다.[11]Orc1의 ATPase 활동은 사전 RC 형성에 관여한다.[21][22][23]Orc5에 의한 ATP 바인딩은 ORC 전체의 안정성을 위해 중요하다.Orc1-5 서브유닛만 원점 바인딩에 필요하며, Orc6는 일단 형성된 사전 RC의 유지보수를 위해 필수적이다.[24]ORC 내의 상호작용은 Orc2-3-6이 핵심 복합체를 형성할 수 있음을 시사한다.[4]또한, 최근 한 보고서는 효모 ORC 싹이 면허를 통제하기 위해 세포 주기에 의존하는 방식으로 약화될 것을 제안한다.[25][26]
단백질
ORC에는 다음과 같은 단백질이 존재한다.
| S. 세레비시아과 | S. 폼비 | D. 멜라노가스터 | 척추동물 |
|---|---|---|---|
| ORC 1-6 | ORC 1-6 | ORC 1-6 | ORC 1-6 |
| Cdc6 | Cdc18 | Cdc6 | Cdc6 |
| Cdt1/Tah11/Sid2 | Cdt1 | DUP | Cdt1/RLF-B |
| 맥m2 | Mcm2/Cdc19/Nda1 | 맥m2 | 맥m2 |
| 맥엠3 | 맥엠3 | 맥엠3 | 맥엠3 |
| Cdc54/Mcm4 | Cdc21 | DPA | 맥엠4 |
| Cdc46/Mcm5 | Mcm5/Nda4 | 맥름5 | 맥름5 |
| 맥름6 | Mcm6/Mis5 | 맥름6 | 맥름6 |
| Cdc47/Mcm7 | 맥엠7 | 맥엠7 | mcm7 |
아르케아는 ORC, Mcm의 단순화된 버전을 특징으로, 그 결과 결합된 사전RC가 특징이다.6개의 서로 다른 mcm 단백질을 사용하여 사이비대칭 이단백질을 형성하는 대신 고고학적 MCM에서 6개의 서브유닛은 모두 동일하다.그들은 보통 Cdc6와 Orc1 둘 다에 동질인 여러 단백질을 가지고 있는데, 그 중 일부는 둘 다의 기능을 수행한다.진핵산 오크와 달리, 그들은 항상 콤플렉스를 형성하지는 않는다.사실, 그것들은 형성될 때 서로 다른 복잡한 구조를 가지고 있다.Sulfolobus islandicus는 또한 그것의 복제 기원들 중 하나를 인식하기 위해 Cdt1 호몰로그를 사용한다.[28]
자동으로 시퀀스 복제
싹트기 효모
싹이 트는 효모에서 처음 발견된 자율 복제 시퀀스(ARS)는 ORC의 성공에 필수적이다.이러한 100-200bp 시퀀스는 S 단계 동안 복제 활동을 촉진한다.ARS는 싹이 트는 효모 염색체의 새로운 위치에 배치될 수 있으며 이러한 부위로부터의 복제를 촉진할 것이다.11bp(A 원소로 알려져 있음)의 보존도가 높은 순서는 싹이 돋아나는 효모에서 기원 기능에 필수적이라고 생각된다.[27]ORC는 원래 효모 싹트기에서 ARS의 A 요소에 바인딩할 수 있는 능력에 의해 식별되었다.
동물
동물 세포는 ARS의 훨씬 더 암호화된 버전을 포함하고 있으며, 아직 보존된 염기서열은 발견되지 않았다.그러나 동물 세포에서는 복제 기원이 replicon clusters라고 불리는 묶음으로 모인다.각 클러스터의 복제자는 길이가 유사하지만, 개별 클러스터는 다양한 길이의 복제본을 가지고 있다.이러한 복제자는 모두 ORC가 결합하는 유사한 기본 잔류물을 가지고 있으며, 이는 여러 면에서 보존된 11bp A 요소를 모방한다.이 모든 클러스터는 S상 동안 동시에 활성화된다.[27]
사전 RC 조립체에서의 역할
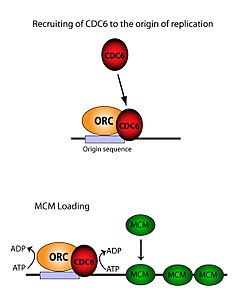
ORC는 MCM 복합체(Pre-RC)를 DNA에 로드하는 데 필수적이다.이 프로세스는 ORC, Noc3, Cdc6 및 Cdt1에 따라 달라진다. 여기에는 여러 ATP가 관리하는 모집 이벤트가 포함된다.첫째, ORC, Noc3p, Cdc6는 (ARS 유형 영역으로 표시) 원점 DNA에 콤플렉스를 형성한다.이어 새로운 ORC/Noc3/Cdc6 복합체가 현장에 Cdt1/Mcm2-7 분자를 모집한다.일단 이 거대한 ORC/Noc3/Cdc6/Cdt1/Mcm2-7 복합체가 형성되면, ORC/Noc3/Cdc6/Cdt1 분자는 Cdc6에 의한 ATP의 가수 분해에 의해 Mcm2-7을 DNA 자체에 탑재하기 위해 함께 작용한다.Cdc6의 인광 활성도는 ORC와 원본 DNA 모두에 의존한다.이로 인해 Cdt1은 DNA의 안정성이 저하되고 콤플렉스에서 떨어져 나가 맥m2-7이 DNA에 탑재되게 된다.[29][27][30][31]ORC, MCM의 구조는 물론 중간 OCCM 단지의 구조도 해결됐다.[32]
오리진 바인딩 활동
ORC는 6개의 분리된 하위 유니트로 구성되지만, 이 중 오직 하나의 하위 유니트만이 유의미한 것으로 밝혀졌다 - ORC.생체내 연구에서는 Lys-263과 Arg-367이 충실한 ORC 하중을 담당하는 기본 잔류물임을 밝혀냈다.이 분자들은 위에서 언급한 ARS를 나타낸다.[33]ORC1은 ATP 및 이러한 기본 잔류물과 상호 작용하여 ORC를 원래 DNA에 바인딩한다.이는 복제 훨씬 이전에 발생하며, ORC 자체는 Mcm2-7 로딩이 발생할 때 이미 오리진 DNA에 바인딩되어 있다는 것이 확인되었다.[31]Mcm2-7을 처음 장착할 때 DNA를 완전히 감싸고 헬리케아제 활성을 억제한다.S상에서는 Mcm2-7 콤플렉스가 헬리코아제 공동 인자 Cdc45와 GINS와 상호작용하여 하나의 DNA 가닥을 분리하고 원점을 풀고 염색체 아래로 복제를 시작한다.양방향 복제를 하기 위해서, 이 과정은 오리진에서 두 번 일어난다.두 로딩 이벤트는 첫 번째와 동일한 프로세스를 통해 하나의 ORC에 의해 조정된다.[34]
참고 항목
참조
- ^ 미국 국립 의학 라이브러리 의료 과목 제목(MSH)의 오리진+인식+복합
- ^ Dutta A, Bell SP (1997). "Initiation of DNA replication in eukaryotic cells". Annual Review of Cell and Developmental Biology. 13: 293–332. doi:10.1146/annurev.cellbio.13.1.293. PMID 9442876.
- ^ Chesnokov IN (2007). "Multiple functions of the origin recognition complex". International Review of Cytology. 256: 69–109. doi:10.1016/S0074-7696(07)56003-1. ISBN 9780123737007. PMID 17241905.
{{cite journal}}:Cite 저널은 필요로 한다.journal=(도움말) - ^ a b c Matsuda K, Makise M, Sueyasu Y, Takehara M, Asano T, Mizushima T (December 2007). "Yeast two-hybrid analysis of the origin recognition complex of Saccharomyces cerevisiae: interaction between subunits and identification of binding proteins". FEMS Yeast Research. 7 (8): 1263–9. doi:10.1111/j.1567-1364.2007.00298.x. PMID 17825065.
- ^ a b Bell SP, Stillman B (May 1992). "ATP-dependent recognition of eukaryotic origins of DNA replication by a multiprotein complex". Nature. 357 (6374): 128–34. Bibcode:1992Natur.357..128B. doi:10.1038/357128a0. PMID 1579162. S2CID 4346767.
- ^ a b Bell SP, Mitchell J, Leber J, Kobayashi R, Stillman B (November 1995). "The multidomain structure of Orc1p reveals similarity to regulators of DNA replication and transcriptional silencing". Cell. 83 (4): 563–8. doi:10.1016/0092-8674(95)90096-9. PMID 7585959.
- ^ a b c Gibson DG, Bell SP, Aparicio OM (June 2006). "Cell cycle execution point analysis of ORC function and characterization of the checkpoint response to ORC inactivation in Saccharomyces cerevisiae". Genes to Cells. 11 (6): 557–73. doi:10.1111/j.1365-2443.2006.00967.x. PMID 16716188. S2CID 22439595.
- ^ Zhang Y, Yu Z, Fu X, Liang C (June 2002). "Noc3p, a bHLH Protein, Plays an Integral Role in the Initiation of DNA Replication in Budding Yeast". Cell. 109 (7): 849–860. doi:10.1016/s0092-8674(02)00805-x. PMID 12110182.
- ^ Rao H, Stillman B (March 1995). "The origin recognition complex interacts with a bipartite DNA binding site within yeast replicators". Proceedings of the National Academy of Sciences of the United States of America. 92 (6): 2224–8. Bibcode:1995PNAS...92.2224R. doi:10.1073/pnas.92.6.2224. PMC 42456. PMID 7892251.
- ^ Rowley A, Cocker JH, Harwood J, Diffley JF (June 1995). "Initiation complex assembly at budding yeast replication origins begins with the recognition of a bipartite sequence by limiting amounts of the initiator, ORC". The EMBO Journal. 14 (11): 2631–41. doi:10.1002/j.1460-2075.1995.tb07261.x. PMC 398377. PMID 7781615.
- ^ a b Speck C, Chen Z, Li H, Stillman B (November 2005). "ATPase-dependent cooperative binding of ORC and Cdc6 to origin DNA". Nature Structural & Molecular Biology. 12 (11): 965–71. doi:10.1038/nsmb1002. PMC 2952294. PMID 16228006.
- ^ Kelly TJ, Brown GW (2000). "Regulation of chromosome replication". Annual Review of Biochemistry. 69: 829–80. doi:10.1146/annurev.biochem.69.1.829. PMID 10966477.
- ^ Bell SP, Dutta A (2002). "DNA replication in eukaryotic cells". Annual Review of Biochemistry. 71: 333–74. doi:10.1146/annurev.biochem.71.110601.135425. PMID 12045100.
- ^ Stillman B (February 2005). "Origin recognition and the chromosome cycle". FEBS Letters. 579 (4): 877–84. doi:10.1016/j.febslet.2004.12.011. PMID 15680967. S2CID 33220937.
- ^ Weinreich M, Liang C, Chen HH, Stillman B (September 2001). "Binding of cyclin-dependent kinases to ORC and Cdc6p regulates the chromosome replication cycle". Proceedings of the National Academy of Sciences of the United States of America. 98 (20): 11211–7. doi:10.1073/pnas.201387198. PMC 58709. PMID 11572976.
- ^ Nguyen VQ, Co C, Li JJ (June 2001). "Cyclin-dependent kinases prevent DNA re-replication through multiple mechanisms". Nature. 411 (6841): 1068–73. Bibcode:2001Natur.411.1068N. doi:10.1038/35082600. PMID 11429609. S2CID 4393812.
- ^ Archambault V, Ikui AE, Drapkin BJ, Cross FR (August 2005). "Disruption of mechanisms that prevent rereplication triggers a DNA damage response". Molecular and Cellular Biology. 25 (15): 6707–21. doi:10.1128/MCB.25.15.6707-6721.2005. PMC 1190345. PMID 16024805.
- ^ Triolo T, Sternglanz R (May 1996). "Role of interactions between the origin recognition complex and SIR1 in transcriptional silencing". Nature. 381 (6579): 251–3. Bibcode:1996Natur.381..251T. doi:10.1038/381251a0. PMID 8622770. S2CID 4309206.
- ^ Fox CA, Ehrenhofer-Murray AE, Loo S, Rine J (June 1997). "The origin recognition complex, SIR1, and the S phase requirement for silencing". Science. 276 (5318): 1547–51. doi:10.1126/science.276.5318.1547. PMID 9171055.
- ^ Klemm RD, Austin RJ, Bell SP (February 1997). "Coordinate binding of ATP and origin DNA regulates the ATPase activity of the origin recognition complex". Cell. 88 (4): 493–502. doi:10.1016/S0092-8674(00)81889-9. PMID 9038340.
- ^ Klemm RD, Bell SP (July 2001). "ATP bound to the origin recognition complex is important for preRC formation". Proceedings of the National Academy of Sciences of the United States of America. 98 (15): 8361–7. Bibcode:2001PNAS...98.8361K. doi:10.1073/pnas.131006898. PMC 37444. PMID 11459976.
- ^ Bowers JL, Randell JC, Chen S, Bell SP (December 2004). "ATP hydrolysis by ORC catalyzes reiterative Mcm2-7 assembly at a defined origin of replication". Molecular Cell. 16 (6): 967–78. doi:10.1016/j.molcel.2004.11.038. PMID 15610739.
- ^ Randell JC, Bowers JL, Rodríguez HK, Bell SP (January 2006). "Sequential ATP hydrolysis by Cdc6 and ORC directs loading of the Mcm2-7 helicase". Molecular Cell. 21 (1): 29–39. doi:10.1016/j.molcel.2005.11.023. PMID 16387651.
- ^ Semple JW, Da-Silva LF, Jervis EJ, Ah-Kee J, Al-Attar H, Kummer L, et al. (November 2006). "An essential role for Orc6 in DNA replication through maintenance of pre-replicative complexes". The EMBO Journal. 25 (21): 5150–8. doi:10.1038/sj.emboj.7601391. PMC 1630405. PMID 17053779.
- ^ Yin YC, Prasanth SG (July 2021). "Replication initiation: Implications in genome integrity". DNA Repair. 103. doi:10.1016/j.dnarep.2021.103131.
- ^ Amin A, Wu, R, Cheung MH, Scott JF, Wang Z, Zhou Z, Liu C, Zhu G, Wong KC, Yu Z, Liang C (March 2020). "An Essential and Cell-Cycle-Dependent ORC Dimerization Cycle Regulates Eukaryotic Chromosomal DNA Replication". Cell Reports. 30 (10): 3323-3338.e6. doi:10.1016/j.celrep.2020.02.046. PMID 32160540.
- ^ a b c d Morgan, David (2007). The Cell Cycle: Principles of Control. Primers in Biology. pp. 62–75. ISBN 978-0878935086.
- ^ Ausiannikava D, Allers T (January 2017). "Diversity of DNA Replication in the Archaea". Genes. 8 (2): 56. doi:10.3390/genes8020056. PMC 5333045. PMID 28146124.
- ^ Fernández-Cid A, Riera A, Tognetti S, Herrera MC, Samel S, Evrin C, et al. (May 2013). "An ORC/Cdc6/MCM2-7 complex is formed in a multistep reaction to serve as a platform for MCM double-hexamer assembly". Molecular Cell. 50 (4): 577–88. doi:10.1016/j.molcel.2013.03.026. PMID 23603117.
- ^ Randell JC, Bowers JL, Rodríguez HK, Bell SP (January 2006). "Sequential ATP hydrolysis by Cdc6 and ORC directs loading of the Mcm2-7 helicase". Molecular Cell. 21 (1): 29–39. doi:10.1016/j.molcel.2005.11.023. PMID 16387651.
- ^ a b Speck C, Chen Z, Li H, Stillman B (November 2005). "ATPase-dependent cooperative binding of ORC and Cdc6 to origin DNA". Nature Structural & Molecular Biology. 12 (11): 965–71. doi:10.1038/nsmb1002. PMC 2952294. PMID 16228006.
- ^ Yuan Z, Riera A, Bai L, Sun J, Nandi S, Spanos C, et al. (March 2017). "Structural basis of Mcm2-7 replicative helicase loading by ORC-Cdc6 and Cdt1". Nature Structural & Molecular Biology. 24 (3): 316–324. doi:10.1038/nsmb.3372. PMC 5503505. PMID 28191893.
- ^ Kawakami H, Ohashi E, Kanamoto S, Tsurimoto T, Katayama T (October 2015). "Specific binding of eukaryotic ORC to DNA replication origins depends on highly conserved basic residues". Scientific Reports. 5: 14929. doi:10.1038/srep14929. PMC 4601075. PMID 26456755.
- ^ Chistol G, Walter JC (April 2015). "Single-Molecule Visualization of MCM2-7 DNA Loading: Seeing Is Believing". Cell. 161 (3): 429–430. doi:10.1016/j.cell.2015.04.006. PMID 25910200.
추가 읽기
- Bell SP, Dutta A (July 2002). "DNA replication in eukaryotic cells". Annual Review of Biochemistry. Annual Reviews. 71: 333–74. doi:10.1146/annurev.biochem.71.110601.135425. PMID 12045100.
A comprehensive review of molecular DNA replication