1000개의 게놈 프로젝트
1000 Genomes Project2008년 1월에 시작된 1000개의 게놈 프로젝트(1KGP)는 지금까지 가장 상세한 인간 유전자 변형 카탈로그를 확립하기 위한 국제적인 연구 활동이었다.과학자들은 더 빠르고 저렴한 새로 개발된 기술을 사용하여 앞으로 3년 안에 여러 민족 집단에서 온 적어도 1,000명의 익명의 참가자들의 게놈을 배열할 계획을 세웠다.2010년에 이 프로젝트는 파일럿 단계를 마쳤으며,[1] 자세한 내용은 네이처 저널에 게재되었습니다.2012년에는 1092개의 게놈 배열이 [2]네이처지에 발표됐다.2015년 Nature지에서는 두 개의 논문이 결과와 프로젝트 완료 및 향후 [3]연구의 기회를 보고했습니다.[4]
밀접하게 관련된 그룹으로 제한되는 많은 희귀 변이가 확인되었고, 8개의 구조 변동 클래스가 [5]분석되었다.
이 프로젝트에는 중국, 이탈리아, 일본, 케냐, 나이지리아, 페루, 영국 및 미국을 포함한 전 세계 연구소의 다원적 연구팀이 참여하고 있습니다.각각은 방대한 염기서열 데이터 집합과 정교한 인간 게놈 지도에 기여할 것이며, 공공 데이터베이스를 통해 과학계와 일반 대중 [2]모두 자유롭게 접근할 수 있게 될 것이다.
모든 인간 유전자 변이에 대한 개요를 제공함으로써, 컨소시엄은 특히 유전학, 의학, 약리학, 생화학,[6] 생물정보학 분야의 모든 생물과학 분야를 위한 가치 있는 도구를 만들어 낼 것입니다.
배경
인간 게놈 프로젝트의 완료 이후, 인간 집단 유전학의 진보와 비교 유전학은 유전적 [7]다양성의 본질에 대한 점점 더 많은 통찰력을 얻는 것을 가능하게 했다.하지만 우리는 프로세스 생식체의 임의 추출처럼 이해하기, 구조적 변화(insertions/deletions(indels)시작하고 있copy(불확정 음성 변동), retroelements),single-nucleotide polymorphisms(SNPs)자연 선택은 수준과 변화의 생물과 또한 종들 사이에 패턴의 번호.[8][9][10][11]
인간 유전자 변이
성적 생식 중 배우자의 무작위 표본 추출은 후속 세대에 유전적 표류(특성의 모집단 빈도의 무작위 변동)로 이어지고 외부 영향이 없을 때 모든 변이를 잃게 될 것이다.유전적 표류 속도는 인구 규모에 반비례하며, 일정 기간 인구 규모가 감소하는 병목현상이나 창시자 효과(소수의 창시자 [8]개체로 거슬러 올라가는 개체)와 같은 특정 상황에서 가속화될 수 있다고 가정한다.
안자이 등은 인델이 인간과 침팬지 사이의 주요 조직적합성 궤적(MHC) 순서에서 관찰된 모든 변이의 90.4%를 차지한다는 것을 입증했다.여러 인델을 고려한 후, 두 종 사이의 높은 게놈 유사도(98.6% 뉴클레오티드 배열 동일성)는 86.7%로 떨어졌다.예를 들어 인간의 MICA와 MICB 유전자 위치 사이에 95킬로베이스(kb)가 대량 결실되면 단일 잡종 침팬지 MIC 유전자가 생성되며, 이 영역은 여러 레트로바이러스 감염의 특정 종별 처리와 다양한 자가면역질환에 대한 영향을 받기 쉽다.저자들은 보다 미묘한 SNP 대신 인델이 영장류 분화의 [9]원동력이었다고 결론지었다.
돌연변이 외에도 SNP와 복사 번호 변종(CNV)과 같은 다른 구조적 변종은 인간 집단의 유전적 다양성에 기여하고 있다.HapMap 샘플 컬렉션에서는 마이크로 어레이를 사용하여 게놈의 약 12%를 커버하고 수백 개의 유전자, 질병 위치, 기능 요소 및 세그먼트 복제를 포함하는 거의 1,500개의 복사 번호 가변 영역이 확인되었다.CNVs의 구체적인 기능은 아직 밝혀지지 않았지만, CNVs가 SNPs보다 게놈당 더 많은 뉴클레오티드 함량을 포괄한다는 사실은 유전적 다양성과 [10]진화에 있어 CNVs의 중요성을 강조한다.
인간 게놈 변이를 조사하는 것은 내병성(예: MHC 영역) 또는 약물 [12]대사의 차이에 기초할 수 있는 유전자를 식별할 수 있는 큰 잠재력을 가지고 있다.
자연선택
특성의 자연 선택 진화는 세 가지로 나눌 수 있다.방향성 또는 양성 선택은 특정 대립 유전자가 다른 대립 유전자에 비해 적합성이 높아 결과적으로 모집단 빈도(예: 박테리아 항생제 내성)가 증가하는 상황을 말한다.반대로 안정화 또는 음성 선택(정제 선택이라고도 함)은 다른 대립 유전자와 관련된 단점 때문에 빈도를 낮추거나 집단에서 대립 유전자를 제거한다.마지막으로, 균형 선택의 많은 형태가 존재한다; 그것들은 과우성(예를 들어 용혈성 빈혈과 말라리아 내성 모두에 관여하는 유전자인 G6PD와 같은 동종 접합 개인보다 적합함)으로써 종 내에서 유전적 변화를 증가시키거나, 다른 니치에 서식하는 종 내에서 공간적으로 변화할 수 있다.따라서 다른 [13]대립 유전자를 선호합니다.몇몇 유전자 차이는 체력에 영향을 미치지 않을 수 있다.이전에는 "정크" DNA로 생각되었던 중성 변이는 자연 도태의 영향을 받지 않아 [14]변이가 적합성에 영향을 미치는 부위와 비교했을 때 그러한 부위에서 더 높은 유전적 변이를 일으킨다.
자연 선택이 인구 차이를 어떻게 형성했는지는 완전히 명확하지 않지만,[11] 최근 선택 중인 유전자 후보 지역이 확인되었다.DNA 다형성 패턴은 선택의 신호를 확실하게 검출하기 위해 사용될 수 있으며 질병 저항성이나 약물 [13][14]대사의 기초가 될 수 있는 유전자를 식별하는데 도움을 줄 수 있다.Barreiro 등은 음성 선택이 아미노산 변화 수준(특히 질병 관련 유전자)에서 모집단 분화를 감소시켰다는 증거를 발견했으며, 양성 선택은 유전자 영역(주로 익명이 아닌 5'-번역되지 않은 r)에서 모집단 분화를 증가시켜 인간 모집단의 지역적 적응을 보장했다.egion variants).[11]
대부분의 복잡하고 멘델식 질병(노인 개인이 더 이상 자손의 적합성에 기여하지 않는다고 가정하는 후발병을 제외한)은 생존 및/또는 번식에 영향을 미칠 것으로 생각되며, 따라서 이러한 질병의 근저에 있는 유전적 요인은 자연 선택에 의해 영향을 받아야 한다.그러나 오늘날 늦게 발병하는 질병은 질병의 진행을 지연시키는 유전자가 선택을 받았을 수 있기 때문에 과거에는 소아 질환일 수 있다.가우처병(GBA 유전자의 변형), 크론병(NOD2의 변형), 가족성 비후성 심근증(MYH7, TNNT2, TPM1, MYBPC3의 변형)은 모두 음성 선택의 예이다.이러한 질병 돌연변이는 주로 열성이고 낮은 빈도로 예상대로 분리되며, 가설화된 음성 선택을 뒷받침한다.제1형 당뇨병의 유전적 기반이 양성 [15]선택을 받았을 수 있다는 증거가 있다.균형 잡힌 선택에 의해 지원되는 높은 빈도로 질병을 유발하는 돌연변이가 나타나는 사례는 거의 보고되지 않았다.가장 두드러진 예는 동종 접합성 G6PD 효소 결핍과 결과적으로 용혈성 빈혈이 발생하는 경우, 그러나 헤테로 접합 상태에서는 말라리아에 대해 부분적으로 보호되는 G6PD 궤적의 돌연변이이다.중간 또는 높은 빈도의 질병 대립 유전자의 분리에 대한 다른 가능한 설명으로는 유전적 표류와 다이어트 또는 유전적 히치하이킹과 [12]같은 환경 변화로 인한 최근의 양성 선택으로의 변화가 있다.
종간(예를 들어 인간과 침팬지)뿐만 아니라 다른 개체군의 게놈 전체 비교 분석은 질병과 선택 사이의 관계를 이해하고 제약된 유전자의 돌연변이가 유전성 질병 표현형과 불균형적으로 연관되어 있다는 증거를 제공하는 데 도움을 주고 있다.복잡한 장애에 관여하는 유전자는 멘델병 유전자나 비질환 [12]유전자에 비해 덜 부정적인 선택을 받는 경향이 있다.
프로젝트 설명
이 섹션은 업데이트해야 합니다. 이 하는 데 을 주십시오. (2021년 4월) |
목표들
질병과 관련된 두 가지 종류의 유전자 변이가 있다.첫 번째는 주로 단순한 특징(예: 낭포성 섬유증, 헌팅턴병)에 심각한 영향을 미치는 희귀한 유전자 변형이다.두 번째, 보다 일반적인 유전적 변종은 가벼운 영향을 미치며 복잡한 특성(예: 인지, 당뇨병, 심장병)에 관여하는 것으로 생각된다.이 두 가지 유형의 유전자 변형 사이에는 상당한 지식의 차이가 있으며, 1000개의 게놈 프로젝트가 이에 [6]대처하기 위해 고안되었습니다.
이 프로젝트의 주요 목표는 인간 유전자 변이의 완전하고 상세한 카탈로그를 만드는 것이며, 이는 다시 질병에 대한 유전자 변이와 관련된 연관 연구에 사용될 수 있다.이를 통해 컨소시엄은 유전자 전체에서 1%만큼 낮은 경미한 대립 유전자 빈도를 가진 변종([16]예: SNP, CNV, 인델)의 95% 이상을 발견하고 유전자 영역에서 개체 빈도, 하플로타입 배경 및 관련 불균형 패턴을 추정하는 것을 목표로 한다.
2차 목표에는 향후 연구에서 유전자형 플랫폼을 위한 더 나은 SNP와 프로브 선택을 지원하고 인간 기준 시퀀스의 개선이 포함될 것이다.또한 완성된 데이터베이스는 선택 대상 지역, 여러 모집단의 변동을 연구하고 돌연변이와 [16]재조합의 기초 과정을 이해하는 데 유용한 도구가 될 것이다.
개요
인간 게놈은 약 30억 개의 DNA 염기쌍으로 구성되어 있으며 약 20,000개의 단백질 코드 유전자를 가지고 있는 것으로 추정된다.연구를 설계할 때 컨소시엄은 기술 과제, 데이터 품질 표준 및 시퀀스 [16]적용 범위와 같은 프로젝트 지표와 관련된 몇 가지 중요한 문제에 대처해야 했습니다.
향후 3년 [clarification needed]동안, 생어 연구소, BGI 선전, 국립 인간 게놈 연구소의 대규모 염기서열 분석 네트워크의 과학자들은 최소 1,000개의 인간 게놈을 배열할 계획이다.생성 및 분석해야 하는 시퀀스 데이터가 많기 때문에 시간이 [6]지남에 따라 다른 참가자를 모집할 수 있습니다.
생산 단계 2년 동안 하루에 100억 개 가까운 베이스가 배열될 것입니다.이는 24시간마다 두 개 이상의 인간 게놈을 가진 획기적인 용량에 해당한다.생물정보학 및 통계유전학 분야의 선도적인 전문가들에게 도전장을 내밀며, 시퀀스 데이터 세트는 6조 개의 DNA 베이스로 구성될 것이며, 이는 지난 25년 동안 [6]DNA 데이터베이스에 발표된 것보다 60배 더 많은 시퀀스 데이터로 구성될 것이다.
전체 프로젝트의 최종 설계를 결정하기 위해 3개의 파일럿 스터디가 설계되었으며 프로젝트 첫 해 내에 실시될 예정입니다.첫 번째 파일럿은 3대 지리적 그룹의 180명을 낮은 범위(2배)로 유전자형화할 예정이다.두 번째 시범 연구에서는 두 개의 핵가족(부모와 성인 자녀 모두)의 게놈 배열을 심층 조사한다(게놈당 20배).세 번째 시범 연구는 심층 커버리지(20배)[6][16]를 가진 1,000명 중 1,000명의 유전자의 코드 영역(exons)의 염기서열을 분석하는 것을 포함한다.
표준 DNA 염기서열 분석 기술을 사용할 경우 이 프로젝트는 5억 달러 이상이 소요될 것으로 추정되고 있다.따라서 몇 가지 새로운 기술(Solexa, 454, SOLiD 등)이 적용되어 예상 비용이 3,000만 달러에서 5,000만 달러로 낮아집니다.주요 지원은 영국 힌스턴에 있는 웰컴 트러스트 생어 연구소, 베이징 유전체학 연구소, 중국 선전(BGI 선전) 및 국립보건원([6]NIH)의 일부인 NHGRI에 의해 제공될 것이다.
Wayback Machine에서 Archived 2013-12-28의 Fort Lauderdale 원칙에 따라 프로젝트가 진행됨에 따라 모든 게놈 시퀀스 데이터(변종 호출 포함)를 자유롭게 사용할 수 있으며 1000개의 게놈 프로젝트 웹 페이지에서 FTP를 통해 다운로드할 수 있습니다.
인간 게놈 샘플
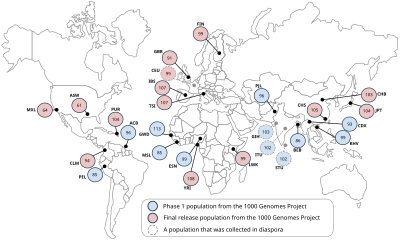
프로젝트의 전체 목표에 기초하여 샘플은 일반 질병에 대한 연관성 연구가 수행되고 있는 모집단에서 전력을 제공하기 위해 선택될 것이다.또한 제안된 카탈로그는 인간 [16]변이에 대한 기본 자원이 될 것이기 때문에 샘플은 의료 또는 표현형 정보를 가질 필요가 없다.
파일럿 스터디의 경우 HapMap 컬렉션의 인간 게놈 샘플이 배열됩니다.다른 [16]프로젝트의 결과와 비교할 수 있도록 추가 데이터(ENCODE 시퀀스, 게놈 전체 유전자형, fosmid-end 시퀀스, 구조적 변화 분석 및 유전자 발현 등)를 가진 샘플에 초점을 맞추는 것이 유용할 것이다.
광범위한 윤리 절차를 준수하여, 1000 Genetomes Project는 자원봉사자 기증자의 샘플을 사용합니다.다음 모집단이 연구에 포함됩니다.나이지리아 이바단의 Yoruba(YRI), 도쿄의 일본인(JPT), 베이징의 중국인(CHB), 북유럽 및 서유럽(CEU), 케냐 웨부예의 Luhya(LWK), 케냐의 Kinyawa의 Maasai(K)로스앤젤레스(MXL)의 멕시코계 혈통, 미국 남서부(ASW)[6]의 아프리카계 혈통.
| 아이디 | 장소 | 인구. | 세부 사항 |
|---|---|---|---|
| ASW | SW USA의 아프리카 조상 | 세부 사항 | |
| ACB | 바베이도스의 아프리카 카리브해 | 세부 사항 | |
| BEB | 방글라데시의 벵골어 | 세부 사항 | |
| GBR | 잉글랜드와 스코틀랜드에서 온 영국인 | 세부 사항 | |
| CDX | 중국 시샹반나의 중국 다이 | 세부 사항 | |
| CLM | 콜롬비아 메데인의 콜롬비아인 | 세부 사항 | |
| ESN | 나이지리아 에산 | 세부 사항 | |
| 핀 | 핀란드어(핀란드어) | 세부 사항 | |
| GWD | 감비아 서부 지역 – 만딩카 | 세부 사항 | |
| GIH | 미국 텍사스 휴스턴의 구자라티 인디언 | 세부 사항 | |
| CHB | 중국 베이징의 한족 | 세부 사항 | |
| CHS | 한족남, 중국 | 세부 사항 | |
| IBS | 스페인의 이베리아 인구 | 세부 사항 | |
| ITU | 영국의 인도 텔루구 | 세부 사항 | |
| JPT | 일본 도쿄의 일본인 | 세부 사항 | |
| KHV | 베트남 호찌민시의 킨 | 세부 사항 | |
| LWK | 케냐 웨부예의 루히야 | 세부 사항 | |
| MSL | 시에라리온의 멘데 | 세부 사항 | |
| MXL | 미국 로스앤젤레스 캘리포니아의 멕시코계 조상 | 세부 사항 | |
| 펠 | 페루, 리마의 페루인 | 세부 사항 | |
| PUR | 푸에르토리코의 푸에르토리코인 | 세부 사항 | |
| PJL | 파키스탄 라호르의 펀자비 | 세부 사항 | |
| 스튜 | 스리랑카 타밀어(영국) | 세부 사항 | |
| TSI | 이탈리아 토스카니 | 세부 사항 | |
| YRI | 나이지리아 이바단의 요루바 | 세부 사항 | |
| CEU | CEPH 컬렉션에서 나온 북유럽과 서유럽의 조상을 가진 유타 거주자 | 세부 사항 |
* 디아스포라에서 수집된 인구
커뮤니티 미팅
1000개의 게놈 프로젝트에서 생성된 데이터는 유전학계에서 널리 사용되고 있으며, 최초의 1000개의 게놈 프로젝트는 [18]생물학에서 가장 많이 인용된 논문 중 하나입니다.이 사용자 커뮤니티를 지원하기 위해 이 프로젝트는 2012년 7월에 커뮤니티 분석 회의를 개최하여 주요 프로젝트 발견, 인구 유전학 및 인간 질병 연구에 미치는 영향, 기타 대규모 시퀀싱 [19]연구 요약 등을 중점적으로 다루었습니다.
프로젝트 결과
파일럿 단계
파일럿 단계는 다음 3개의 프로젝트로 구성되었습니다.
- 4개 모집단에서 179명의 개인에 대한 저농도 전립선 배열
- 2개의 트리오(어머니-아버지-자녀)의 고대역 시퀀싱
- 7개 모집단에서 697명의 개체 엑손에 의한 배열
평균적으로 각 사람은 주석이 달린 유전자에서 약 250-300개의 기능 상실 변종과 이전에 유전 장애와 관련된 50-100개의 변종을 가지고 있는 것으로 밝혀졌다.2개의 트리오를 바탕으로 de novo geramine 돌연변이의 발생률은 [1]세대당 염기당 약 10으로−8 추정된다.
「 」를 참조해 주세요.
레퍼런스
- ^ a b Abecasis GR, Altshuler D, Auton A, Brooks LD, Durbin RM, Gibbs RA, et al. (October 2010). "A map of human genome variation from population-scale sequencing". Nature. 467 (7319): 1061–73. Bibcode:2010Natur.467.1061T. doi:10.1038/nature09534. PMC 3042601. PMID 20981092.
- ^ a b Abecasis GR, Auton A, Brooks LD, DePristo MA, Durbin RM, Handsaker RE, et al. (November 2012). "An integrated map of genetic variation from 1,092 human genomes". Nature. 491 (7422): 56–65. Bibcode:2012Natur.491...56T. doi:10.1038/nature11632. PMC 3498066. PMID 23128226.
- ^ Auton A, Brooks LD, Durbin RM, Garrison EP, Kang HM, Korbel JO, et al. (October 2015). "A global reference for human genetic variation". Nature. 526 (7571): 68–74. Bibcode:2015Natur.526...68T. doi:10.1038/nature15393. PMC 4750478. PMID 26432245.
- ^ Sudmant PH, Rausch T, Gardner EJ, Handsaker RE, Abyzov A, Huddleston J, et al. (October 2015). "An integrated map of structural variation in 2,504 human genomes". Nature. 526 (7571): 75–81. Bibcode:2015Natur.526...75.. doi:10.1038/nature15394. PMC 4617611. PMID 26432246.
- ^ "Variety of life". Nature News & Comment. 2015-09-30. Retrieved 2015-10-15.
- ^ a b c d e f g G Spencer, 국제 컨소시엄, 1000 게놈 프로젝트 발표, 엠바고드(2008) http://www.nih.gov/news/health/jan2008/nhgri-22.htm
- ^ Nielsen R (October 2010). "Genomics: In search of rare human variants". Nature. 467 (7319): 1050–1. Bibcode:2010Natur.467.1050N. doi:10.1038/4671050a. PMID 20981085.
- ^ a b JC 길이, 인간 유전자 변이:미세진화의 메커니즘과 결과, 미국 인류학회(2004)
- ^ a b Anzai T, Shiina T, Kimura N, Yanagiya K, Kohara S, Shigenari A, et al. (June 2003). "Comparative sequencing of human and chimpanzee MHC class I regions unveils insertions/deletions as the major path to genomic divergence". Proceedings of the National Academy of Sciences of the United States of America. 100 (13): 7708–13. Bibcode:2003PNAS..100.7708A. doi:10.1073/pnas.1230533100. PMC 164652. PMID 12799463.
- ^ a b Redon R, Ishikawa S, Fitch KR, Feuk L, Perry GH, Andrews TD, et al. (November 2006). "Global variation in copy number in the human genome". Nature. 444 (7118): 444–54. Bibcode:2006Natur.444..444R. doi:10.1038/nature05329. PMC 2669898. PMID 17122850.
- ^ a b c Barreiro LB, Laval G, Quach H, Patin E, Quintana-Murci L (March 2008). "Natural selection has driven population differentiation in modern humans". Nature Genetics. 40 (3): 340–5. doi:10.1038/ng.78. PMID 18246066. S2CID 205357396.
- ^ a b c Nielsen R, Hellmann I, Hubisz M, Bustamante C, Clark AG (November 2007). "Recent and ongoing selection in the human genome". Nature Reviews. Genetics. 8 (11): 857–68. doi:10.1038/nrg2187. PMC 2933187. PMID 17943193.
- ^ a b EE Harris 등인간 적응의 기초가 되는 선택의 분자 시그니처, 물리 인류학 연감 49: 89-130 (2006)
- ^ a b Bamshad M, Wooding SP (February 2003). "Signatures of natural selection in the human genome". Nature Reviews. Genetics. 4 (2): 99–111. doi:10.1038/nrg999. PMID 12560807. S2CID 13722452.
- ^ Corona E, Dudley JT, Butte AJ (August 2010). Hawks J (ed.). "Extreme evolutionary disparities seen in positive selection across seven complex diseases". PLOS ONE. 5 (8): e12236. Bibcode:2010PLoSO...512236C. doi:10.1371/journal.pone.0012236. PMC 2923198. PMID 20808933.
- ^ a b c d e f 회의 보고서: 인간 유전자 변이의 심층 카탈로그를 계획하기 위한 워크숍, (2007) http://www.1000genomes.org/sites/1000genomes.org/files/docs/1000Genomes-MeetingReport.pdf
- ^ Oleksyk TK, Brukhin V, O'Brien SJ (2015). "The Genome Russia project: closing the largest remaining omission on the world Genome map". GigaScience. 4: 53. doi:10.1186/s13742-015-0095-0. PMC 4644275. PMID 26568821.
- ^ C. 킹 (2012) 2011년 가장 핫한 리서치.Science Watch http://archive.sciencewatch.com/newsletter/2012/201203/hottest_research_2012/
- ^ 1000 게놈 프로젝트 커뮤니티 분석 회의 http://1000gconference.sph.umich.edu/
외부 링크
- 1000개의 게놈 - 인간 유전자 변이의 심층 카탈로그 - 공식 웹 페이지
- International HapMap Project - 공식 웹 페이지
- 인간 게놈 프로젝트 정보




