생물정보학에서의 머신러닝
Machine learning in bioinformatics생물정보학에서 머신러닝(machine learning)은 유전체학, 단백질학, 마이크로레이, 시스템 생물학, 진화, 텍스트 마이닝 등 [1]생물정보학에 머신러닝 알고리즘을 적용하는 것이다.[2][3]
머신러닝이 등장하기 전에는 생물정보학 알고리즘을 손으로 프로그래밍해야 했는데, 단백질 구조 예측과 같은 문제에서는 이것이 어렵다는 것이 입증되었다.[4] 딥러닝과 같은 머신러닝 기법은 프로그래머가 데이터 세트를 개별적으로 정의하도록 요구하는 대신 데이터 세트의 특징을 학습할 수 있다. 알고리즘은 낮은 수준의 특징을 좀 더 추상적인 특징으로 결합하는 방법 등을 더 자세히 배울 수 있다. 이러한 다층 접근법은 그러한 시스템이 적절한 훈련을 받았을 때 정교한 예측을 할 수 있게 한다. 이러한 방법들은 기존 데이터 집합을 이용하면서 데이터가 예상치 못한 방식으로 해석되고 분석되는 것을 허용하지 않는 다른 계산 생물학적 접근법과 대조된다. 최근 몇 년간 사용 가능한 생물학적 데이터 집합의 크기와 수는 급증했다.[2]
임무들
생물정보학에서의 머신러닝 알고리즘은 예측, 분류, 특징 선택에 사용할 수 있다. 이 과제를 달성하는 방법은 다양하고 여러 분야에 걸쳐 있다. 그중에서 가장 잘 알려진 것은 기계 학습과 통계학이다. 분류 및 예측 작업은 향후 예측을 위해 등급이나 개념을 기술하고 구분하는 모델을 구축하는 것을 목표로 한다. 이들 간의 차이는 다음과 같다.
- 분류/인식은 범주형 클래스를 출력하는 반면 예측은 숫자 값 형상을 출력한다.
- 아날로그, 규칙, 신경 네트워크, 확률 및/또는 통계를 사용하여 데이터로부터 예측 모델을 구축하는 데 사용되는 알고리즘 유형 또는 프로세스.
인공지능, 데이터 마이닝 등 정보기술과 해당 모델이 기하급수적으로 성장함에 따라 더욱 포괄적인 데이터 세트에 접근하는 것 외에도 학습 능력을 바탕으로 보다 새롭고 우수한 정보 분석 기법이 탄생했다. 그러한 모델은 설명할 수 없는 범위를 허용하고 시험 가능한 모델의 형태로 통찰력을 제공한다.
머신러닝 접근 방식
인공신경망
생물정보학에서 인공신경망은 다음을 위해 사용되어 왔다.[5]
- RNA, 단백질, DNA 염기서열 비교 및 정렬.
- 촉진자 식별 및 DNA와 관련된 시퀀스로부터 유전자를 찾는다.
- 표현식-gene 및 마이크로 어레이 데이터 해석
- 유전자의 네트워크(규제)를 확인한다.
- 계통생성 나무를 구축하여 진화적 관계를 학습한다.
- 단백질 구조 분류 및 예측.
- 분자 설계 및 도킹.
피쳐 엔지니어링
다차원 공간의 벡터인 특징들이 도메인 데이터에서 추출되는 방법은 학습 시스템의 중요한 구성 요소다.[6] 게놈학에서 수열의 대표적인 표현은 k-메르 주파수의 벡터로서, 치수 4의 벡터로서, 입력 항목은 일정한 순서에 따라 길이 의 각 수열의 모양을 계산한다. = 만큼 작은 값의 경우 이러한 벡터의 치수성은 매우 크기 때문에(예: 이 경우 치수는 4 16× 약 주성분 분석과 같은 기법을 사용하여 데이터를 더 낮은 치수 공간에 투영하므로 선택한다. 시퀀스 중에서 더 작은 형상의 [6][additional citation(s) needed]세트
분류
이러한 유형의 기계 학습 과제에서 출력은 이산형 변수다. 생물정보학에서 이러한 유형의 과제의 한 예는 이미 라벨이 붙어 있는 데이터 모델에 기초하여 새로운 게놈 데이터(예: 헤아릴 수 없는 박테리아의 게놈)에 라벨을 붙이는 것이다.[6]
히든 마르코프 모델
Hidden Markov 모델(HM)은 순차적 데이터를 위한 통계 모델의 일종이다(종종 시간이 지남에 따라 진화하는 시스템과 관련된다). An HMM is composed of two mathematical objects: an observed state‐dependent process , and an unobserved (hidden) state process . In an HMM, the state process is not directly observed – it '수치'(또는 '수치') 변수지만, 관찰은 기본 상태 프로세스에 의해 구동되는 상태 추적 프로세스(또는 관찰 프로세스)로 이루어진다(따라서 관심 있는 시스템 상태에 대한 소음이 있는 측정으로 간주될 수 있다).[7] HMM은 연속적인 시간에 공식화될 수 있다.[8][9]
HMM은 다중 시퀀스 정렬을 프로파일링하고 원격으로 동질 시퀀스를 데이터베이스 검색에 적합한 위치별 점수 시스템으로 변환하는 데 사용할 수 있다.[10] 또한 생태학적 현상은 HMMs에 의해 설명될 수 있다.[11]
콘볼루션 신경망
경련신경망(CNN)은 깊은 신경망의 한 종류로, 경련 커널이나 입력 형상을 따라 미끄러지는 필터의 공유 가중치에 기초하여 피쳐 맵이라고 하는 번역 등가 반응을 제공한다.[12][13] CNN은 데이터의 계층적 패턴을 활용하고 필터를 통해 발견된 더 작고 단순한 패턴을 사용하여 복잡성이 증가하는 패턴을 취합한다. 따라서 연결성과 복잡성의 규모에서 더 낮다.[citation needed]
콘볼루션 네트워크는 뉴런들 사이의 연결 패턴이 동물 시각 피질의 조직과 닮았다는 점에서 생물학적 과정에[14][15][16][17] 의해 영감을 받았다. 개별 피질 뉴런은 수용성 장으로 알려진 시야의 제한된 영역에서만 자극에 반응한다. 서로 다른 뉴런의 수용적인 장은 부분적으로 겹쳐서 전체 시야를 덮는다.
CNN은 다른 영상 분류 알고리즘에 비해 상대적으로 전처리를 거의 사용하지 않는다. 즉, 네트워크는 자동화된 학습을 통해 필터(또는 커널)를 최적화하는 방법을 학습하는 반면, 기존의 알고리즘에서는 이러한 필터는 수작업으로 설계된다. 이러한 사전 지식으로부터의 독립성과 형상 추출에 대한 인간의 개입은 주요한 장점이다.[citation needed]
계통성 경련 신경망
Ph-CNN(Phylogenetic convolutional neural neural neural neural neural network)은 피오란티 외 연구진이 메타게노믹스 데이터를 분류하기 위해 제안한 새로운 콘볼루션 신경 네트워크 아키텍처다.[18] 이 접근법에서 계통생성 데이터는 패트리스틱 거리(운영 분류학 단위[OTU]를 연결하는 모든 분기의 길이를 합한 값)를 부여받아 각 OTU와 그 주변은 경련 필터로 처리된다. Ph-CNN은 완전히 연결된 신경망, 무작위 포리스트 및 지원 벡터 기계와 비교했을 때 유망한 결과를 얻는다.[18][verification needed]
랜덤 포리스트
무작위 숲(RF)은 의사결정 나무의 앙상블을 구성하고 개별 나무의 평균 예측을 출력하여 분류한다.[19] 이것은 부트스트랩 집계(많은 의사결정 나무의 집합)의 수정이며 분류 또는 회귀에 사용할 수 있다.[20][21]
무작위 숲은 일반화 오류의 내부 추정치를 제공하므로 교차 검증은 불필요하다. 또한 그들은 누락된 값을 귀속시키는 데 사용할 수 있고 새로운 데이터 시각화를 가능하게 하는 대용물을 생산한다.[22]
계산적으로 임의의 숲은 자연적으로 퇴행과 (다중) 분류를 모두 처리하고, 훈련과 예측이 비교적 빠르며, 한두 개의 튜닝 매개변수에만 의존하며, 일반화 오류에 대한 추정치가 내장되어 있으며, 고차원적인 문제에 직접 사용될 수 있으며, 쉽게 구현될 수 있기 때문에 매력적이다. 병행하여 통계적으로 랜덤 포리스트는 가변 중요도 측정, 차등 등급 가중치 측정, 누락된 값 귀속, 시각화, 특이치 감지, 무감독 학습과 같은 추가 특징을 호소하고 있다.[22]
클러스터링
군집화 - 각 부분 집합의 데이터가 어떤 정의된 거리 또는 유사성 함수에 따라 가능한 한 서로 가깝고 다른 부분 집합의 데이터로부터 가능한 한 멀리 떨어지도록 데이터 집합을 분할하는 것은 통계 데이터 분석의 일반적인 기법이다.
클러스터링은 많은 데이터 기반 생물정보학 연구의 중심이며, 계층적, 중심적, 분포 기반, 밀도 기반 및 자가 조직 지도 분류에 의해 오랫동안 연구되고 고전적 기계 학습 환경에서 사용되어 온 강력한 계산 방법의 역할을 한다. 특히 클러스터링은 구조화되지 않은 데이터와 고차원 데이터를 시퀀스, 표현, 텍스트, 이미지 등의 형태로 분석하는 데 도움이 된다. 군집화는 유전자 기능, 세포 과정, 세포의 하위 유형, 유전자 조절, 대사 과정과 같은 유전학적 수준에서 생물학적 과정에 대한 통찰력을 얻는 데도 사용된다.[23]
생물정보학에서 사용되는 군집화 알고리즘
데이터 클러스터링 알고리즘은 계층적 또는 분할적일 수 있다. 계층 알고리즘은 이전에 설정된 클러스터를 사용하여 연속적인 클러스터를 찾는 반면, 파티션 알고리즘은 모든 클러스터를 한 번에 결정한다. 계층 알고리즘은 응집성(상향) 또는 분열성(상향)일 수 있다.
응집 알고리즘은 각 요소에서 별도의 클러스터로 시작하여 이들을 연속적으로 더 큰 클러스터로 병합한다. 분열 알고리즘은 전체 집합에서 시작하여 연속적으로 더 작은 클러스터로 나뉜다. 계층적 군집화는 유클리드 공간에 대한 지표를 사용하여 계산되는데, 가장 일반적으로 사용되는 것은 각 변수의 차이의 제곱을 찾고, 모든 제곱을 추가하고, 해당 합계의 제곱근을 찾아 계산한 유클리드 거리다. 계층적 군집화 알고리즘의 예로는 BACH가 있는데, 일반적으로 큰 데이터 집합이 주어지는 거의 선형 시간 복잡성으로 인해 생물정보학에서 특히 좋다.[24] 파티셔닝 알고리즘은 초기 그룹 수를 지정하고, 그룹 간에 개체를 반복적으로 재할당하여 정합화한다. 이 알고리즘은 일반적으로 모든 클러스터를 한 번에 결정한다. 대부분의 애플리케이션은 k-means 알고리즘이나 k-medoids의 두 가지 인기 있는 경험적 접근법 중 하나를 채택한다. 다른 알고리즘은 선호도 전파와 같은 초기 그룹 수가 필요하지 않다. 유전학적 설정에서 이 알고리즘은 유전자 군집(GCF)의 생합성 유전자 군집과 해당 GCF 군집 모두에 사용되어 왔다.[25]
워크플로우
일반적으로 생물학적 데이터에 머신러닝을 적용하는 워크플로는 다음 4단계를 거친다.[2]
- 캡처 및 저장을 포함한 기록. 이 단계에서는 서로 다른 정보원을 하나의 집합으로 통합할 수 있다.
- 사전 처리, 청소 및 분석 가능한 형태로의 구조 조정. 이 단계에서 수정되지 않은 데이터는 제거되거나 수정되며, 누락된 데이터는 귀속되고 관련 변수를 선택할 수 있다.
- 감독되거나 감독되지 않은 알고리즘을 사용하여 데이터 분석, 평가 알고리즘은 일반적으로 데이터의 서브셋에 대해 훈련되고 파라미터를 최적화하며 별도의 테스트 서브셋에 대해 평가된다.
- 다양한 방법을 사용하여 지식을 효과적으로 표현하여 소견의 중요성과 중요성을 평가하는 시각화 및 해석.
데이터 오류
- 중복된 데이터는 생물정보학에서 중요한 문제다. 공개적으로 이용할 수 있는 데이터는 품질이 불확실한 것일 수 있다.[26]
- 실험 중 오류.[26]
- 잘못된 해석.[26]
- 타이핑 실수.[26]
- 비표준화 방법(다중 소스에서 나오는 PDB의 3D 구조, X선 회절, 이론 모델링, 핵 자기 공명 등)이 실험에 사용된다.[26]
적용들
일반적으로 기계학습 시스템은 충분한 샘플이 주어진 특정 클래스의 요소를 인식하도록 훈련될 수 있다.[27] 예를 들어, 기계 학습 방법은 스플라이스 사이트와 같은 특정한 시각적 특징을 식별하도록 훈련될 수 있다.[28]
지원 벡터 기계는 암 유전학 연구에 광범위하게 사용되어 왔다.[29] 또한 딥러닝은 생물정보학 알고리즘에 통합되었다. 딥러닝 애플리케이션은 규제 유전체학 및 세포 영상촬영에 사용되어 왔다.[30] 그 밖에 의료영상 분류, 유전체 시퀀스 분석, 단백질 구조 분류 및 예측 등이 응용된다.[31] 딥러닝은 규제 유전체학, 변종 호출, 병원성 점수에 적용되었다.[32] 자연 언어 처리와 텍스트 마이닝은 생체 분자 구조와 기능을 예측하는 것뿐만 아니라 단백질과 단백질 상호작용, 유전자-질병 관계를 포함한 현상을 이해하는 데 도움을 주었다.[33]
정밀/개인화된 의학
자연 언어 처리 알고리즘은 환자가 사용할 수 있는 임상 정보와 유전 데이터를 결합하여 유전 질환을 앓고 있는 환자를 위한 개인화된 의학이다. 보건기금 약리유전체학 연구 네트워크와 같은 기관들은 유방암 치료법을 찾는 데 초점을 맞추고 있다.[34]
정밀 의학은 대규모 생물학적 데이터베이스에 의해 가능한 개별 유전적 가변성을 고려한다. 머신러닝은 (환자군)과 특정 치료양식 사이의 매칭 기능을 수행하기 위해 적용할 수 있다.[35]
컴퓨터 기법은 PCR을 위한 효율적인 프라이머 설계, 생물학적 이미지 분석, 단백질의 역번역(즉, 유전 코드의 퇴화로 인해 복합 결합형 문제가 발생하는 경우)과 같은 다른 문제를 해결하기 위해 사용된다.[2]
게노믹스
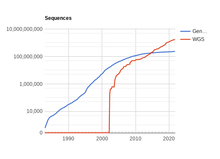
유전체 시퀀스 데이터는 DNA 조각을 염기서열화하는 기술적 어려움 때문에 역사적으로 희박했지만, 이용 가능한 시퀀스의 수는 기하급수적으로 증가하고 있다.[36] 그러나 원시 데이터가 점점 더 이용가능해지고 접근가능해지고 있는 반면, 이 데이터에 대한 생물학적 해석은 훨씬 느린 속도로 일어나고 있다.[37] 이는 주어진 DNA 순서 내에서 단백질을 암호화하는 유전자의 위치를 자동으로 결정할 수 있는 머신러닝 시스템을 포함한 계산 유전체학 도구를 개발해야 하는 필요성을 증가시킨다(즉, 유전자 예측).[37]
유전자 예측은 일반적으로 외적 검색과 내적 검색을 통해 이루어진다.[37] 외인 검색의 경우, 입력 DNA 시퀀스는 유전자가 이전에 발견되고 그 위치에 주석을 달아서 알려진 유전자 시퀀스와 일치하는 염기 문자열을 결정함으로써 대상 시퀀스의 유전자를 식별하는 대규모 데이터베이스를 통해 실행된다. 그러나 알려진 유전자 배열의 데이터베이스 크기 및 주석 처리된 유전자 배열의 한계로 인해 특정 입력 순서의 모든 유전자를 호몰로학만을 통해 식별할 수 있는 것은 아니다. 따라서 유전자 예측 프로그램이 DNA 염기서열에서만 남은 유전자를 가려내는 내적 탐색이 필요하다.[37]
기계학습은 또한 많은 DNA 또는 아미노산 염기서열을 정렬하는 문제를 수반하는 다중 염기서열 정렬 문제에도 사용되어 왔으며, 이는 공유된 진화역사를 나타낼 수 있는 유사성의 영역을 결정하기 위한 것이다.[2] 또한 게놈 재배열을 감지하고 시각화하는 데도 사용할 수 있다.[38]
프로테오믹스
아미노산의 끈인 단백질은 단백질 접힘으로부터 많은 기능을 얻는데, 1차 구조, 2차 구조(알파 나선과 베타 시트), 3차 구조, 4차 구조 등 3차원 구조로 순응한다.
단백질 2차 구조 예측은 3차 구조와 4차 구조물이 2차 구조에 기초하여 결정되기 때문에 이 하위 영역의 주요 초점이다.[4] 단백질의 진정한 구조를 해결하는 것은 비용이 많이 들고 시간이 많이 소요되기 때문에 아미노산 염기서열을 직접 분석하여 단백질의 구조를 정확하게 예측할 수 있는 시스템의 필요성이 더욱 커지고 있다.[4][2] 머신러닝에 앞서 연구자들은 이러한 예측을 수동으로 수행할 필요가 있었다. 이러한 경향은 1951년 폴링과 코리가 폴리펩타이드 체인의 단백질의 수소 결합 구성을 예측하는 연구를 발표하면서 시작되었다.[39] 자동 피쳐 학습이 82-84%[4][40]의 정확도에 도달함 현재의 2차 구조 예측의 최첨단은 인공신경망의 기계학습 모델에 의존하는 DeepCNF(딥콘볼루션 신경장)라는 시스템을 사용하여 단백질 시퀀스의 아미노산을 세 가지 구조 등급(헬릭스) 중 하나로 분류할 때 약 84%의 정확도를 달성한다., 시트 또는 코일).[40] [needs update] 3-상태 단백질 2차 구조에 대한 이론적 한계는 88-90%[4]이다.
단백질 사이드체인 예측, 단백질 루프 모델링, 단백질 접점 맵 예측 등 단백질학 문제에도 머신러닝이 적용됐다.[2]
메타게노믹스
메타게노믹스는 환경 DNA 샘플로부터 미생물 집단을 연구하는 학문이다.[41] 현재 머신러닝 툴의 구현에는 환경 샘플의 데이터 양으로 인해 한계와 과제가 우세하다.[42] 슈퍼컴퓨터와 웹서버는 이러한 도구에 더 쉽게 접근할 수 있게 했다.[43] 마이크로바이옴 데이터 집합의 높은 차원성은 마이크로바이옴을 연구하는 데 있어 중요한 과제로서, 이는 진정한 차이를 식별하기 위한 현재 접근법의 힘을 크게 제한하고 잘못된 발견의 가능성을 증가시킨다.[44]
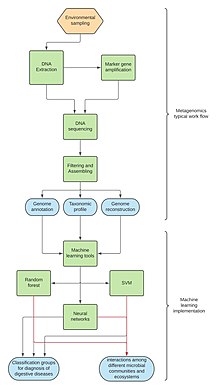
메타게노믹스와 관련된 기계학습 도구는 그 중요성에도 불구하고 내트 마이크로바이오타 연구와 염증성 장질환(IBD), 클로스트리디오이드 디피실 감염(CDI), 대장암, 당뇨병 등 소화기 질환과의 관계에 초점을 맞춰 보다 나은 진단과 치료를 모색해 왔다.[43] 최소절대수축 및 선택 연산자 분류자, 무작위 포리스트, 감독된 분류 모델, 그라데이션 부스트 등의 방법을 사용하여 시퀀스 데이터의 종류에 관계없이 호스트의 건강 상태에 따라 미생물 집단을 분류하기 위해 많은 알고리즘이 개발되었다.에드 트리 모델 재발 신경망(RNN), 경동신경망(CNN), 홉필드 신경망 등 신경망이 추가됐다.[43] 일례로 2018년 피오라반티 외에서는 Ph-CNN이라는 알고리즘을 개발하여 (건강과 병든 환자를 구분하기 위해) 족유전 트리와 경련 신경망을 이용하여 건강한 환자 및 IBD 증상이 있는 환자로부터 데이터 샘플을 분류했다.[45]
또한 무작위 포리스트(RF) 방법과 중요도 대책을 구현하면 병든 시료와 비병든 시료를 구별하는 데 사용할 수 있는 마이크로바이옴 종의 식별에 도움이 된다. 그러나 의사결정 트리의 성능과 앙상블 내 의사결정 트리의 다양성은 RF 알고리즘의 성능에 상당한 영향을 미친다. RF에 대한 일반화 오류는 개별 분류자의 정확성과 상호의존성을 측정한다. 따라서 마이크로바이옴 데이터 집합의 높은 차원성 문제는 도전을 제기한다. 효과적인 접근법은 가능한 많은 변수 조합이 필요하며, 이것은 특징의 수가 증가함에 따라 계산 부담이 기하급수적으로 증가한다.[44]
2020년 마이크로바이옴 분석을 위해 당앤키시노가[44] 참신한 분석 파이프라인을 개발했다. 송유관의 핵심은 포워딩 변수 선택(RF-FVS)과 결합된 RF 분류기로 예측 분류기 성능을 극대화하는 최소 크기의 미생물 종 핵심 세트나 기능서명을 선택한다. 프레임워크는 다음을 결합한다.
- 대규모 병렬 전방 변수 선택 절차를 통해 몇 가지 중요한 특성 식별
- 계통생성 트리에 선택된 종을 매핑하고
- 메타게놈 16S rRNA 데이터에서 기능 유전자 농축 분석을 통한 기능 프로파일 예측.
이들은 대규모 사례 제어 연구에서 발표된 두 데이터셋을 분석하여 성능을 입증했다.
- C. difficile 감염을 위한 16S rRNA 유전자 증폭기 데이터(CDI) 및
- 인간 대장암(CRC)에 대한 엽총 메타게노믹스 데이터.
제안된 접근방식은 CDI의 경우 81%에서 99.01%로, CRC의 경우 75.14%에서 90.17%로 정확도를 개선했다.
특히 WGS에서 데이터 복잡성 때문에 환경 샘플에서의 머신러닝의 사용은 덜 탐구되었다. 어떤 작품들은 이러한 도구들을 환경 샘플에 적용하는 것이 가능하다는 것을 보여준다. 2021년, Dhungel 외,[46] MegaR이라는 R 패키지를 디자인했다. 이 패키지는 16S rRNA 및 전체 메타게놈 시퀀스와의 협업을 통해 머신러닝 모델별 분류 프로파일과 분류 모델을 만들 수 있다. MegaR은 사용자 경험을 개선하기 위한 편안한 시각화 환경을 포함한다. 환경 메타게놈학에서의 머신러닝은 토양, 미생물 생물다양성, 생태계 안정성에 대한 통찰력을 제공하는 2021년의[47] 쉰 등의 연구와 같이 미생물 집단과 생태계 사이의 상호작용과 관련된 질문에 답하는 데 도움이 될 수 있다.
마이크로레이
마이크로레이는 많은 양의 생물학적 물질에 대한 데이터를 자동으로 수집하는 데 사용된다. 머신러닝은 분석에 도움이 될 수 있으며, 표현 패턴 식별, 분류, 유전적 네트워크 유도에 적용되어 왔다.[2]
이 기술은 특히 유전자 발현을 모니터링하는 데 유용하며, 어떤 유전자가 발현되는지 검사하여 암을 진단하는 데 도움을 준다.[48] 주요 과제 중 하나는 수집된 데이터를 바탕으로 어떤 유전자가 표현되는지를 파악하는 것이다.[2] 게다가 마이크로 어레이에 의해 데이터가 수집되는 유전자의 수가 엄청나게 많기 때문에, 많은 양의 관련 없는 데이터를 표현된 유전자 식별이라는 과제에 포함시키는 것은 어려운 일이다. 머신러닝은 이러한 식별을 수행하기 위해 다양한 분류 방법을 사용할 수 있기 때문에 잠재적인 해결책을 제시한다. 가장 일반적으로 사용되는 방법은 방사상 기반 기능 네트워크, 딥러닝, 베이시안 분류, 의사결정 트리, 랜덤 포레스트 등이다.[48]
시스템 생물학
시스템 생물학은 시스템에서 단순한 생물학적 구성요소의 복잡한 상호작용에서 발생하는 새로운 행동에 대한 연구에 초점을 맞춘다. 그러한 성분은 DNA, RNA, 단백질, 대사물을 포함할 수 있다.[49]
기계 학습은 유전 네트워크, 신호 전달 네트워크 및 대사 경로와 같은 영역에서 이러한 상호작용을 모델링하는 데 도움을 주기 위해 사용되어 왔다.[2] 서로 다른 변수들 간의 관계를 결정하기 위한 기계 학습 기법인 확률론적 그래픽 모델은 유전적 네트워크를 모델링하는 데 가장 흔히 사용되는 방법 중 하나이다.[2] 또한 기계학습은 마르코프 체인 최적화를 이용하여 전사 계수 결합 사이트를 식별하는 등 시스템 생물학 문제에도 적용되었다.[2] 유전 알고리즘, 즉 진화의 자연적 과정에 기초한 기계 학습 기법은 유전적 네트워크와 규제 구조를 모형화하는 데 사용되어 왔다.[2]
머신러닝의 다른 시스템 생물학 애플리케이션으로는 효소 함수 예측, 고처리량 마이크로 어레이 데이터 분석, 질병 표지를 더 잘 이해하기 위한 게놈 전체 연관 연구 분석, 단백질 함수 예측 등이 있다.[50]
진화
이 영역, 특히 계통생성 트리 재구성은 기계 학습 기법의 특징을 이용한다. 계통생식 나무는 유기체의 진화를 개략적으로 나타낸 것이다. 처음에는 형태학적, 신진대사적 특징과 같은 특징을 사용하여 구성되었다. 이후 게놈 서열의 이용가능성 때문에 계통생성 트리 알고리즘의 구축은 게놈 비교에 기초한 개념을 사용했다. 최적화 기법의 도움으로 다중 시퀀스 정렬을 통해 비교가 수행되었다.[51]
뇌졸중 진단
신경영상 데이터 분석을 위한 머신러닝 방법을 사용하여 뇌졸중을 진단한다. 역사적으로 이 문제에 대한 다중 접근법은 신경망을 포함한다.[52][53]
스트로크를 감지하는 다중 접근법은 기계 학습을 사용했다. 미르트스크훌라바가 제안한 대로, 피드-포워드 네트워크는 신경영상을 이용한 스트로크를 검출하기 위해 시험되었다.[54] 타이타노[55] 3D-CNN 기법은 급성 신경학적 사건에 대한 머리 CT 영상을 선별하기 위해 감독된 분류로 테스트되었다. 3차원 CNN과 SVM 방식이 자주 사용된다.[53]
텍스트 마이닝
생물학적 출판물의 증가로 주어진 주제에 대해 이용 가능한 관련 정보를 검색하고 편집하는 데 어려움이 커졌다. 이 작업을 지식 추출이라고 한다. 생물학적 데이터 수집은 새로운 생물학적 지식을 생성하기 위해 머신러닝 알고리즘으로 공급될 수 있는 것이 필요하다.[2][56] 머신러닝(machine learning)은 자연어 처리와 같은 기법을 사용하여 데이터베이스에 있는 사람이 작성한 보고서로부터 유용한 정보를 추출할 수 있다. 임상 서술 노트에서 특징을 추출할 수 있는 머신러닝(machine learning)의 대안적 접근법인 '텍스트 네일링(text Nailing)'이 2017년 도입됐다.
이 기법은 생물학적 데이터베이스와 저널에 저장된 정보의 검사가 필요하기 때문에 새로운 약물 표적을 찾는 데 적용되었다.[56] 단백질 데이터베이스에 있는 단백질의 주석들은 종종 각 단백질에 대해 알려진 완전한 지식 집합을 반영하지 못하므로, 추가적인 정보는 생물 의학 문헌에서 추출해야 한다. 기계학습은 유전자와 단백질 기능의 자동 주석, 단백질 아세포 국산화 결정, DNA-표현 배열 분석, 대규모 단백질 상호작용 분석, 분자 상호작용 분석 등에 적용됐다.[56]
텍스트 마이닝의 또 다른 적용 분야는 충분한 참조 데이터가 주어진 구별되는 DNA 영역의 검출과 시각화다.[57]
BGC의 군집화 및 풍부성 프로파일링
미생물 집단은 다양한 미생물의 복잡한 집합체로서 [58]공생 파트너들이 1차 및 2차(전문) 신진대사로부터 파생된 다양한 대사물을 끊임없이 생산하며, 그 결과 신진대사가 미생물 상호작용에 중요한 역할을 한다.[59] 메타게놈과 메타트랜스펙터콤 데이터는 통신신호 해독에 중요한 원천이다.
분자 메커니즘은 다양한 방법으로 전문화된 대사물을 생산한다. Biosynthetic Gene Clusters (BGCs) attract attention, since several metabolites are clinically valuable, anti-microbial, anti-fungal, anti-parasitic, anti-tumor and immunosuppressive agents produced by the modular action of multi-enzymatic, multi-domains gene clusters, such as Nonribosomal peptide synthetases (NRPSs) and polyketide synthases (PKSs)다양한 연구에[61][62][63][64][65][66][67][68] 따르면 동질성 핵심 유전자를 유전자 군집성분군(GCF)으로 공유하는 BGC를 유전자 군집성분군(Gene cluster family, GCFs)으로 분류하면 분석된 변종의 화학적 다양성에 대한 유용한 통찰력을 얻을 수 있으며, BGC와 그들의 2차 대사물과의 연계를 지원할 수 있다.[60][62][64] GCF는 인간의 건강 연구에서 기능적 지표로 사용되어 왔고 곰팡이 병원균을 억제하는 토양의 능력을 연구하기 위해 사용되었다.[71] 촉매 효소 및 그 암호화된 경로에서 생성된 화합물과의 직접적인 관계를 고려할 때, BGC/GCFs는 미생물 2차 대사의 화학적 공간을 탐구하는 대용물 역할을 할 수 있다. GCF를 시퀀싱된 미생물 게놈으로 분류하면 기존의 화학적 다양성에 대한 개요를 얻을 수 있으며 향후 우선순위에 대한 통찰력을 얻을 수 있다.[61][63] BiG-SLiCE, BIG-MAP과[72] 같은 툴은 자연환경에서 BGC의 중요성을 밝히기 위한 유일한 목적으로 등장했다.
비지-SLCE
BiG-SLCE(BiG-Synthetic Genes Super-Linear Clustering Engine)는 방대한 수의 BGC를 클러스터링하도록 설계된 자동화된 파이프라인 툴이다. BiG-SLiCE는 이들을 유클리드 공간에서 대표함으로써 다양한 유기체의 게놈 데이터와 메타게놈 데이터로부터 BGC를 비파상적이고 선형에 가까운 방식으로 GCF로 그룹화할 수 있다.[73]
그 BiG-SLiCE 워크 플로 벡터화(특징 추출)에서 제공한 클러스터 GenBank파일의 데이터 세트에서 MIBiGantiSMASH에서 숫자로 나타낸 형상의 벡터 히트 곡 absence/presence과 bitscores 도서관 프로필 Hidden마르코프 Model[74](의 엄선된에서 주정 단장 유전자 서열 조회 조사 내용에 입력 BGCs으로 시작한다.pHMMs) BGC의 생합성 영역의 그런 다음 이러한 형상은 BACH 클러스터링에 기반한 초선형 클러스터링 알고리즘에 의해 처리되어 [24]GCF 모델을 나타내는 중심 형상 벡터가 된다. 데이터 집합의 모든 BGC는 각 BGC에 대한 GCF 멤버십 값 목록을 출력하여 해당 모델에 대해 쿼리된다. 그런 다음 글로벌 클러스터 매핑은 k-평균을 사용하여 GCF 빈의 모든 GCF 중심 기능을 그룹화한다. 그 후에 BGC 기능의 전체 집합을 결과 GCF 빈에 일치시키기 위해 또 다른 멤버십 할당이 수행된다. 결국, 그것은 아카이브를 생산하고, 그 다음 (외부 스크립트를 통해) 추가적인 분석을 수행하거나 사용자 상호 작용 애플리케이션에서 결과를 시각화하는 데 사용할 수 있다.
사트리아 외 연구진은 120만 개의 생물합성 유전자 군집의 미지의 생합성 잠재력을 식별하기 위해 분류학 전반에 걸친 2차 대사 다양성의 글로벌 지도를 재구성하여 그러한 분석의 효용성을 입증했다.[73] 이것은 천연물 발견을 가속화할 수 있는 새로운 가능성을 열어주고, BGC의 전세계적이고 검색 가능한 상호연결 네트워크를 구축하기 위한 첫 단계를 제공한다. 더 많은 게놈들이 연구되지 않은 세금에서 서열화됨에 따라, 그들의 잠재적으로 새로운 화학작용을 강조하기 위해 더 많은 정보를 캐낼 수 있다.[73]
BiG-MAP
BGC는 대사물 생산의 중요한 원천이기 때문에, BGC를 식별하기 위한 현재의 도구는 게놈의 풍부함과 표현 수준에 대한 관련 정보를 무시한 채 게놈의 채굴에 초점을 맞추고 있으며, 실제로 표현형 의존 메타볼라이트 동심원을 유발하는 데 중요한 생태학적 역할을 한다.그것이 바로 2020년 BGC의 풍부함(메타게놈 데이터)과 표현(메타크릭 데이터)을 미생물 집단에 걸쳐 결정하는 데 도움을 주는 자동화된 파이프라인인 [72]생물합성 유전자 군집 메타오믹스 풍부함 프로파일러(BiG-MAP, BiG-MAP)가 그것이다. 그것은 antiSMASH나 gutSMASH에 의해 예측된 유전자 군집들에 대한 산탄총 염기서열 판독을 한다.
BiG-MAP는 4개의 주요 모듈로 워크플로우를 분할한다.
- BiG-MAP. 패밀리: 계산 시간을 줄이고 모호한 매핑을 방지하기 위해 유전자 클러스터 컬렉션의 중복 필터링. MASH-MAP는 MinHash 기반 알고리즘을 사용하여 단백질 시퀀스 사이의 거리를 추정하며,[76] 이는 k-medoids 클러스터링을 이용하여 대표적인 유전자 클러스터를 선택하는 데 사용된다.[75] 마지막으로 선택한 유전자 군집은 BiG-SCAPE를 사용하여 GCF로 군집화되며,[61] 이는 구조 유사성을 고려함으로써 서로 다른 유기체에서 동일한 화학 제품을 생산하는 더 먼 관련 유전자 군집과 관련이 있다.
- BiG-MAP.download: SRA(Sequence Read Archive) 데이터베이스 목록을 사용하는 선택적 모듈
- BiG-MAP.map: 첫 번째 모듈에서 얻은 대표적인 GCF 집합을 읽는다. 지도는 GCF에 별도로 표시되며, 가족당 결합된 풍부함 또는 표현 수준을 보고한다. 읽기는 짧은 판독기 얼라이너 Bowtie2를 사용하여 GCF의 대표자에게 매핑되며,[77] 이는 이후 RPKM(Rades Per Kilobase Million)으로 변환되어 GCF 크기에 걸쳐 평균화된다.
- BiG-MAP.analyse: 프로파일링 풍부함. RPKM 값은 간격의 고려를 위해 누적 합계 스케일링[78](CSS)을 사용하여 정규화된다. 차등식 분석은 제로 인플레이션 가우스 분포 혼합물 모델(ZIGH-models) 또는 크러스칼-월리스(Kruskal-Wallis)를 사용한다. 파이프라인에는 유전자 군집(hatmaps), 로그 접힘 변화(bar fold change, 막대 그림), 적용 범위 값 및 전위적 데이터(heatmap)에 대한 하우스키핑 유전자(hatmap) 표현 값을 보여주는 결과 그래프가 표시된다.
RiPPs 화학구조물의 해독
BAGEL, BTIBASE, MIBIG, THIOBASE와 같은 데이터베이스에서 선택한 순서와 화학 구조에 대한 정보의 가용성과 함께 실험적으로 특징지어지는 리보솜 합성 및 후번역 변형 펩타이드(RIPP)의 증가는 디코딩을 위한 기계 학습 도구를 개발할 수 있는 기회를 제공한다.화학적 구조와 분류.
2017년 인도 뉴델리 국립면역학연구소 연구진이 유전자 채굴에 의한 RiPP 화학구조 해독을 위한 생물정보학 자원인 RiPPminer 소프트웨어를 개발했다. RiPPminer 웹 서버는 쿼리 인터페이스와 RiPPDB 데이터베이스로 구성된다. RiPPminer는 리더 펩타이드의 갈라짐 현장과 RiPP 화학 구조의 최종 교차 링크를 예측하는 12개의 RiPP 하위 등급을 정의한다.
RIPP 식별 및 RIPP 등급 예측
AntiSMASH와 RiPP-PRISM과 같은 RiPP 분석 도구는 RiPP의 생체합성 유전자 클러스터에 존재하는 효소를 수정하는 HMM을[74] 사용하여 RiPP 하위 등급을 예측한다. 이러한 도구와 달리 RiPPminer는 513 RiPP로 훈련된 머신러닝 모델을 사용하여 RiPP 유전자의 아미노산 시퀀스를 고유하게 사용하여 식별하고 하위 분류한다. RIPPminer는 293년에 훈련된 서포트 벡터 머신 모델을 사용하여 RIPP를 다른 단백질과 펩타이드와 구별하여 실험적으로 특징지은 RIPPs를 양의 데이터 세트로, 8140개의 게놈으로 부호화 비 RIPPs 폴리펩타이드들을 음의 데이터 세트로 분류한다. 음성 데이터 세트에는 리보솜 단백질, 매트릭스 단백질, 사이토크롬 B 단백질 등 리보솜과 유사한 길이의 스위스프로트 항목이 포함되었다. 서포트 벡터는 아미노산 구성과 디펩티드 주파수로 구성된다.
2중 교차 검증 접근법을 사용한 독립적인 데이터 집합(훈련에 포함되지 않음)을 벤치마킹한 결과 각각 0.93, 0.90, 0.90, 0.90, 0.85의 민감도, 특수도, 정밀도 및 MCC 값은 각각 0.93, 0.90, 0.85로 나타났다. 이것은 RIPP와 비 RIPP를 구별하기 위한 모델의 예측 능력을 나타낸다. RiPP 클래스 또는 하위 클래스의 예측을 위해, 아미노산 구성과 디펩티드 주파수를 특징 벡터로 사용하여 Multi-Class SVM을 훈련했다. Multi-Class SVM 훈련 중, 주어진 클래스에 속하는 가용 RiPP 전구 시퀀스(예: 라소 펩타이드)가 양의 집합으로 사용되었고, 다른 모든 클래스에 속하는 RiPP는 음의 집합으로 사용되었다.
갈라진 부위의 예측
RiPPDB에서 50개 이상의 RiPP를 실험적으로 특징지었던 4대 RiPP 클래스 중, 쇄골 부위 예측을 위한 SVM 모델이 란티펩타이드, 시아노박틴, 라소펩타이드에 대해 개발되었다. 란티펩타이드의 갈라짐 부위 예측을 위한 SVM을 개발하기 위해, 알려진 갈라짐 패턴이 있는 115개의 란티펩타이드 전구체 시퀀스 세트에서 12개의 메르 펩타이드 시퀀스를 추출했다. 그 결과 103개의 고유한 12개의 메르펩타이드의 양성 데이터 집합이 골절 부위가 중심부에 있는 반면, 나머지 12개는 음의 데이터 집합을 구성했다. 각각의 메르스 특징 벡터는 20개의 아미노산 각각에 해당하는 20차원 벡터의 결합으로 구성되었다. 갈라진 부위를 예측하기 위한 SVM 모델을 개발하여 2배 교차 검증을 사용하여 벤치마킹했으며, 데이터의 절반은 교육에, 나머지 절반은 테스트에 사용하였다. SVM 모델은 시아노박틴과 라소펩타이드의 갈라진 부위의 예측을 위해 개발되었다. ROC 곡선의 분석에 기초하여 란티펩타이드와 라소펩타이드의 갈라진 부위 예측에 적합한 점수 컷오프를 선택했다.
교차 링크 예측
RiPP의 교차연계 예측과 완전한 화학구조 해석 알고리즘은 란티펩타이드, 라소펩타이드, 시아노박틴, 티오펩타이드에 대해 구현되었다. 란티펩타이드 내 란티오닌 연계 예측은 머신러닝을 이용해 진행했다. 화학 구조가 알려진 93개의 란티펩타이드 데이터 세트는 RiPPDB에서 가져갔다. 이 세트의 각 란티펩타이드에 대해, 코어 펩타이드의 시퀀스에 Ser/Thr-(X)n-Cys 또는 Cys-(X)n-Ser/Thr 형식의 문자열 또는 하위 시퀀스를 스캔하여 이론적으로 가능한 모든 사이클화 패턴을 열거했다. 이러한 시퀀스 문자열 중 란시오닌 브리지로 연결된 Ser/Thr-Cys 또는 Cys-Ser/Thr 쌍에 해당하는 문자열은 양수 집합에 포함되었고, 다른 문자열은 모두 음수 집합에 포함되었다.
질량 스펙트럼 유사도 점수
라이브러리 매칭과 분자 네트워킹과 같은 많은 탠덤 질량 분광학(MS/MS) 기반 대사물학 연구는 구조 유사성의 대용품으로 스펙트럼 유사성을 사용한다. Spec2vec[80] 알고리즘은 Word2Vec에 기초한 스펙트럼 유사성 점수의 새로운 방법을 제공한다. Spec2Vec은 분자 간의 스펙트럼 유사성을 평가하고 이러한 비교를 통해 알려지지 않은 분자를 분류하기 위해 대규모 스펙트럼 데이터 집합 내에서 파괴적인 관계를 학습한다.
전신 주석의 경우 일부 대사물학 연구는 측정된 단편화 질량 스펙트럼을 라이브러리 스펙트럼에 적합시키거나 네트워크 분석을 통해 대조되는 스펙트럼에 의존한다. 스코어링 함수는 이러한 프로세스의 일부로서 조각 스펙트럼 쌍 사이의 유사성을 결정하는 데 사용된다. 지금까지, 어떤 연구도 일반적으로 이용되는 코사인 기반 유사성과 현저하게 다른 점수를 제시하지 못했다.[81]
데이터베이스
생물정보학의 중요한 부분은 참조 데이터베이스라고 알려진 큰 데이터셋의 관리다. 데이터베이스는 생물합성 유전자 군집과 메타게놈과 같은 생물학적 데이터의 각 유형에 대해 존재한다.
생물정보학을 이용한 일반 데이터베이스
국립생명공학정보센터
국립생명공학정보센터(NCBI)[82]는 GenBank 핵산 염기서열 데이터베이스와 PubMed 데이터베이스 등 생물학적 정보와 데이터를 위한 대규모 온라인 자원군을 제공한다. 많은 웹 애플리케이션을 강화하는 것은 전문화된 데이터 세트를 검색하기 위해 최적화된 블라스트 프로그램의 맞춤형 구현이다. 리소스에는 PubMed 데이터 관리, RefSeq 기능 요소, 게놈 데이터 다운로드, 변형 서비스 API, Magic-BLAST, QuickB가 포함된다.LASTp 및 동일한 단백질 그룹. 이 모든 자원은 NCBI를 통해 접근할 수 있다.[83]
생합성 유전자 군집을 위한 생물정보학 분석
안티SMASH
AntiSMASH는 박테리아 및 곰팡이 유전체에서 2차 대사물 생합성 유전자 군집을 신속하게 식별, 주석 및 분석할 수 있다. 그것은 많은 수의 실리카 2차 대사물 분석 도구와 통합되고 교차 링크된다.[84]
gutSMASH
gutSMASH는 내장 마이크로바이옴으로부터 알려진 것과 새로운 혐기성 대사유전자 클러스터(MGCs)를 모두 예측하여 박테리아 대사 잠재력을 체계적으로 평가하는 도구다.
MIBiG
MIBiG는 [85]생합성 유전자 군집 사양에 대한 최소 정보로서 생합성 유전자 군집과 그 분자 생산물에 대한 주석과 메타데이터에 대한 표준을 제공한다. MIBiG는 모든 시퀀스(MIxS) 프레임워크에 대한 최소 정보를 기반으로 구축되는 Genomic Standards Consortium 프로젝트다.[86]
MIBiG는 생합성 유전자 군집 데이터의 표준화된 축적과 검색을 촉진하고 종합적인 비교 분석 도구를 개발한다. 강력한 실험 근거와 풍부한 메타데이터 구성요소에 의해 사회적으로 관련이 있는 광범위한 생물 활성 2차 대사물의 생합성, 화학 및 생태에 대한 차세대 연구를 가능하게 한다.[87]
실바
SIVA는[88] 작은(16S,18S, SSU)과 큰(23S, 28S, LSU) 부유닛 둘 다, 박테리아, 고고학, 진카리아 영역에 속하는 RNA 리보솜(RNA, RNA) 시퀀스의 완전한 데이터베이스를 조립하는 생물학자와 컴퓨터 과학자들 사이의 학제간 프로젝트다. 이 자료들은 학술적, 상업적으로 자유롭게 이용할 수 있다.[89]
그렝게네스
그렝게네스는[90] 키메라 스크리닝, 표준 정렬, 그리고 de novo tree 추론에 기초한 커리큘럼 분류법을 제공하는 16S rRNA 유전자 데이터베이스다.[91][92] 개요:
- 92,684개의 유기체에서 나온 1,012,863개의 RNA 염기서열은 RNA중심에 기여했다.
- 최단 시퀀스는 뉴클레오티드가 1,253개로 가장 긴 2,368개다.
- 평균 길이는 1,402 뉴클레오티드다.
- 데이터베이스 버전: 13.5.
열린 생명의 나무 분류법
오픈 트리 오브 라이프 택사노미(OTT)[93]는 출판된 계통생성 나무를 분류 데이터와 함께 합성해 완전하고 역동적이며 디지털로 이용 가능한 생명의 트리(Tree of Life)를 구축하는 것을 목표로 한다. 계통생식 나무는 분류, 정렬, 합병되었다. 분류법은 유전자가 남긴 희박한 지역과 공백을 메우기 위해 사용되어 왔다. OTT는 그동안 16S 영역의 시퀀스 분석에는 거의 사용되지 않았던 기반이지만, SIVA나 그렝게네스에 비해 분류학적으로 속 수준으로 분류된 시퀀스 수가 더 많다. 단, 가장자리 수준의 분류에 있어서는, 적은 양의 정보를 포함한다.
리보솜 데이터베이스 프로젝트
RDP(Ribosomal Database Project)[95]는 영역 박테리아와 고고학(16S)의 작은 서브유닛의 RRNA(RNA) 시퀀스와 대형 서브유닛(28S)의 곰팡이 rRNA 시퀀스를 제공하는 데이터베이스다.[96]
참조
- ^ Chicco D (December 2017). "Ten quick tips for machine learning in computational biology". BioData Mining. 10 (35): 35. doi:10.1186/s13040-017-0155-3. PMC 5721660. PMID 29234465.
- ^ a b c d e f g h i j k l m n Larrañaga P, Calvo B, Santana R, Bielza C, Galdiano J, Inza I, et al. (March 2006). "Machine learning in bioinformatics". Briefings in Bioinformatics. 7 (1): 86–112. doi:10.1093/bib/bbk007. PMID 16761367.
- ^ Pérez-Wohlfeil E, Torrenoa O, Bellis LJ, Fernandes PL, Leskosek B, Trellesa O (December 2018). "Training bioinformaticians in High Performance Computing". Heliyon. 4 (12): e01057. doi:10.1016/j.heliyon.2018.e01057. PMC 6299036. PMID 30582061.
- ^ a b c d e Yang Y, Gao J, Wang J, Heffernan R, Hanson J, Paliwal K, Zhou Y (May 2018). "Sixty-five years of the long march in protein secondary structure prediction: the final stretch?". Briefings in Bioinformatics. 19 (3): 482–494. doi:10.1093/bib/bbw129. PMC 5952956. PMID 28040746.
- ^ Shastry KA, Sanjay HA (2020). "Machine Learning for Bioinformatics". In Srinivasa K, Siddesh G, Manisekhar S (eds.). Statistical Modelling and Machine Learning Principles for Bioinformatics Techniques, Tools, and Applications. Algorithms for Intelligent Systems. Singapore: Springer. pp. 25–39. doi:10.1007/978-981-15-2445-5_3. ISBN 978-981-15-2445-5. S2CID 214350490. Retrieved June 28, 2021.
- ^ a b c Soueidan H, Nikolski M (2019). "Machine learning for metagenomics: methods and tools" (PDF). Metagenomics. 1. arXiv:1510.06621. doi:10.1515/metgen-2016-0001. S2CID 17418188.
- ^ Rabiner L, Juang B (January 1986). "An introduction to hidden Markov models". IEEE ASSP Magazine. 3 (1): 4–16. doi:10.1109/MASSP.1986.1165342. ISSN 1558-1284. S2CID 11358505.
- ^ Jackson CH, Sharples LD, Thompson SG, Duffy SW, Couto E (July 2003). "Multistate Markov models for disease progression with classification error". Journal of the Royal Statistical Society, Series D (The Statistician). 52 (2): 193–209. doi:10.1111/1467-9884.00351.
- ^ Amoros R, King R, Toyoda H, Kumada T, Johnson PJ, Bird TG (May 30, 2019). "A continuous-time hidden Markov model for cancer surveillance using serum biomarkers with application to hepatocellular carcinoma". Metron. 77 (2): 67–86. doi:10.1007/s40300-019-00151-8. PMC 6820468. PMID 31708595.
- ^ Eddy SR (October 1, 1998). "Profile hidden Markov models". Bioinformatics. 14 (9): 755–63. doi:10.1093/bioinformatics/14.9.755. PMID 9918945.
- ^ McClintock BT, Langrock R, Gimenez O, Cam E, Borchers DL, Glennie R, Patterson TA (December 2020). "Uncovering ecological state dynamics with hidden Markov models". Ecology Letters. 23 (12): 1878–1903. arXiv:2002.10497. doi:10.1111/ele.13610. PMC 7702077. PMID 33073921.
- ^ Zhang W (1988). "Shift-invariant pattern recognition neural network and its optical architecture". Proceedings of Annual Conference of the Japan Society of Applied Physics.
- ^ Zhang W, Itoh K, Tanida J, Ichioka Y (November 1990). "Parallel distributed processing model with local space-invariant interconnections and its optical architecture". Applied Optics. 29 (32): 4790–7. Bibcode:1990ApOpt..29.4790Z. doi:10.1364/AO.29.004790. PMID 20577468.
- ^ Fukushima K (2007). "Neocognitron". Scholarpedia. 2 (1): 1717. Bibcode:2007SchpJ...2.1717F. doi:10.4249/scholarpedia.1717.
- ^ Hubel DH, Wiesel TN (March 1968). "Receptive fields and functional architecture of monkey striate cortex". The Journal of Physiology. 195 (1): 215–43. doi:10.1113/jphysiol.1968.sp008455. PMC 1557912. PMID 4966457.
- ^ Fukushima K (1980). "Neocognitron: a self organizing neural network model for a mechanism of pattern recognition unaffected by shift in position". Biological Cybernetics. 36 (4): 193–202. doi:10.1007/BF00344251. PMID 7370364. S2CID 206775608.
- ^ Matsugu M, Mori K, Mitari Y, Kaneda Y (2003). "Subject independent facial expression recognition with robust face detection using a convolutional neural network". Neural Networks. 16 (5–6): 555–9. doi:10.1016/S0893-6080(03)00115-1. PMID 12850007.
- ^ a b Fioravanti D, Giarratano Y, Maggio V, Agostinelli C, Chierici M, Jurman G, Furlanello C (March 2018). "Phylogenetic convolutional neural networks in metagenomics". BMC Bioinformatics. 19 (Suppl 2): 49. doi:10.1186/s12859-018-2033-5. PMC 5850953. PMID 29536822.
- ^ Ho TK (1995). Random Decision Forests. Proceedings of the 3rd International Conference on Document Analysis and Recognition, Montreal, QC, 14–16 August 1995. pp. 278–282.
- ^ Dietterich T (2000). An Experimental Comparison of Three Methodsfor Constructing Ensembles of Decision Trees:Bagging, Boosting, and Randomization. Kluwer Academic Publishers. pp. 139–157.
- ^ Breiman L (2001). Random Forest (45 ed.). Machine Learning: Kluwer Academic Publisers. pp. 5–32.
- ^ a b Zhang C, Ma Y (2012). Ensemble machine learning: methods and applications. New York: Springer New York Dordrecht Heidelberg London. pp. 157–175. ISBN 978-1-4419-9325-0.
- ^ Karim MR, Beyan O, Zappa A, Costa IG, Rebholz-Schuhmann D, Cochez M, Decker S (January 2021). "Deep learning-based clustering approaches for bioinformatics". Briefings in Bioinformatics. 22 (1): 393–415. doi:10.1093/bib/bbz170. PMC 7820885. PMID 32008043.
- ^ a b Lorbeer B, Kosareva A, Deva B, Softić D, Ruppel P, Küpper A (March 1, 2018). "Variations on the Clustering Algorithm BIRCH". Big Data Research. 11: 44–53. doi:10.1016/j.bdr.2017.09.002.
- ^ Navarro-Muñoz JC, Selem-Mojica N, Mullowney MW, Kautsar SA, Tryon JH, Parkinson EI, et al. (January 2020). "A computational framework to explore large-scale biosynthetic diversity". Nature Chemical Biology. 16 (1): 60–68. doi:10.1038/s41589-019-0400-9. PMC 6917865. PMID 31768033.
- ^ a b c d e Shastry KA, Sanjay HA (2020). "Machine Learning for Bioinformatics". Statistical Modelling and Machine Learning Principles for Bioinformatics Techniques, Tools, and Applications. Algorithms for Intelligent Systems. Springer Singapore. pp. 25–39. doi:10.1007/978-981-15-2445-5_3. ISBN 978-981-15-2444-8. S2CID 214350490.
- ^ Libbrecht MW, Noble WS (June 2015). "Machine learning applications in genetics and genomics". Nature Reviews. Genetics. 16 (6): 321–32. doi:10.1038/nrg3920. PMC 5204302. PMID 25948244.
- ^ Degroeve S, De Baets B, Van de Peer Y, Rouzé P (2002). "Feature subset selection for splice site prediction". Bioinformatics. 18 Suppl 2: S75-83. doi:10.1093/bioinformatics/18.suppl_2.s75. PMID 12385987.
- ^ Huang S, Cai N, Pacheco PP, Narrandes S, Wang Y, Xu W (January 2018). "Applications of Support Vector Machine (SVM) Learning in Cancer Genomics". Cancer Genomics & Proteomics. 15 (1): 41–51. doi:10.21873/cgp.20063. PMC 5822181. PMID 29275361.
- ^ Angermueller C, Pärnamaa T, Parts L, Stegle O (July 2016). "Deep learning for computational biology". Molecular Systems Biology. 12 (7): 878. doi:10.15252/msb.20156651. PMC 4965871. PMID 27474269.
- ^ Cao C, Liu F, Tan H, Song D, Shu W, Li W, et al. (February 2018). "Deep Learning and Its Applications in Biomedicine". Genomics, Proteomics & Bioinformatics. 16 (1): 17–32. doi:10.1016/j.gpb.2017.07.003. PMC 6000200. PMID 29522900.
- ^ Zou J, Huss M, Abid A, Mohammadi P, Torkamani A, Telenti A (January 2019). "A primer on deep learning in genomics". Nature Genetics. 51 (1): 12–18. doi:10.1038/s41588-018-0295-5. PMID 30478442. S2CID 205572042.
- ^ Zeng Z, Shi H, Wu Y, Hong Z (2015). "Survey of Natural Language Processing Techniques in Bioinformatics". Computational and Mathematical Methods in Medicine. 2015 (D1): 674296. doi:10.1155/2015/674296. PMC 4615216. PMID 26525745.
- ^ Zeng Z, Shi H, Wu Y, Hong Z (2012). "Survey of Natural Language Processing Techniques in Bioinformatics". Computational and Mathematical Methods in Medicine. 2015 (D1): 674296. doi:10.1016/B978-0-12-385467-4.00006-3. PMC 4615216. PMID 26525745.
- ^ Zeng Z, Shi H, Wu Y, Hong Z (2017). "Survey of Natural Language Processing Techniques in Bioinformatics". Computational and Mathematical Methods in Medicine. 2015 (D1): 674296. doi:10.1155/2015/674296. PMC 4615216. PMID 26525745.
- ^ "GenBank and WGS Statistics". www.ncbi.nlm.nih.gov. Retrieved May 6, 2017.
- ^ a b c d Mathé C, Sagot MF, Schiex T, Rouzé P (October 2002). "Current methods of gene prediction, their strengths and weaknesses". Nucleic Acids Research. 30 (19): 4103–17. doi:10.1093/nar/gkf543. PMC 140543. PMID 12364589.
- ^ Pratas D, Silva RM, Pinho AJ, Ferreira PJ (May 2015). "An alignment-free method to find and visualise rearrangements between pairs of DNA sequences". Scientific Reports. 5 (10203): 10203. Bibcode:2015NatSR...510203P. doi:10.1038/srep10203. PMC 4434998. PMID 25984837.
- ^ Pauling L, Corey RB, Branson HR (April 1951). "The structure of proteins; two hydrogen-bonded helical configurations of the polypeptide chain". Proceedings of the National Academy of Sciences of the United States of America. 37 (4): 205–11. Bibcode:1951PNAS...37..205P. doi:10.1073/pnas.37.4.205. PMC 1063337. PMID 14816373.
- ^ a b Wang S, Peng J, Ma J, Xu J (January 2016). "Protein Secondary Structure Prediction Using Deep Convolutional Neural Fields". Scientific Reports. 6: 18962. arXiv:1512.00843. Bibcode:2016NatSR...618962W. doi:10.1038/srep18962. PMC 4707437. PMID 26752681.
- ^ Riesenfeld CS, Schloss PD, Handelsman J (2004). "Metagenomics: genomic analysis of microbial communities". Annual Review of Genetics. 38 (1): 525–52. doi:10.1146/annurev.genet.38.072902.091216. PMID 15568985.
- ^ Soueidan H, Nikolski M (March 8, 2016). "Machine learning for metagenomics: methods and tools". arXiv:1510.06621 [q-bio.GN].
- ^ a b c Lin Y, Wang G, Yu J, Sung JJ (April 2021). "Artificial intelligence and metagenomics in intestinal diseases". Journal of Gastroenterology and Hepatology. 36 (4): 841–847. doi:10.1111/jgh.15501. PMID 33880764. S2CID 233312307.
- ^ a b c Dang T, Kishino H (January 2020). "Detecting significant components of microbiomes by random forest with forward variable selection and phylogenetics". bioRxiv 10.1101/2020.10.29.361360.
- ^ Fioravanti D, Giarratano Y, Maggio V, Agostinelli C, Chierici M, Jurman G, Furlanello C (March 2018). "Phylogenetic convolutional neural networks in metagenomics". BMC Bioinformatics. 19 (Suppl 2): 49. doi:10.1186/s12859-018-2033-5. PMC 5850953. PMID 29536822.
- ^ Dhungel E, Mreyoud Y, Gwak HJ, Rajeh A, Rho M, Ahn TH (January 2021). "MegaR: an interactive R package for rapid sample classification and phenotype prediction using metagenome profiles and machine learning". BMC Bioinformatics. 22 (1): 25. doi:10.1186/s12859-020-03933-4. PMC 7814621. PMID 33461494.
- ^ Xun W, Liu Y, Li W, Ren Y, Xiong W, Xu Z, et al. (January 2021). "Specialized metabolic functions of keystone taxa sustain soil microbiome stability". Microbiome. 9 (1): 35. doi:10.1186/s40168-020-00985-9. PMC 7849160. PMID 33517892.
- ^ a b Pirooznia M, Yang JY, Yang MQ, Deng Y (2008). "A comparative study of different machine learning methods on microarray gene expression data". BMC Genomics. 9 Suppl 1 (1): S13. doi:10.1186/1471-2164-9-S1-S13. PMC 2386055. PMID 18366602.
- ^ "Machine Learning in Molecular Systems Biology". Frontiers. Retrieved June 9, 2017.
- ^ d'Alché-Buc F, Wehenkel L (December 2008). "Machine learning in systems biology". BMC Proceedings. 2 Suppl 4 (4): S1. doi:10.1186/1753-6561-2-S4-S1. PMC 2654969. PMID 19091048.
- ^ Bhattacharya M (2020). "Unsupervised Techniques in Genomics". In Srinivasa MG, Siddesh GM, MAnisekhar SR (eds.). Statistical Modelling and Machine Learning Principles for Bioinformatics Techniques, Tools, and Applications. Springer Singapore. pp. 164–188. ISBN 978-981-15-2445-5.
- ^ Topol EJ (January 2019). "High-performance medicine: the convergence of human and artificial intelligence". Nature Medicine. 25 (1): 44–56. doi:10.1038/s41591-018-0300-7. PMID 30617339. S2CID 57574615.
- ^ a b Jiang F, Jiang Y, Zhi H, Dong Y, Li H, Ma S, et al. (December 2017). "Artificial intelligence in healthcare: past, present and future". Stroke and Vascular Neurology. 2 (4): 230–243. doi:10.1136/svn-2017-000101. PMC 5829945. PMID 29507784.
- ^ Mirtskhulava L, Wong J, Al-Majeed S, Pearce G (March 2015). "Artificial Neural Network Model in Stroke Diagnosis" (PDF). 2015 17th UKSim-AMSS International Conference on Modelling and Simulation (UKSim): 50–53. doi:10.1109/UKSim.2015.33. ISBN 978-1-4799-8713-9. S2CID 6391733.
- ^ Titano JJ, Badgeley M, Schefflein J, Pain M, Su A, Cai M, et al. (September 2018). "Automated deep-neural-network surveillance of cranial images for acute neurologic events". Nature Medicine. 24 (9): 1337–1341. doi:10.1038/s41591-018-0147-y. PMID 30104767. S2CID 51976344.
- ^ a b c Krallinger M, Erhardt RA, Valencia A (March 2005). "Text-mining approaches in molecular biology and biomedicine". Drug Discovery Today. 10 (6): 439–45. doi:10.1016/S1359-6446(05)03376-3. PMID 15808823.
- ^ Pratas D, Hosseini M, Silva R, Pinho A, Ferreira P (June 20–23, 2017). "Visualization of Distinct DNA Regions of the Modern Human Relatively to a Neanderthal Genome". Pattern Recognition and Image Analysis. Iberian Conference on Pattern Recognition and Image Analysis. Springer. Lecture Notes in Computer Science. Vol. 10255. pp. 235–242. doi:10.1007/978-3-319-58838-4_26. ISBN 978-3-319-58837-7.
- ^ Bardgett RD, Caruso T (March 2020). "Soil microbial community responses to climate extremes: resistance, resilience and transitions to alternative states". Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences. 375 (1794): 20190112. doi:10.1098/rstb.2019.0112. PMC 7017770. PMID 31983338.
- ^ Deveau A, Bonito G, Uehling J, Paoletti M, Becker M, Bindschedler S, et al. (May 2018). "Bacterial-fungal interactions: ecology, mechanisms and challenges". FEMS Microbiology Reviews. 42 (3): 335–352. doi:10.1093/femsre/fuy008. PMID 29471481.
- ^ Ansari MZ, Yadav G, Gokhale RS, Mohanty D (July 2004). "NRPS-PKS: a knowledge-based resource for analysis of NRPS/PKS megasynthases". Nucleic Acids Research. 32 (Web Server issue): W405-13. doi:10.1093/nar/gkh359. PMC 441497. PMID 15215420.
- ^ a b c Navarro-Muñoz JC, Selem-Mojica N, Mullowney MW, Kautsar SA, Tryon JH, Parkinson EI, et al. (January 2020). "A computational framework to explore large-scale biosynthetic diversity". Nature Chemical Biology. 16 (1): 60–68. doi:10.1038/s41589-019-0400-9. PMC 6917865. PMID 31768033.
- ^ a b Doroghazi JR, Albright JC, Goering AW, Ju KS, Haines RR, Tchalukov KA, et al. (November 2014). "A roadmap for natural product discovery based on large-scale genomics and metabolomics". Nature Chemical Biology. 10 (11): 963–8. doi:10.1038/nchembio.1659. PMC 4201863. PMID 25262415.
- ^ a b Cimermancic P, Medema MH, Claesen J, Kurita K, Wieland Brown LC, Mavrommatis K, et al. (July 2014). "Insights into secondary metabolism from a global analysis of prokaryotic biosynthetic gene clusters". Cell. 158 (2): 412–421. doi:10.1016/j.cell.2014.06.034. PMC 4123684. PMID 25036635.
- ^ a b Goering AW, McClure RA, Doroghazi JR, Albright JC, Haverland NA, Zhang Y, et al. (February 2016). "Metabologenomics: Correlation of Microbial Gene Clusters with Metabolites Drives Discovery of a Nonribosomal Peptide with an Unusual Amino Acid Monomer". ACS Central Science. 2 (2): 99–108. doi:10.1021/acscentsci.5b00331. PMC 4827660. PMID 27163034.
- ^ Amiri Moghaddam J, Crüsemann M, Alanjary M, Harms H, Dávila-Céspedes A, Blom J, et al. (November 2018). "Analysis of the Genome and Metabolome of Marine Myxobacteria Reveals High Potential for Biosynthesis of Novel Specialized Metabolites". Scientific Reports. 8 (1): 16600. Bibcode:2018NatSR...816600A. doi:10.1038/s41598-018-34954-y. PMC 6226438. PMID 30413766.
- ^ Duncan KR, Crüsemann M, Lechner A, Sarkar A, Li J, Ziemert N, et al. (April 2015). "Molecular networking and pattern-based genome mining improves discovery of biosynthetic gene clusters and their products from Salinispora species". Chemistry & Biology. 22 (4): 460–471. doi:10.1016/j.chembiol.2015.03.010. PMC 4409930. PMID 25865308.
- ^ Nielsen JC, Grijseels S, Prigent S, Ji B, Dainat J, Nielsen KF, et al. (April 2017). "Global analysis of biosynthetic gene clusters reveals vast potential of secondary metabolite production in Penicillium species". Nature Microbiology. 2 (6): 17044. doi:10.1038/nmicrobiol.2017.44. PMID 28368369. S2CID 22699928.
- ^ McClure RA, Goering AW, Ju KS, Baccile JA, Schroeder FC, Metcalf WW, et al. (December 2016). "Elucidating the Rimosamide-Detoxin Natural Product Families and Their Biosynthesis Using Metabolite/Gene Cluster Correlations". ACS Chemical Biology. 11 (12): 3452–3460. doi:10.1021/acschembio.6b00779. PMC 5295535. PMID 27809474.
- ^ Cao L, Shcherbin E, Mohimani H (August 2019). "A Metabolome- and Metagenome-Wide Association Network Reveals Microbial Natural Products and Microbial Biotransformation Products from the Human Microbiota". mSystems. 4 (4): e00387–19, /msystems/4/4/msys.00387–19.atom. doi:10.1128/mSystems.00387-19. PMC 6712304. PMID 31455639.
- ^ Olm MR, Bhattacharya N, Crits-Christoph A, Firek BA, Baker R, Song YS, et al. (December 2019). "Necrotizing enterocolitis is preceded by increased gut bacterial replication, Klebsiella, and fimbriae-encoding bacteria". Science Advances. 5 (12): eaax5727. Bibcode:2019SciA....5.5727O. doi:10.1126/sciadv.aax5727. PMC 6905865. PMID 31844663.
- ^ Carrión VJ, Perez-Jaramillo J, Cordovez V, Tracanna V, de Hollander M, Ruiz-Buck D, et al. (November 2019). "Pathogen-induced activation of disease-suppressive functions in the endophytic root microbiome". Science. 366 (6465): 606–612. Bibcode:2019Sci...366..606C. doi:10.1126/science.aaw9285. PMID 31672892. S2CID 207814746.
- ^ a b Andreu VP, Augustijn HE, van den Berg K, van der Hooft JJ, Fischbach MA, Medema MH (December 15, 2020). "BiG-MAP: an automated pipeline to profile metabolic gene cluster abundance and expression in microbiomes". bioRxiv 10.1101/2020.12.14.422671.
- ^ a b c Kautsar SA, van der Hooft JJ, de Ridder D, Medema MH (January 2021). "BiG-SLiCE: A highly scalable tool maps the diversity of 1.2 million biosynthetic gene clusters". GigaScience. 10 (1): giaa154. doi:10.1093/gigascience/giaa154. PMC 7804863. PMID 33438731.
- ^ a b Medema MH, Blin K, Cimermancic P, de Jager V, Zakrzewski P, Fischbach MA, et al. (July 2011). "antiSMASH: rapid identification, annotation and analysis of secondary metabolite biosynthesis gene clusters in bacterial and fungal genome sequences". Nucleic Acids Research. 39 (Web Server issue): W339-46. doi:10.1093/nar/gkr466. PMC 3125804. PMID 21672958.
- ^ "MinHash". Wikipedia. April 17, 2021.
- ^ Ondov BD, Treangen TJ, Melsted P, Mallonee AB, Bergman NH, Koren S, Phillippy AM (June 2016). "Mash: fast genome and metagenome distance estimation using MinHash". Genome Biology. 17 (1): 132. doi:10.1186/s13059-016-0997-x. PMC 4915045. PMID 27323842.
- ^ Langmead B, Salzberg SL (March 2012). "Fast gapped-read alignment with Bowtie 2". Nature Methods. 9 (4): 357–9. doi:10.1038/nmeth.1923. PMC 3322381. PMID 22388286.
- ^ "CSS - Metagenomics". www.metagenomics.wiki.
- ^ Agrawal P, Khater S, Gupta M, Sain N, Mohanty D (July 2017). "RiPPMiner: a bioinformatics resource for deciphering chemical structures of RiPPs based on prediction of cleavage and cross-links". Nucleic Acids Research. 45 (W1): W80–W88. doi:10.1093/nar/gkx408. PMC 5570163. PMID 28499008.
- ^ Huber F, Ridder L, Verhoeven S, Spaaks JH, Diblen F, Rogers S, van der Hooft JJ (February 2021). "Spec2Vec: Improved mass spectral similarity scoring through learning of structural relationships". PLOS Computational Biology. 17 (2): e1008724. Bibcode:2021PLSCB..17E8724H. doi:10.1371/journal.pcbi.1008724. PMC 7909622. PMID 33591968.
- ^ Huber F, Ridder L, Verhoeven S, Spaaks JH, Diblen F, Rogers S, van der Hooft JJ (February 2021). "Spec2Vec: Improved mass spectral similarity scoring through learning of structural relationships". PLOS Computational Biology. 17 (2): e1008724. Bibcode:2021PLSCB..17E8724H. doi:10.1371/journal.pcbi.1008724. PMC 7909622. PMID 33591968.
- ^ National Center for Biotechnology Information; U.S. National Library of Medicine. "National Center for Biotechnology Information". www.ncbi.nlm.nih.gov. Retrieved July 30, 2021.
- ^ Agarwala R, Barrett T, Beck J, Benson DA, Bollin C, Bolton E, et al. (NCBI Resource Coordinators) (January 2018). "Database resources of the National Center for Biotechnology Information". Nucleic Acids Research. 46 (D1): D8–D13. doi:10.1093/nar/gkx1095. PMC 5753372. PMID 29140470.
- ^ "antiSMASH database". antismash-db.secondarymetabolites.org.
- ^ "MIBiG: Minimum Information about a Biosynthetic Gene cluster". mibig.secondarymetabolites.org. Retrieved July 30, 2021.
- ^ 미비지
- ^ Kautsar SA, Blin K, Shaw S, Navarro-Muñoz JC, Terlouw BR, van der Hooft JJ, et al. (January 2020). "MIBiG 2.0: a repository for biosynthetic gene clusters of known function". Nucleic Acids Research. 48 (D1): D454–D458. doi:10.1093/nar/gkz882. PMC 7145714. PMID 31612915.
- ^ "Silva". www.arb-silva.de. Retrieved July 30, 2021.
- ^ Quast C, Pruesse E, Yilmaz P, Gerken J, Schweer T, Yarza P, et al. (January 2013). "The SILVA ribosomal RNA gene database project: improved data processing and web-based tools". Nucleic Acids Research. 41 (Database issue): D590-6. doi:10.1093/nar/gks1219. PMC 3531112. PMID 23193283.
- ^ "greengenes.secondgenome.com". greengenes.secondgenome.com. Retrieved July 30, 2021.
- ^ DeSantis TZ, Hugenholtz P, Larsen N, Rojas M, Brodie EL, Keller K, et al. (July 2006). "Greengenes, a chimera-checked 16S rRNA gene database and workbench compatible with ARB". Applied and Environmental Microbiology. 72 (7): 5069–72. Bibcode:2006ApEnM..72.5069D. doi:10.1128/AEM.03006-05. PMC 1489311. PMID 16820507.
- ^ McDonald D, Price MN, Goodrich J, Nawrocki EP, DeSantis TZ, Probst A, et al. (March 2012). "An improved Greengenes taxonomy with explicit ranks for ecological and evolutionary analyses of bacteria and archaea". The ISME Journal. 6 (3): 610–8. doi:10.1038/ismej.2011.139. PMC 3280142. PMID 22134646.
- ^ "opentree". tree.opentreeoflife.org. Retrieved July 30, 2021.
- ^ Hinchliff CE, Smith SA, Allman JF, Burleigh JG, Chaudhary R, Coghill LM, et al. (October 2015). "Synthesis of phylogeny and taxonomy into a comprehensive tree of life". Proceedings of the National Academy of Sciences of the United States of America. 112 (41): 12764–9. Bibcode:2015PNAS..11212764H. doi:10.1073/pnas.1423041112. PMC 4611642. PMID 26385966.
- ^ "RDP Release 11 -- Sequence Analysis Tools". rdp.cme.msu.edu. Retrieved July 30, 2021.
- ^ Cole JR, Wang Q, Fish JA, Chai B, McGarrell DM, Sun Y, et al. (January 2014). "Ribosomal Database Project: data and tools for high throughput rRNA analysis". Nucleic Acids Research. 42 (Database issue): D633-42. doi:10.1093/nar/gkt1244. PMC 3965039. PMID 24288368.