피루브산탈탄산화효소
Pyruvate decarboxylase| 피루브산탈탄산화효소 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
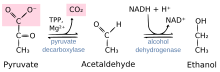 피루브산 탈탄산화효소에 의해 촉매되는 반응: 피루브산+티아민피로인산(TPP)→히드록시에틸-TPP+CO2 | |||||||||
| 식별자 | |||||||||
| EC 번호 | 4.1.1.1 | ||||||||
| CAS 번호 | 9001-04-1 | ||||||||
| 데이터베이스 | |||||||||
| 인텐츠 | IntEnz 뷰 | ||||||||
| 브렌다 | 브렌다 엔트리 | ||||||||
| ExPASy | NiceZyme 뷰 | ||||||||
| 케그 | KEGG 엔트리 | ||||||||
| 메타사이크 | 대사 경로 | ||||||||
| 프라이머리 | 프로필 | ||||||||
| PDB 구조 | RCSB PDB PDBe PDBum | ||||||||
| 진 온톨로지 | AmiGO / QuickGO | ||||||||
| |||||||||
피루브산 탈탄산화효소는 피루브산의 탈탄산을 아세트알데히드로 촉매하는 효소(EC 4.1.1.1)이다.그것은 또한 2-옥소산 카르복실화효소, 알파-케토산 [1]카르복실화효소, 피루빅 탈탄산화효소라고도 한다.혐기성 조건에서, 이 효소는 발효에 의해 에탄올을 생산하기 위해 효모, 특히 사카로미세스에서 발생하는 발효 과정에 참여한다.또한 일부 어종([2]금붕어 및 잉어 포함)에서 산소가 부족할 때 에탄올 발효(유산 발효와 함께)를 수행할 수 있습니다.피루브산 탈탄산화효소는 피루브산을 아세트알데히드와 [3]이산화탄소로 변환함으로써 이 과정을 시작한다.피루브산 탈탄산화효소는 티아민 피로인산염(TPP)과 마그네슘에 의존합니다.이 효소는 피루브산의 산화적 탈탄산을 아세틸-CoA로 촉매하는 산화환원효소(EC 1.2.4.1)인 비관련 효소인 피루브산 탈수소효소로 오해해서는 안 된다.
구조.
피루브산 탈카르복실화효소는 각 이합체의 단량체 간에 공유되는 두 활성 부위의 이합체로서 발생한다.이 효소는 베타-알파-베타 구조를 포함하고 있으며, 평행 베타-시트를 생성한다.그것은 각 이합체에 563개의 잔류 서브유닛을 포함합니다; 효소는 강한 단량체 간 흡인력을 가지고 있지만, 이합체는 느슨하게 상호작용하여 느슨한 사량체를 [4]형성합니다.
활성 부위 잔류물
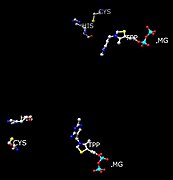
각 활성부위에는 TPP 고리와 상호작용하는 산성 Glu-477과 보조인자의 결합에 관여하는 글루-51을 포함한 20개의 아미노산 잔기가 있다.이러한 글루타메이트는 또한 양성자 기증자의 역할을 하면서 TPP의 일리드를 안정화시킨다.이 Glu 477 주변의 비극성 환경은 일반 pK보다a 높은 pK에 기여하는 비극성 환경이다(일반적인 Glu와 Asp pKa는 작은 [5]단백질에서 약 4.6이다).
친유성 잔류물 Ile-476, Ile-480 및 Pro-26은 글루-477 주변 영역의 비극성에 기여한다.TPP 코엔자임 외에 음전하를 띤 유일한 잔류물은 Asp-28이며, 이는 또한 글루-477의 pK를a 증가시키는 데 도움을 준다.따라서 효소의 환경은 글루-477의 감마 카복실기의 양성자가 [5]pH 6 정도가 되도록 허용해야 한다.
TPP의 아미노피리미딘 고리는 일단 이민 형태로 TPP에서 C2 양성자를 추출하여 친핵성 [4]일라이드를 형성하는 염기 역할을 한다.이는 효소가 TPP C2의 탈양성자를 위한 기본 곁사슬을 가지고 있지 않기 때문에 발생해야 한다. 이러한 글루를 포함하는 활성 부위의 돌연변이는 효소의 비효율 또는 비활성화를 초래할 수 있다.이러한 비활성성은 N1' 및/또는 4'-아미노 그룹이 누락된 실험에서 입증되었습니다.NMR 분석에서는 TPP가 서브스테이트-아날로그 피루브아미드와 함께 효소에 결합되어 있을 때 이리드 형성 속도가 정상 효소 속도보다 높은 것으로 확인되었다.또, Glu51~Gln의 돌연변이율은,[4] 이 비율을 큰폭으로 저감 한다.
잔류물 Asp-444 및 Asp-28은 Mg. Two Cys-221(각 부위로부터 20옹스트롬 이상 떨어져 있음)과 His-92와 결합하며2+, 이는 기질 가용성에 따라 효소를 억제하거나 활성화하는 구조 변화를 일으킨다.활성부위에 결합된 기질이 피루브산인 경우, 효소는 이 [6]조절부위의 배좌변화에 의해 활성화된다.구조 변화는 1,2의 친핵성 첨가를 포함한다.이 반응, 티오케탈의 형성은 효소를 비활성 상태에서 활성 상태로 변화시킨다.
사이트의 억제는 XCHCH64=CHCOOOH 클래스의 억제제/유사체 및 계피알데하이드와 같은 화합물로부터의 탈탄산화 생성물에 의해 이루어진다.억제제의 다른 잠재적 친핵성 부위로는 Cys-152, Asp-28, His-114, His-115 및 Gln-477이 [6]있다.
피루브산 탈탄산화효소의 정상 촉매 속도는cat k = 10초이다−1.단, Gln에 대한 Glu-51 돌연변이를 가진 효소의 속도는 1.7초이다−1.[4]
TPP 보철기
보조인자 TPP는 효소에 대한 보철기이다.티아졸 고리의 황과 질소 원자 사이에 위치한 CH 중심은 산성이다.탈양성자화 시 이라이드를 생성하며, 동일한 탄소 중심은 카르바니온 특성을 획득한다(즉, 음전하가 된다).피루브산의 케톤 탄소는 이 [3]친핵체에 결합한다.피루브산의 탈탄산화 중에 TPP는 비공유결합에 [4]의해 카르바니온 중간체를 친전자성 물질로 안정화시킨다.구체적으로는 TPP의 피리질 [5]질소 N1' 및 4'-아미노기는 효소-TDP 복합체의 촉매 기능에 필수적이다.
메커니즘
이 효소는 피루브산을 이산화탄소와 아세트알데히드로 분해한다.반응은 케토기에 대한 친핵성 카베노이드 탄소의 공격에 의해 진행된다.이 중간체는 CO를 손실하여2 아세트알데히드의 에놀 부가물을 생성합니다.이 단계는 되돌릴 수 없습니다.이어서 유리 아세트알데히드가 [7]방출된다.
효모균
효모에서 피루브산탈탄산화효소는 혐기발효 중에 독립적으로 작용하여 아세트알데히드와 이산화탄소로 2-탄소단편을 방출한다.피루브산 탈탄산화효소는 세포가 배출하는 CO 제거 수단을2 만든다.이 효소는 또한 경쟁하는 [4]유기체를 제거하기 위한 항생제로 사용되는 에탄올을 만드는 수단이다.이 효소는 전이 상태에서 카르보닐 탄소 원자에 음전하가 축적되기 때문에 알파-케토산의 탈탄산화를 돕는 데 필요합니다. 따라서 이 효소는 TPP와 알파-케토산([4]피루브산)이 충족될 수 있는 적절한 환경을 제공합니다.
레퍼런스
- ^ "NiceZyme View of ENZYME: EC 4.1.1.1". ExPASy Proteomics Server.
- ^ Aren van Waarde; G. Van den Thillart; Maria Verhagen (1993). "Ethanol Formation and pH-Regulation in Fish". Surviving Hypoxia. pp. 157−170. hdl:11370/3196a88e-a978-4293-8f6f-cd6876d8c428. ISBN 0-8493-4226-0.
- ^ a b Tadhg P. Begley; McMurry, John (2005). The organic chemistry of biological pathways. Roberts and Co. Publishers. p. 179. ISBN 0-9747077-1-6.
- ^ a b c d e f g PDB: 1pyd; Dyda F, Furey W, Swaminathan S, Sax M, Farrenkopf B, Jordan F (June 1993). "Catalytic centers in the thiamin diphosphate dependent enzyme pyruvate decarboxylase at 2.4-A resolution". Biochemistry. 32 (24): 6165–70. doi:10.1021/bi00075a008. PMID 8512926.
- ^ a b c Lobell M, Crout DH (1996). "Pyruvate Decarboxylase: A Molecular Modeling Study of Pyruvate Decarboxylation and Acyloin Formation". J. Am. Chem. Soc. 118 (8): 1867–1873. doi:10.1021/ja951830t.
- ^ a b Baburina I, Dikdan G, Guo F, Tous GI, Root B, Jordan F (February 1998). "Reactivity at the substrate activation site of yeast pyruvate decarboxylase: inhibition by distortion of domain interactions". Biochemistry. 37 (5): 1245–55. doi:10.1021/bi9709912. PMID 9477950.
- ^ H., Garrett, Reginald (2013). Biochemistry. Grisham, Charles M. (5th ed.). Belmont, CA: Brooks/Cole, Cengage Learning. ISBN 9781133106296. OCLC 777722371.