촉매삼각형
Catalytic triad촉매 삼합체는 일부 효소의 활성 부위에서 발견될 수 있는 세 가지 조정된 아미노산의 집합이다.[1][2]촉매 트라이아드는 하이드롤라아제 및 전달효소(예: 프로테아제, 아드레아제, 에스테라제, 아킬라제, 리파제, β-락타마제)에서 가장 흔히 발견된다.산-기초핵세포 3종(acid-base-nucleophile triad)은 공동 촉매 촉매용 핵분열 잔류물을 생성하기 위한 일반적인 모티프다.잔류물은 전하-릴레이 네트워크를 형성하여 기질을 공격하는 핵분열체를 편극화 및 활성화시켜 공밸런트 중간을 형성하고, 그 다음 가수분해하여 제품을 방출하고 자유효소를 재생시킨다.뉴클레오필드는 세린이나 시스테인 아미노산이 가장 흔하지만, 때로는 세로닌이나 셀레노시스테인이 되기도 한다.효소의 3D 구조는 삼합체 잔여물이 순서(일차 구조)에서 멀리 떨어져 있어도 정확한 방향으로 모인다.[3]
함수(그리고 삼합체 핵분열체)의 상이한 진화뿐만 아니라 촉매 삼합체도 수렴 진화의 가장 좋은 예들을 보여준다.촉매변환에 대한 화학적 제약으로 인해 동일한 촉매 용액이 최소 23개의 개별적인 슈퍼 패밀리에서 독립적으로 진화하고 있다.[2]그들의 행동 메커니즘은 결과적으로 생화학 분야에서 가장 잘 연구된 것 중 하나이다.[4][5]
역사
효소 트립신과 치모트리핀은 1930년대에 처음으로 정화되었다.[6]트립신과 키모트립신 각각에 있는 세린은 1950년대에 촉매핵종(디이소프로필 불소인산염 개조에 의해)으로 확인되었다.[7]치모트립신의 구조는 1960년대 X선 결정학에 의해 해결되어 활성 부위에서 촉매 삼합체의 방향을 보여주었다.[8]현재 S1 계열로 불리는 관련 보호막의 가족이 드러나도록 다른 보호막들을 배열하고 정렬했다.[9][10][11]동시에, 진화적으로 무관한 파판과 미분비인 보호제의 구조는 유사한 3종류를 포함하고 있는 것으로 밝혀졌다.다른 3종류의 구성원에 의한 핵물질의 활성화를 위한 '충전-릴레이' 메커니즘은 1960년대 후반에 제안되었다.[12]1970~80년대 X선 결정학으로 더 많은 프로테아제 구조가 해결되면서 동음이의어(TEV 프로테아제 등), 유사어(파파인 등) 트라이애드가 발견됐다.[13][14][15]1990년대와 2000년대에 MEROPS 분류 체계는 구조적으로 관련된 효소 슈퍼 패밀리로 단백질 분류를 분류하기 시작했으며, 따라서 20개 이상의 슈퍼 패밀리에서 3종류의 융합적 진화의 데이터베이스 역할을 한다.[16][17]진화에 대한 화학적 제약이 어떻게 그렇게 많은 효소 계열이 같은 3중 기하학적 기하학적 구조로 융합되게 되었는지를 이해하는 것은 2010년대에 발전해 왔다.[2]
그들의 최초 발견 이후, 그들의 정확한 촉매 메커니즘에 대한 점점 더 상세한 조사가 있었다.1990년대와 2000년대에는 저배리어 수소 본딩이 촉매에 기여했는지 [18][19][20]또는 일반적인 수소 본딩이 메커니즘을 설명하기에 충분한지 여부에 대한 논쟁이 있었다.[21][22]촉매 트리아드에 의해 사용되는 전하-릴레이, 공밸런스 카탈루션에 대한 방대한 작업 본체는 메커니즘을 모든 생화학에서 가장 잘 특징지워지게 했다.[4][5][21]
함수
촉매 3중합체를 포함하는 효소는 기질을 분할하거나(유압) 기질의 한 부분을 제2 기질(전달)으로 옮기는 두 가지 반응 유형 중 하나에 사용한다.트라이애드는 효소의 활성 부위의 상호 의존적인 잔류물 집합이며 핵포화 강직성을 달성하기 위해 다른 잔류물(예: 결합 부위와 옥산온 구멍)과 함께 작용한다.이러한 삼합체 잔류물은 핵소성 부재를 고반응성으로 만들기 위해 함께 작용하여 기질과의 공밸런트 중간을 생성하여 완전한 촉매로 분해된다.[citation needed]
메커니즘
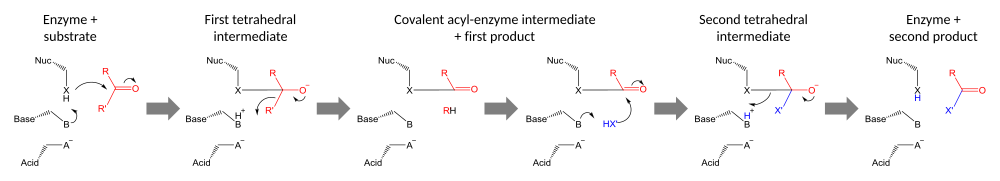
촉매 트라이애드는 잔류물을 핵물질로 사용하여 공동 촉매 촉매제를 수행한다.핵소독 잔류물의 반응성은 다른 3개 부재의 기능 그룹에 의해 증가한다.핵분열체는 분극화되고 염기에 의해 방향이 잡히는데, 염기 자체는 산성에 의해 결합되고 안정화된다.[citation needed]
카탈루션은 두 단계로 수행된다.먼저 활성 핵소체는 카보닐 탄소를 공격하고 카보닐 산소를 전자쌍을 받아들이도록 강제하여 사면중간으로 이어진다.이 중간의 음전하 증가는 일반적으로 활성 사이트 내의 옥시anion 구멍에 의해 안정화된다.그 후 중간은 다시 카보닐로 붕괴되어 기질의 전반부를 배출하지만, 후반부는 여전히 아킬-엔자임 중간체로 효소에 공동 결합되어 있다.제1차 및 제2차 사면중간파괴에 대한 일반 산산성 강직은 다이어그램에 표시된 경로에 의해 발생할 수 있지만, 이 메커니즘을 뒷받침하는 키모트립신(chymotrypsin[23])[24]이 사용된 증거가 논란이 되고 있다.
제2단계 카탈리틱은 제2기판 공격에 의한 아킬-엔자임 중간 분해능이다.만약 이 기질이 물이라면, 그 결과는 가수분해다. 만약 그것이 유기 분자라면, 그 결과는 그 분자를 첫번째 기질로 옮기는 것이다.이 두 번째 기질에 의한 공격은 새로운 사면체 중간을 형성하는데, 이것은 효소의 핵소체를 배출하여 두 번째 생산물을 방출하고 자유효소를 재생함으로써 해결된다.[25]
트라이애드 멤버의 정체성
뉴클레오필레
핵소독 잔류물의 측면 사슬은 기질에 공밸런스 카탈루션을 수행한다.산소나 유황에 존재하는 한 쌍의 전자는 전기적 카보닐 탄소를 공격한다.[3]자연적으로 발생하는 20개의 생물학적 아미노산은 많은 어려운 촉매 반응을 위한 충분한 핵성 기능 그룹을 포함하고 있지 않다.3중창에 핵물질을 내장하면 반응성이 높아져 효율적인 촉매제가 된다.가장 일반적으로 사용되는 핵물질은 세린의 히드록실(OH)과 시스테인의 티올/티올레이트 이온(SH/S−)이다.[2]대신, threonine 프로테아제는 threonine의 2차 히드록실(hydroxyl)을 사용하지만, 사이드 체인의 여분의 메틸 그룹의 강력한 방해로 인해 그러한 프로테아제는 별도의 아미노산이 아닌 그들의 N-단자 아미드를 베이스로 사용한다.[1][26]
핵포화 원자로 산소나 황을 사용하면 촉매에 약간의 차이가 생긴다.산소에 비해 유황의 여분의 d 궤도 때문에 더 크고(0.4 å)[27] 부드러워져 더 긴 결합(d와C-X d를X-H 1.3배)을 형성할 수 있으며 pK(5단위a)를 낮춘다.[28]따라서 세린은 강직과 결합된 감응을 이루기 위해 pK를a[28] 줄이기 위해 산 기반 삼합체 부재의 최적 방향에 시스틴보다 더 의존적이다.[2]시스틴의 낮은 pK는a 원래의 핵포함 공격의 비생산적인 반전이 보다 바람직한 분해산물이기 때문에 첫 번째 사면중간의 분해능에서 불리하게 작용한다.[2]따라서 삼합체 베이스는 효소 유황이 기질 N-terminus에 균일하게 결합되도록 배출되도록 하기 위해 이탈 그룹 아미드를 양성하는 것을 우선적으로 지향한다.마지막으로 아킬-엔자임(기질 C-terminus를 방출하기 위해)의 분해능은 세린을 재프로토닝해야 하는 반면, 사이스틴은 S로− 떠날 수 있다.시스틴의 황은 또한 긴 결합을 형성하고 더 큰 반 데르 발스 반경을[2] 가지고 있으며 세린으로 변이된 경우 활성 현장의 비생산적인 방향에 갇힐 수 있다.[27]
매우 드물게, 희귀한 아미노산 셀레노시스테인의 셀레늄 원자가 핵소포체로 사용된다.[29]감압된 SE− 상태는 촉매 3중창일 때 강하게 선호된다.[29]
베이스
천연 아미노산은 강한 핵성 아미노산이 없기 때문에 촉매 3종류의 기저부는 핵성분의 반응성을 높이기 위해 극성을 내고 탈핵한다.[3]게다가, 그것은 집단 탈출을 돕기 위한 첫 번째 제품을 개발한다.[citation needed]
베이스는 히스티딘으로, 그것의 pK는a 효과적인 베이스 카탈루션, 산 잔여물에 대한 수소 결합, 핵분열 잔여물의 감응을 가능하게 하기 때문에 가장 흔하게 히스티딘이다.[1]TEM-1과 같은 β-락타마아제는 리신 잔류물을 베이스로 사용한다.라이신의 pK가a 너무 높기 때문에(pKa=11), 글루탐산염과 그 밖의 여러 잔류물이 촉매 사이클 동안 그것의 탈염화 상태를 안정시키는 산 역할을 한다.[30][31]세레오닌 보호제는 촉매 세레오닌의 메틸에 의해 황색 덩어리가 몰려 다른 잔여물이 충분히 가까이 있는 것을 방지하므로 N-단자 아미드를 베이스로 사용한다.[32][33]
산성
산성 삼합체 부재는 기본 잔류물과 수소 결합을 형성한다.이것은 사이드 체인 회전을 제한하여 기본 잔류물을 정렬하고, 양극 전하를 안정화하여 극성을 낸다.[3]두 개의 아미노산은 생리학적 pH(아스파테이트 또는 글루탐산염)에 산성측 사슬을 가지고 있으며, 이 삼합체 구성원에 가장 일반적으로 사용된다.[3]시토메갈로바이러스 프로테아제는[b] 히스티딘 한 쌍을 사용하는데 하나는 평소와 같이 베이스로, 하나는 산으로 사용한다.[1]두 번째 히스티딘은 아스파테이트나 글루탐산염보다 더 일반적인 산처럼 효과적인 산성이 아니어서 촉매 효율이 낮아진다.일부 효소에서는 삼합체의 산성 성분이 덜 필요하며 일부 효소는 다이아드 역할만 한다.예를 들어, 파파린은[c] 히스티딘 베이스의 방향을 잡지만 산으로 작용하지 않는 세 번째 삼합체로 아스파라진을 사용한다.마찬가지로, A형 간염 바이러스 프로테아제는[d] 산성 잔류물이 있어야 하는 위치에 주문한 물을 함유하고 있다.[citation needed]
트라이애드의 예

세르히스아스프
세린-히스티딘-아스파테이트 모티브는 생화학에서 가장 철저하게 특징화된 촉매 모티브 중 하나이다.[3]이 삼합창은 그것의 삼합창으로 단백질 등뼈를 가수분해하는 것을 사용하는 PA 슈퍼 패밀리의 모델 세린 프로테아제인 [e]키모트리핀에 의해 예시된다.아스파라테는 히스티딘에 수소가 결합되어 이미다졸 질소의 pK가a 7에서 12 정도까지 증가한다.이를 통해 히스티딘이 강력한 일반 염기 역할을 하고 세린 핵소체를 활성화할 수 있다.그것은 또한 중간자에서의 전하 증가를 안정시키는 몇 개의 백본 아미드들로 구성된 옥시아니온 구멍을 가지고 있다.히스티딘 베이스는 양성자를 기증해 첫 번째 떠나는 그룹을 보조하고, 나머지 OH가− 아킬-엔자임 중간을 공격하자 양성자를 추상화하여 수성수 기질을 활성화하기도 한다.[citation needed]
같은 3중주형도 일부 지질이나 에스테라제와 같은 α/β 하이드롤레이즈에서 수렴하여 진화하였지만, 3중주형 부재의 방향은 역전된다.[34][35]또한 뇌 아세틸 하이드롤라아제(작은 G단백질과 같은 접이식)도 이 3중주를 가지고 있는 것으로 밝혀졌다.등가 Ser-His-Glu 3ad는 아세틸콜린세테라아제에 사용된다.[citation needed]
시스-히스-아스프
두번째로 가장 많이 연구된 삼합체는 시스테인-히스티딘-아스파테이트 모티브다.[2]몇몇 사이스틴 프로테아제 제품군은 TEV 프로테아제와[a] 파파인과 같은 이 3종 세트를 사용한다.[c]삼합창은 세린 프로테아제 삼합창과 유사하게 작용하며, 몇 가지 눈에 띄는 차이가 있다.시스틴의 낮은 pK로a 인해, 촉매에 대한 아스펜의 중요성은 다양하며, 몇몇 시스틴 프로테아제는 효과적으로 시스-히스 디아드(예: A형 간염 바이러스 프로테아제)인 반면, 다른 것에서는 시스틴은 카탈로이트가 시작되기 전에 이미 디프로토닌이 된다(예: 파파인).[36]이 삼합체는 N글리카나아제와 같은 일부 미드레아제에 의해 비펩타이드 C-N 결합을 가수분해하는 데도 사용된다.[37]
세르히스히스
시토메갈로바이러스 프로테아제[b] 트라이아드는 히스티딘을 산성 및 염기성 삼합체로 사용한다.산 히스티딘을 제거하면 10배 활성이 손실될 뿐이다(아스파테이트를 키모트립신에서 제거할 때 10,000배 이상과 비교).이 삼합회는 갈라진 비율을 조절하기 위해 덜 활동적인 효소를 생성하는 가능한 방법으로 해석되어 왔다.[26]
세르-글루-아스프
셀돌리신 프로테아제에서는 특이한 3종류가 발견된다.[f]글루탐산염 카르복실산군의 pK가a 낮다는 것은 매우 낮은 pH에서 삼합창의 베이스 역할만 한다는 것을 의미한다.삼합창은 산성 온천(예: 쿠마몰리신)이나 세포 리소솜(예: 트리펩티딜 펩티다아제)과 같은 특정 환경에 적응하는 것으로 가정한다.[26]
시스히스서
내피 단백질 분해효소인 바소히빈은[g] 핵성분으로서 시스테인을 사용하지만 히스티딘 기지를 조정하기 위해 세린을 사용한다.[38][39]세린은 산성이 약함에도 불구하고 촉매 3중창의 히스티딘 방향을 잡는 데 여전히 효과적이다.[38]일부 호몰로게이션은 산성 위치에 세린 대신 트레오닌이 있다.[38]
스르-엔터, 세르-엔터 및 시스-엔터
프로테아솜 프로테아제 서브유닛[h] 및 오르니틴 아킬트랜스퍼레이즈와[i] 같은 트레오닌 프로테아제는 세린 1차 히드록실 사용과 유사한 방식으로 트레오닌의 2차 히드록실(hydroxyl)을 사용한다.[32][33]그러나, 트레오닌의 여분의 메틸 그룹의 강한 간섭으로 인해, 삼합체의 기본 구성원은 N-단자 아미드인데, 이것은 순수를 극화시키고 촉매 수산화물을 감압시켜 반응성을 증가시킨다.[1][26]마찬가지로 프로테아솜 프로테아제와 진화적으로 관련이 있는 페니실린 아실라제 G, 페니실린[j] 아실라제 V와 같은 동등한[k] '세린 전용'과 '시스테인 전용' 구성이 존재한다.다시 말하지만, 이것들은 N단자 아미드를 베이스로 사용한다.[26]
세르-시스서-라이스
이 특이한 삼합회는 오직 하나의 거대한 무드패밀리에서만 일어난다.이 경우 리신은 중간 세린을 분극시키는 작용을 한다.[40]이어 중간 세린은 핵소필 세린에 두 개의 강한 수소 결합을 형성하여 그것을 활성화시킨다(한 개는 측사슬 수산화물과 다른 하나는 등뼈 아미드).중간 세린은 다른 두 3중 잔류물과의 정밀한 접촉을 용이하게 하기 위해 특이한 시스 방향으로 고정된다.삼합창은 리신과 시스세린 둘 다 촉매 세린을 활성화하는 기초 역할을 한다는 점에서 더욱 특이한데, 같은 리신이 주요 구조 접점을 만들 뿐 아니라 산성 부재의 역할도 수행한다.[40][41]
세히스글루
희귀하지만 자연적으로 발생하는 아미노산 셀레노시스테인(Sec)은 일부 촉매 트라이애드에서도 핵세포로 발견될 수 있다.[29]셀레노시스테인은 시스테인과 비슷하지만 유황 대신 셀레늄 원자를 함유하고 있다.예를 들어 티오레독신 환원효소의 활성 부위에서 이황화물의 감소를 위해 셀레늄을 사용한다.[29]
엔지니어링된 트라이애드
자연적으로 발생하는 촉매 3종 외에도 단백질 공학을 이용해 비원성 아미노산, 즉 완전히 합성 아미노산을 가진 효소 변형을 만들어 왔다.[42]촉매 트라이어드는 다른 비 촉매 단백질, 즉 단백질 모사에도 삽입되었다.[citation needed]
아미틸리신(세린 프로테아제)은 각각의 황,[43][44] 셀레늄 [45]또는 텔루륨으로 산소 핵물질을 대체했다.[46]키스테인과 셀레노시스테인은 돌연변이 유발에 의해 삽입된 반면, 비천연 아미노산인 텔루로시스테인은 합성 텔루로시스테인이 공급된 보조세포를 사용하여 삽입되었다.이 원소들은 모두 제16회 주기율표기둥(찰코균)에 있으므로 성질이 비슷하다.[47][48]각각의 경우에서 핵소포체를 변화시키면 효소의 프로테아제 활성은 감소했지만, 다른 활성은 증가하였다.유황 핵소체는 효소 전달효소 활성을 향상시켰다(중간효소라고도 한다.셀레늄과 텔루륨 뉴클레오파일즈는 효소를 산화효소로 변환시켰다.[45][46]TEV 프로테아제의 핵소포체가 시스테인에서 세린으로 변환되었을 때 프로테아제 활성도는 강하게 감소하였으나 유도된 진화에 의해 회복될 수 있었다.[49]
비 촉매성 단백질은 그 안에 촉매성 트라이애드가 삽입된 후 지시된 진화에 의해 개선된, 비 촉매성 단백질로 사용되어 왔다.세르히스-아스프 삼합창은 항체에 삽입되었고,[50] 다양한 단백질도 포함되어 있다.[51]마찬가지로 촉매 삼합체 모형은 일지 이델렌화 같은 작은 유기 분자에서 생성되어 메리필드 레진과 같은 더 큰 폴리머에 표시되며,[52][53][54] 자가 조립되는 짧은 펩타이드 나노 구조물에 표시된다.[55]
다이버전트 진화
활성 사이트 네트워크의 고도화로 인해 촉매에 관련된 잔류물(및 이와 접촉하는 잔류물)이 고도로 진화적으로 보존된다.[56]그러나 촉매 3종에서 카탈리틱한 반응과 카탈리틱에 사용된 잔류물 모두 분리한 진화의 예가 있다.삼합창은 여전히 활동 현장의 핵심으로 남아 있지만, 다른 기능을 제공하도록 진화적으로 적응하고 있다.[57][58]가성질이라고 불리는 일부 단백질은 비촉성 기능(예: 억제 결합에 의한 규제)을 가지고 있으며 촉매 3중주를 불활성화하는 변이가 누적되어 있다.[59]
반응 변화
촉매 트라이애드는 아킬-엔자임 중간을 통해 공동 촉매 촉매 촉매제를 수행한다.만약 이 중간이 물에 의해 해결된다면, 그 결과는 기질의 가수분해다.그러나 제2 기질에 의한 공격에 의해 중간이 해결되면 효소는 전이효소 역할을 한다.예를 들어, 아킬 그룹에 의한 공격은 아킬전달효소 반응을 일으킨다.몇몇 전이효소 효소 제품군은 물을 배제하고 두 번째 기질 공격을 선호하기 위해 적응에 의해 수화물로부터 진화해왔다.[60]α/β-히드롤라아제 슈퍼 패밀리의 다른 멤버에서 세르히스-아스프 3중주는 주변 잔류물에 의해 튜닝되어 최소 17개의 다른 반응을 수행한다.[35][61]이러한 반응들 중 일부는 아킬-엔자임 중간의 형성 또는 분해능을 변경하거나 아킬-엔자임 중량을 통해 진행되지 않는 메커니즘으로도 달성된다.[35]
또한 대체 전달효소 메커니즘은 두 개의 활성 부위가 있는 아미도인스포리보실 트랜스퍼레이즈에 의해 진화되었다.[l]첫 번째 활성 부위에서 시스틴 삼합체가 글루타민 기질을 가수분해하여 자유 암모니아를 방출한다.암모니아는 효소의 내부 터널을 통해 두 번째 활성 부위로 확산되어 두 번째 기질에 전달된다.[62][63]
핵분열체 변화
강한 화학적 제약으로 인해 활성 현장 잔류물의 다양한 진화가 느리다.그럼에도 불구하고, 일부 프로테아제 슈퍼 패밀리는 한 핵종에서 다른 핵종으로 진화했다.이는 (같은 접힌) 슈퍼 패밀리가 서로 다른 핵물질을 사용하는 패밀리를 포함하고 있을 때 추론할 수 있다.[49]그러한 핵분열 스위치는 진화 역사 동안 여러 번 발생했지만, 이것이 일어나는 메커니즘은 여전히 불분명하다.[17][49]
핵성분 혼합물을 포함하는 protease superfamily(예: PA 씨족) 내에서 가족은 촉매 핵성분(C=cystine protease, S=serine protease)에 의해 지정된다.
| 슈퍼 패밀리 | 가족들 | 예 |
|---|---|---|
| PA씨 | C3, C4, C24, C30, C37, C62, C74, C99 | TEV 프로테아제(담배 에치 바이러스) |
| S1, S3, S6, S7, S29, S30, S31, S32, S39, S46, S55, S64, S65, S75 | 키모트립신(모말, Bos 황소자리 등) | |
| PB씨 | C44, C45, C59, C69, C89, C95 | 아미노포스포리보실전달효소 전구체(호모 사피엔스) |
| S45, S63 | 페니실린 G 아실라아제 전구체(대장균) | |
| T1, T2, T3, T6 | 프로테아솜, 베타 성분(테르모플라즈마 산도필름) | |
| PC 클랜 | C26, C56 | 감마-글루타밀 하이드롤라아제(랫투스 노르베기우스) |
| S51 | 디펩티다아제 E(대장균) | |
| PD 씨족 | C46 | 고슴도치 단백질(드로소필라 멜라노가스터) |
| N9, N10, N11 | 내장형 V형 양성자 ATPase 촉매 서브유닛 A(성체 세레비시아) | |
| PE 씨 | P1 | DmpA아미노펩티아제(Ochrobactrum waei) |
| T5 | 오르니틴 아세틸전달효소 전구체(사카로마이오스 세레비시아아) |
가성어목
촉매 3종 변형의 또 다른 하위 등급은 유사성인데, 3종 돌연변이를 가지고 있어 촉매적으로 활동하지 않지만 결합 또는 구조 단백질로서 기능할 수 있다.[65][66]예를 들어 헤파린 결합 단백질인 아즈로시딘은 PA씨족의 일원이지만 핵성분 대신 글리신을, 히스티딘 대신 세린을 쓴다.[67]마찬가지로 RHBDF1은 뉴클레오필릭세린 대신 알라닌이 있는 S54 계열 rhomboid protease의 호몰로어다.[68][69]어떤 경우에는 가성비가 여전히 촉매 3중주를 가질 수 있지만 나머지 단백질에서 돌연변이가 발생하면 촉매 활성도가 제거된다.CA 클랜은 돌연변이 트라이애드와 온전한 트라이애드를 가지고 있지만 다른 곳에서 돌연변이를 활성화하지 않는(랫드 테스틴은 Cys-His-Asn triad는 유지한다.[70]
| 슈퍼 패밀리 | 가성질을 가진 가족 | 예 |
|---|---|---|
| CA 씨족 | C1, C2, C19 | 칼파모둘린 |
| 씨디 씨족 | C14 | CFLAR |
| SC 씨 | S9, S33 | 노이로리긴 |
| SK 씨 | S14 | ClpR |
| SR씨 | S60 | 세로트란스페린 도메인 2 |
| 성씨 | S54 | RHBDF1 |
| PA씨 | S1 | 아즈로시딘1길 |
| PB씨 | T1 | PSMB3 |
수렴 진화
프로테아제의 효소는 융합 진화의 가장 명확한 예시를 제공한다.20개 이상의 분리된 효소 슈퍼패밀리에서 삼합 잔류물의 동일한 기하학적 배치가 발생한다.이러한 각각의 초가족은 다른 구조 접힘 내에서 동일한 3중배열을 위한 융합적 진화의 결과물이다.3가지 삼합 잔류물인 효소 등뼈와 기질을 정리하는 생산적인 방법이 제한적이기 때문이다.이러한 예는 효소에 대한 본질적인 화학적 물리적 제약조건을 반영하여 진화를 반복적으로 그리고 독립적으로 동등한 용액에 수렴하도록 이끈다.[1][2]
시스틴 및 세린 하이드롤라제
같은 3중 기하학적[e] 구조는 키모트립신이나 아미틸리신 슈퍼패밀리와 같은 세린 보호에 의해 융합되었다.바이러스 C3 프로테아제나 파파인[c] 슈퍼패밀리와 같은 사이스틴 프로테아제에서도 유사한 융합적 진화가 일어났다.이 3종류들은 사이스틴과 세린 프로톨리시스 메커니즘의 기계론적 유사성 때문에 거의 동일한 배열로 수렴되었다.[2]
시스틴 프로테아제 패밀리
| 슈퍼 패밀리 | 가족들 | 예 |
|---|---|---|
| CA | C1, C2, C6, C10, C12, C16, C19, C28, C31, C32, C33, C39, C47, C51, C54, C58, C64, C65, C66, C67, C70, C71, C76, C78, C83, C85, C86, C87, C93, C96, C98, C101 | 파파인 (카리카 파파야)과 칼페인 (호모 사피엔스) |
| CD | C11, C13, C14, C25, C50, C80, C84 | 카스파아제-1(Rattus novigus)과 분리아제(Saccharomes serebisiae) |
| CE | C5, C48, C55, C57, C63, C79 | 아데닌(인간 아데노바이러스 타입 2) |
| CF | C15 | 피로글루타밀-펩티아제 I(바실러스 아밀롤리케파시엔스) |
| CL | C60, C82 | 분류효소 A(Staphylococcus aureus |
| CM | C18 | C형 간염바이러스 펩티다아제2(뇌염 C형 바이러스) |
| 씨엔 | C9 | 신드비스 바이러스 타입 nsP2 펩티다아제(신드비스 바이러스) |
| CO | C40 | 디펩티딜-펩티다아제 6세(리시니바실러스 스파헤리쿠스) |
| CP | C97 | DeSI-1 펩티다아제(Muss musculus) |
| PA | C3, C4, C24, C30, C37, C62, C74, C99 | TEV 프로테아제(담배 에치 바이러스) |
| PB | C44, C45, C59, C69, C89, C95 | 아미노포스포리보실전달효소 전구체(호모 사피엔스) |
| PC | C26, C56 | 감마-글루타밀 하이드롤라아제(랫투스 노르베기우스) |
| PD | C46 | 고슴도치 단백질(드로소필라 멜라노가스터) |
| PE | P1 | DmpA아미노펩티아제(Ochrobactrum waei) |
| 미지정의 | C7, C8, C21, C23, C27, C36, C42, C53, C75 |
세린 보호대 가족
| 슈퍼 패밀리 | 가족들 | 예 |
|---|---|---|
| SB | S8, S53 | 아빌리신 (바실러스 이끼니폼균) |
| SC | S9, S10, S15, S28, S33, S37 | 프롤릴 올리고프티다아제(Sus scrofa) |
| SE | S11, S12, S13 | D-알라-D-알라 펩티다아제 C(대장균) |
| SF | S24, S26 | 시그널 펩티다아제 I(대장균) |
| SH | S21, S73, S77, S78, S80 | 사이토메갈로바이러스 조립린(인간 헤르페스바이러스 5) |
| SJ | S16, S50, S69 | Lon-A 펩티다아제(대장균) |
| SK | S14, S41, S49 | 클프프로테아제(대장균) |
| 그렇게 | S74 | 페이지 GA-1 목 추가 CIMCD 자가 제거 단백질(바실러스 페이지 GA-1) |
| SP | S59 | 뉴클레오폴린 145 (호모 사피엔스) |
| SR. | S60 | 락토페린 (호모 사피엔스) |
| SS | S66 | 무린 테트라펩티아제 LD-카복시펩티아제(페스두도모나스 에어로기노사) |
| 세인트 | S54 | 롬보이드-1 (드로소필라 멜라노가스터) |
| PA | S1, S3, S6, S7, S29, S30, S31, S32, S39, S46, S55, S64, S65, S75 | 키모트립신 A(보스 황소자리) |
| PB | S45, S63 | 페니실린 G 아실라아제 전구체(대장균) |
| PC | S51 | 디펩티다아제 E(대장균) |
| PE | P1 | DmpA아미노펩티아제(Ochrobactrum waei) |
| 미지정의 | S48, S62, S68, S71, S72, S79, S81 |
트레오닌 프로테아제
트레오닌 프로테아제는 아미노산 트레오닌을 촉매 핵물질로 사용한다.시스틴이나 세린과는 달리, 트레오닌은 2차 히드록실(즉, 메틸군이 있다)이다.이 메틸 그룹은 메틸이 효소 백본 또는 히스티딘 베이스와 충돌함에 따라 삼합체 및 기질로의 가능한 방향을 크게 제한한다.[2]세린 프로테아제의 핵소화제가 테레오닌으로 변이되었을 때 메틸은 위치의 혼합물을 차지했고, 그 대부분은 기질 결합을 막았다.[71]결과적으로, Threonine protease의 촉매 잔여물은 그것의 N-terminus에 위치한다.[2]
단백질 주름이 서로 다른 두 개의 진화적으로 독립된 효소 슈퍼패밀리는 N단자 잔류물을 핵분열체로 사용하는 것으로 알려져 있다.슈퍼 패밀리 PB(Ntn 폴드를 사용한 단백질)[32] 및 슈퍼 패밀리 PE(DOM 폴드를 사용한 아세틸 전송)[33]완전히 다른 단백질 접힘에서 활성 사이트 구조의 공통성은 활성 사이트가 그러한 슈퍼 패밀리에서 수렴하여 진화했음을 나타낸다.[2][26]
트레오닌 보호제 가족
| 슈퍼 패밀리 | 가족들 | 예 |
|---|---|---|
| PB씨 | T1, T2, T3, T6 | 프로테아솜, 베타 성분(테르모플라즈마 산도필름) |
| PE 씨 | T5 | 오르니틴 아세틸전달효소(사카로마이오스 세레비시아아) |
참고 항목
참조
메모들
- ^ a b c d TEV 프로테아제 MEROPS: 클랜 PA, 패밀리 C4
- ^ a b 시토메갈로바이러스 프로테아제 MEROPS: 씨족 SH, 가족 S21
- ^ a b c d 파파인 MEROPS: CA 클랜, C1 패밀리
- ^ A형 간염 바이러스 프로테아제 MEROPS: 클랜 PA, 패밀리 C3
- ^ a b c 키모트립신 MEROPS: PA 씨족, S1 가족
- ^ 셀돌리신 프로테아제 MEROPS: 클랜 SB, 패밀리 53
- ^ 바소히빈 프로테아제 MEROPS: 씨족 CA
- ^ a b 프로테아솜 MEROPS: 클랜 PB, 패밀리 T1
- ^ a b Ornithine acyltransferases MEROPS: 클랜 PE, 패밀리 T5
- ^ 페니실린 아실라제 G MEROPS: 클랜 PB, 패밀리 S45
- ^ 페니실린 아킬라아제 V MEROPS: 클랜 PB, 패밀리 C59
- ^ 아미노포시보실전달효소 MEROPS: 클랜 PB, 패밀리 C44
- ^ 서브틸리신 MEROPS: 클랜 SB, 패밀리 S8
- ^ 프롤릴 올리고프티다제 MEROPS: 씨족SC, 패밀리 S9
인용구
- ^ a b c d e f Dodson G, Wlodawer A (1998). "Catalytic triads and their relatives". Trends Biochem. Sci. 23 (9): 347–52. doi:10.1016/S0968-0004(98)01254-7. PMID 9787641.
- ^ a b c d e f g h i j k l m Buller AR, Townsend CA (2013). "Intrinsic evolutionary constraints on protease structure, enzyme acylation, and the identity of the catalytic triad". Proc. Natl. Acad. Sci. U.S.A. 110 (8): E653–61. Bibcode:2013PNAS..110E.653B. doi:10.1073/pnas.1221050110. PMC 3581919. PMID 23382230.
- ^ a b c d e f Stryer L, Berg JM, Tymoczko JL (2002). "9 Catalytic Strategies". Biochemistry (5th ed.). San Francisco: W.H. Freeman. ISBN 9780716749554.
- ^ a b Perutz M (1992). Protein structure. New approaches to disease and therapy. New York: W.H. Freeman and Co. ISBN 9780716770213.
- ^ a b Neurath H (1994). "Proteolytic enzymes past and present: the second golden era. Recollections, special section in honor of Max Perutz". Protein Sci. 3 (10): 1734–9. doi:10.1002/pro.5560031013. PMC 2142620. PMID 7849591.
- ^ Ohman KP, Hoffman A, Keiser HR (1990). "Endothelin-induced vasoconstriction and release of atrial natriuretic peptides in the rat". Acta Physiol. Scand. 138 (4): 549–56. doi:10.1111/j.1748-1716.1990.tb08883.x. PMID 2141214.
- ^ Dixon GH, Kauffman DL, Neurath H (1958). "Amino Acid Sequence in the Region of Diisopropyl Phosphoryl Binding in Dip-Trypsin". J. Am. Chem. Soc. 80 (5): 1260–1. doi:10.1021/ja01538a059.
- ^ Matthews BW, Sigler PB, Henderson R, et al. (1967). "Three-dimensional structure of tosyl-α-chymotrypsin". Nature. 214 (5089): 652–656. Bibcode:1967Natur.214..652M. doi:10.1038/214652a0. PMID 6049071. S2CID 4273406.
- ^ Walsh KA, Neurath H (1964). "Trypsinogen and Chymotrypsinogen as Homologous Proteins". Proc. Natl. Acad. Sci. U.S.A. 52 (4): 884–9. Bibcode:1964PNAS...52..884W. doi:10.1073/pnas.52.4.884. PMC 300366. PMID 14224394.
- ^ de Haën C, Neurath H, Teller DC (1975). "The phylogeny of trypsin-related serine proteases and their zymogens. New methods for the investigation of distant evolutionary relationships". J. Mol. Biol. 92 (2): 225–59. doi:10.1016/0022-2836(75)90225-9. PMID 1142424.
- ^ Lesk AM, Fordham WD (1996). "Conservation and variability in the structures of serine proteinases of the chymotrypsin family". J. Mol. Biol. 258 (3): 501–37. doi:10.1006/jmbi.1996.0264. PMID 8642605.
- ^ Blow DM, Birktoft JJ, Hartley BS (1969). "Role of a buried acid group in the mechanism of action of chymotrypsin". Nature. 221 (5178): 337–40. Bibcode:1969Natur.221..337B. doi:10.1038/221337a0. PMID 5764436. S2CID 4214520.
- ^ Gorbalenya AE, Blinov VM, Donchenko AP (1986). "Poliovirus-encoded proteinase 3C: a possible evolutionary link between cellular serine and cysteine proteinase families". FEBS Lett. 194 (2): 253–7. doi:10.1016/0014-5793(86)80095-3. PMID 3000829. S2CID 23268152.
- ^ Bazan JF, Fletterick RJ (1988). "Viral cysteine proteases are homologous to the trypsin-like family of serine proteases: structural and functional implications". Proc. Natl. Acad. Sci. U.S.A. 85 (21): 7872–6. Bibcode:1988PNAS...85.7872B. doi:10.1073/pnas.85.21.7872. PMC 282299. PMID 3186696.
- ^ Phan J, Zdanov A, Evdokimov AG, et al. (2002). "Structural basis for the substrate specificity of tobacco etch virus protease". J. Biol. Chem. 277 (52): 50564–72. doi:10.1074/jbc.M207224200. PMID 12377789.
- ^ Rawlings ND, Barrett AJ (1993). "Evolutionary families of peptidases". Biochem. J. 290 (1): 205–18. doi:10.1042/bj2900205. PMC 1132403. PMID 8439290.
- ^ a b Rawlings ND, Barrett AJ, Bateman A (2010). "MEROPS: the peptidase database". Nucleic Acids Res. 38 (supl_1): D227–33. doi:10.1093/nar/gkp971. PMC 2808883. PMID 19892822.
- ^ Frey PA, Whitt SA, Tobin JB (1994). "A low-barrier hydrogen bond in the catalytic triad of serine proteases". Science. 264 (5167): 1927–30. Bibcode:1994Sci...264.1927F. doi:10.1126/science.7661899. PMID 7661899.
- ^ Ash EL, Sudmeier JL, De Fabo EC, et al. (1997). "A low-barrier hydrogen bond in the catalytic triad of serine proteases? Theory versus experiment". Science. 278 (5340): 1128–32. Bibcode:1997Sci...278.1128A. doi:10.1126/science.278.5340.1128. PMID 9353195.
- ^ Agback P, Agback T (2018). "Direct evidence of a low barrier hydrogen bond in the catalytic triad of a Serine protease". Sci. Rep. 8 (1): 10078. Bibcode:2018NatSR...810078A. doi:10.1038/s41598-018-28441-7. PMC 6031666. PMID 29973622.
- ^ a b Schutz CN, Warshel A (2004). "The low barrier hydrogen bond (LBHB) proposal revisited: the case of the Asp... His pair in serine proteases". Proteins. 55 (3): 711–23. doi:10.1002/prot.20096. PMID 15103633. S2CID 34229297.
- ^ Warshel A, Papazyan A (1996). "Energy considerations show that low-barrier hydrogen bonds do not offer a catalytic advantage over ordinary hydrogen bonds". Proc. Natl. Acad. Sci. U.S.A. 93 (24): 13665–70. Bibcode:1996PNAS...9313665W. doi:10.1073/pnas.93.24.13665. PMC 19385. PMID 8942991.
- ^ Fersht, A.R.; Requena, Y (1971). "Mechanism of the Chymotrypsin-Catalyzed Hydrolysis of Amides. pH Dependence of kc and Km.' Kinetic Detection of an Intermediate". J. Am. Chem. Soc. 93 (25): 7079–87. doi:10.1021/ja00754a066. PMID 5133099.
- ^ Zeeberg, B; Caswell, M; Caplow, M (1973). "Concerning a reported change in rate-determining step in chymotrypsin catalysis". J. Am. Chem. Soc. 95 (8): 2734–5. doi:10.1021/ja00789a081. PMID 4694533.
- ^ Shafee T (2014). Evolvability of a viral protease: experimental evolution of catalysis, robustness and specificity (PhD thesis). University of Cambridge. doi:10.17863/CAM.16528.
- ^ a b c d e f Ekici OD, Paetzel M, Dalbey RE (2008). "Unconventional serine proteases: variations on the catalytic Ser/His/Asp triad configuration". Protein Sci. 17 (12): 2023–37. doi:10.1110/ps.035436.108. PMC 2590910. PMID 18824507.
- ^ a b McGrath ME, Wilke ME, Higaki JN, et al. (1989). "Crystal structures of two engineered thiol trypsins". Biochemistry. 28 (24): 9264–70. doi:10.1021/bi00450a005. PMID 2611228.
- ^ a b Polgár L, Asbóth B (1986). "The basic difference in catalyses by serine and cysteine proteinases resides in charge stabilization in the transition state". J. Theor. Biol. 121 (3): 323–6. Bibcode:1986JThBi.121..323P. doi:10.1016/s0022-5193(86)80111-4. PMID 3540454.
- ^ a b c d Brandt W, Wessjohann LA (2005). "The functional role of selenocysteine (Sec) in the catalysis mechanism of large thioredoxin reductases: proposition of a swapping catalytic triad including a Sec-His-Glu state". ChemBioChem. 6 (2): 386–94. doi:10.1002/cbic.200400276. PMID 15651042. S2CID 25575160.
- ^ Damblon C, Raquet X, Lian LY, et al. (1996). "The catalytic mechanism of beta-lactamases: NMR titration of an active-site lysine residue of the TEM-1 enzyme". Proc. Natl. Acad. Sci. U.S.A. 93 (5): 1747–52. Bibcode:1996PNAS...93.1747D. doi:10.1073/pnas.93.5.1747. PMC 39852. PMID 8700829.
- ^ Jelsch C, Lenfant F, Masson JM, et al. (1992). "Beta-lactamase TEM1 of E. coli. Crystal structure determination at 2.5 A resolution". FEBS Lett. 299 (2): 135–42. doi:10.1016/0014-5793(92)80232-6. PMID 1544485.
- ^ a b c Brannigan JA, Dodson G, Duggleby HJ, et al. (1995). "A protein catalytic framework with an N-terminal nucleophile is capable of self-activation". Nature. 378 (6555): 416–9. Bibcode:1995Natur.378..416B. doi:10.1038/378416a0. PMID 7477383. S2CID 4277904.
- ^ a b c Cheng H, Grishin NV (2005). "DOM-fold: a structure with crossing loops found in DmpA, ornithine acetyltransferase, and molybdenum cofactor-binding domain". Protein Sci. 14 (7): 1902–10. doi:10.1110/ps.051364905. PMC 2253344. PMID 15937278.
- ^ Sun Y, Yin S, Feng Y, et al. (2014). "Molecular basis of the general base catalysis of an α/β-hydrolase catalytic triad". J. Biol. Chem. 289 (22): 15867–79. doi:10.1074/jbc.m113.535641. PMC 4140940. PMID 24737327.
- ^ a b c Rauwerdink A, Kazlauskas RJ (2015). "How the Same Core Catalytic Machinery Catalyzes 17 Different Reactions: the Serine-Histidine-Aspartate Catalytic Triad of α/β-Hydrolase Fold Enzymes". ACS Catal. 5 (10): 6153–6176. doi:10.1021/acscatal.5b01539. PMC 5455348. PMID 28580193.
- ^ Beveridge AJ (1996). "A theoretical study of the active sites of papain and S195C rat trypsin: implications for the low reactivity of mutant serine proteinases". Protein Sci. 5 (7): 1355–65. doi:10.1002/pro.5560050714. PMC 2143470. PMID 8819168.
- ^ Allen MD, Buchberger A, Bycroft M (2006). "The PUB domain functions as a p97 binding module in human peptide N-glycanase". J. Biol. Chem. 281 (35): 25502–8. doi:10.1074/jbc.M601173200. PMID 16807242.
- ^ a b c Sanchez-Pulido L, Ponting CP (2016). "Vasohibins: new transglutaminase-like cysteine proteases possessing a non-canonical Cys-His-Ser catalytic triad". Bioinformatics. 32 (10): 1441–5. doi:10.1093/bioinformatics/btv761. PMC 4866520. PMID 26794318.
- ^ Sato Y, Sonoda H (2007). "The vasohibin family: a negative regulatory system of angiogenesis genetically programmed in endothelial cells". Arterioscler. Thromb. Vasc. Biol. 27 (1): 37–41. doi:10.1161/01.atv.0000252062.48280.61. PMID 17095714.
- ^ a b Shin S, Yun YS, Koo HM, et al. (2003). "Characterization of a novel Ser-cisSer-Lys catalytic triad in comparison with the classical Ser-His-Asp triad". J. Biol. Chem. 278 (27): 24937–43. doi:10.1074/jbc.M302156200. PMID 12711609.
- ^ Cerqueira NM, Moorthy H, Fernandes PA, et al. (2017). "The mechanism of the Ser-(cis)Ser-Lys catalytic triad of peptide amidases". Phys. Chem. Chem. Phys. 19 (19): 12343–12354. Bibcode:2017PCCP...1912343C. doi:10.1039/C7CP00277G. PMID 28453015. Archived from the original on 2017-12-24. Retrieved 2017-12-24.
- ^ Toscano MD, Woycechowsky KJ, Hilvert D (2007). "Minimalist active-site redesign: teaching old enzymes new tricks". Angew. Chem. 46 (18): 3212–36. doi:10.1002/anie.200604205. PMID 17450624.
- ^ Abrahmsén L, Tom J, Burnier J, et al. (1991). "Engineering subtilisin and its substrates for efficient ligation of peptide bonds in aqueous solution". Biochemistry. 30 (17): 4151–9. CiteSeerX 10.1.1.461.9606. doi:10.1021/bi00231a007. PMID 2021606.
- ^ Jackson DY, Burnier J, Quan C, et al. (1994). "A designed peptide ligase for total synthesis of ribonuclease A with unnatural catalytic residues". Science. 266 (5183): 243–7. Bibcode:1994Sci...266..243J. doi:10.1126/science.7939659. JSTOR 2884761. PMID 7939659.
- ^ a b Syed R, Wu ZP, Hogle JM, et al. (1993). "Crystal structure of selenosubtilisin at 2.0-A resolution". Biochemistry. 32 (24): 6157–64. doi:10.1021/bi00075a007. PMID 8512925.
- ^ a b Mao S, Dong Z, Liu J, et al. (2005). "Semisynthetic tellurosubtilisin with glutathione peroxidase activity". J. Am. Chem. Soc. 127 (33): 11588–9. doi:10.1021/ja052451v. PMID 16104720.
- ^ Devillanova FA, Du Mont W (2013). Handbook of Chalcogen Chemistry. Vol. 1: new perspectives in sulfur, selenium and tellurium (2nd ed.). Cambridge: RSC. ISBN 9781849736237. OCLC 868953797.
- ^ Bouroushian M (2010). "Electrochemistry of the Chalcogens". Electrochemistry of Metal Chalcogenides. Monographs in Electrochemistry. Berlin, Heidelberg: Springer. pp. 57–75. doi:10.1007/978-3-642-03967-6_2. ISBN 9783642039669.
- ^ a b c Shafee T, Gatti-Lafranconi P, Minter R, et al. (2015). "Handicap-Recover Evolution Leads to a Chemically Versatile, Nucleophile-Permissive Protease". ChemBioChem. 16 (13): 1866–9. doi:10.1002/cbic.201500295. PMC 4576821. PMID 26097079.
- ^ Okochi N, Kato-Murai M, Kadonosono T, et al. (2007). "Design of a serine protease-like catalytic triad on an antibody light chain displayed on the yeast cell surface". Appl. Microbiol. Biotechnol. 77 (3): 597–603. doi:10.1007/s00253-007-1197-0. PMID 17899065. S2CID 35590920.
- ^ Rajagopalan S, Wang C, Yu K, et al. (2014). "Design of activated serine-containing catalytic triads with atomic-level accuracy". Nat. Chem. Biol. 10 (5): 386–91. doi:10.1038/nchembio.1498. PMC 4048123. PMID 24705591.
- ^ Bhowmick D, Mugesh G (2015). "Introduction of a catalytic triad increases the glutathione peroxidase-like activity of diaryl diselenides". Org. Biomol. Chem. 13 (34): 9072–82. doi:10.1039/C5OB01294E. PMID 26220806.
- ^ Bhowmick D, Mugesh G (2015). "Insights into the catalytic mechanism of synthetic glutathione peroxidase mimetics". Org. Biomol. Chem. 13 (41): 10262–72. doi:10.1039/c5ob01665g. PMID 26372527.
- ^ Nothling MD, Ganesan A, Condic-Jurkic K, et al. (2017). "Simple Design of an Enzyme-Inspired Supported Catalyst Based on a Catalytic Triad". Chem. 2 (5): 732–745. doi:10.1016/j.chempr.2017.04.004.
- ^ Gulseren G, Khalily MA, Tekinay AB, et al. (2016). "Catalytic supramolecular self-assembled peptide nanostructures for ester hydrolysis". J. Mater. Chem. B. 4 (26): 4605–4611. doi:10.1039/c6tb00795c. hdl:11693/36666. PMID 32263403.
- ^ Halabi N, Rivoire O, Leibler S, et al. (2009). "Protein sectors: evolutionary units of three-dimensional structure". Cell. 138 (4): 774–86. doi:10.1016/j.cell.2009.07.038. PMC 3210731. PMID 19703402.
- ^ Murzin AG (1998). "How far divergent evolution goes in proteins". Current Opinion in Structural Biology. 8 (3): 380–387. doi:10.1016/S0959-440X(98)80073-0. PMID 9666335.
- ^ Gerlt JA, Babbitt PC (2001). "Divergent evolution of enzymatic function: mechanistically diverse superfamilies and functionally distinct suprafamilies". Annu. Rev. Biochem. 70 (1): 209–46. doi:10.1146/annurev.biochem.70.1.209. PMID 11395407.
- ^ Murphy JM, Farhan H, Eyers PA (2017). "Bio-Zombie: the rise of pseudoenzymes in biology". Biochem. Soc. Trans. 45 (2): 537–544. doi:10.1042/bst20160400. PMID 28408493.
- ^ Stehle F, Brandt W, Stubbs MT, et al. (2009). "Sinapoyltransferases in the light of molecular evolution". Phytochemistry. Evolution of Metabolic Diversity. 70 (15–16): 1652–62. doi:10.1016/j.phytochem.2009.07.023. PMID 19695650.
- ^ Dimitriou PS, Denesyuk A, Takahashi S, et al. (2017). "Alpha/beta-hydrolases: A unique structural motif coordinates catalytic acid residue in 40 protein fold families". Proteins. 85 (10): 1845–1855. doi:10.1002/prot.25338. PMID 28643343. S2CID 25363240.
- ^ Smith JL (1998). "Glutamine PRPP amidotransferase: snapshots of an enzyme in action". Current Opinion in Structural Biology. 8 (6): 686–94. doi:10.1016/s0959-440x(98)80087-0. PMID 9914248.
- ^ Smith JL, Zaluzec EJ, Wery JP, et al. (1994). "Structure of the allosteric regulatory enzyme of purine biosynthesis". Science. 264 (5164): 1427–33. Bibcode:1994Sci...264.1427S. doi:10.1126/science.8197456. PMID 8197456.
- ^ "Clans of Mixed (C, S, T) Catalytic Type". www.ebi.ac.uk. MEROPS. Retrieved 20 Dec 2018.
- ^ a b Fischer K, Reynolds SL (2015). "Pseudoproteases: mechanisms and function". Biochem. J. 468 (1): 17–24. doi:10.1042/BJ20141506. PMID 25940733.
- ^ Todd AE, Orengo CA, Thornton JM (2002). "Sequence and structural differences between enzyme and nonenzyme homologs". Structure. 10 (10): 1435–51. doi:10.1016/s0969-2126(02)00861-4. PMID 12377129.
- ^ Iversen LF, Kastrup JS, Bjørn SE, et al. (1997). "Structure of HBP, a multifunctional protein with a serine proteinase fold". Nat. Struct. Biol. 4 (4): 265–8. doi:10.1038/nsb0497-265. PMID 9095193. S2CID 19949043.
- ^ Zettl M, Adrain C, Strisovsky K, et al. (2011). "Rhomboid family pseudoproteases use the ER quality control machinery to regulate intercellular signaling". Cell. 145 (1): 79–91. doi:10.1016/j.cell.2011.02.047. PMC 3149277. PMID 21439629.
- ^ Lemberg MK, Adrain C (2016). "Inactive rhomboid proteins: New mechanisms with implications in health and disease". Semin. Cell Dev. Biol. 60: 29–37. doi:10.1016/j.semcdb.2016.06.022. hdl:10400.7/759. PMID 27378062.
- ^ Cheng CY, Morris I, Bardin CW (1993). "Testins Are Structurally Related to the Mouse Cysteine Proteinase Precursor But Devoid of Any Protease/Anti-Protease Activity". Biochem. Biophys. Res. Commun. 191 (1): 224–231. doi:10.1006/bbrc.1993.1206. PMID 8447824.
- ^ Pelc LA, Chen Z, Gohara DW, et al. (2015). "Why Ser and not Thr brokers catalysis in the trypsin fold". Biochemistry. 54 (7): 1457–64. doi:10.1021/acs.biochem.5b00014. PMC 4342846. PMID 25664608.