인공 유전자 합성
Artificial gene synthesis인공 유전자 합성, 또는 간단히 유전자 합성은 뉴클레오티드 드 노보로부터 유전자를 구성하고 조립하기 위해 합성 생물학에서 사용되는 방법들의 그룹을 말한다.살아있는 세포의 DNA 합성과는 달리, 인공 유전자 합성은 템플릿 DNA를 필요로 하지 않으며, 사실상 모든 DNA 서열을 실험실에서 합성할 수 있게 한다.이것은 두 가지 주요 단계로 구성되어 있는데, 첫 번째 단계는 DNA 인쇄로 알려져 [1]있는 고체상 DNA 합성입니다.이것은 일반적으로 200개의 염기쌍 아래에 있는 올리고뉴클레오티드 단편을 생성한다.두 번째 단계는 다양한 DNA 조립 방법을 사용하여 이러한 올리고뉴클레오티드 단편을 연결하는 것을 포함한다.인공 유전자 합성은 템플릿 DNA를 필요로 하지 않기 때문에 이론적으로 뉴클레오티드 배열이나 크기 제한 없이 완전한 합성 DNA 분자를 만드는 것이 가능하다.
최초의 완전한 유전자인 효모 tRNA의 합성은 1972년 [2]Har Gobind Khorana와 동료들에 의해 증명되었다.첫 번째 펩타이드 및 단백질 코드 유전자의 합성은 각각 [3][4]허버트 보이어와 알렉산더 마캄의 연구실에서 수행되었다.최근에는 전체 염색체와 게놈을 조립할 수 있는 인공 유전자 합성 방법이 개발되고 있다.첫 번째 합성 효모 염색체는 2014년에 합성되었으며, 전체 기능성 세균 염색체도 [5]합성되었다.또한, 인공 유전자 합성은 미래에 새로운 핵염기쌍(비자연 염기쌍)[6][7][8]을 사용할 수 있다.
DNA 합성을 위한 표준 방법
올리고뉴클레오티드합성
올리고뉴클레오티드는 뉴클레오시드 포스포라미다이트라고 불리는 구성 요소를 사용하여 화학적으로 합성됩니다.이것들은 정상적이거나 변형된 뉴클레오시드일 수 있으며, 그들의 아민, 수산기 및 인산기가 잘못 상호작용하는 것을 방지하기 위한 보호기를 가지고 있다.한 번에 1개의 인산화아미다이트를 첨가하여 5' 하이드록실기를 탈보호하고 새로운 염기를 첨가하는 등의 처리를 한다.사슬은 생합성에 비해 역방향인 3'에서 5' 방향으로 성장한다.마지막에 모든 보호 그룹이 제거됩니다.그럼에도 불구하고 화학 공정이기 때문에 여러 가지 잘못된 상호작용이 발생하여 일부 불량품이 발생합니다.합성되는 올리고뉴클레오티드 배열이 길어질수록 결함이 많아지므로, 이 과정은 뉴클레오티드의 짧은 염기서열을 생성하는 데만 실용적이다.현재 실제 한계는 생물학적 응용에 직접 사용하기에 충분한 품질을 가진 올리고뉴클레오티드에 대해 약 200bp(염기쌍)이다.HPLC를 사용하여 올바른 시퀀스를 가진 제품을 분리할 수 있습니다.한편, 유전자 칩상에서 다수의 올리고를 병렬로 합성할 수 있다.후속 유전자 합성 절차에서 최적의 성능을 위해 개별적으로 더 큰 규모로 준비되어야 한다.
올리고뉴클레오티드의 아닐화 기반 결합
일반적으로 개별적으로 설계된 올리고뉴클레오티드 세트는 자동화된 고체상 신시사이저에서 만들어지고 정제된 다음 특정 아닐링 및 표준 결합 또는 중합효소 반응에 의해 연결된다.올리고뉴클레오티드 아닐의 특이성을 개선하기 위해, 합성 단계는 일련의 내열성 DNA 연결효소 및 중합효소 효소에 의존합니다.지금까지 유전자 합성을 위한 몇 가지 방법이 설명되었는데, 예를 들어 인산화 중복 올리고뉴클레오티드의 [2][3]결찰, Fok I 방법[4] 및 유전자 합성을 위한 리게아제 연쇄 반응의 변형 형태 등이 있다.또한 몇 가지 PCR 조립 접근법이 [9]설명되었습니다.그들은 보통 서로 겹치는 40-50 뉴클레오티드 길이의 올리고뉴클레오티드를 사용한다.이들 올리고뉴클레오티드는 양쪽 가닥의 배열 대부분을 포함하도록 설계되었으며, 전장 분자는 오버랩 익스텐션(OE) PCR,[9] 열역학적으로 균형 잡힌 내부외(TBIO) PCR[10] 또는 복합 접근법에 [11]의해 점진적으로 생성된다.가장 일반적으로 합성되는 유전자는 크기가 600에서 1,200 bp 사이이지만, 이전에 조립된 1,000 bp 이하의 조각들을 연결함으로써 훨씬 더 긴 유전자가 만들어졌다.이 크기 범위에서는 자동화된 염기서열결정법에 의해 복제된 합성 유전자의 염기서열을 확인하는 여러 후보 클론을 시험할 필요가 있다.
제한 사항
더욱이, 전장 유전자 생성물의 조립은 긴 단일 가닥 올리고뉴클레오티드의 효율적이고 특정한 정렬에 의존하기 때문에, 합성 성공을 위한 중요한 매개 변수는 반전 반복, 비정상적으로 높은 또는 낮은 GC 함량 또는 반복 구조에 의해 야기되는 2차 구조를 포함하는 확장된 배열 영역을 포함한다.통상, 특정 유전자의 이러한 세그먼트는, 몇개의 연속적인 스텝과 짧은 서브 시퀀스의 최종적인 조립에 의해서만 합성할 수 있습니다.그 결과, 제조에 필요한 시간과 노동이 큰폭으로 증가합니다.유전자 합성 실험의 결과는 사용된 올리고뉴클레오티드의 품질에 크게 좌우된다.이러한 아닐링 기반 유전자 합성 프로토콜의 경우, 제품의 품질은 사용된 올리고뉴클레오티드의 정확성에 직접적이고 기하급수적으로 의존한다.또는 낮은 품질의 올리고로 유전자 합성을 수행한 후, 클론 분석 중에 다운스트림 품질 보장에 더 많은 노력을 기울여야 하며, 이는 보통 시간이 많이 걸리는 표준 클로닝 및 염기서열 처리 절차에 의해 이루어진다.현재의 모든 유전자 합성 방법과 관련된 또 다른 문제는 화학적으로 합성된 올리고뉴클레오티드의 사용으로 인한 높은 배열 오류 빈도이다.오류 빈도는 올리고뉴클레오티드가 길어질수록 증가하며, 그 결과 올리고뉴클레오티드가 많이 사용될수록 정확한 생성물의 비율이 급격히 감소한다.돌연변이 문제는 유전자를 조립하는 데 사용되는 짧은 올리고뉴클레오티드에 의해 해결될 수 있다.그러나 모든 아닐 기반 조립 방법에서는 프라이머를 하나의 튜브에 혼합해야 합니다.이 경우, 짧은 겹침이 상보적 프라이머의 정확하고 특정한 아닐링을 항상 가능하게 하는 것은 아니며, 결과적으로 전체 길이의 생성물 형성을 억제한다.올리고뉴클레오티드의 수동 설계는 힘든 절차이며 원하는 유전자의 성공적인 합성을 보장하지 않습니다.거의 모든 아닐링 기반 방법의 최적 성능을 위해 중복 영역의 용해 온도는 모든 올리고뉴클레오티드에 대해 유사해야 한다.필요한 프라이머 최적화는 전문화된 올리고뉴클레오티드 설계 프로그램을 사용하여 수행해야 한다.유전자 합성을 위한 자동화된 프라이머 설계를 위한 몇 가지 솔루션이 지금까지 [12][13][14]제시되었다.
오류 정정 절차
올리고뉴클레오티드 품질과 관련된 문제를 극복하기 위해, 여러 정교한 전략이 개발되었으며, 별도로 준비된 어획 올리고뉴클레오티드,[15] mutS[16] 계열의 불일치 결합 효소 또는 박테리아 또는 [17]페이징으로부터의 특정 엔도뉴클레오타아제 중 하나를 사용한다.그럼에도 불구하고, 이러한 모든 전략은 화학적으로 합성된 올리고뉴클레오티드의 어닐링을 기반으로 유전자 합성에 시간과 비용을 증가시킨다.
대규모 병렬 시퀀싱은 복잡한 올리고뉴클레오티드 라이브러리를 선별하고 정확한 분자의 검색을 가능하게 하는 도구로도 사용되어 왔다.하나의 어프로치에서는 올리고뉴클레오티드가 454개의 파이로시퀀싱 플랫폼 상에 배열되어 로봇시스템에 의해 촬영되어 정확한 [18]시퀀스에 대응하는 개별 비즈를 추출한다.또 다른 접근법에서는 복합 올리고뉴클레오티드 라이브러리는 대규모 병렬 시퀀싱 전에 고유한 측면 태그에 의해 수정된다.다음으로 태그 다이렉트프라이머는 다이얼아웃 PCR에 [19]의해 원하는 시퀀스를 가진 분자의 취득을 가능하게 합니다.
점점 더 많은 유전자들이 기능적으로 관련된 유전자 또는 단일 유전자에 대한 다중 배열 변형을 포함하는 세트로 정렬된다.모노클로널 항체와 같은 개발 중인 거의 모든 치료 단백질은 개선된 기능 또는 발현을 위해 많은 유전자 변형을 테스트함으로써 최적화된다.
부자연스러운 염기쌍
기존의 핵산 합성은 아데닌, 티민, 구아닌, 시토신 등 4개의 염기쌍만을 사용하는 반면, 향후 올리고뉴클레오티드 합성은 자연에서 발생하지 않는 인공적으로 설계되고 합성된 핵산염기쌍의 사용을 포함할 수 있다.
2012년, 캘리포니아 샌디에고에 있는 스크립스 연구소의 화학 생물학자인 플로이드 롬스버그가 이끄는 미국 과학자 그룹은 그의 팀이 부자연스러운 염기쌍을 설계했다고 발표했다.두 개의 새로운 인공 뉴클레오티드 또는 부자연스러운 염기쌍(UBP)은 d5SICS와 dNaM으로 명명되었다.좀 더 기술적으로, 소수성 핵산염기를 가진 이러한 인공 뉴클레오티드는 DNA에서 (d5SICS–dNaM) 복합체 또는 염기쌍을 형성하는 두 개의 융합 방향족 고리를 특징으로 한다.2014년 스크립스 연구소의 같은 팀은 자연 T-A와 C-G 염기쌍을 포함하는 플라스미드로 알려진 원형 DNA를 UBP 롬스버그의 연구소가 설계한 가장 뛰어난 성능의 UBP Romesberg의 실험실과 합성하여 부자연스러운 복제를 성공적으로 수행한 일반적인 박테리아 대장균의 세포에 삽입했다고 보고했습니다.기본 쌍을 여러 세대에 걸쳐 만듭니다.이것은 살아있는 유기체가 확장된 유전자 코드를 따라 다음 세대에 전달되는 최초의 알려진 사례이다.이는 부분적으로 d5SICSTP와 dNaMTP의 트리포스페이트를 대장균에 효율적으로 수입하는 뉴클레오티드 삼인산 트랜스포터를 발현하는 지지 조류 유전자를 추가함으로써 달성되었다.그런 다음 자연 세균 복제 경로는 d5SICS–dNaM을 포함하는 플라스미드를 정확하게 복제하기 위해 이들을 사용한다.
세 번째 염기쌍의 성공적인 통합은 DNA에 의해 암호화될 수 있는 아미노산의 수를 기존의 20개의 아미노산에서 이론적으로 가능한 172개로 크게 확장함으로써 생물들이 새로운 [20]단백질을 생산할 수 있는 가능성을 확장하는 목표를 향한 중요한 돌파구이다.미래에는 이러한 부자연스러운 염기쌍이 합성되어 DNA 인쇄 방법을 통해 올리고뉴클레오티드에 통합될 수 있다.
DNA조립체
따라서 DNA 인쇄는 특정 생물학적 기능(예: 촉진제, 전사 조절 시퀀스 또는 개방형 판독 프레임)[21]을 인코딩하는 DNA의 배열로 정의되는 DNA 부분을 생산하는데 사용될 수 있습니다.그러나 올리고뉴클레오티드 합성은 일반적으로 수백 개의 염기쌍보다 긴 올리고뉴클레오티드 배열을 정확하게 생산할 수 없기 때문에 기능성 유전자, 다중 유전자 회로 또는 전체 합성 염색체 또는 게놈을 만들기 위해 DNA 조립 방법이 사용되어야 한다.일부 DNA 조립 기술은 DNA 부분을 결합하기 위한 프로토콜만 정의하는 반면, 다른 기술은 또한 그들과 호환되는 DNA 부분의 형식에 대한 규칙을 정의합니다.이 과정들은 전체 염색체나 게놈을 조립할 수 있도록 확장될 수 있다.최근 몇 년 동안 서로 다른 DNA 조립 표준의 수가 급증하고 있으며, 2015년 현재 14개의 다른 조립 표준이 개발되었으며,[22] 각각 장단점이 있습니다.전반적으로, DNA 조립 표준의 개발은 합성 생물학의 흐름을 크게 촉진하고, 연구 그룹 간의 물질 교환을 지원하며, 모듈러형 및 재사용 가능한 DNA 부품을 [22]만들 수 있게 했다.
다양한 DNA 조립 방법은 크게 엔도핵산가수분해효소 매개 조립체, 부위 특이적 재조합 및 긴 겹침 [22]기반 조립체의 세 가지 범주로 분류될 수 있다.각 방법 그룹에는 고유한 특성과 장단점이 있습니다.
엔도핵산가수분해효소 매개 어셈블리
엔도핵산가수분해효소는 핵산 세그먼트를 인식하고 분해하는 효소이며 DNA 조립을 지시하는 데 사용될 수 있다.다양한 유형의 제한 효소 중 II형 제한 효소는 가장 일반적으로 사용 가능하며, 그 이유는 그 절단 부위가 인식 부위 근처에 위치하기 때문이다.따라서 엔도핵산가수분해효소 매개 조립법은 DNA 부분과 조립 프로토콜을 정의하기 위해 이 특성을 이용한다.
바이오브릭스
BioBricks 어셈블리 표준은 Tom Knight에 의해 2003년에 설명되고 소개되었으며 [23]그 이후로 지속적으로 업데이트되고 있습니다.현재 가장 일반적으로 사용되는 BioBricks 표준은 조립 표준 10 또는 BBF RFC 10입니다.BioBricks는 DNA 부품이 BioBricks 어셈블리 방법과 호환되기 위해 필요한 접두사 및 접미사 시퀀스를 정의하여 BioBricks 형식의 모든 DNA 부품을 결합할 수 있도록 합니다.
프리픽스에는 EcoRI, NotI 및 XBaI 제한 사이트가 포함되어 있으며, 서픽스에는 SpI, NotI 및 PstI 제한 사이트가 포함되어 있습니다.프리픽스 및 서픽스 영역 이외에서는 DNA 부분에 이러한 제한 사이트를 포함할 수 없습니다.두 개의 BioBrick 부품을 결합하기 위해 플라스미드 중 하나는 EcoRI 및 SpeI로 소화되고 두 번째 플라스미드는 EcoRI 및 XbaI로 소화됩니다.2개의 EcoRI 오버행은 상보적이며, 따라서 함께 아닐되며 SpeI와 XbaI는 상보적인 오버행도 생성되어 함께 결합할 수 있습니다.결과 플라스미드는 원래 접두사 및 접미사 시퀀스를 포함하므로 더 많은 BioBricks [24]부품과 결합하는 데 사용할 수 있습니다.이러한 특성 때문에, BioBricks 조립 표준은 사실상 무의미하다고 알려져 있습니다.그러나 두 개의 융합된 BioBrick 사이에 "scar" 배열(TACTAG 또는 TACTAG)이 형성될 것이다.이는 티로신과 정지 코돈에 대한 6bp 흉터 배열 코드로서 BioBricks가 융합 단백질 생성에 사용되는 것을 방지하여 첫 번째 도메인이 발현된 후 번역이 종료되는 반면, 8bp 흉터 배열은 프레임 시프트를 유발하여 코돈의 지속적인 판독을 방해한다.예를 들어 6bp 흉터를 주는 흉터 시퀀스 또는 정지 코돈을 포함하지 않는 흉터 시퀀스를 제공하기 위해 BB-2 어셈블리, BglBricks 어셈블리, Silver 어셈블리 및 Freiburg 어셈블리와 같은 다른 어셈블리 표준을 설계했습니다.[25][26][27][28]
BioBrick 부품을 조립하는 가장 쉬운 방법은 위에서 설명했지만, 표준 어셈블리에 비해 몇 가지 이점을 제공하는 다른 일반적으로 사용되는 조립 방법도 몇 가지 있습니다.3개의 항생제(3A) 어셈블리를 사용하면 항생제 선택을 통해 올바른 어셈블리를 선택할 수 있으며, 증폭된 삽입 어셈블리는 3A [29][30]어셈블리에서 볼 수 있는 낮은 변환 효율성을 극복하고자 합니다.
BioBrick 조립 표준은 또한 DNA 조립을 위해 다른 유형의 엔도핵산 분해 효소를 사용하는 데 영감을 주는 역할을 했습니다.예를 들어 iBrick 표준과 HomeRun 벡터 조립 표준 모두 II형 제한 [31][32]효소 대신 호밍 엔도핵산가수분해효소를 사용한다.
타입 II 제한 핵산가수분해효소 어셈블리
일부 조립 방법은 타입 II 제한 핵산가수분해효소를 사용하기도 한다.이것들은 인식 부위에서 몇 개의 염기쌍을 절단하기 때문에 다른 타입 II 핵산 분해 효소와 다르다.그 결과 원하는 시퀀스를 포함하도록 오버행 시퀀스를 변경할 수 있다.이를 통해 타입 II의 어셈블리 방법에는 두 가지 장점이 있습니다. 즉, "스칼리스" 어셈블리가 가능하고, 1포트 멀티 부품 어셈블리가 가능합니다.타입 II 엔도핵산가수분해효소를 사용하는 조립 방법에는 골든 게이트와 그 관련 변종들이 있다.
골든 게이트 복제
골든 게이트 조립 프로토콜은 흉터 배열 없이 최종 구조를 제공하는 DNA 조립 방법을 정의하기 위해 Engler 등에 의해 정의되었으며, 원래의 제한 부위도 결여되어 있다.이것은 단백질 접힘이나 발현에 부정적인 영향을 미칠 수 있는 원치 않는 단백질 서열을 포함하지 않고 단백질이 발현될 수 있도록 한다.4 염기쌍의 오버행을 생성하는 BsaI 제한 효소를 사용함으로써 최대 240개의 고유한 비회문 배열이 [33]조립에 사용될 수 있습니다.
플라스미드 설계 및 조립
Golden Gate 복제에서 조립되는 각 DNA 조각은 플라스미드에 배치되며, 프로그램된 돌출된 염기서열을 포함하는 안쪽을 향한 BsaI 제한 부위에 의해 측면으로 배치됩니다.각 DNA 조각에 대해, 3' 돌출 배열은 다음 하류 DNA 조각의 5' 돌출을 보완합니다.첫 번째 단편에 대해 5' 돌출부는 목적 플라스미드의 5' 돌출부를 보완하고, 마지막 단편의 3' 돌출부는 목적 플라스미드의 3' 돌출부를 보완한다.이러한 설계를 통해 모든 DNA 조각이 올바른 순서로 배열된 상태에서 (모든 반응물이 함께 혼합된) 1포트 반응으로 조립될 수 있습니다.원래 타깃 플라스미드에 [33]있던 스크리닝 카세트의 기능 상실을 검출함으로써 조립에 성공한 구조물을 선택한다.
MoClo 및 Golden Braid
원래 Golden Gate Assembly는 대상 벡터에서 단일 구성만 만들 수 있습니다.이 구조가 이후 반응에서 진입 벡터로 사용될 수 있도록 MoClo 및 Golden Braid 표준을 [34]설계했습니다.
MoClo 표준에는 여러 계층의 DNA 어셈블리가 정의되어 있습니다.
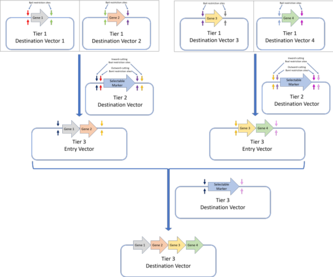
- 계층 1: 계층 1 어셈블리는 표준 Golden Gate 어셈블리이며, 유전자는 구성 요소(UTR, 프로모터, 리보솜 결합 부위 또는 터미네이터 배열과 같은 유전 요소를 코드하는 DNA 부품)에서 조립됩니다.계층 1의 행선지 벡터의 삽입 부위 옆에는 한 쌍의 내향 절단 BpiI 제한 부위가 있다.이것에 의해, 이러한 플라스미드를 계층 2 행선지 벡터의 엔트리 벡터로 사용할 수 있습니다.MoClo 조립 표준을 통해 Golden Gate 구조물을 후속 계층에서 추가로 조립할 수 있습니다.이 예에서 계층 1 Golden Gate 어셈블리를 통해 조립된 4개의 유전자는 계층 2 어셈블리에서 다중 유전자 구조로 조립된다.또한 Golden Braid 어셈블리 표준은 Golden Gate 어셈블리의 첫 번째 계층에 구축되며 쌍으로 구성된 프로토콜을 통해 추가 계층을 조립합니다.4개의 Tier 1 행선지 벡터(Golden Gate 어셈블리를 통해 조립됨)는 2개의 Tier 2 행선지 벡터에 조립되어 계층 3 행선지 벡터의 계층 3 엔트리 벡터로 사용됩니다.교대제한효소(계층2의 BpiI와 계층3의 BsaI)를 사용한다.MoClo 및 Golden Braid 조립 표준은 원래 Golden Gate 조립 표준의 파생 모델입니다.
- 계층 2: 계층 2 어셈블리는 계층 1 어셈블리에서 조립된 유전자를 다중 유전자 구조로 더 조립하는 것을 포함합니다.한층 더 상위층의 어셈블리가 필요한 경우는, 삽입 부위의 측면에 내측 절삭 BsaI 제한 부위를 추가할 수 있다.그런 다음 이러한 벡터를 상위 계층 구조의 엔트리 벡터로 사용할 수 있습니다.
각 조립 계층은 금지된 사이트의 수를 최소화하기 위해 BsaI 및 BpiI 제한 사이트를 번갈아 사용하며, 각 계층의 순차 조립은 Golden Gate 플라스미드 설계에 따라 이루어집니다.전반적으로 MoClo 표준을 사용하면 여러 개의 전사 유닛을 포함하는 구조를 조립할 수 있으며, 모두 다른 DNA 부품에서 조립되어 일련의 원포트 Golden Gate 반응에 의해 조립할 수 있습니다.그러나 MoClo 표준의 한 가지 단점은 최종 구조가 4개 미만의 구성 [35]요소를 필요로 하는 경우 생물학적 기능이 없는 '더미 부품'을 사용해야 한다는 것입니다.한편, 골든 브레이드 표준은 쌍으로 구성된 골든 게이트 조립 표준을 도입했습니다.
Golden Braid 표준에서는 MoClo와 동일한 계층형 어셈블리를 사용하지만 각 계층에는 2개의 DNA 단편(쌍별 접근법)의 조합만 포함됩니다.따라서 각 계층에서 유전자 쌍을 원하는 배열로 목적지 단편으로 복제하고, 이어서 두 개씩 순차적으로 조립한다.MoClo와 마찬가지로 Golden Braid 표준은 각 계층 간에 BsaI 및 BpiI 제한 효소를 번갈아 사용합니다.
Golden Gate 조립 방법과 그 변형체의 개발로 연구원들은 합성 생물학 작업 흐름을 가속화하기 위한 도구 키트를 설계할 수 있게 되었습니다.예를 들어 EcoFlex는 E를 위한 툴킷으로 개발되었습니다. DNA 부품에 MoClo 표준을 사용하는 대장균과 Chlamydomonas reinhardtii mircoalgae [36][37]엔지니어링을 위해 유사한 툴킷이 개발되었습니다.
사이트 고유의 재조합
부위 특이적 재조합은 제한 효소 대신 파이지 인테그레이스를 사용하여 DNA 조각에 제한 부위를 가질 필요성을 없앱니다.대신, 인테그라아제들은 고유한 부착(att) 부위를 사용하고 표적 단편과 대상 벡터 사이의 DNA 재배열을 촉매한다.Invitrogen Gateway 복제 시스템은 1990년대 말에 발명되었으며 두 가지 독점 효소 혼합물인 BP 클로나제와 LR 클로나제를 사용합니다.BP 클로나아제 혼합물은 attB와 attP 부위 간의 재결합을 촉매하여 하이브리드 attL과 attR 부위를 생성하고, LR 클로나아제 혼합물은 attL과 attR 부위의 재결합을 촉매하여 attB와 attP 부위를 제공한다.각 효소 혼합물은 특정 att 부위만을 인식하기 때문에 재조합이 매우 특이하고 원하는 [38]순서로 단편을 조립할 수 있다.
벡터 설계 및 조립
게이트웨이 클로닝은 독자적인 테크놀로지이기 때문에 모든 게이트웨이 대응은 제조원이 제공하는 게이트웨이 키트를 사용하여 수행해야 합니다.반응은 두 단계로 요약할 수 있습니다.첫 번째 단계는 대상 DNA 조각을 포함하는 엔트리 클론을 조립하는 것이고, 두 번째 단계는 대상 클론에 해당 조각을 삽입하는 것입니다.
- 엔트리 클론은 attP 사이트 옆에 있는 게이트웨이 카세트가 포함된 제공된 "공여자" 벡터를 사용하여 작성해야 합니다.게이트웨이 카세트에는 성공적으로 재조합된 엔트리 클론을 선택하고 생존할 수 있는 박테리아 자살 유전자(예: ccdB)가 포함되어 있습니다.한 쌍의 attB 부위가 대상 DNA 조각의 측면에 추가되며, BP 클로나아제 혼합물이 추가될 때 attP 부위와 재결합할 수 있습니다.엔트리 클론이 생성되고 관심의 단편은 attL 사이트에 의해 측면 처리됩니다.
- 행선지 벡터에는 게이트웨이 카세트도 부속되어 있습니다만, 그 대신에 attR 사이트의 페어가 붙어 있습니다.이 대상 플라스미드를 엔트리 클론 및 LR 클로나아제 혼합물과 혼합하면 attR 부위와 attL 부위 간에 재조합이 발생할 수 있습니다.대상 fragment가 정상적으로 삽입된 상태에서 대상 클론이 생성됩니다.치사 유전자가 원래 벡터에 삽입되고 이 플라스미드로 변형된 박테리아는 죽게 된다.따라서 원하는 벡터를 쉽게 선택할 수 있습니다.
게이트웨이 복제 방식의 초기 반복에서는 생성된 각 대상 복제에 대해 하나의 엔트리 복제만 사용할 수 있습니다.그러나 추가 연구에 따르면 4개의 직교 att 시퀀스가 추가로 생성되어 최대 4개의 서로 다른 DNA 조각을 조립할 수 있으며, 이 프로세스는 현재 멀티사이트 게이트웨이 [39]기술로 알려져 있습니다.
게이트웨이 복제 외에도 다른 인테그레이스를 사용하는 비상업적 방법도 개발되었습니다.예를 들어 Serine Integrational Assembly(SIRA) 방법은 δC31 Integrase를 사용하고, Site-Specific Reconnation-based Tandem Assembly(SSRTA) 방법은 Streptomyces page δBT1 Integrase를 사용한다.[40][41]HVAS(HomeRun Vector Assembly System)와 같은 다른 방법은 게이트웨이 클로닝 시스템을 기반으로 하며 합성 DNA [31]구조의 산업적 합성을 잠재적으로 지원할 수 있는 프로토콜을 설계하기 위해 호밍 엔듀클레이스를 추가로 통합합니다.
롱오버랩베이스어셈블리
최근 몇 년 동안 다양한 오버랩 기반 조립 방법이 개발되었습니다.가장 일반적으로 사용되는 방법 중 하나인 깁슨 조립법은 2009년에 개발되었으며 제한 효소나 인테그레이스를 사용하지 [42]않아도 되는 원포트 DNA 조립 방법을 제공합니다.다른 유사한 중복 기반 조립 방법으로는 원형 중합효소 확장 클로닝(CPEC), 배열 및 리가제 독립 클로닝(SLIC) 및 이음새 없는 결합 클로닝 추출물(SLicE)[43][44][45]이 있습니다.많은 오버랩 조립 방법이 존재함에도 불구하고, 깁슨 조립 방법이 여전히 가장 [46]인기 있습니다.위에 열거된 방법 외에도, 다른 연구자들은 Gibson 어셈블리와 다른 어셈블리 방법에 사용된 개념을 바탕으로 MODAL(Modular Overlap-Directed Assembly with Linkers) 전략 또는 BASIC(Biopart Assembly Standard for Idempotent Cloning) [47][48]방법과 같은 새로운 어셈블리 전략을 개발했습니다.
깁슨 조립체
깁슨 조립 방법은 비교적 간단한 DNA 조립 방법으로, 5' T5 핵산가수분해효소, 퓨전 DNA 중합효소 및 Taq DNA 연결효소 등 몇 가지 시약만 추가로 필요로 한다.조립되는 DNA 조각은 조립되는 순서대로 겹치는 5'와 3'의 끝을 가지도록 합성된다.이러한 시약은 DNA 조각과 함께 혼합되어 50°C에서 조립되며 다음과 같은 반응이 발생합니다.
- T5핵산가수분해효소는 각 DNA 조각의 5' 끝에서 DNA를 씹어먹어 각 DNA 조각의 3' 돌출부를 노출시킵니다.
- 인접한 DNA 조각의 상보적인 돌출부는 상보적인 염기쌍을 통해 풀린다.
- 퓨전 DNA 중합효소는 파편들이 풀리는 틈새를 메운다.
- Taq DNA 연결효소는 양쪽 DNA 가닥의 상처를 치료합니다.
T5 엑소핵산가수분해효소는 열연성이기 때문에 최초 씹기 단계 후 50 °C에서 비활성화됩니다.따라서 제품은 안정적이며 원하는 순서로 파편을 조립할 수 있습니다.이 원포트 프로토콜은 최대 5개의 서로 다른 조각들을 정확하게 조립할 수 있는 반면, 몇몇 상용 공급자들은 2단계 [49]반응으로 최대 15개의 서로 다른 조각들을 정확하게 조립할 수 있는 키트를 가지고 있습니다.그러나 Gibson 조립 프로토콜은 빠르고 비교적 적은 시약을 사용하는 반면, 각 조각은 인접한 조각과 겹치는 시퀀스를 포함하고 PCR을 통해 증폭되도록 설계되어야 하므로 맞춤형 DNA 합성이 필요합니다.긴 fragment, GC 함량이 높은 fragment 또는 반복 시퀀스를 [48]사용하는 경우 PCR에 대한 의존도는 반응의 충실도에 영향을 줄 수 있습니다.
모듈
MODAL 전략은 각 DNA 조각에 대해 수행해야 하는 커스터마이즈의 양을 줄이기 위해 "링커"로 알려진 오버랩 시퀀스를 정의합니다.링커는 R2oDNA Designer 소프트웨어를 사용하여 설계되었으며 오버랩 영역은 깁슨 어셈블리 및 기타 오버랩 어셈블리 방법과 호환되도록 45bp 길이로 설계되었습니다.이러한 링커를 조립하는 부품에 장착하기 위해 PCR은 15bp 프리픽스 및 서픽스 어댑터 시퀀스를 포함하는 부품 고유의 프라이머를 사용하여 반송됩니다.그런 다음 링커는 두 번째 PCR 반응을 통해 어댑터 시퀀스에 부착됩니다.DNA fragment를 배치하기 위해 원하는 업스트림 fragment의 서픽스와 원하는 다운스트림 fragment의 프리픽스에 동일한 링커를 연결합니다.링커를 연결하면 Gibson 어셈블리, CPEC 또는 기타 오버랩 어셈블리 방법을 모두 사용하여 원하는 순서로 DNA 조각을 조립할 수 있습니다.
기본의
BASIC 조립 전략은 2015년에 개발되었으며 이전 조립 기술의 한계를 해결하기 위해 6가지 핵심 개념을 통합했습니다. 즉, 표준 재사용 가능 부품, 단일 계층 형식(모든 부품은 동일한 형식으로 동일한 공정을 사용하여 조립됨), 유휴 복제, 병렬(멀티파트) DNA 조립, 크기 인데페입니다.ndence;자동화성.[48]
DNA 부품 및 링커 설계
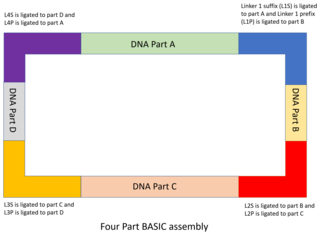
DNA 부분은 설계되어 저장 플라스미드로 복제되며, 그 부분은 통합 접두사(iP)와 통합 접미사(iS) 시퀀스로 양쪽에 배치됩니다.iP 및 iS 시퀀스에는 BASIC [48]링커를 보완하는 오버행이 포함된 내향 BSAI 제한 사이트가 포함됩니다.MODAL과 마찬가지로 BASIC에서 사용되는 7개의 표준 링커는 R2oDNA Designer 소프트웨어로 설계되어 섀시 게놈에 대한 상동성을 갖는 배열을 포함하지 않으며 2차 구조 배열, 제한 부위 또는 리보솜 결합 부위 등 원하지 않는 배열을 포함하지 않도록 스크리닝되었다.각 링커 시퀀스는 BsaI 제한 사이트를 보완하는4 bp 오버행, 12 bp 더블트루드시퀀스 및 21 bp 오버랩시퀀스를 나머지 절반과 공유합니다.업스트림 DNA 부분에 바인드되는 절반은 서픽스링커부(L1S 등)라고 불리며 다운스트림 부분에 바인드되는 절반은 프리픽스링커부(L1P 등)라고 불립니다.이 링커들은 DNA 부품들을 함께 조립하는 기초를 형성합니다.
표준 BASIC 링커는 조립 순서를 지시하는 것 외에 다른 기능을 수행하도록 변경할 수도 있습니다.Idempotent 어셈블리를 가능하게 하기 위해 링커는 추가 메틸화 iP 및 iS 시퀀스를 삽입하여 BsaI에 의해 인식되지 않도록 설계되어 있습니다.이 메틸화는 형질전환 및 생체내 플라스미드 복제에 따라 손실되며 플라스미드는 추출, 정제 및 추가 반응에 사용될 수 있다.
링커 배열은 비교적 길기 때문에(표준 링커의 경우 45bp), 조립 중에 필요한 DNA 부품의 수를 줄이기 위해 기능성 DNA 배열을 통합할 수 있습니다.BASIC 어셈블리 표준은 다양한 강도의 RBS가 내장된 여러 링커를 제공합니다.마찬가지로, 여러 단백질 도메인을 포함하는 융합 단백질의 구축을 용이하게 하기 위해, DNA 구조의 완전한 판독을 가능하게 하는 여러 융합 링커도 설계되었다.이러한 융합 링커는 15개의 아미노산 글리신과 세린 폴리펩타이드를 코드화하며, 이는 다중 도메인을 가진 융합 단백질에 대한 이상적인 링커 펩타이드이다.
어셈블리
최종 구성의 조립에는 크게 세 가지 단계가 있습니다.
- 우선 저장 플라스미드에서 DNA 부분을 절제하여 3' 및 5' 끝에 BsaI가 돌출된 DNA 단편을 얻는다.
- 다음으로 각 링커부를 T4 DNA 리가아제로 배양하여 각각의 DNA부에 부착한다.각 DNA 부품에는 조립 순서를 지시하기 위해 2개의 다른 링커로부터의 접미사와 접두사 링커 부분이 있습니다.예를 들어 시퀀스의 첫 번째 부분에는 L1P와 L2S가 있고 두 번째 부분에는 L2P와 L3S가 연결되어 있습니다.링커 부품을 변경하여 조립 순서를 변경할 수 있습니다.
- 마지막으로 링커가 부착된 부분을 50℃에서 배양하여 플라스미드로 조립한다.P와 S 링커의 21 bp 돌출부와 최종 구성은 복제를 위한 박테리아 세포로 변형될 수 있습니다.단일 가닥 닉은 변환 후 생체 내에서 수리되어 플라스미드로 복제된 안정적인 최종 구조를 생성한다.
적용들
DNA 인쇄와 DNA 조립 방법이 지난 몇 [50]년 동안 상업적 유전자 합성이 점진적으로 그리고 기하급수적으로 저렴해짐에 따라, 인공 유전자 합성은 새로운 DNA 배열과 단백질 기능을 만들고 설계하기 위한 강력하고 유연한 엔지니어링 도구이다.합성 생물학 외에도, 이종 유전자 발현, 백신 개발, 유전자 치료, 분자 공학과 같은 다양한 연구 분야는 단백질과 펩타이드를 [51]코드하기 위해 DNA를 합성하는 빠르고 저렴한 방법을 갖는 것으로부터 큰 이익을 얻을 수 있을 것이다.DNA 인쇄와 조립에 사용된 방법들은 DNA를 정보 저장 매체로 사용할 수 있게 해주기도 했다.
세균 게놈 합성
신시아와 마이코플라스마 연구실
2007년 6월 28일, J. Craig Venter Institute의 한 팀은 Science Express에 그들이 마이코플라스마 마이코이데스 박테리아의 자연 DNA를 마이코플라스마 카프리콜룸 세포에 성공적으로 이식하여, 마이코이데스처럼 [52]행동하는 박테리아를 만들었다는 기사를 실었다.
2007년 10월 6일, 크레이그 벤터는 영국의 가디언지와의 인터뷰에서 같은 팀이 마이코플라스마 제니탈리움 단일 염색체의 변형된 버전을 인공적으로 합성했다고 발표했다.그 염색체는 살아있는 박테리아에서 실험할 필요가 없는 모든 유전자를 제거하기 위해 수정되었다.이 최소 게놈 프로젝트의 다음 계획된 단계는 합성된 최소 게놈을 오래된 DNA가 제거된 박테리아 세포에 이식하는 것이다; 그 결과 생기는 박테리아는 마이코플라스마 라보토리엄이라고 불리게 될 것이다.다음날 캐나다 생명윤리단체인 ETC 그룹은 그들의 대표인 Pat Mooney를 통해 Venter의 "창작"은 "거의 모든 것을 만들 수 있는 섀시"라고 성명서를 발표했다.합성된 게놈은 아직 작동하는 [53]세포에 이식되지 않았다.
2010년 5월 21일 사이언스는 벤터 그룹이 컴퓨터 기록에서 마이코플라스마 마이코이데스 박테리아의 게놈을 성공적으로 합성해 DNA를 제거한 마이코플라스마 카프리콜럼 박테리아의 기존 세포에 이식했다고 보도했다."합성" 박테리아는 생존할 수 있었고, 즉 수십억 번 복제할 수 있었다.연구팀은 원래 이전에 연구해 온 M. 제니탈리움 박테리아를 사용할 계획이었으나 M.마이코이데스 박테리아가 훨씬 더 빨리 자라기 때문에 [54]M.마이코이데스균으로 바꿨다.벤터는 그것을 "첫 번째 종…"이라고 표현했다.부모가 컴퓨터가 되는 것"이라고 말했다.[55]이 변형된 박테리아는 ETC에 의해 "신티아"라고 불립니다.Venter 대변인은 이 글의 작성 시점에서는 돌파구를 확인하는 것을 거부했습니다.
합성 효모 2.0
합성효모 2.0 프로젝트의 일환으로 전 세계 다양한 연구단체가 참여해 합성효모 게놈을 합성하고 있으며, 이 과정을 통해 모델 생물인 Saccharomyces cerevisae의 [56]게놈을 최적화한다.효모 2.0 프로젝트는 위에서 논의한 다양한 DNA 조립 방법을 적용했으며, 2014년 3월 뉴욕대 랭곤 메디컬 센터의 제프 보케는 그의 팀이 [57][58]세레비새 염색체 III를 합성했다고 밝혔다.이 절차는 원래 염색체의 유전자를 합성 버전으로 교체하는 것을 포함했고 완성된 합성 염색체는 효모 세포에 통합되었다.원래 염색체의 316,667쌍보다 적은 273,871쌍의 DNA 염기쌍을 설계하고 생성해야 했다.2017년 3월, 16개의 염색체 중 6개의 합성이 완료되었고, 나머지 염색체들의 합성은 아직 [59]진행 중이다.
「 」를 참조해 주세요.
메모들
- ^ Stein R (7 May 2015). "DNA 'Printing' A Big Boon To Research, But Some Raise Concerns". All Things Considered. National Public Radio.
- ^ a b Khorana HG, Agarwal KL, Büchi H, Caruthers MH, Gupta NK, Kleppe K, et al. (December 1972). "Studies on polynucleotides. 103. Total synthesis of the structural gene for an alanine transfer ribonucleic acid from yeast". Journal of Molecular Biology. 72 (2): 209–17. doi:10.1016/0022-2836(72)90146-5. PMID 4571075.
- ^ a b Itakura K, Hirose T, Crea R, Riggs AD, Heyneker HL, Bolivar F, Boyer HW (December 1977). "Expression in Escherichia coli of a chemically synthesized gene for the hormone somatostatin". Science. 198 (4321): 1056–63. Bibcode:1977Sci...198.1056I. doi:10.1126/science.412251. PMID 412251.
- ^ a b Edge MD, Green AR, Heathcliffe GR, Meacock PA, Schuch W, Scanlon DB, et al. (August 1981). "Total synthesis of a human leukocyte interferon gene". Nature. 292 (5825): 756–62. Bibcode:1981Natur.292..756E. doi:10.1038/292756a0. PMID 6167861. S2CID 4330168.
- ^ Shukman D (2014-03-27). "Synthetic DNA advance is hailed". BBC News. Retrieved 2020-04-11.
- ^ Kimoto M, Yamashige R, Matsunaga K, Yokoyama S, Hirao I (May 2013). "Generation of high-affinity DNA aptamers using an expanded genetic alphabet". Nature Biotechnology. 31 (5): 453–7. doi:10.1038/nbt.2556. PMID 23563318. S2CID 23329867.
- ^ Malyshev DA, Dhami K, Quach HT, Lavergne T, Ordoukhanian P, Torkamani A, Romesberg FE (July 2012). "Efficient and sequence-independent replication of DNA containing a third base pair establishes a functional six-letter genetic alphabet". Proceedings of the National Academy of Sciences of the United States of America. 109 (30): 12005–10. Bibcode:2012PNAS..10912005M. doi:10.1073/pnas.1205176109. PMC 3409741. PMID 22773812.
- ^ Malyshev DA, Dhami K, Lavergne T, Chen T, Dai N, Foster JM, et al. (May 2014). "A semi-synthetic organism with an expanded genetic alphabet". Nature. 509 (7500): 385–8. Bibcode:2014Natur.509..385M. doi:10.1038/nature13314. PMC 4058825. PMID 24805238.
- ^ a b Fuhrmann M, Oertel W, Hegemann P (August 1999). "A synthetic gene coding for the green fluorescent protein (GFP) is a versatile reporter in Chlamydomonas reinhardtii". The Plant Journal. 19 (3): 353–61. doi:10.1046/j.1365-313X.1999.00526.x. PMID 10476082.
- ^ Mandecki W, Bolling TJ (August 1988). "FokI method of gene synthesis". Gene. 68 (1): 101–7. doi:10.1016/0378-1119(88)90603-8. PMID 3265397.
- ^ Stemmer WP, Crameri A, Ha KD, Brennan TM, Heyneker HL (October 1995). "Single-step assembly of a gene and entire plasmid from large numbers of oligodeoxyribonucleotides". Gene. 164 (1): 49–53. doi:10.1016/0378-1119(95)00511-4. PMID 7590320.
- ^ Gao X, Yo P, Keith A, Ragan TJ, Harris TK (November 2003). "Thermodynamically balanced inside-out (TBIO) PCR-based gene synthesis: a novel method of primer design for high-fidelity assembly of longer gene sequences". Nucleic Acids Research. 31 (22): 143e–143. doi:10.1093/nar/gng143. PMC 275580. PMID 14602936.
- ^ Young L, Dong Q (April 2004). "Two-step total gene synthesis method". Nucleic Acids Research. 32 (7): e59. doi:10.1093/nar/gnh058. PMC 407838. PMID 15087491.
- ^ Hillson NJ, Rosengarten RD, Keasling JD (January 2012). "j5 DNA assembly design automation software". ACS Synthetic Biology. 1 (1): 14–21. doi:10.1021/sb2000116. PMID 23651006.
- ^ Hoover DM, Lubkowski J (May 2002). "DNAWorks: an automated method for designing oligonucleotides for PCR-based gene synthesis". Nucleic Acids Research. 30 (10): 43e–43. doi:10.1093/nar/30.10.e43. PMC 115297. PMID 12000848.
- ^ Villalobos A, Ness JE, Gustafsson C, Minshull J, Govindarajan S (June 2006). "Gene Designer: a synthetic biology tool for constructing artificial DNA segments". BMC Bioinformatics. 7: 285. doi:10.1186/1471-2105-7-285. PMC 1523223. PMID 16756672.
- ^ Tian J, Gong H, Sheng N, Zhou X, Gulari E, Gao X, Church G (December 2004). "Accurate multiplex gene synthesis from programmable DNA microchips" (PDF). Nature. 432 (7020): 1050–4. Bibcode:2004Natur.432.1050T. doi:10.1038/nature03151. hdl:2027.42/62677. PMID 15616567. S2CID 4373350.
- ^ Matzas M, Stähler PF, Kefer N, Siebelt N, Boisguérin V, Leonard JT, et al. (December 2010). "High-fidelity gene synthesis by retrieval of sequence-verified DNA identified using high-throughput pyrosequencing". Nature Biotechnology. 28 (12): 1291–4. doi:10.1038/nbt.1710. PMC 3579223. PMID 21113166.
- ^ Schwartz JJ, Lee C, Shendure J (September 2012). "Accurate gene synthesis with tag-directed retrieval of sequence-verified DNA molecules". Nature Methods. 9 (9): 913–5. doi:10.1038/nmeth.2137. PMC 3433648. PMID 22886093.
- ^ Weidman C (2017-12-06). "Expanding the genetic alphabet". Blog. Harvard University. Retrieved 2020-04-17.
- ^ "Help:Synthetic Biology - parts.igem.org". parts.igem.org. Retrieved 2020-04-11.
- ^ a b c Casini A, Storch M, Baldwin GS, Ellis T (September 2015). "Bricks and blueprints: methods and standards for DNA assembly". Nature Reviews. Molecular Cell Biology. 16 (9): 568–76. doi:10.1038/nrm4014. hdl:10044/1/31281. PMID 26081612. S2CID 3502437.
- ^ Knight T (2003). "Idempotent Vector Design for Standard Assembly of Biobricks". hdl:1721.1/21168.
{{cite journal}}:Cite 저널 요구 사항journal=(도움말) - ^ Røkke G, Korvald E, Pahr J, Oyås O, Lale R (2014). "BioBrick assembly standards and techniques and associated software tools". In Valla S, Lale R (eds.). DNA Cloning and Assembly Methods. Methods in Molecular Biology. Vol. 1116. Clifton, N.J. pp. 1–24. doi:10.1007/978-1-62703-764-8_1. ISBN 978-1-62703-763-1. PMID 24395353.
- ^ Knight T (2008-11-19). "Draft Standard for Biobrick BB-2 Biological Parts". hdl:1721.1/45139.
{{cite journal}}:Cite 저널 요구 사항journal=(도움말) - ^ Anderson JC, Dueber JE, Leguia M, Wu GC, Goler JA, Arkin AP, Keasling JD (January 2010). "BglBricks: A flexible standard for biological part assembly". Journal of Biological Engineering. 4 (1): 1. doi:10.1186/1754-1611-4-1. PMC 2822740. PMID 20205762.
- ^ Phillips I, Silver P (2006-04-20). "A New Biobrick Assembly Strategy Designed for Facile Protein Engineering". hdl:1721.1/32535.
{{cite journal}}:Cite 저널 요구 사항journal=(도움말) - ^ Grünberg R, Arndt K, Müller K (2009-04-18). "Fusion Protein (Freiburg) Biobrick assembly standard". hdl:1721.1/45140.
{{cite journal}}:Cite 저널 요구 사항journal=(도움말) - ^ Shetty R, Lizarazo M, Rettberg R, Knight TF (2011). "Assembly of BioBrick standard biological parts using three antibiotic assembly". Methods in Enzymology. 498: 311–26. doi:10.1016/B978-0-12-385120-8.00013-9. hdl:1721.1/65066. ISBN 9780123851208. PMID 21601683.
- ^ Speer MA, Richard TL (December 2011). "Amplified insert assembly: an optimized approach to standard assembly of BioBrickTM genetic circuits". Journal of Biological Engineering. 5 (1): 17. doi:10.1186/1754-1611-5-17. PMC 3287150. PMID 22176971.
- ^ a b Li MV, Shukla D, Rhodes BH, Lall A, Shu J, Moriarity BS, Largaespada DA (2014-06-24). "HomeRun Vector Assembly System: a flexible and standardized cloning system for assembly of multi-modular DNA constructs". PLOS ONE. 9 (6): e100948. Bibcode:2014PLoSO...9j0948L. doi:10.1371/journal.pone.0100948. PMC 4069157. PMID 24959875.
- ^ Liu JK, Chen WH, Ren SX, Zhao GP, Wang J (2014-10-20). "iBrick: a new standard for iterative assembly of biological parts with homing endonucleases". PLOS ONE. 9 (10): e110852. Bibcode:2014PLoSO...9k0852L. doi:10.1371/journal.pone.0110852. PMC 4203835. PMID 25329380.
- ^ a b Engler C, Kandzia R, Marillonnet S (2008-11-05). "A one pot, one step, precision cloning method with high throughput capability". PLOS ONE. 3 (11): e3647. Bibcode:2008PLoSO...3.3647E. doi:10.1371/journal.pone.0003647. PMC 2574415. PMID 18985154.
- ^ Weber E, Engler C, Gruetzner R, Werner S, Marillonnet S (February 2011). "A modular cloning system for standardized assembly of multigene constructs". PLOS ONE. 6 (2): e16765. Bibcode:2011PLoSO...616765W. doi:10.1371/journal.pone.0016765. PMC 3041749. PMID 21364738.
- ^ Klein CA, Emde L, Kuijpers A, Sobetzko P (2019-10-17). "MoCloFlex: A Modular Yet Flexible Cloning System". Frontiers in Bioengineering and Biotechnology. 7: 271. doi:10.3389/fbioe.2019.00271. PMC 6843054. PMID 31750294.
- ^ Moore SJ, Lai HE, Kelwick RJ, Chee SM, Bell DJ, Polizzi KM, Freemont PS (October 2016). "EcoFlex: A Multifunctional MoClo Kit for E. coli Synthetic Biology". ACS Synthetic Biology. 5 (10): 1059–1069. doi:10.1021/acssynbio.6b00031. PMID 27096716.
- ^ Crozet P, Navarro FJ, Willmund F, Mehrshahi P, Bakowski K, Lauersen KJ, et al. (September 2018). "Birth of a Photosynthetic Chassis: A MoClo Toolkit Enabling Synthetic Biology in the Microalga Chlamydomonas reinhardtii". ACS Synthetic Biology. 7 (9): 2074–2086. doi:10.1021/acssynbio.8b00251. PMID 30165733. S2CID 52131500.
- ^ Reece-Hoyes JS, Walhout AJ (January 2018). "Gateway Recombinational Cloning". Cold Spring Harbor Protocols. 2018 (1): pdb.top094912. doi:10.1101/pdb.top094912. PMC 5935001. PMID 29295908.
- ^ Sasaki Y, Sone T, Yoshida S, Yahata K, Hotta J, Chesnut JD, et al. (February 2004). "Evidence for high specificity and efficiency of multiple recombination signals in mixed DNA cloning by the Multisite Gateway system". Journal of Biotechnology. 107 (3): 233–43. doi:10.1016/j.jbiotec.2003.10.001. PMID 14736459.
- ^ Colloms SD, Merrick CA, Olorunniji FJ, Stark WM, Smith MC, Osbourn A, et al. (February 2014). "Rapid metabolic pathway assembly and modification using serine integrase site-specific recombination". Nucleic Acids Research. 42 (4): e23. doi:10.1093/nar/gkt1101. PMC 3936721. PMID 24225316.
- ^ Zhang L, Zhao G, Ding X (2011-11-03). "Tandem assembly of the epothilone biosynthetic gene cluster by in vitro site-specific recombination". Scientific Reports. 1 (1): 141. Bibcode:2011NatSR...1E.141Z. doi:10.1038/srep00141. PMC 3216622. PMID 22355658.
- ^ Gibson DG, Young L, Chuang RY, Venter JC, Hutchison CA, Smith HO (May 2009). "Enzymatic assembly of DNA molecules up to several hundred kilobases". Nature Methods. 6 (5): 343–5. doi:10.1038/nmeth.1318. PMID 19363495. S2CID 1351008.
- ^ Quan J, Tian J (July 2009). "Circular polymerase extension cloning of complex gene libraries and pathways". PLOS ONE. 4 (7): e6441. Bibcode:2009PLoSO...4.6441Q. doi:10.1371/journal.pone.0006441. PMC 2713398. PMID 19649325.
- ^ Li MZ, Elledge SJ (March 2007). "Harnessing homologous recombination in vitro to generate recombinant DNA via SLIC". Nature Methods. 4 (3): 251–6. doi:10.1038/nmeth1010. PMID 17293868. S2CID 30893882.
- ^ Zhang Y, Werling U, Edelmann W (April 2012). "SLiCE: a novel bacterial cell extract-based DNA cloning method". Nucleic Acids Research. 40 (8): e55. doi:10.1093/nar/gkr1288. PMC 3333860. PMID 22241772.
- ^ "How Gibson Assembly® is Changing Synthetic Biology". New England Biolabs. Retrieved 2020-04-14.
- ^ Casini A, MacDonald JT, De Jonghe J, Christodoulou G, Freemont PS, Baldwin GS, Ellis T (January 2014). "One-pot DNA construction for synthetic biology: the Modular Overlap-Directed Assembly with Linkers (MODAL) strategy". Nucleic Acids Research. 42 (1): e7. doi:10.1093/nar/gkt915. PMC 3874208. PMID 24153110.
- ^ a b c d Storch M, Casini A, Mackrow B, Fleming T, Trewhitt H, Ellis T, Baldwin GS (July 2015). "BASIC: A New Biopart Assembly Standard for Idempotent Cloning Provides Accurate, Single-Tier DNA Assembly for Synthetic Biology". ACS Synthetic Biology. 4 (7): 781–7. doi:10.1021/sb500356d. PMID 25746445.
- ^ "Gibson Assembly Protocol". Addgene. Retrieved 2020-04-14.
- ^ El Karoui M, Hoyos-Flight M, Fletcher L (2019). "Future Trends in Synthetic Biology-A Report". Frontiers in Bioengineering and Biotechnology. 7: 175. doi:10.3389/fbioe.2019.00175. PMC 6692427. PMID 31448268.
- ^ Kosuri S, Church GM (May 2014). "Large-scale de novo DNA synthesis: technologies and applications" (PDF). Nature Methods. 11 (5): 499–507. doi:10.1038/nmeth.2918. PMC 7098426. PMID 24781323.
- ^ Lartigue C, Glass JI, Alperovich N, Pieper R, Parmar PP, Hutchison CA, et al. (August 2007). "Genome transplantation in bacteria: changing one species to another". Science. 317 (5838): 632–8. Bibcode:2007Sci...317..632L. CiteSeerX 10.1.1.395.4374. doi:10.1126/science.1144622. PMID 17600181. S2CID 83956478.
- ^ Pilkington E (2009-10-06). "I am creating artificial life, declares US gene pioneer". The Guardian. London. Archived from the original on 28 May 2010. Retrieved 2010-05-22.
- ^ Pennisi, E. (2010-05-21). "Synthetic Genome Brings New Life to Bacterium" (PDF). Science. 328 (5981): 958–9. doi:10.1126/science.328.5981.958. PMID 20488994. Archived (PDF) from the original on 25 May 2010. Retrieved 2010-05-21.
- ^ "How scientists made 'artificial life'". BBC News. 2010-05-20. Archived from the original on June 1, 2013. Retrieved 2010-05-21.
- ^ "Yeast 2.0". Nature Communications Collection. Springer Nature Limited. Retrieved 2020-04-17.
- ^ Shukman D (27 March 2014). "Scientists hail synthetic chromosome advance". BBC News. Retrieved 2014-03-28.
- ^ Annaluru N, Muller H, Mitchell LA, Ramalingam S, Stracquadanio G, Richardson SM, et al. (April 2014). "Total synthesis of a functional designer eukaryotic chromosome". Science. 344 (6179): 55–8. Bibcode:2014Sci...344...55A. doi:10.1126/science.1249252. PMC 4033833. PMID 24674868.
- ^ 특별호 합성 효모 게놈 사이언스 2017년 3월 10일 제355권 제6329호