프라택신
Frataxin| FXN | |||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
 | |||||||||||||||||||||||||
| |||||||||||||||||||||||||
| 식별자 | |||||||||||||||||||||||||
| 별칭 | FXN, CyaY, FA, FARR, FRDA, X25, Frataxin | ||||||||||||||||||||||||
| 외부 ID | OMIM: 606829 MGI: 1096879 HomoloGene: 47908 GeneCard: FXN | ||||||||||||||||||||||||
| |||||||||||||||||||||||||
| |||||||||||||||||||||||||
| |||||||||||||||||||||||||
| 직교체 | |||||||||||||||||||||||||
| 종 | 인간 | 마우스 | |||||||||||||||||||||||
| 엔트레스 | |||||||||||||||||||||||||
| 앙상블 | |||||||||||||||||||||||||
| 유니프로트 | |||||||||||||||||||||||||
| RefSeq(mRNA) | |||||||||||||||||||||||||
| RefSeq(단백질) | |||||||||||||||||||||||||
| 위치(UCSC) | Cr 9: 69.04 – 69.08Mb | Cr 19: 24.26 – 24.28Mb | |||||||||||||||||||||||
| PubMed 검색 | [3] | [4] | |||||||||||||||||||||||
| 위키다타 | |||||||||||||||||||||||||
| |||||||||||||||||||||||||
프라택신은 인간에게 FXN 유전자에 의해 암호화된 단백질이다.[5][6]
미토콘드리온에 위치하며 프라탁신 mRNA는 대부분 대사율이 높은 조직으로 표현된다. 프라탁신의 기능은 명확하지 않지만 철황 성단의 조립에 관여한다. 철분 보호판이나 철분 저장 단백질로 활동할 것을 제안했다. 프리드레이히의 아탁시아의 원인은 프락신(frataxin)의 감소된 발현이다.
구조
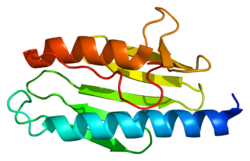
X선 결정학에서 인간 프락신은 한 쌍의 평행 α-헬리케스를 지지하는 β-시트로 이루어져 콤팩트 αβ 샌드위치를 형성한다.[7] 다른 종의 프라탁신 동음이의어는 유사하며, 동일한 핵심 구조를 공유한다. 그러나 한 나선의 끝에서 확장된 프래택신 꼬리 순서는 순서에 따라 갈라지며 길이가 다르다. 인간의 프락신은 박테리아나 효모에서 발견되는 프락신보다 꼬리의 순서가 더 길다. 꼬리의 목적은 단백질을 안정시키기 위한 것이라는 가설이 있다.[7]
대부분의 미토콘드리아 단백질과 마찬가지로 프리탁신은 세포질 리보솜에서 미토콘드리아를 표적으로 하는 대형 전구체 분자로 합성된다. 미토콘드리아에 들어가자마자 분자들은 성숙한 프락신을 생산하기 위한 단백질 반응에 의해 분해된다.[8]
함수
프라탁신은 미토콘드리온에 국부적이다. 프라탁신의 기능이 완전히 명확한 것은 아니지만 철황 성단의 조립에 관여하고 있는 것 같다. 철분 보호판이나 철분 저장 단백질로 활동할 것을 제안했다.[9]
프라탁신 mRNA는 주로 대사율이 높은 조직(간, 신장, 갈색 지방, 심장 포함)에서 발현된다. 생쥐와 효모 프리탁신 호몰로그램에는 잠재적 N단자 미토콘드리아 시퀀스를 대상으로 하는 것이 들어 있으며, 인간의 프리탁신은 미토콘드리아 단백질과 공동 국소화하는 것이 관찰되어 왔다. 게다가, 효모 유전자의 붕괴는 미토콘드리아 기능 장애를 초래하는 것으로 밝혀졌다. 따라서 프리드리히의 아탁시아는 핵 게놈의 돌연변이에 의해 생기는 미토콘드리아병(특히 단백질 프라탁신을 인코딩하는 FXN유전자에서 인트로닉 GAA 3중 반복의 팽창)[5][10][11]으로 여겨진다.
임상적 유의성
프리드레이히의 신경퇴행성질환인 아탁시아(FRDA)의 발현이 줄어든 것이 원인이다. 프리탁신 유전자 발현 감소는 염색체 실체의[12] 후생유전적 수정으로 인한 프라탁신 유전자의 전사의 음소거 또는[13] 박테리아와 인간 세포[14] 또는 둘 다에서 볼 수 있는 바와 같이 mRNA 이전의 첫 번째 인트론에서 확장된 GAA 반복을 분할할 수 없기 때문에 기인할 수 있다. 인트로닉 트리뉴클레오티드 확장은 GAA를 반복하여 프리드리히의 아탁시아를 발생시킨다.[15] 이렇게 확장된 반복은 R-루프 형성을 유발하며, 반복 대상인 올리고뉴클레오티드를 사용하여 R-루프를 교란시키면 프락신 표현을 재활성화할 수 있다.[16]
FRDA 환자의 96%는 그들의 FXN 유전자의 양쪽 알레르기의 인트론 1에서 GAA 트리뉴클레오티드 반복 팽창을 가지고 있다.[17] 전반적으로, 이것은 FRDA를 가진 사람들의 Frataxin mRNA 합성의 감소와 FRDA를 가진 사람들의 Frataxin 단백질의 감소로 이어진다. (FRDA 환자의 하위 집합은 한 염색체에서는 GAA 확장, 다른 염색체에서는 FXN exon에서는 점 돌연변이를 가진다.) 일반적인 경우, 짧은 GAA 확장에 따른 알레르기의 길이는 반대로 프라택신 수준과 상관관계가 있다. FRDA 환자들의 말초 조직은 일반적으로 영향을 받지 않은 사람들이 보여주는 프라택신 수준의 10% 미만이다.[17] 프라택신 수치가 낮으면 질병이 더 빨리 발병하고 더 빨리 진행되게 된다.
FRDA는 아탁시아, 감각 상실, 심근증이 특징이다. 프라택신 결핍이 이러한 증상을 일으키는 이유는 완전히 명확하지 않다. 세포 수준에서 미토콘드리아에 철분이 축적되고 산화제 민감성이 증가되는 것과 연결된다. 잘 이해되지 않는 이유들로 인해, 이것은 주로 등뿌리 갱년기, 소뇌, 심장 근육의 조직에 영향을 미친다.[8]
동물학
생쥐에서 FXN 유전자의 완전 불활성화는 초기 배아 단계에서 치명적이다.[18] 거의 모든 유기체가 프리탁신 호몰로뉴를 표현하지만, 인트론 1의 GAA 반복은 인간과 다른 영장류에서만 존재하기 때문에 FDRA를 일으키는 돌연변이는 다른 동물에서 자연적으로 일어날 수 없다. 과학자들은 쥐를 대상으로 이 질병을 모형화하기 위한 몇 가지 옵션을 개발했다. 한 가지 접근방식은 프리택신 표현을 단지 하나의 특정 조직 유형으로 침묵시키는 것이다: 심장(이 방법으로 변형된 것을 맥키라고 한다), 모든 뉴런(NSE), 또는 척수와 소뇌(PRP)[19]이다. 또 다른 접근방법은 인간처럼 프락신 생성을 억제해야 하는 마우스 FXN 유전자의 첫 번째 인트론에 GAA 확장을 삽입하는 것이다. 이 변형 유전자에 대해 동질성이 있는 생쥐를 KIKI(knock-in knock-in knock-in)라고 하며, KIKI 생쥐와 Frataxin knock-out 생쥐를 교차시켜 형성된 복합 이질체를 KIKO(kock-in knock-out)라고 한다. 그러나 KIKO 생쥐도 여전히 정상 프락신 수준의 25-36%를 표현하고 있으며, 매우 가벼운 증상을 보인다. 최종 접근법은 인간 프리택신 유전자의 GAA 확장 버전을 가진 유전자 변형 생쥐를 만드는 것을 포함한다. 이들 마우스는 YG22R(하나의 GAA 시퀀스 190회 반복)과 YG22R(두 개의 GAA 시퀀스 90회, 190회 반복)로 불린다. 이 쥐들은 인간 환자와 비슷한 증상을 보인다.[19]
드로소필라에서 프락신 과다 압착은 항산화 능력, 산화 스트레스 모욕 및 장수에 대한 저항력의 증가를 보여,[20] 프락신의 역할은 산화 스트레스와 그에 따른 세포 손상으로부터 미토콘드리아를 보호하는 것이라는 이론을 뒷받침한다.
FRDA와 FRDA 환자 섬유로블라스트의 마우스 모델에서 나온 섬유로블라스트는 DNA 이중 스트랜드 파손의 수치가 증가했음을 보여준다.[21] 렌티바이러스 유전자 전달 시스템은 FRDA 마우스 모델과 인간 환자 세포에 프래택신 유전자를 전달하는데 사용되었고, 이로 인해 프래택신 mRNA와 프래택신 단백질의 발현이 장기간 복원되었다. 이 복원된 프리택신 유전자의 발현에는 DNA 이중 가닥의 파괴 횟수가 상당히 줄어들었다.[21] FRDA 세포의 손상된 프락신은 DNA 손상을 복구하기 위한 용량이 감소하는 것으로 보이며 이는 신경퇴행의 원인이 될 수 있다.[21]
상호작용
프라택신은 PMPCB 효소와 생물학적으로 상호작용하는 것으로 밝혀졌다.[22]
참조
- ^ a b c GRCh38: 앙상블 릴리스 89: ENSG00000165060 - 앙상블, 2017년 5월
- ^ a b c GRCm38: 앙상블 릴리스 89: ENSMUSG000059363 - 앙상블, 2017년 5월
- ^ "Human PubMed Reference:". National Center for Biotechnology Information, U.S. National Library of Medicine.
- ^ "Mouse PubMed Reference:". National Center for Biotechnology Information, U.S. National Library of Medicine.
- ^ a b Campuzano V, Montermini L, Moltò MD, Pianese L, Cossée M, Cavalcanti F, Monros E, Rodius F, Duclos F, Monticelli A, Zara F, Cañizares J, Koutnikova H, Bidichandani SI, Gellera C, Brice A, Trouillas P, De Michele G, Filla A, De Frutos R, Palau F, Patel PI, Di Donato S, Mandel JL, Cocozza S, Koenig M, Pandolfo M (Mar 1996). "Friedreich's ataxia: autosomal recessive disease caused by an intronic GAA triplet repeat expansion". Science. 271 (5254): 1423–7. Bibcode:1996Sci...271.1423C. doi:10.1126/science.271.5254.1423. PMID 8596916. S2CID 20303793.
- ^ Carvajal JJ, Pook MA, dos Santos M, Doudney K, Hillermann R, Minogue S, Williamson R, Hsuan JJ, Chamberlain S (Oct 1996). "The Friedreich's ataxia gene encodes a novel phosphatidylinositol-4- phosphate 5-kinase". Nature Genetics. 14 (2): 157–62. doi:10.1038/ng1096-157. PMID 8841185. S2CID 6324358.
- ^ a b Dhe-Paganon S, Shigeta R, Chi YI, Ristow M, Shoelson SE (Oct 2000). "Crystal structure of human frataxin". The Journal of Biological Chemistry. 275 (40): 30753–6. doi:10.1074/jbc.C000407200. PMID 10900192.
- ^ a b Stemmler TL, Lesuisse E, Pain, Dancis (August 2010). "Frataxin and Mitochondrial FeS Cluster Biogenesis". Journal of Biological Chemistry. 285 (35): 26737–26743. doi:10.1074/jbc.R110.118679. PMC 2930671. PMID 20522547.
- ^ Adinolfi S, Iannuzzi C, Prischi F, Pastore C, Iametti S, Martin SR, Bonomi F, Pastore A (Apr 2009). "Bacterial frataxin CyaY is the gatekeeper of iron-sulfur cluster formation catalyzed by IscS". Nature Structural & Molecular Biology. 16 (4): 390–6. doi:10.1038/nsmb.1579. PMID 19305405. S2CID 205522816.
- ^ Dürr A, Cossee M, Agid Y, Campuzano V, Mignard C, Penet C, Mandel JL, Brice A, Koenig M (Oct 1996). "Clinical and genetic abnormalities in patients with Friedreich's ataxia". The New England Journal of Medicine. 335 (16): 1169–75. doi:10.1056/NEJM199610173351601. PMID 8815938.
- ^ Koutnikova H, Campuzano V, Foury F, Dollé P, Cazzalini O, Koenig M (Aug 1997). "Studies of human, mouse and yeast homologues indicate a mitochondrial function for frataxin". Nature Genetics. 16 (4): 345–51. doi:10.1038/ng0897-345. PMID 9241270. S2CID 5883249.
- ^ Kim E, Napierala M, Dent SY (Oct 2011). "Hyperexpansion of GAA repeats affects post-initiation steps of FXN transcription in Friedreich's ataxia". Nucleic Acids Research. 39 (19): 8366–77. doi:10.1093/nar/gkr542. PMC 3201871. PMID 21745819.
- ^ Pan X, Ding Y, Shi L (Nov 2009). "The roles of SbcCD and RNaseE in the transcription of GAA x TTC repeats in Escherichia coli". DNA Repair. 8 (11): 1321–7. doi:10.1016/j.dnarep.2009.08.001. PMID 19733517.
- ^ Baralle M, Pastor T, Bussani E, Pagani F (Jul 2008). "Influence of Friedreich ataxia GAA noncoding repeat expansions on pre-mRNA processing". American Journal of Human Genetics. 83 (1): 77–88. doi:10.1016/j.ajhg.2008.06.018. PMC 2443835. PMID 18597733.
- ^ "Entrez Gene: FXN frataxin".
- ^ Li L, Matsui M, Corey DR (2016-01-01). "Activating frataxin expression by repeat-targeted nucleic acids". Nature Communications. 7: 10606. Bibcode:2016NatCo...710606L. doi:10.1038/ncomms10606. PMC 4742999. PMID 26842135.
- ^ a b Clark E, Johnson J, Dong YN, Mercado-Ayon, Warren N, Zhai M, McMillan E, Salovin A, Lin H, Lynch DR (November 2018). "Role of frataxin protein deficiency and metabolic dysfunction in Friedreich ataxia, an autosomal recessive mitochondrial disease". Neuronal Signaling. 2 (4): NS20180060. doi:10.1042/NS20180060. PMC 7373238. PMID 32714592.
- ^ Cossée M, Puccio H, Gansmuller A, Koutnikova H, Dierich A, LeMeur M, Fischbeck K, Dollé P, Kœnig M (May 2000). "Inactivation of the Friedreich ataxia mouse gene leads to early embryonic lethality without iron accumulation". Human Molecular Genetics. 9 (8): 1219–1226. doi:10.1093/hmg/9.8.1219. PMID 10767347. Archived from the original on 2 June 2018. Retrieved 5 April 2019.
- ^ a b Perdomini M, Hick A, Puccio H (17 July 2013). "Animal and cellular models of Friedreich ataxia". Journal of Neurochemistry. 126: 65–79. doi:10.1111/jnc.12219. PMID 23859342. S2CID 1427817.
- ^ Runko AP, Griswold AJ, Min KT (March 2008). "Overexpression of frataxin in the mitochondria increases resistance to oxidative stress and extends lifespan in Drosophila". FEBS Letters. 582 (5): 715–9. doi:10.1016/j.febslet.2008.01.046. PMID 18258192. S2CID 207603250.
- ^ a b c Khonsari H, Schneider M, Al-Mahdawi S, Chianea YG, Themis M, Parris C, Pook MA, Themis M (December 2016). "Lentivirus-meditated frataxin gene delivery reverses genome instability in Friedreich ataxia patient and mouse model fibroblasts". Gene Ther. 23 (12): 846–856. doi:10.1038/gt.2016.61. PMC 5143368. PMID 27518705.
- ^ Koutnikova H, Campuzano V, Koenig M (Sep 1998). "Maturation of wild-type and mutated frataxin by the mitochondrial processing peptidase". Human Molecular Genetics. 7 (9): 1485–9. doi:10.1093/hmg/7.9.1485. PMID 9700204.
추가 읽기
- Thierbach R, Drewes G, Fusser M, Voigt A, Kuhlow D, Blume U, Schulz TJ, Reiche C, Glatt H, Epe B, Steinberg P, Ristow M (Nov 2010). "The Friedreich's ataxia protein frataxin modulates DNA base excision repair in prokaryotes and mammals". The Biochemical Journal. 432 (1): 165–72. doi:10.1042/BJ20101116. PMC 2976068. PMID 20819074.
- Montermini L, Rodius F, Pianese L, Moltò MD, Cossée M, Campuzano V, Cavalcanti F, Monticelli A, Palau F, Gyapay G (Nov 1995). "The Friedreich ataxia critical region spans a 150-kb interval on chromosome 9q13". American Journal of Human Genetics. 57 (5): 1061–7. PMC 1801369. PMID 7485155.
- Bidichandani SI, Ashizawa T, Patel PI (May 1997). "Atypical Friedreich ataxia caused by compound heterozygosity for a novel missense mutation and the GAA triplet-repeat expansion". American Journal of Human Genetics. 60 (5): 1251–6. PMC 1712428. PMID 9150176.
- Babcock M, de Silva D, Oaks R, Davis-Kaplan S, Jiralerspong S, Montermini L, Pandolfo M, Kaplan J (Jun 1997). "Regulation of mitochondrial iron accumulation by Yfh1p, a putative homolog of frataxin". Science. 276 (5319): 1709–12. doi:10.1126/science.276.5319.1709. PMID 9180083.
- Koutnikova H, Campuzano V, Foury F, Dollé P, Cazzalini O, Koenig M (Aug 1997). "Studies of human, mouse and yeast homologues indicate a mitochondrial function for frataxin". Nature Genetics. 16 (4): 345–51. doi:10.1038/ng0897-345. PMID 9241270. S2CID 5883249.
- Wilson RB, Roof DM (Aug 1997). "Respiratory deficiency due to loss of mitochondrial DNA in yeast lacking the frataxin homologue". Nature Genetics. 16 (4): 352–7. doi:10.1038/ng0897-352. PMID 9241271. S2CID 22652291.
- Campuzano V, Montermini L, Lutz Y, Cova L, Hindelang C, Jiralerspong S, Trottier Y, Kish SJ, Faucheux B, Trouillas P, Authier FJ, Dürr A, Mandel JL, Vescovi A, Pandolfo M, Koenig M (Oct 1997). "Frataxin is reduced in Friedreich ataxia patients and is associated with mitochondrial membranes". Human Molecular Genetics. 6 (11): 1771–80. doi:10.1093/hmg/6.11.1771. PMID 9302253.
- Rötig A, de Lonlay P, Chretien D, Foury F, Koenig M, Sidi D, Munnich A, Rustin P (Oct 1997). "Aconitase and mitochondrial iron-sulphur protein deficiency in Friedreich ataxia". Nature Genetics. 17 (2): 215–7. doi:10.1038/ng1097-215. PMID 9326946. S2CID 23151137.
- Jiralerspong S, Liu Y, Montermini L, Stifani S, Pandolfo M (1997). "Frataxin shows developmentally regulated tissue-specific expression in the mouse embryo". Neurobiology of Disease. 4 (2): 103–13. doi:10.1006/nbdi.1997.0139. PMID 9331900. S2CID 6520439.
- Koutnikova H, Campuzano V, Koenig M (Sep 1998). "Maturation of wild-type and mutated frataxin by the mitochondrial processing peptidase". Human Molecular Genetics. 7 (9): 1485–9. doi:10.1093/hmg/7.9.1485. PMID 9700204.
- Zühlke C, Laccone F, Cossée M, Kohlschütter A, Koenig M, Schwinger E (Jul 1998). "Mutation of the start codon in the FRDA1 gene: linkage analysis of three pedigrees with the ATG to ATT transversion points to a unique common ancestor". Human Genetics. 103 (1): 102–5. doi:10.1007/s004390050791. PMID 9737785. S2CID 26999143.
- Bartolo C, Mendell JR, Prior TW (Oct 1998). "Identification of a missense mutation in a Friedreich's ataxia patient: implications for diagnosis and carrier studies". American Journal of Medical Genetics. 79 (5): 396–9. doi:10.1002/(SICI)1096-8628(19981012)79:5<396::AID-AJMG13>3.0.CO;2-M. PMID 9779809.
- Cossée M, Dürr A, Schmitt M, Dahl N, Trouillas P, Allinson P, Kostrzewa M, Nivelon-Chevallier A, Gustavson KH, Kohlschütter A, Müller U, Mandel JL, Brice A, Koenig M, Cavalcanti F, Tammaro A, De Michele G, Filla A, Cocozza S, Labuda M, Montermini L, Poirier J, Pandolfo M (Feb 1999). "Friedreich's ataxia: point mutations and clinical presentation of compound heterozygotes". Annals of Neurology. 45 (2): 200–6. doi:10.1002/1531-8249(199902)45:2<200::AID-ANA10>3.0.CO;2-U. PMID 9989622.
- Coppola G, De Michele G, Cavalcanti F, Pianese L, Perretti A, Santoro L, Vita G, Toscano A, Amboni M, Grimaldi G, Salvatore E, Caruso G, Filla A (May 1999). "Why do some Friedreich's ataxia patients retain tendon reflexes? A clinical, neurophysiological and molecular study". Journal of Neurology. 246 (5): 353–7. doi:10.1007/s004150050362. PMID 10399865. S2CID 7367457.
- Branda SS, Cavadini P, Adamec J, Kalousek F, Taroni F, Isaya G (Aug 1999). "Yeast and human frataxin are processed to mature form in two sequential steps by the mitochondrial processing peptidase". The Journal of Biological Chemistry. 274 (32): 22763–9. doi:10.1074/jbc.274.32.22763. PMID 10428860.
- Gordon DM, Shi Q, Dancis A, Pain D (Nov 1999). "Maturation of frataxin within mammalian and yeast mitochondria: one-step processing by matrix processing peptidase". Human Molecular Genetics. 8 (12): 2255–62. doi:10.1093/hmg/8.12.2255. PMID 10545606.
- Forrest SM, Knight M, Delatycki MB, Paris D, Williamson R, King J, Yeung L, Nassif N, Nicholson GA (Aug 1998). "The correlation of clinical phenotype in Friedreich ataxia with the site of point mutations in the FRDA gene". Neurogenetics. 1 (4): 253–7. doi:10.1007/s100480050037. PMID 10732799. S2CID 7463903.
- Al-Mahdawi S, Pook M, Chamberlain S (Jul 2000). "A novel missense mutation (L198R) in the Friedreich's ataxia gene". Human Mutation. 16 (1): 95. doi:10.1002/1098-1004(200007)16:1<95::AID-HUMU29>3.0.CO;2-E. PMID 10874325.









