생물학적 소각 산란
Biological small-angle scattering생물학적 소각 산란은 생물 물질의 구조 분석을 위한 소각 산란 방법이다.소각 산란은 생물학적 고분자, 나노콤포사이트, 합금, 합성 고분자 등의 다양한 물체의 구조를 연구하는 데 사용된다.[1]소각 X선 산란(SAXS)과 소각 중성자 산란(SANS)은 공동으로 소각 산란(SAS)으로 알려진 두 가지 보완 기법이다.SAS는 정적 광 산란뿐만 아니라 X선 및 중성자 회절, 광각 X선 산란과 유사하다.다른 X선 및 중성자 산란 방법과 달리 SAS는 결정 입자와 비결정 입자의 크기와 모양에 대한 정보를 제공한다.수성 용액에서 매우 자주 사용되는 생물학적 물질을 연구하기 위해 사용될 때 산란 패턴은 평균 방향이다.[2][3]null
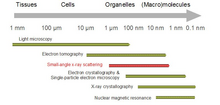
SAS 패턴은 몇 도 정도의 작은 각도로 수집된다.SAS는 최대 150nm 크기의 부분적으로 정렬된 시스템에서 1 ~ 25nm의 분해능 범위와 반복 거리의 구조 정보를 제공할 수 있다.초소각 산란(USAS)은 더 큰 치수를 해결할 수 있다.방목-침입 소각 산란(GISAS)은 표면의 생물학적 분자층을 연구하는 데 강력한 기술이다.null
생물학적 응용에서 SAS는 입자의 구조를 평균 입자 크기와 형태 면에서 결정하는데 사용된다.또한 표면 대 부피 비율에 대한 정보를 얻을 수 있다.전형적으로 생물학적 고분자는 액체로 분산된다.방법은 정확하고 대부분 비파괴적이며 일반적으로 최소한의 검체 준비만 하면 된다.그러나 생물학적 분자는 항상 방사선 손상에 취약하다.null
솔루션 NMR이나 X선 결정학 등 다른 구조 결정 방법과 비교하면 SAS는 어느 정도 제약을 극복할 수 있다.예를 들어, 용액 NMR은 단백질 크기로 제한되는 반면 SAS는 다분자 조립체뿐만 아니라 작은 분자에도 사용될 수 있다.솔리드 스테이트 NMR은 40 kDa 이상의 고분자 또는 아밀로이드 섬유질과 같은 비결정성 샘플의 원자 수준 정보를 결정하는 데 여전히 필수적인 도구다.X선 결정학에 의한 구조 결정은 몇 주 또는 심지어 몇 년이 걸릴 수 있는 반면 SAS 측정은 며칠이 걸릴 수 있다.SAS는 또한 크기 제외 크로마토그래피와 같은 다른 분석 기법과 결합되어 이기종 샘플을 연구할 수 있다.[4]그러나 SAS로는 분자 내 원자의 위치를 측정할 수 없다.null
방법
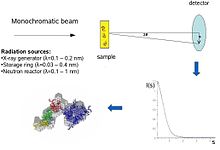
개념적으로, 소각 산란 실험은 간단하다: 표본은 X선이나 중성자에 노출되고 산란 방사선은 검출기에 의해 등록된다.SAS 측정은 1차 빔("작은 각도")에 매우 가깝게 수행되므로 이 기술은 시준되거나 집중된 X선 또는 중성자 빔을 필요로 한다.생물학적 소각 X선 산란은 보통 생물학적 분자가 약하게 산란하고 측정 용액이 희석되기 때문에 싱크로트론 방사선원에서 종종 수행된다.생물학적 SACS 방법은 싱크로트론 저장 링이 제공하는 X선 광자 빔의 고강도로 이익을 얻는다.X선 또는 중성자 산란 곡선(강도 대 산란 각도)은 단백질의 저해상도 모델을 만드는 데 사용되며, 여기 오른쪽 그림에 나와 있다.X선 또는 중성자 산란 데이터를 추가로 사용하여 별도의 도메인(X선 또는 NMR 구조)을 "SAXS 봉투"에 장착할 수 있다.null
산란 실험에서 고분자 용액은 X선(파장 λ은 일반적으로 0.15nm 전후)이나 열 중성자(λλ0.5nm)에 노출된다.산란 강도 I(s)는 운동력 전달 s의 함수로 기록된다(s=44sinsin//, 여기서 2θ은 입사 및 산란 방사선의 각도임).용액의 강도로부터 용매로부터만 산란되는 것을 뺀다.입자의 무작위 위치 및 방향은 비 상호작용 입자의 경우 모든 방향에서 평균화된 단일 입자로부터의 산란과 비례하는 등방성 강도 분포를 초래한다.순입자 산란은 입자와 용매 사이의 산란 길이 밀도 차이(X선의 경우 전자 밀도, 중성자의 경우 핵/스핀 밀도)에 비례한다. 즉, 소위 대비.대비는2 HO2/DO 혼합물 또는 추가 정보를 얻기 위해 선택적 분해를 사용하는 중성자 산란에서 변경될 수 있다.[1]SAS 데이터의 정보 내용은 오른쪽 그림에서 설명되며, 주름과 분자 질량이 다른 단백질에서 발생하는 X선 산란 패턴을 보여준다.낮은 각도(2-3nm 분해능)에서 곡선은 입자 모양에 따라 결정되는 s의 급속한 붕괴 함수로서, 이는 분명히 다르다.중간 분해능(2 ~ 0.5 nm)에서는 차이가 이미 덜 뚜렷하고 0.5 nm 분해능 이상에서는 모든 곡선이 매우 유사하다.[5]따라서 SAS는 형태, 2차 및 3차 구조 등 총 구조 특성에 대한 정보를 포함하지만 원자 구조 분석에는 적합하지 않다.null
역사
첫 적용은 1930년대 후반으로 거슬러 올라간다. 당시 SACS의 주요 원리는 금속 합금에 대한 그의 연구에 이어 기니어의 기초 연구로 개발되었다.기니어와 포넷의 SACS에 관한 첫 번째 모노그래프에서는 그 방법이 입자의 크기와 모양뿐만 아니라 질서 정연하고 부분적으로 순서화된 시스템의 내부 구조에 관한 정보를 산출한다는 것이 이미 증명되었다.null
1960년대에 이 방법은 결정체가 없을 때 전체적인 모양과 내부 구조에 대한 저해상도 구조 정보를 얻을 수 있게 되면서 용해 중인 생물학적 고분자 연구에서 점점 더 중요해졌다.1970년대에 싱크로트론 방사선과 중성자 선원의 이용가능성 덕분에 SACS와 SANS 실험에서 획기적인 발전이 이루어졌으며, 후자는 DO용22 HO의 용매 교환과 특정 디utation 방법에 의한 대조 변동의 길을 열었다.솔루션에 대한 분산 연구는 시간과 노력을 최소로 투자하여 비결정 생화학적 시스템의 구조에 대한 유용한 통찰력을 제공한다는 것이 밝혀졌다.또한, 색스/샌스는 고분자 어셈블리의 조립 및 대규모 순응적 변화를 포함하여 분자 간 상호작용에 대한 실시간 조사를 가능하게 했다.null
구조 방법으로서의 SAS의 주요 과제는 1차원 실험 데이터에서 물체의 3차원 구조에 관한 정보를 추출하는 것이다.과거에는 고분자의 전체 입자 매개변수(예: 부피, 교정의 반지름)만 실험 데이터에서 직접 결정되는 반면, 3차원 모델의 분석은 단순한 기하학적 본체(예: 타원체, 실린더 등)로 제한되거나 임시 시행착오 기준으로 수행되었다.전자현미경 검사는 종종 컨센서스 모델을 구축하는 데 제약으로 사용되었다.1980년대에는 다른 구조 방법의 발전으로 SAS 연구에 대한 생화학자들의 관심의 감소를 가져왔는데, 이는 단지 두어 가지 전체 매개변수로부터 구조적인 결론을 이끌어내거나 시행착오 모델에 기초하였다.null
1990년대는 형상과 영역 구조의 세부 결정과 강체 신체 정교화 기법의 적용 등 고분자 복합체의 신뢰성 있는 초기 모델링을 위한 길을 열어준 SACS/SANS 데이터 분석 방법에 획기적인 발전을 가져왔다.이러한 진전은 계측기의 추가적인 발전을 동반하여 단백질과 핵산 접힘 연구에서 3세대 SR 선원에 대한 서브ms 시간 분해능을 달성할 수 있었다.[1]null
2005년에 4년짜리 프로젝트가 시작되었다.SAXIER(Small-Angle X-Ray 산란 이니셔티브 for Europe)는 SAXIER(Small-Angle X-Ray 산란 이니셔티브)와 SACS 방법을 다른 분석 기법과 결합하고 대량의 데이터를 신속하게 분석하는 자동화된 소프트웨어를 만드는 것을 목표로 한다.이 프로젝트는 사용 가능한 가장 진보된 방법을 사용하여 통합된 유럽 SACS 인프라를 만들었다.[6]null
데이터 분석
좋은 품질의 SAS 실험에서는 조사 중인 고분자의 농도가 다양한 여러 솔루션을 측정한다.다른 농도에서 측정한 산란 곡선을 0 농도로 추론함으로써 무한 희석을 나타내는 산란 곡선을 얻을 수 있다.그러면 농도 영향이 산란 곡선에 영향을 미치지 않아야 한다.외삽 산란 곡선의 데이터 분석은 s = 0 주위에 있는 지역의 산란 곡선의 시작에 대한 검사로 시작한다. 지역이 기니어 근사치(일명 기니어 법칙)를 따른다면 표본은 집계되지 않는다.그러면 문제의 입자의 모양은 여러 가지 방법에 의해 결정될 수 있는데, 그 중 일부는 다음과 같은 참고문헌에 기술되어 있다.[1]null
간접 푸리에 변환
첫 번째 단계는 일반적으로 산란 곡선의 푸리에 변환을 계산하는 것이다.변환된 곡선은 입자 내부의 거리 분포 함수로 해석할 수 있다.이러한 변환은 또한 입력 데이터의 정규화의 이점을 제공한다.[citation needed]null
저해상도 모델
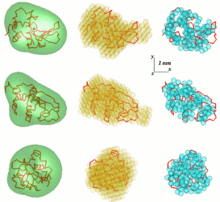
SAS 데이터 분석의 한 가지 문제는 1차원 산란 패턴에서 3차원 구조를 얻는 것이다.SAS 데이터는 단일 솔루션을 의미하지 않는다.예를 들어, 많은 다른 단백질들은 같은 산란 곡선을 가질 수 있다.3D 구조의 재구성은 많은 수의 다른 모델을 야기할 수 있다.이 문제를 피하기 위해 여러 가지 단순화를 고려할 필요가 있다.null
또 다른 접근방식은 소각 X선 및 중성자 산란 데이터와 모델을 MONSA 프로그램과 결합하는 것이다.
자유롭게 이용할 수 있는 SAS 분석 컴퓨터 프로그램이 EMBL에서 집중적으로 개발되었다.첫 번째 일반 아비니시오 접근법에서 입자 r=F(Ω)의 각도 봉투 함수는 여기서 (r, Ω)는 구면 좌표로서 일련의 구면 고조파로 설명된다.따라서 낮은 분해능 모양은 산란 데이터에 적합한 몇 개의 매개변수(이 시리즈의 계수)로 정의된다.이 접근법은 컴퓨터 프로그램인 사샤(Small Angle Disclanizing Shape Decision)에서 더욱 발전되어 구현되었다.[7][8]특정 상황에서는 산란 데이터에서 고유한 봉투를 추출할 수 있다는 것이 입증되었다.이 방법은 형상이 비교적 단순하고 내부 빈도가 유의미한 입자에만 적용된다.이러한 한계를 극복하기 위해 다른 유형의 몬테카를로 검색을 사용하는 또 다른 접근법이 개발되었다.달라이_GA는 산란 데이터에서 결정되는 최대 입자 크기 Dmax와 같은 직경의 구를 취하여 구슬로 채우는 우아한 프로그램이다.각 비드는 입자(index=1) 또는 용매(index=0)에 속한다.따라서 모양은 길이 M의 이진 문자열로 설명된다. 무작위 문자열에서 시작하여 유전 알고리즘은 데이터에 적합한 모델을 검색한다.컴팩트함과 연결성 제약은 DAMMIN 프로그램에서 구현된 검색에서 부과된다.[9][10] 만약 입자 대칭성이 알려져 있다면, 사샤와 DAMMIN은 그것을 유용한 제약조건으로 활용할 수 있다.상호 연결된 타원체를 기반으로 하는 'give-n-take' 절차인 SACS3D와 프로그램 SASMODEL은 검색 공간에서 제한 없이 접근하는 이니시오 몬테카를로 접근이다.[5]null
Dmax 직경 영역 내에 로컬로 "체인 호환" DR 모델을 구축하기 위해 Dummy Legentues(DRs)와 시뮬레이션 어닐링의 앙상블을 사용하는 접근방식은 SCS 데이터에서 더 많은 세부 정보를 추출할 수 있게 한다.이 방법은 GASBOR 프로그램에서 구현된다.[11][12]null
다영역 단백질과 고분자 복합체의 용액 산란 패턴은 3차 구조가 보존된다고 가정할 때 개별 도메인이나 서브유닛의 고해상도(NMR 또는 X선) 구조로부터 구축된 모델을 사용하여 장착할 수도 있다.객체의 복잡성에 따라, 실험 데이터에 적합한 하위 유니트의 최적 구성을 전세계적으로 검색하기 위해 다양한 접근방식을 채택한다.null
컨센서스 모델
몬테카를로 기반 모델은 수백, 수천 개의 매개변수를 포함하고 있으며, 과도한 해석을 방지하기 위해 주의가 필요하다.일반적인 접근방식은 독립적인 형상 재구성 실행으로 인한 일련의 모델을 정렬하여 가장 지속적이고 가장 신뢰할 수 있는 기능(예: SUPCOMB 프로그램 사용)을 유지하는 평균 모델을 구하는 것이다.[5][13][14]null
누락된 루프를 추가하는 중
비순열 표면 아미노산("loops")은 NMR 및 결정학 연구에서 자주 관찰되지 않으며 보고된 모델에서 누락될 수 있다.이와 같이 질서 정연한 원소는 산란 강도에 기여하며, 구조물의 알려진 부분을 고정하고 전체 입자로부터 SAS 패턴에 맞게 누락된 부분을 추가함으로써 발생 가능한 위치를 찾을 수 있다.더미 잔류물 접근법이 확장되었고 누락된 루프나 도메인을 추가하기 위한 알고리즘이 프로그램 세트 크레도에서 구현되었다.[5]
혼성법
최근에 몇몇 방법들은 SACS 데이터를 제약조건으로 사용할 것을 제안했다.저자들은 접이식 인식과[15] 디노보 단백질 구조 예측[16] 방법의 결과 개선을 목표로 삼았다.SACS 데이터는 주어진 단백질에 대한 원자 쌍 거리 히스토그램의 푸리에 변환을 제공한다.이것은 단백질의 고유 순응 접종을 결정하는 데 사용되는 방법의 구조적 제약으로 작용할 수 있다.나사산이나 접이식 인식은 3D 구조가 시퀀스보다 보존성이 높다고 가정한다.따라서 매우 상이한 시퀀스는 유사한 구조를 가질 수 있다.반면에 아비니시오 방법은 분자생물학에서 가장 큰 문제들 중 하나인, 즉 "원래부터" 단백질의 접힘을 예측하는 것으로서, 동음이의 순서나 구조를 사용하지 않는다.저자들은 'SAXS 필터'를 이용해 드노보 단백질 모델 세트를 대폭 정화할 수 있었다.[16]이것은 구조 동종학 검색에 의해 더욱 증명되었다.또한 나사산 방식으로 사용되는 SACS 점수와 점수의 조합이 접이식 인식 성능을 크게 향상시키는 것으로 나타났다.[15]한 예에서 모듈형 단백질의 대략적인 3차 구조가 어떻게 변환 자유도를 구속하는 SCS 데이터를 사용하여 도메인의 고해상도 NMR 구조에서 조립될 수 있는지 입증했다.[17]또 다른 예는 다단백질의 2분위 구조를 재구성하기 위해 어떻게 SACS 데이터를 NMR, X선 결정학 및 전자 현미경 검사와 결합할 수 있는지를 보여준다.[18]null
유연한 시스템
본질적으로 질서 정연하지 않거나 유연한 링커로 다중 도메인 단백질 문제를 해결할 수 있는 우아한 방법이 최근 제안되었다.[19]그것은 단백질의 서로 다른 순응의 공존을 가능하게 하며, 이것은 함께 평균적인 실험 산란 패턴에 기여한다.초기에 UM(Enexemble Optimization Method)은 단백질 구성 공간을 커버하는 모델 풀을 생성한다.그런 다음 각 모델에 대해 산란 곡선을 계산한다.두 번째 단계에서 프로그램은 단백질 모델의 서브셋을 선택한다.평균 실험 산란은 각 서브셋에 대해 계산되며 실험 SACS 데이터에 적합된다.최적 적합도를 찾지 못하면 다른 하위 집합으로 모델을 개편하고 새로운 평균 산란 계산 및 실험 데이터에 대한 적합을 수행한다.이 방법은 변성 리소자임과 브루턴의 단백질 키나아제라는 두 가지 단백질로 실험되었다.그것은 흥미롭고 전도유망한 결과를 주었다.[19]null
생물학적 분자층 및 GISAS
생체 분자의 코팅은 방목-증인 X선과 중성자 산란으로 연구할 수 있다.IsGISAXS(Grazing Incidention small angle X선 산란)는 나노구조체에서 GISAXS의 시뮬레이션과 분석에 전념하는 소프트웨어 프로그램이다.IsGISAXS는 나노미터 크기의 입자에 의한 산란만을 포괄하는데, 이 입자는 표면 아래 매트릭스에 묻히거나 기질에 받치거나 기판의 얇은 층에 묻힌다.구멍의 케이스도 취급한다.그 기하학은 입자의 평면으로 제한된다.산란 단면은 간섭함수와 입자형성 인자의 관점에서 분해된다."빔 굴절 효과"를 유도하는 방목 발생 기하학에 중점을 둔다.입자 형태 인자는 왜곡파 Born 근사치(DWBA) 내에서 계산되며, 날카로운 인터페이스를 가진 불침투 상태 또는 굴절 지수의 실제 수직 프로필로 시작한다.다양한 종류의 간단한 기하학적 형상은 디커플링 근사치(DA), 국소 모노디사이즈 근사치(LMA) 및 크기 간격 상관관계 근사치(SSCA)에서 크기와 형상 분포의 전체 계정으로 이용할 수 있다.입자-입자 쌍 상관관계 함수에 의해 정의된 입자의 질서 정연한 시스템과 2차원 결정 또는 파라 결정 둘 다 고려된다.[20]null
참고 항목
참조
- ^ a b c d Svergun DI, Koch MH (2003). "Small-angle scattering studies of biological macromolecules in solution". Rep. Prog. Phys. 66 (10): 1735–82. Bibcode:2003RPPh...66.1735S. doi:10.1088/0034-4885/66/10/R05. S2CID 9305500.
- ^ Ho DL, Byrnes WM, Ma WP, Shi Y, Callaway DJ, Bu Z (September 2004). "Structure-specific DNA-induced conformational changes in Taq polymerase revealed by small angle neutron scattering". The Journal of Biological Chemistry. 279 (37): 39146–54. doi:10.1074/jbc.M404565200. PMID 15247286.
- ^ Lipfert J, Doniach S (1 June 2007). "Small-angle X-ray scattering from RNA, proteins, and protein complexes". Annual Review of Biophysics and Biomolecular Structure. 36 (1): 307–27. doi:10.1146/annurev.biophys.36.040306.132655. PMID 17284163.
- ^ Meisburger SP, Thomas WC, Watkins MB, Ando N (June 2017). "X-ray Scattering Studies of Protein Structural Dynamics". Chemical Reviews. 117 (12): 7615–7672. doi:10.1021/acs.chemrev.6b00790. PMC 5562295. PMID 28558231.
- ^ a b c d Svergun DI, Koch MH (October 2002). "Advances in structure analysis using small-angle scattering in solution". Current Opinion in Structural Biology. 12 (5): 654–60. doi:10.1016/S0959-440X(02)00363-9. PMID 12464319.
- ^ "SAXIER: Small-Angle X-ray Scattering Initiative for Europe".
- ^ "SASHA: Small Angle Scattering Shape Determination". Biological small angle scattering group. EMBL Hamburg.
- ^ Svergun DI, Volkov VV, Kozin MB, Stuhrmann HB (1996). "New developments in direct shape determination from small-angle scattering. 2. Uniqueness". Acta Crystallogr. A52 (6): 419–426. doi:10.1107/S0108767391006414.
- ^ "DAMMIN: Ab initio shape determination by simulated annealing using a single phase dummy atom model". Biological small angle scattering group. EMBL Hamburg.
- ^ Svergun DI (June 1999). "Restoring low resolution structure of biological macromolecules from solution scattering using simulated annealing". Biophysical Journal. 76 (6): 2879–86. Bibcode:1999BpJ....76.2879S. doi:10.1016/S0006-3495(99)77443-6. PMC 1300260. PMID 10354416.
- ^ "GASBOR: Ab initio reconstruction of a protein structure by a chain-like ensemble of dummy residues". Biological small angle scattering group. EMBL Hamburg.
- ^ Svergun DI, Petoukhov MV, Koch MH (June 2001). "Determination of domain structure of proteins from X-ray solution scattering". Biophysical Journal. 80 (6): 2946–53. Bibcode:2001BpJ....80.2946S. doi:10.1016/S0006-3495(01)76260-1. PMC 1301478. PMID 11371467.
- ^ "SUPCOMB". Biological small angle scattering group. EMBL Hamburg.
- ^ Kozin MB, Svergun DI (2001). "Automated matching of high- and low-resolution structural models". J. Appl. Crystallogr. 34: 33–41. doi:10.1107/S0021889800014126.
- ^ a b Zheng W, Doniach S (May 2005). "Fold recognition aided by constraints from small angle X-ray scattering data". Protein Engineering, Design & Selection. 18 (5): 209–19. doi:10.1093/protein/gzi026. PMID 15845555.
- ^ a b Zheng W, Doniach S (February 2002). "Protein structure prediction constrained by solution X-ray scattering data and structural homology identification". Journal of Molecular Biology. 316 (1): 173–87. doi:10.1006/jmbi.2001.5324. PMID 11829511. S2CID 2970219.
- ^ Mattinen ML, Pääkkönen K, Ikonen T, Craven J, Drakenberg T, Serimaa R, Waltho J, Annila A (August 2002). "Quaternary structure built from subunits combining NMR and small-angle x-ray scattering data". Biophysical Journal. 83 (2): 1177–83. Bibcode:2002BpJ....83.1177M. doi:10.1016/S0006-3495(02)75241-7. PMC 1302219. PMID 12124297.
- ^ Tidow H, Melero R, Mylonas E, Freund SM, Grossmann JG, Carazo JM, Svergun DI, Valle M, Fersht AR (July 2007). "Quaternary structures of tumor suppressor p53 and a specific p53 DNA complex". Proceedings of the National Academy of Sciences of the United States of America. 104 (30): 12324–9. Bibcode:2007PNAS..10412324T. doi:10.1073/pnas.0705069104. PMC 1941468. PMID 17620598.
- ^ a b Bernadó P, Mylonas E, Petoukhov MV, Blackledge M, Svergun DI (May 2007). "Structural characterization of flexible proteins using small-angle X-ray scattering". Journal of the American Chemical Society. 129 (17): 5656–64. doi:10.1021/ja069124n. PMID 17411046.
- ^ "IsGISAXS: a program for analyzing Grazing Incidence Small Angle X-ray Scattering from nanostructures". Archived from the original on May 22, 2012.
추가 읽기
- Koch MH, Vachette P, Svergun DI (May 2003). "Small-angle scattering: a view on the properties, structures and structural changes of biological macromolecules in solution". Quarterly Reviews of Biophysics. 36 (2): 147–227. doi:10.1017/S0033583503003871. PMID 14686102. S2CID 5432509.
- Petoukhov MV, Svergun DI (August 2005). "Global rigid body modeling of macromolecular complexes against small-angle scattering data". Biophysical Journal. 89 (2): 1237–50. Bibcode:2005BpJ....89.1237P. doi:10.1529/biophysj.105.064154. PMC 1366608. PMID 15923225.
- Bernadó P, Blackledge M (December 2010). "Structural biology: Proteins in dynamic equilibrium". Nature. 468 (7327): 1046–8. Bibcode:2010Natur.468.1046B. doi:10.1038/4681046a. PMID 21179158.
외부 링크
| 위키북스는 엑스레이 결정학이라는 주제에 관한 책을 가지고 있다. |
- SACS/WAXS Beamline Australian Synchrotron, 오스트레일리아 멜버른
- SIBYLS – 미국 버클리 어드밴스트 광원(Advanced Light Source)의 빔라인
- SACS – 이탈리아 트리에스테에 있는 ELETTRA 싱크로트론 빛 연구소의 빔라인
- X33 – 독일 함부르크 DESY의 빔 라인
- D11A[dead link] – 브라질 캄피나스 주 브라질 싱크로트론 빛 연구소의 빔라인
- X21 및 X9 – 미국 Upton의 Brookhaven National Laboratory에 있는 National Synchrotron 광원의 빔 라인
- F2 및 G1 – 미국 이타카 주, 가속기 기반 과학 및 교육 연구소의 빔라인
- Bio-SANS – 미국 TN, Oak Ridge의 Oak Ridge National Laboratory에 있는 고유속 동위원소 원자로의 빔라인