5-메틸시토신
5-Methylcytosine | |
| 이름 | |
|---|---|
| 우선 IUPAC 이름 4-아미노-5-메틸피리미딘-2(1H)-원 | |
| 식별자 | |
3D 모델(JSmol) | |
| 3 DMet | |
| 120387 | |
| 체비 | |
| 켐스파이더 | |
| ECHA 정보 카드 | 100.008.236 |
| EC 번호 |
|
| 케그 | |
| 메쉬 | 5-메틸시토신 |
PubChem CID | |
| 유니 | |
CompTox 대시보드 (EPA ) | |
| |
| |
| 특성. | |
| C5H7N3O | |
| 몰 질량 | 125.150 g/140−1 |
| 위험 요소 | |
| GHS 라벨링: | |
 | |
| 경고 | |
| H317, H319 | |
| P261, , , , , , , , , | |
달리 명시되지 않은 한 표준 상태(25°C[77°F], 100kPa)의 재료에 대한 데이터가 제공됩니다. | |
5-메틸시토신은 유전자 전사를 조절하고 여러 가지 다른 생물학적 [1]역할을 하는 DNA 염기 시토신(C)의 메틸화된 형태이다.시토신이 메틸화 될 때, DNA는 같은 염기서열을 유지하지만 메틸화 유전자의 발현은 바뀔 수 있다(이에 대한 연구는 후생유전학 분야의 일부이다.5-메틸시토신은 뉴클레오시드 5-메틸시티딘에 함유되어 있다.
5-메틸시토신에서 메틸기는 6-원자환의 5번째 원자에 부착되어 있으며, 6시 위치에서 NH결합질소에서 시계 반대방향으로 계수된다.이 메틸기는 5-메틸시토신과 시토신을 구별한다.
검출
W.G. Ruppel은 결핵을 일으키는 박테리아 독소를 분리하려고 노력하면서 1898년 결핵균에서 [2]투베르쿨린산이라는 새로운 핵산을 분리했다.이 핵산은 티민, 구아닌, 메틸화 뉴클레오티드인 시토신 외에 함유되어 있다는 점에서 특이한 것으로 밝혀졌다.1925년 존슨과 코길은 황산과 [3][4]투베르쿨린산의 가수분해 산물로 소량의 메틸화 시토신 유도체를 성공적으로 검출했다.이 보고서는 결정성 피크레이트의 광학적 특성만을 근거로 이들의 식별이 이뤄졌고 다른 과학자들은 같은 결과를 [5]재현하지 못했기 때문에 혹독한 비판을 받았다.하지만 그 존재는 1948년, 호치키스가 종이 크로마토그래피를 사용하여 송아지 흉선에서 DNA의 핵산을 분리했을 때, 종래의 시토신이나 우라실과는 [6]상당히 다른 독특한 메틸화 시토신을 검출했을 때, 최종적으로 증명되었다.70년 후, 비록 정확한 역할이 [7]불확실하지만, 그것은 또한 다른 RNA 분자들의 공통적인 특징이라는 것이 밝혀졌다.
생체내
이 화학물질의 기능은 [8]종에 따라 크게 다릅니다.
- 박테리아에서 5-메틸시토신은 다양한 부위에서 발견될 수 있으며, 종종 천연 메틸화 감수성 제한 효소에 의해 DNA가 절단되지 않도록 보호하는 마커로 사용된다.
- 식물에서 5-메틸사이토신은 CpG, CpHpG 및 CpHpH 배열(여기서 H = A, C 또는 T)에서 발생한다.
- 균류와 동물에서 5-메틸시토신은 주로 CpG 디뉴클레오티드에서 발생한다.대부분의 진핵생물은 이러한 부위의 극히 일부만 메틸화하지만, CpG 세포질의 70-80%는 척추동물에서 메틸화된다.포유동물 세포에서 유전자의 5' 말단에 있는 CpG 군집을 CpG [9]섬이라고 한다.포유류 DNA의 1%는 [10]5mC이다.
시토신의 자발적 탈아미노화는 DNA 복구 효소에 의해 인식되고 제거되는 우라실을 형성하는 반면, 5-메틸시토신의 탈아미노화는 티민을 형성한다.시토신(C)에서 티민(T)으로 DNA 염기의 전환은 전이 [11]돌연변이를 일으킬 수 있다.또한 APOBEC 계열의 시토신 탈아미나제에 의한 시토신 또는 5-메틸시토신의 활성 효소 탈아미나화는 다양한 세포 과정 및 생물 [12]진화에 유익한 영향을 미칠 수 있다.반면, 5-히드록시메틸시토신에 대한 탈아미네이션의 영향은 덜 알려져 있다.
시험관내
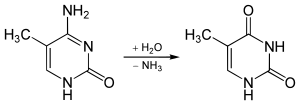
NH2 그룹은 아질산과 같은 시약을 사용하여 5-메틸시토신에서 제거(탈아미네이션)하여 티민을 형성할 수 있다. 시토신은 유사한 조건에서 유라실(U)로 탈아미네이트를 사용한다.
5-메틸시토신은 시토신 잔기를 탈아미네이트화하는 중황산염 처리에 의한 탈아미네이션에 내성이 있다.이 특성은 종종 DNA 시토신 메틸화 패턴을 분석하기 위해 이용된다.[13]
DNMT(Eukaryotes)에 의한 추가 및 규제
5mC 마크는 DNA메틸전달효소(DNMT)를 통해 게놈 DNA에 배치된다.인간에게는 DNMT1, DNMT2, DNMT3A, DNMT3B, DNMT3L의 5가지 DNMT가 있으며 조류와 곰팡이에는 3가지(DNMT4, DNMT5, DNMT6)[14]가 더 존재한다.DNMT1는 CXXC 메틸화의 DNA.[15]DNMT1에 실제로 새로운 추가에 아연 손가락 도메인이 포함된 무선 주파수 시험기 DNA복제의 장소에 5mC의 딸 가닥에 DNA복제 중에 정비를 지원하기 위해서, DNMT1을 시도한다가 복제 FOCUS의 복수 시퀀스(무선 주파수 시험기)을 목표로 하고 5mC 마크의 추가 촉매제 역할은 CXXC 도메인을 포함하고 있다.was는 모든 인체 [16]조직에서 지배적인 DNA 메틸전달효소인 것으로 밝혀졌다.주로 DNMT3A와 DNMT3B가 de novo 메틸화를 담당하며 DNMT1은 [1]복제 후에도 5mC 마크를 유지합니다.DNMT는 서로 상호작용하여 메틸화 능력을 높일 수 있습니다.예를 들어 2 DNMT3L은 2 DNMT3A와 복합체를 형성하여 DNA와의 상호작용을 개선하여 메틸화를 [17]촉진할 수 있다.DNMT의 발현 변화는 비정상적인 메틸화를 초래한다.과잉발현은 메틸화를 증가시키는 반면, 효소의 파괴는 메틸화 [16]수준을 감소시킨다.
첨가 메커니즘은 다음과 같다. 먼저 DNMT의 PCQ 모티브에 있는 시스테인 잔류물은 메틸화되어야 하는 시토신 뉴클레오티드의 탄소 6에 핵친균 공격을 일으킨다.다음으로 S-아데노실메티오닌은 메틸기를 탄소5에 공여한다.DNMT 효소 중 염기는 고리 중 탄소5와 탄소6의 이중결합을 복원하는 탄소5 위의 잔류수소를 탈양성자화하여 5-메틸시토신 염기쌍을 [15]생성한다.
탈메틸화
시토신은 5mC까지 메틸화된 후 여러 메커니즘을 통해 초기 상태로 되돌릴 수 있다.DNMT에 의한 유지 부족에 의한 복제를 통한 수동적 DNA 탈메틸화는 점진적으로 표식을 제거한다. 활성 DNA 탈메틸화에서는 일련의 산화가 이를 5-히드록시메틸시토신(5hmC), 5-포르밀시토신(5fC) 및 5-카르복실시토신(5C)으로 변환하고, 최종적으로 2-카르복실시토신(5C)으로 변환한다.시토신을 복원하기 위해 BER([1]Base 절제 복구)를 수행합니다.TDG 녹아웃은 통계적으로 유의한 수준인 5hmC의 변화 없이 5fC의 2배가량 증가하였다. 이는 5mC가 완전히 [18]탈메틸화되기 전에 최소 2회 반복 산화되어야 함을 의미한다.산화는 5mC, 5hmC, 5fC를 산화 형태로 변환할 수 있는 TET(Ten-Eleven translocation) 패밀리 디옥시게나아제(TET 효소)를 통해 발생합니다.단, 효소는 5mC에 대한 선호도가 가장 높으며 TET2와의 5hmC 및 5fC 전환 초기 반응 속도는 4.9~7.6배 [19]느리다.TET는 보조인자로서 Fe(II)를, 기질로서 산소와 α-케토글루타르산(α-KG)을 필요로 하며, 후자의 기질은 이소시트레이트탈수소효소(IDH)[20]에 의해 이소시트레이트로부터 생성된다.그러나 암은 α-KG와 경쟁하는 2-히드록시글루타르산(2HG)을 생성해 TET 활성을 감소시키고 5mC에서 [21]5hmC로의 전환을 감소시킬 수 있다.
인간의 역할
암에서
암의 경우 DNA는 과잉메틸화(hypermethylation)와 과소메틸화(hypomethylation)[22] 둘 다 될 수 있다.중복되는 CpG섬 유전자 프로모터는 탈노보메틸화되어 종양의 성장저해와 통상 관련된 유전자의 비정상적인 불활성화를 초래한다(과메틸화의 [23]예).종양과 정상 조직을 비교하면 전자는 메틸전달효소 DNMT1, DNMT3A 및 대부분 DNMT3B의 수치가 높았으며, 모두 [16]암의 5mC 이상 수치와 관련이 있었다.위성 DNA, Alu, 그리고 긴 인터스펙트 요소(LINE)를 포함한 게놈의 반복 배열은 암에서 종종 저메틸화 되는 것으로 보여지며, 이러한 정상 침묵 유전자의 발현을 초래하고, 수준은 종종 종양 [22]진행의 중요한 지표이다.이상 데노보 5mC 메틸화를 생성하는 DNA 메틸전달효소의 과잉활성은 후생유전학적 수리의 일종인 메틸화를 제거함으로써 보상될 수 있다.그러나 메틸화의 제거는 비효율적이어서 게놈 전체의 저메틸화가 과다하게 발생한다.그 반대도 가능하다; 저메틸화의 과잉 발현은 게놈 전체의 고메틸화에 의해 [22]침묵될 수 있다.암 특징 기능은 암세포와 종양 미세 환경 [24]내 주변 종양 관련 스트로마 모두에서 5mC를 변화시키는 후생유전학적 변화를 통해 획득될 수 있다.항암제인 시스플라틴은 [25]5mC와 반응하는 것으로 보고되었다.
노화의 바이오마커로서
"[26]유전 연령"은 게놈의 DNA 메틸화 수준과 연대기 나이 사이의 연관성을 의미한다."clock CpGs"라고 불리는 특정 세트의 CpGs에서 DNA 메틸화 수준을 특정 연대기에 집단 게놈 전체 메틸화의 전형적인 수준을 회귀시키는 알고리즘과 결합하면 후생유전적 나이를 예측할 수 있다.청년기(0~20세)에는 발달과 성장이 진행됨에 따라 DNA 메틸화의 변화가 더 빠른 속도로 일어나며, 나이가 들수록 변화가 느려지기 시작한다.복수의 후생유전 연령 추정치가 존재한다.Horvath의 시계는 353개의 CpGs의 다중 조직 세트를 측정하는데, 이 중 절반은 나이와 양의 상관관계가 있고 나머지 절반은 음의 상관관계가 있어 후생연대를 [27]추정한다.Hannum의 시계는 71CpGs의 [28]직교 기준으로 나이를 계산하기 위해 성인 혈액 샘플을 사용합니다.DNAm PhenoAge로 알려진 Levine의 시계는 513CpGs에 의존하며 사망률과 수명을 예측하는 데 있어 다른 연령 추정치를 능가하지만 비혈액 조직에 [29]대한 편향을 보인다.유전자 ELOVL2에서 [30]오직 하나의 CpG의 메틸화 상태를 가진 연령 추정기에 대한 보고가 있다.연령 추정은 5mC 메틸화 마커에 기초하여 개인이 대상일 수 있는 연령 관련 조건의 예상을 통해 예측 수명을 허용한다.
레퍼런스
- ^ a b c Wu, Xiaoji; Zhang, Yi (2017-05-30). "TET-mediated active DNA demethylation: mechanism, function and beyond". Nature Reviews Genetics. 18 (9): 517–534. doi:10.1038/nrg.2017.33. ISSN 1471-0056. PMID 28555658. S2CID 3393814.
- ^ Matthews AP (2012). Physiological Chemistry. Williams & Wilkins Company/. p. 167. ISBN 978-1130145373.
- ^ Johnson TB, Coghill RD (1925). "The discovery of 5-methyl-cytosine in tuberculinic acid, the nucleic acid of the Tubercle bacillus". J Am Chem Soc. 47 (11): 2838–2844. doi:10.1021/ja01688a030.
- ^ Grosjean H(2009).핵산은 네 가지 유형의 뉴클레오티드로만 구성된 긴 중합체를 지루하게 만들지 않습니다. 가이드 투어Landes Bioscience.
- ^ Vischer E, Zamenhof S, Chargaff E (1949). "Microbial nucleic acids: the desoxypentose nucleic acids of avian tubercle bacilli and yeast". J Biol Chem. 177 (1): 429–438. doi:10.1016/S0021-9258(18)57100-3. PMID 18107446.
- ^ Hotchkiss RD (1948). "The quantitative separation of purines, pyrimidines and nucleosides by paper chromatography". J Biol Chem. 175 (1): 315–332. doi:10.1016/S0021-9258(18)57261-6. PMID 18873306.
- ^ Squires JE, Patel HR, Nousch M, Sibbritt T, Humphreys DT, Parker BJ, Suter CM, Preiss T (2012). "Widespread occurrence of 5-methylcytosine in human coding and non-coding RNA". Nucleic Acids Res. 40 (11): 5023–5033. doi:10.1093/nar/gks144. PMC 3367185. PMID 22344696.
- ^ Colot V, Rossignol JL (1999). "Eukaryotic DNA methylation as an evolutionary device". BioEssays. 21 (5): 402–411. doi:10.1002/(SICI)1521-1878(199905)21:5<402::AID-BIES7>3.0.CO;2-B. PMID 10376011. S2CID 10784130.
- ^ Bird, Adrian P. (May 1986). "CpG-rich islands and the function of DNA methylation". Nature. 321 (6067): 209–213. Bibcode:1986Natur.321..209B. doi:10.1038/321209a0. ISSN 0028-0836. PMID 2423876. S2CID 4236677.
- ^ Ehrlich, M.; Wang, R. Y. (1981-06-19). "5-Methylcytosine in eukaryotic DNA". Science. 212 (4501): 1350–1357. Bibcode:1981Sci...212.1350E. doi:10.1126/science.6262918. ISSN 0036-8075. PMID 6262918.
- ^ Sassa A, Kanemaru Y, Kamoshita N, Honma M, Yasui M (2016). "Mutagenic consequences of cytosine alterations site-specifically embedded in the human genome". Genes and Environment. 38 (1): 17. doi:10.1186/s41021-016-0045-9. PMC 5007816. PMID 27588157.
- ^ Chahwan R, Wontakal SN, Roa S (2010). "Crosstalk between genetic and epigenetic information through cytosine deamination". Trends in Genetics. 26 (10): 443–448. doi:10.1016/j.tig.2010.07.005. PMID 20800313.
- ^ Clark SJ, Harrison J, Paul CL, Frommer M (1994). "High sensitivity mapping of methylated cytosines". Nucleic Acids Res. 22 (15): 2990–2997. doi:10.1093/nar/22.15.2990. PMC 310266. PMID 8065911.
- ^ Ponger, Loïc; Li, Wen-Hsiung (2005-04-01). "Evolutionary Diversification of DNA Methyltransferases in Eukaryotic Genomes". Molecular Biology and Evolution. 22 (4): 1119–1128. doi:10.1093/molbev/msi098. ISSN 0737-4038. PMID 15689527.
- ^ a b Lyko, Frank (February 2018). "The DNA methyltransferase family: a versatile toolkit for epigenetic regulation". Nature Reviews Genetics. 19 (2): 81–92. doi:10.1038/nrg.2017.80. ISSN 1471-0064. PMID 29033456. S2CID 23370418.
- ^ a b c Robertson, K D; Uzvolgyi, E; Liang, G; Talmadge, C; Sumegi, J; Gonzales, F A; Jones, P A (1999-06-01). "The human DNA methyltransferases (DNMTs) 1, 3a and 3b: coordinate mRNA expression in normal tissues and overexpression in tumors". Nucleic Acids Research. 27 (11): 2291–2298. doi:10.1093/nar/27.11.2291. ISSN 0305-1048. PMC 148793. PMID 10325416.
- ^ Jia, Da; Jurkowska, Renata Z.; Zhang, Xing; Jeltsch, Albert; Cheng, Xiaodong (September 2007). "Structure of Dnmt3a bound to Dnmt3L suggests a model for de novo DNA methylation". Nature. 449 (7159): 248–251. Bibcode:2007Natur.449..248J. doi:10.1038/nature06146. ISSN 1476-4687. PMC 2712830. PMID 17713477.
- ^ Song, Chun-Xiao; Szulwach, Keith E.; Dai, Qing; Fu, Ye; Mao, Shi-Qing; Lin, Li; Street, Craig; Li, Yujing; Poidevin, Mickael; Wu, Hao; Gao, Juan (2013-04-25). "Genome-wide profiling of 5-formylcytosine reveals its roles in epigenetic priming". Cell. 153 (3): 678–691. doi:10.1016/j.cell.2013.04.001. ISSN 1097-4172. PMC 3657391. PMID 23602153.
- ^ Ito, Shinsuke; Shen, Li; Dai, Qing; Wu, Susan C.; Collins, Leonard B.; Swenberg, James A.; He, Chuan; Zhang, Yi (2011-09-02). "Tet Proteins Can Convert 5-Methylcytosine to 5-Formylcytosine and 5-Carboxylcytosine". Science. 333 (6047): 1300–1303. Bibcode:2011Sci...333.1300I. doi:10.1126/science.1210597. ISSN 0036-8075. PMC 3495246. PMID 21778364.
- ^ Lu, Xingyu; Zhao, Boxuan Simen; He, Chuan (2015-02-12). "TET Family Proteins: Oxidation Activity, Interacting Molecules, and Functions in Diseases". Chemical Reviews. 115 (6): 2225–2239. doi:10.1021/cr500470n. ISSN 0009-2665. PMC 4784441. PMID 25675246.
- ^ Xu, Wei; Yang, Hui; Liu, Ying; Yang, Ying; Wang, Ping; Kim, Se-Hee; Ito, Shinsuke; Yang, Chen; Wang, Pu; Xiao, Meng-Tao; Liu, Li-xia (2011-01-18). "Oncometabolite 2-Hydroxyglutarate Is a Competitive Inhibitor of α-Ketoglutarate-Dependent Dioxygenases". Cancer Cell. 19 (1): 17–30. doi:10.1016/j.ccr.2010.12.014. ISSN 1535-6108. PMC 3229304. PMID 21251613.
- ^ a b c Ehrlich, Melanie (2009-12-01). "DNA hypomethylation in cancer cells". Epigenomics. 1 (2): 239–259. doi:10.2217/epi.09.33. ISSN 1750-1911. PMC 2873040. PMID 20495664.
- ^ Jones, Peter A. (1996-06-01). "DNA Methylation Errors and Cancer". Cancer Research. 56 (11): 2463–2467. ISSN 0008-5472. PMID 8653676.
- ^ Hanahan, Douglas; Weinberg, Robert A. (2011-03-04). "Hallmarks of Cancer: The Next Generation". Cell. 144 (5): 646–674. doi:10.1016/j.cell.2011.02.013. ISSN 0092-8674. PMID 21376230.
- ^ Menke, Annika; Dubini, Romeo C.A.; Mayer, Peter; Rovó, Petra; Daumann, Lena (2020-10-23). "Formation of Cisplatin Adducts with the Epigenetically-relevant Nucleobase 5-Methylcytosine". European Journal of Inorganic Chemistry. 2021: 30–36. doi:10.1002/ejic.202000898. ISSN 1434-1948.
- ^ Horvath, Steve; Raj, Kenneth (June 2018). "DNA methylation-based biomarkers and the epigenetic clock theory of ageing". Nature Reviews Genetics. 19 (6): 371–384. doi:10.1038/s41576-018-0004-3. ISSN 1471-0064. PMID 29643443. S2CID 4709691.
- ^ Horvath, Steve (2013-12-10). "DNA methylation age of human tissues and cell types". Genome Biology. 14 (10): 3156. doi:10.1186/gb-2013-14-10-r115. ISSN 1474-760X. PMC 4015143. PMID 24138928.
- ^ Hannum, Gregory; Guinney, Justin; Zhao, Ling; Zhang, Li; Hughes, Guy; Sadda, SriniVas; Klotzle, Brandy; Bibikova, Marina; Fan, Jian-Bing; Gao, Yuan; Deconde, Rob (2013-01-24). "Genome-wide Methylation Profiles Reveal Quantitative Views of Human Aging Rates". Molecular Cell. 49 (2): 359–367. doi:10.1016/j.molcel.2012.10.016. ISSN 1097-2765. PMC 3780611. PMID 23177740.
- ^ Levine, Morgan E.; Lu, Ake T.; Quach, Austin; Chen, Brian H.; Assimes, Themistocles L.; Bandinelli, Stefania; Hou, Lifang; Baccarelli, Andrea A.; Stewart, James D.; Li, Yun; Whitsel, Eric A. (2018-04-17). "An epigenetic biomarker of aging for lifespan and healthspan". Aging (Albany NY). 10 (4): 573–591. doi:10.18632/aging.101414. ISSN 1945-4589. PMC 5940111. PMID 29676998.
- ^ Garagnani, Paolo; Bacalini, Maria G.; Pirazzini, Chiara; Gori, Davide; Giuliani, Cristina; Mari, Daniela; Blasio, Anna M. Di; Gentilini, Davide; Vitale, Giovanni; Collino, Sebastiano; Rezzi, Serge (2012). "Methylation of ELOVL2 gene as a new epigenetic marker of age". Aging Cell. 11 (6): 1132–1134. doi:10.1111/acel.12005. hdl:11585/128353. ISSN 1474-9726. PMID 23061750. S2CID 8775590.
문학.
- Griffiths, Anthony J. F. (1999). An Introduction to genetic analysis. San Francisco: W.H. Freeman. Chapter 15: Gene Mutation. ISBN 0-7167-3520-2. (미국 국립생명공학정보센터에서 온라인으로 입수 가능)