탄수화물결합모듈
Carbohydrate-binding module| CBM_1 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 트리코더마 리세이, nmr, 18개 구조물의 셀로비오하이드롤라아제 i의 3개 공학적 셀룰로오스계 영역 3차원 구조물 | |||||||||
| 식별자 | |||||||||
| 기호 | CBM_1 | ||||||||
| Pfam | PF00734 | ||||||||
| 인터프로 | IPR000254 | ||||||||
| 프로사이트 | PDOC00486 | ||||||||
| SCOP2 | 1cel / SCOPe / SUPFAM | ||||||||
| CAZY | CBM1 | ||||||||
| |||||||||
| CBM_2 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 핵자기 공명 분광법에 의한 셀룰로모나스 피미로부터 셀룰로오스 결합 영역의 솔루션 구조 | |||||||||
| 식별자 | |||||||||
| 기호 | CBM_2 | ||||||||
| Pfam | PF00553 | ||||||||
| Pfam 씨 | CL0203 | ||||||||
| 인터프로 | IPR001919 | ||||||||
| 프로사이트 | PDOC00485 | ||||||||
| SCOP2 | 1exg / SCOPe / SUPFAM | ||||||||
| CAZY | CBM2 | ||||||||
| |||||||||
| CBM_3 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 클로스트리디움 셀룰롤리틱움에서 추출한 2ia cbd 계열의 결정 구조 | |||||||||
| 식별자 | |||||||||
| 기호 | CBM_3 | ||||||||
| Pfam | PF00942 | ||||||||
| Pfam 씨 | CL0203 | ||||||||
| 인터프로 | IPR001956 | ||||||||
| SCOP2 | 1nbc / SCOPe / SUPFAM | ||||||||
| CAZY | CBM3 | ||||||||
| |||||||||
| CBM_5/12 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 18가지 키티나아제와 설계된 억제제 hm508의 상호작용과 그 분해물인 키토비오노-delta-lactone의 상호작용 | |||||||||
| 식별자 | |||||||||
| 기호 | CBM_5_12 | ||||||||
| Pfam | PF02839 | ||||||||
| 인터프로 | IPR003610 | ||||||||
| SCOP2 | 1ed7 / SCOPe / SUPFAM | ||||||||
| CAZY | CBM12 | ||||||||
| |||||||||
| CBM_6 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 xylopentaose 복합체의 클로스트리디움 열전지의 cbm6ct | |||||||||
| 식별자 | |||||||||
| 기호 | CBM_6 | ||||||||
| Pfam | PF03422 | ||||||||
| Pfam 씨 | CL0202 | ||||||||
| 인터프로 | IPR005084 | ||||||||
| SCOP2 | 1gmm / SCOPe / SUPFAM | ||||||||
| CAZY | CBM6 | ||||||||
| |||||||||
| CBM_4/9 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
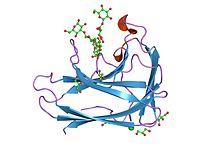 cbm4 구조 및 기능 | |||||||||
| 식별자 | |||||||||
| 기호 | CBM_4_9 | ||||||||
| Pfam | PF02018 | ||||||||
| Pfam 씨 | CL0202 | ||||||||
| 인터프로 | IPR003305 | ||||||||
| SCOP2 | 1ulp / SCOPe / SUPFAM | ||||||||
| CAZY | CBM22 | ||||||||
| |||||||||
| CBM_10 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 x cbm 유형의 솔루션 구조 | |||||||||
| 식별자 | |||||||||
| 기호 | CBM_10 | ||||||||
| Pfam | PF02013 | ||||||||
| 인터프로 | IPR002883 | ||||||||
| SCOP2 | 1qld / SCOPe / SUPFAM | ||||||||
| CAZY | CBM10 | ||||||||
| |||||||||
| CBM_11 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 제품군 11 탄수화물-셀룰로오솜 셀룰로아제 Lic26a-cel5e 클로스트리디움 써모셀럼 | |||||||||
| 식별자 | |||||||||
| 기호 | CBM_11 | ||||||||
| Pfam | PF03425 | ||||||||
| Pfam 씨 | CL0202 | ||||||||
| 인터프로 | IPR005087 | ||||||||
| CAZY | CBM11 | ||||||||
| |||||||||
| CBM_14 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| 식별자 | |||||||||
| 기호 | CBM_14 | ||||||||
| Pfam | PF01607 | ||||||||
| Pfam 씨 | CL0155 | ||||||||
| 인터프로 | IPR002557 | ||||||||
| SCOP2 | 1dqc / SCOPe / SUPFAM | ||||||||
| CAZY | CBM14 | ||||||||
| |||||||||
| CBM_15 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 Xylan-filename 모듈 cbm15 | |||||||||
| 식별자 | |||||||||
| 기호 | CBM_15 | ||||||||
| Pfam | PF03426 | ||||||||
| Pfam 씨 | CL0202 | ||||||||
| 인터프로 | IPR005088 | ||||||||
| SCOP2 | 1gny / SCOPe / SUPFAM | ||||||||
| CAZY | CBM15 | ||||||||
| |||||||||
| CBM_17/28 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 클로스트리디움 셀룰로보란스의 fam17 탄수화물 결합모듈 구조 | |||||||||
| 식별자 | |||||||||
| 기호 | CBM_17_28 | ||||||||
| Pfam | PF03424 | ||||||||
| Pfam 씨 | CL0202 | ||||||||
| 인터프로 | IPR005086 | ||||||||
| SCOP2 | 1g0c / SCOPe / SUPFAM | ||||||||
| CAZY | CBM28 | ||||||||
| |||||||||
| 치틴_bind_1 (CBM18) | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 교차연계wga3/glcnacbeta1,4glcnac 복합체의 결정구조 해석 | |||||||||
| 식별자 | |||||||||
| 기호 | 치틴_bind_1 | ||||||||
| Pfam | PF00187 | ||||||||
| 인터프로 | IPR001002 | ||||||||
| 프로사이트 | PDOC00025 | ||||||||
| SCOP2 | 1wgt / SCOPe / SUPFAM | ||||||||
| CAZY | CBM18 | ||||||||
| |||||||||
| CBM_19 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| 식별자 | |||||||||
| 기호 | CBM_19 | ||||||||
| Pfam | PF03427 | ||||||||
| Pfam 씨 | CL0155 | ||||||||
| 인터프로 | IPR005089 | ||||||||
| CAZY | CBM19 | ||||||||
| |||||||||
| CBM_20 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 글루코아밀라아제, 사이클로덱스트린, nmr, 최소화된 평균 구조를 가진 세분화된 전분 함유 도메인 복합체 | |||||||||
| 식별자 | |||||||||
| 기호 | CBM_20 | ||||||||
| Pfam | PF00686 | ||||||||
| Pfam 씨 | CL0369 | ||||||||
| 인터프로 | IPR002044 | ||||||||
| SCOP2 | 1cdg / SCOPe / SUPFAM | ||||||||
| CAZY | CBM20 | ||||||||
| |||||||||
| CBM_21 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| 식별자 | |||||||||
| 기호 | CBM_21 | ||||||||
| Pfam | PF03370 | ||||||||
| 인터프로 | IPR005036 | ||||||||
| CAZY | CBM21 | ||||||||
| |||||||||
| CBM_25 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| 식별자 | |||||||||
| 기호 | CBM_25 | ||||||||
| Pfam | PF03423 | ||||||||
| 인터프로 | IPR005085 | ||||||||
| CAZY | CBM25 | ||||||||
| |||||||||
| CBM27 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 탄수화물-배출 모듈, tmcbm27에 의한 특정 맨난 인식의 구조 및 열역학 해부 | |||||||||
| 식별자 | |||||||||
| 기호 | CBM27 | ||||||||
| Pfam | PF09212 | ||||||||
| 인터프로 | IPR015295 | ||||||||
| SCOP2 | 1oh4 / SCOPe / SUPFAM | ||||||||
| |||||||||
| 치틴_bind_3 (CBM33) | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 세라티아의 결정 구조 치틴 단백질 cbp21 y54a 돌연변이. | |||||||||
| 식별자 | |||||||||
| 기호 | 치틴_bind_3 | ||||||||
| Pfam | PF03067 | ||||||||
| 인터프로 | IPR004302 | ||||||||
| CAZY | CBM33 | ||||||||
| |||||||||
| CBM_48 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 설포오부스 솔파타리쿠스로부터의 글리코실트레할로스 트레할로하이드롤라아제의 결정구조 | |||||||||
| 식별자 | |||||||||
| 기호 | CBM_48 | ||||||||
| Pfam | PF02922 | ||||||||
| Pfam 씨 | CL0369 | ||||||||
| 인터프로 | IPR004193 | ||||||||
| SCOP2 | 1bf2 / SCOPe / SUPFAM | ||||||||
| CAZY | CBM48 | ||||||||
| |||||||||
| CBM49 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| 식별자 | |||||||||
| 기호 | CBM49 | ||||||||
| Pfam | PF09478 | ||||||||
| Pfam 씨 | CL0203 | ||||||||
| 인터프로 | IPR019028 | ||||||||
| |||||||||
분자생물학에서 탄수화물 결합 모듈(CBM)은 탄수화물-능동 효소(예: 글리코사이드 수산화물)에서 발견되는 단백질 영역이다.이 영역의 대부분은 탄수화물 결합 활동을 가지고 있다.이 영역들 중 일부는 셀룰로오솜 비계 단백질에서 발견된다.CBM은 이전에 셀룰로오스 바인딩 도메인으로 알려져 있었다.[1]CBM은 아미노산 염기서열 유사성에 기초하여 수많은 계열로 분류된다.CAZY 데이터베이스에는 현재 (2011년 6월) 64개 CBM 제품군이 있다.[2]
미생물 글리코사이드 수산화물의 CBM은 특정 식물 구조 다당체에 대한 결합을 통해 광합성 고정 탄소의 재활용에 중심적인 역할을 한다.[3]CBM은 결정성과 비정형 셀룰로오스 형태를 모두 인식할 수 있다.[4]CBM은 식물 세포벽 가수분해에서 활성하는 효소와 관련된 가장 일반적인 비 촉매 모듈이다.많은 Putative CBM은 아미노산 염기서열 정렬에 의해 확인되었지만 소수의 대표자만이 탄수화물 결합 기능을 가지고 있는 것으로 실험적으로 보여졌다.[5]
CBM1
탄수화물 결합 모듈 제품군 1(CBM1)은 36개의 아미노산으로 구성된다.이 영역에는 두 개의 이황화 결합 형성과 관련된 보존된 4개의 시스테인 잔류물이 있다.
CBM2
탄수화물 결합 모듈 제품군 2(CBM2)는 이황화 결합에 관여하는 것으로 밝혀진 두 개의 보존된 사이스테인을 포함한다.또한 보존된 4개의 트립토판도 있는데, 이 중 2개는 셀룰로오스 결합에 관여한다.[7][8][9]
CBM3
탄수화물 결합 모듈 제품군 3(CBM3)은 셀룰로오스 결합에 관여하며, 광범위한 박테리아 글리코실 하이드롤라임과 관련된 것으로 확인된다.이 영역의 구조가 알려져 있다; 그것은 베타 샌드위치를 형성한다.[11]
CBM4
탄수화물 결합 모듈 제품군 4(CBM4)는 셀룰로모나스 피미에서 나온 1,4베타-글루카나아제, CenC의 N 종단부에 나란히 배열된 두 개의 셀룰로오스 결합 도메인 CBD(N1)와 CBD(N2)를 포함한다.이러한 동질 CBM은 결정성 셀룰로오스가 아닌 무형의 결합을 위한 선택성에서 구별된다.[12]다차원 이핵 자기공명(NMR) 분광법을 사용하여 C. fimi 1,4-beta-glucanase CenC(CBDN1)에서 152개의 아미노산 N단자 셀룰로오스 결합 영역의 3차 구조를 결정했다.CBDN1의 3차 구조는 박테리아 1,3-1,4-베타-글루카나제뿐 아니라 젤리 롤 접이식 다른 당밀결합 단백질과 현저하게 유사하다.[13]CBM4와 CBM9는 밀접한 관련이 있다.
CBM5
탄수화물 결합 모듈 제품군 5(CBM5)는 키틴을 결합한다.[14]CBM5와 CBM12는 멀리 관계가 있다.
CBM6
탄수화물 결합 모듈 제품군 6(CBM6)은 기판 결합 부위 2개(구분 A)와 구분 B)를 포함하고 있다는 점에서 이례적이다.Cellvibrio mixtus endoglucanase 5A는 두 개의 CBM6 도메인을 포함하고 있으며, C-terminus의 CBM6 도메인은 각 기질 결합 구획에 뚜렷한 리간드 결합 특이성을 나타낸다.Cleft A와 Cleft B는 모두 첼로-올리고사카리드를 결합할 수 있으며, 라미나린은 우선적으로 분할 A에서 결합할 수 있으며, Xylooligosaccharide는 분할 A에서만 결합되며, 베타1,4,-베타1,3-연계 글루칸은 분할 B에서만 결합할 수 있다.[15]
CBM9
탄수화물 결합 모듈 제품군 9(CBM9)는 결정 셀룰로오스에 결합한다.[16]CBM4와 CBM9는 밀접한 관련이 있다.
CBM10
탄수화물 결합 모듈 제품군 10(CBM10)은 서로 다른 기능을 가진 두 개의 뚜렷한 단백질 세트에서 발견된다.에어로빅 박테리아에서 발견되는 것들은 셀룰로오스(또는 다른 탄수화물)를 결합하지만 혐기성 곰팡이에서는 도케린 영역으로 불리는 단백질 결합 영역이다.도케린 영역은 특정 혐기성 박테리아에서 발견되는 셀룰로솜과 유사한 다단백질 셀룰로아제/헤미셀룰라제 복합체의 조립을 담당하는 것으로 여겨진다.[17][18]
클로스트리디움 열전지로 대표되는 식물 세포벽을 저하시키는 혐기성 박테리아에서 촉매 폴리펩타이드의 도케린 영역은 동일한 유기체의 어떤 응집에도 똑같이 잘 결합될 수 있다.좀 더 최근에는 박테리아와 곰팡이 효소의 도케린 순서는 완전히 다르지만, 피로미세스 에퀴에 의해 특징지어지는 혐기성 곰팡이도 셀룰로솜 콤플렉스를 합성하는 것이 제안되고 있다.[19]예를 들어, 진균 효소는 촉매 폴리펩타이드 내에서 도케린 시퀀스의 1개, 2개 또는 3개의 복사본을 포함한다.이와는 대조적으로 모든 C. 열전지 셀룰로솜 촉매 성분은 단일 도케린 영역을 포함하고 있다.혐기성 박테리아 도케린은 EF 손(칼슘 결합 모티브)에 동질적이며 활동을 위해 칼슘을 필요로 하는 반면 진균 도케린은 칼슘을 필요로 하지 않는다.마지막으로 응집력과 도케린의 상호작용은 박테리아에 특유한 종으로 나타나며, 곰팡이 종 내에서 결합하는 종 특이성이 거의 없으며, 서로 다른 종을 구별하는 확인된 부위도 없다.
P. equi의 도커린에는 2개의 헬리컬 스트레칭과 4개의 짧은 베타 스트랜드가 포함되어 있으며, 이는 추가적인 짧은 꼬임 평행 가닥에 인접한 반팔 시트 구조를 형성한다.N-와 C-termini는 서로 인접해 있다.[19]
CBM11
탄수화물 결합 모듈 제품군 11(CBM11)은 다수의 박테리아 셀룰라아제에서 발견된다.Clostridium Thermocellum Cel26A-Cel5E의 CBM11을 예로 들 수 있는데, 이 도메인은 β-1,4-글루칸과 β-1,3-1,4-혼합된 글루칸을 모두 바인딩하는 것으로 나타났다.[20]CBM11은 오목한 면이 기질 결합 구분을 형성하는 베타-샌드위치 구조를 가지고 있다.[20]
CBM12
탄수화물 결합 모듈 제품군 12(CBM12)는 2개의 베타 시트로 구성되며, 각각 2개와 3개의 항타렐 베타 스트랜드로 구성된다.그것은 트립토판 잔류물의 향기로운 고리를 통해 키틴을 결합한다.[14]CBM5와 CBM12는 멀리 관계가 있다.
CBM14
탄수화물 결합 모듈 패밀리 14(CBM14)는 페리트로핀-A 도메인으로도 알려져 있다.그것은 키틴 결합 단백질, 특히 곤충과 동물 키티나스의 근위축성 기질 단백질에서 발견된다.[21][22][23]도메인의 복사본은 일부 바쿨로바이러스에서 발견되기도 한다.그것은 세포외 영역으로, 아마도 3개의 이황화 다리를 형성하는 6개의 보존된 사이스테인을 포함하고 있다.치틴 결합은 이러한 영역 중 두 개만 포함하는 단백질에 대해 입증되었다.[21]
CBM15
박테리아 효소에서 발견된 탄수화물 결합 모듈 제품군 15(CBM15)는 자일란과 자일리고사카리드에 결합하는 것으로 나타났다.베타 젤리 롤 접이식(beta-jelly roll fold)[3]으로, 베타 시트 중 하나의 오목한 표면에 홈이 있다.
CBM17
Carbohydrate-binding 모듈 가족 17(CBM17)더 셀룰로오스의 체인점에 비정질 섬유소에 가족은 4CBMs.[24]시각적 국면의 보다 깊은 구속력을 억누르고 가족 내에서 CBM17과 CBM28 그들이 유전자 중복 사용함으로써와 진화를 갖고 있다고 말한다 구조적 보존보다 접근할 수 있게 될 수 있는 얕은 바인딩 갈라진 것으로 보인다.sequ난립한 [4]차이CBM17은 비결정 셀룰로오스에 바인딩할 때 CBM28 모듈과 경쟁하지 않는다.서로 다른 CBM이 비정형 셀룰로오스의 서로 다른 거품에 결합하는 것으로 나타났으며, CBM17과 CBM28은 비정형 셀룰로오스의 구별되는 과대파핑 부지를 인식한다.[25]
CBM18
Carbohydrate-binding module family 18 (CBM18) (also known as chitin binding 1 or chitin recognition protein) is found in a number of plant and fungal proteins that bind N-acetylglucosamine (e.g. solanaceous lectins of tomato and potato, plant endochitinases, the wound-induced proteins: hevein, win1 and win2, and the Kluyveromyces lactis killer toxin 알파 서브 유닛.[26]도메인은 하나 이상의 복사본에서 발생할 수 있으며 키틴 서브유닛의 인식 또는 바인딩에 관여하는 것으로 간주된다.[27][28]감자 상처에 의한 단백질뿐만 아니라 키티나스에서 이 43-리시듀 영역은 신호 순서를 직접적으로 따르며 따라서 성숙한 단백질의 N 종단부에 있다; 킬러 독소 알파 서브 유닛에서는 단백질의 중앙 부분에 위치한다.
CBM19
진균 치틴아제에서 발견된 탄수화물 결합 모듈 제품군 19(CBM19)는 키틴을 결합한다.[29]
CBM20
탄수화물 결합 모듈 제품군 20(CBM20)은 전분에 결합한다.[30][31]
CBM21
글리코겐 신진대사에 관여하는 많은 진핵 단백질에서 발견된 탄수화물 결합 모듈 계열 21(CBM21)은 글리코겐에 결합한다.[32]
CBM25
탄수화물 결합 모듈 제품군 25(CBM25)는 알파 글루쿨리고사당류, 특히 알파-1,6 링크와 세분화된 전분을 함유한 것을 결합한다.[33]
CBM27
탄수화물 결합 모듈 제품군 27(CBM27)은 베타-1,4만물리고사카리드, 카로브 갈락토만난, 곤약 글루코만난에 결합하지만 셀룰로오스(불용성 및 용해성)나 수용성 자작나무 자일란에는 결합하지 않는다.CBM27은 13개의 베타 가닥과 하나의 작은 알파-헬릭스, 하나의 금속 원자로 구성된 베타 샌드위치 구조를 채택하고 있다.[34]
CBM28
탄수화물 결합 모듈 제품군 28(CBM28)은 비결정 셀룰로오스에 결합할 때 CBM17 모듈과 경쟁하지 않는다.서로 다른 CBM이 비정형 셀룰로오스의 서로 다른 거품에 결합하는 것으로 나타났으며, CBM17과 CBM28은 비정형 셀룰로오스의 구별되는 과대파핑 부지를 인식한다.CBM28은 CBM17 도메인과 구조가 유사한 "베타젤리 롤" 토폴로지를 가지고 있다.CBM17과 CBM28 계열의 시퀀스와 구조 보존은 이들이 유전자 복제와 그에 따른 분열을 통해 진화했음을 시사한다.[4][25]
CBM32
탄수화물 결합 모듈 제품군 32(CBM32)는 식물 세포벽 다당류에서 복합 글리칸에 이르는 다양한 기질에 결합한다.[35]이 모듈은 지금까지 원형, 에우박테리아, 곰팡이 등 미생물에서 발견되었다.[35]CBM32는 베타-샌드위치 접이식 접이식이고 바운드 금속 원자를 가지고 있으며, 가장 흔히 칼슘으로 관찰된다.[36]CBM32 모듈은 시알리드, B-N-아세틸글루코사미다제, α-N-아세틸글루코사미다제, 마나나나제, 갈락토오스 산화제와 같은 촉매 모듈과 연관되어 있다.[36]
CBM33
탄수화물 결합 모듈 패밀리 33(CBM33)은 치틴 결합 도메인이다.[37]베타 시트 샌드위치로 배열된 2개의 베타 시트로 구성된 피브로넥틴 타입 III 접이식 피브로넥트를 가지고 있으며, 베타 스트랜드 1과 2 사이에 위치한 3개의 짧은 나선형으로 구성된 봉오리를 가지고 있다.그것은 보존된 극지방 아미노산을 통해 키틴을 결합시킨다.[38]이 영역은 바쿨로바이러스 스피로딘과 스핀돌린 단백질에서 격리되어 발견된다.
CBM48
탄수화물 결합 모듈 제품군 48(CBM48)은 글리코실 하이드롤라제 계열 13 촉매 도메인을 포함하는 효소에서 흔히 발견된다.그것은 이소아밀라아제, 풀룰라나제, 분지 효소에 작용하는 다양한 효소에서 발견된다.이소아밀라아제는 글리코겐, 아밀로펙틴, 덱스트린에 1,6-알파-D-글루코시디드 분지 연결, 글리코겐의 1,6-글루코시디드 연결 형성에 1,4-알파-글루칸 분지 효소, 풀라나아제는 전분 분지 효소다.CBM48은 글리코겐을 결합한다.[39][40][41][42]
CBM49
탄수화물 결합 모듈 제품군 49(CBM49)는 셀룰라아제의 C-단자에서 발견되며 체외 결합 연구에서는 결정체 셀룰로오스에 결합하는 것으로 나타났다.[43]
참조
- ^ Gilkes NR, Henrissat B, Kilburn DG, Miller RC, Warren RA (June 1991). "Domains in microbial beta-1, 4-glycanases: sequence conservation, function, and enzyme families". Microbiol. Rev. 55 (2): 303–15. doi:10.1128/MMBR.55.2.303-315.1991. PMC 372816. PMID 1886523.
- ^ Cantarel, B. L.; Coutinho, P. M.; Rancurel, C.; Bernard, T.; Lombard, V.; Henrissat, B. (2009). "The Carbohydrate-Active EnZymes database (CAZy): An expert resource for Glycogenomics". Nucleic Acids Research. 37 (Database issue): D233–D238. doi:10.1093/nar/gkn663. PMC 2686590. PMID 18838391.
- ^ a b Szabo, L.; Jamal, S.; Xie, H.; Charnock, S. J.; Bolam, D. N.; Gilbert, H. J.; Davies, G. J. (2001). "Structure of a Family 15 Carbohydrate-binding Module in Complex with Xylopentaose. Evidence that xylan binds in an approximate 3-fold helical conformation". Journal of Biological Chemistry. 276 (52): 49061–49065. doi:10.1074/jbc.M109558200. PMID 11598143.
- ^ a b c Jamal S, Nurizzo D, Boraston AB, Davies GJ (May 2004). "X-ray crystal structure of a non-crystalline cellulose-specific carbohydrate-binding module: CBM28". J. Mol. Biol. 339 (2): 253–8. doi:10.1016/j.jmb.2004.03.069. PMID 15136030.
- ^ Roske Y, Sunna A, Pfeil W, Heinemann U (July 2004). "High-resolution crystal structures of Caldicellulosiruptor strain Rt8B.4 carbohydrate-binding module CBM27-1 and its complex with mannohexaose". J. Mol. Biol. 340 (3): 543–54. doi:10.1016/j.jmb.2004.04.072. PMID 15210353.
- ^ Gilkes NR, Claeyssens M, Aebersold R, Henrissat B, Meinke A, Morrison HD, Kilburn DG, Warren RA, Miller RC (December 1991). "Structural and functional relationships in two families of beta-1,4-glycanases". Eur. J. Biochem. 202 (2): 367–77. doi:10.1111/j.1432-1033.1991.tb16384.x. PMID 1761039.
- ^ Meinke A, Gilkes NR, Kilburn DG, Miller RC, Warren RA (December 1991). "Bacterial cellulose-binding domain-like sequences in eucaryotic polypeptides". Protein Seq. Data Anal. 4 (6): 349–53. PMID 1812490.
- ^ Simpson PJ, Xie H, Bolam DN, Gilbert HJ, Williamson MP (December 2000). "The structural basis for the ligand specificity of family 2 carbohydrate-binding modules". J. Biol. Chem. 275 (52): 41137–42. doi:10.1074/jbc.M006948200. PMID 10973978.
- ^ Xu, G. Y.; Ong, E.; Gilkes, N. R.; Kilburn, D. G.; Muhandiram, D. R.; Harris-Brandts, M.; Carver, J. P.; Kay, L. E.; Harvey, T. S. (1995). "Solution structure of a cellulose-binding domain from Cellulomonas fimi by nuclear magnetic resonance spectroscopy". Biochemistry. 34 (21): 6993–7009. doi:10.1021/bi00021a011. PMID 7766609.
- ^ Poole DM, Morag E, Lamed R, Bayer EA, Hazlewood GP, Gilbert HJ (December 1992). "Identification of the cellulose-binding domain of the cellulosome subunit S1 from Clostridium thermocellum YS". FEMS Microbiol. Lett. 78 (2–3): 181–6. doi:10.1016/0378-1097(92)90022-g. PMID 1490597.
- ^ Tormo J, Lamed R, Chirino AJ, Morag E, Bayer EA, Shoham Y, Steitz TA (November 1996). "Crystal structure of a bacterial family-III cellulose-binding domain: a general mechanism for attachment to cellulose". EMBO J. 15 (21): 5739–51. doi:10.1002/j.1460-2075.1996.tb00960.x. PMC 452321. PMID 8918451.
- ^ Brun E, Johnson PE, Creagh AL, Tomme P, Webster P, Haynes CA, McIntosh LP (March 2000). "Structure and binding specificity of the second N-terminal cellulose-binding domain from Cellulomonas fimi endoglucanase C". Biochemistry. 39 (10): 2445–58. doi:10.1021/bi992079u. PMID 10704194.
- ^ Johnson PE, Joshi MD, Tomme P, Kilburn DG, McIntosh LP (November 1996). "Structure of the N-terminal cellulose-binding domain of Cellulomonas fimi CenC determined by nuclear magnetic resonance spectroscopy". Biochemistry. 35 (45): 14381–94. doi:10.1021/bi961612s. PMID 8916925.
- ^ a b Akagi, K. -I.; Watanabe, J.; Hara, M.; Kezuka, Y.; Chikaishi, E.; Yamaguchi, T.; Akutsu, H.; Nonaka, T.; Watanabe, T.; Ikegami, T. (2006). "Identification of the Substrate Interaction Region of the Chitin-Binding Domain of Streptomyces griseus Chitinase C". Journal of Biochemistry. 139 (3): 483–493. doi:10.1093/jb/mvj062. PMID 16567413.
- ^ Henshaw, J. L.; Bolam, D. N.; Pires, V. M.; Czjzek, M.; Henrissat, B.; Ferreira, L. M.; Fontes, C. M.; Gilbert, H. J. (2004). "The Family 6 Carbohydrate Binding Module CmCBM6-2 Contains Two Ligand-binding Sites with Distinct Specificities". Journal of Biological Chemistry. 279 (20): 21552–21559. doi:10.1074/jbc.M401620200. PMID 15004011.
- ^ Winterhalter, C.; Heinrich, P.; Candussio, A.; Wich, G.; Liebl, W. (1995). "Identification of a novel cellulose-binding domain within the multidomain 120 kDa xylanase XynA of the hyperthermophilic bacterium Thermotoga maritima". Molecular Microbiology. 15 (3): 431–444. doi:10.1111/j.1365-2958.1995.tb02257.x. PMID 7783614. S2CID 25985173.
- ^ Millward-Sadler SJ, Davidson K, Hazlewood GP, Black GW, Gilbert HJ, Clarke JH (November 1995). "Novel cellulose-binding domains, NodB homologues and conserved modular architecture in xylanases from the aerobic soil bacteria Pseudomonas fluorescens subsp. cellulosa and Cellvibrio mixtus". Biochem. J. 312 (1): 39–48. doi:10.1042/bj3120039. PMC 1136224. PMID 7492333.
- ^ Fanutti C, Ponyi T, Black GW, Hazlewood GP, Gilbert HJ (December 1995). "The conserved noncatalytic 40-residue sequence in cellulases and hemicellulases from anaerobic fungi functions as a protein docking domain". J. Biol. Chem. 270 (49): 29314–22. doi:10.1074/jbc.270.49.29314. PMID 7493964.
- ^ a b Raghothama S, Eberhardt RY, Simpson P, Wigelsworth D, White P, Hazlewood GP, Nagy T, Gilbert HJ, Williamson MP (September 2001). "Characterization of a cellulosome dockerin domain from the anaerobic fungus Piromyces equi". Nat. Struct. Biol. 8 (9): 775–8. doi:10.1038/nsb0901-775. PMID 11524680. S2CID 6442375.
- ^ a b Carvalho, A. L.; Goyal, A.; Prates, J. A.; Bolam, D. N.; Gilbert, H. J.; Pires, V. M.; Ferreira, L. M.; Planas, A.; Romão, M. J.; Fontes, C. M. (2004). "The Family 11 Carbohydrate-binding Module of Clostridium thermocellum Lic26A-Cel5E Accommodates -1,4- and -1,3-1,4-Mixed Linked Glucans at a Single Binding Site". Journal of Biological Chemistry. 279 (33): 34785–34793. doi:10.1074/jbc.M405867200. PMID 15192099.
- ^ a b Shen Z, Jacobs-Lorena M (July 1998). "A type I peritrophic matrix protein from the malaria vector Anopheles gambiae binds to chitin. Cloning, expression, and characterization". J. Biol. Chem. 273 (28): 17665–70. doi:10.1074/jbc.273.28.17665. PMID 9651363.
- ^ Elvin CM, Vuocolo T, Pearson RD, East IJ, Riding GA, Eisemann CH, Tellam RL (April 1996). "Characterization of a major peritrophic membrane protein, peritrophin-44, from the larvae of Lucilia cuprina. cDNA and deduced amino acid sequences". J. Biol. Chem. 271 (15): 8925–35. doi:10.1074/jbc.271.15.8925. PMID 8621536.
- ^ Casu R, Eisemann C, Pearson R, Riding G, East I, Donaldson A, Cadogan L, Tellam R (August 1997). "Antibody-mediated inhibition of the growth of larvae from an insect causing cutaneous myiasis in a mammalian host". Proc. Natl. Acad. Sci. U.S.A. 94 (17): 8939–44. Bibcode:1997PNAS...94.8939C. doi:10.1073/pnas.94.17.8939. PMC 22971. PMID 9256413.
- ^ Notenboom V, Boraston AB, Chiu P, Freelove AC, Kilburn DG, Rose DR (December 2001). "Recognition of cello-oligosaccharides by a family 17 carbohydrate-binding module: an X-ray crystallographic, thermodynamic and mutagenic study". J. Mol. Biol. 314 (4): 797–806. doi:10.1006/jmbi.2001.5153. PMID 11733998.
- ^ a b Jamal, S.; Nurizzo, D.; Boraston, A. B.; Davies, G. J. (2004). "X-ray Crystal Structure of a Non-crystalline Cellulose-specific Carbohydrate-binding Module: CBM28". Journal of Molecular Biology. 339 (2): 253–258. doi:10.1016/j.jmb.2004.03.069. PMID 15136030.
- ^ Wright HT, Sandrasegaram G, Wright CS (September 1991). "Evolution of a family of N-acetylglucosamine binding proteins containing the disulfide-rich domain of wheat germ agglutinin". J. Mol. Evol. 33 (3): 283–94. Bibcode:1991JMolE..33..283W. doi:10.1007/bf02100680. PMID 1757999. S2CID 8327744.
- ^ Butler AR, O'Donnell RW, Martin VJ, Gooday GW, Stark MJ (July 1991). "Kluyveromyces lactis toxin has an essential chitinase activity". Eur. J. Biochem. 199 (2): 483–8. doi:10.1111/j.1432-1033.1991.tb16147.x. PMID 2070799.
- ^ Lerner DR, Raikhel NV (June 1992). "The gene for stinging nettle lectin (Urtica dioica agglutinin) encodes both a lectin and a chitinase". J. Biol. Chem. 267 (16): 11085–91. doi:10.1016/S0021-9258(19)49878-5. PMID 1375935.
- ^ Kuranda, M. J.; Robbins, P. W. (1991). "Chitinase is required for cell separation during growth of Saccharomyces cerevisiae". The Journal of Biological Chemistry. 266 (29): 19758–19767. doi:10.1016/S0021-9258(18)55057-2. PMID 1918080.
- ^ Penninga, D.; Van Der Veen, B. A.; Knegtel, R. M.; Van Hijum, S. A.; Rozeboom, H. J.; Kalk, K. H.; Dijkstra, B. W.; Dijkhuizen, L. (1996). "The raw starch binding domain of cyclodextrin glycosyltransferase from Bacillus circulans strain 251". The Journal of Biological Chemistry. 271 (51): 32777–32784. doi:10.1074/jbc.271.51.32777. PMID 8955113.
- ^ Oyama, T.; Kusunoki, M.; Kishimoto, Y.; Takasaki, Y.; Nitta, Y. (1999). "Crystal structure of beta-amylase from Bacillus cereus var. Mycoides at 2.2 a resolution". Journal of Biochemistry. 125 (6): 1120–1130. doi:10.1093/oxfordjournals.jbchem.a022394. PMID 10348915.
- ^ Armstrong, C. G.; Doherty, M. J.; Cohen, P. T. (1998). "Identification of the separate domains in the hepatic glycogen-targeting subunit of protein phosphatase 1 that interact with phosphorylase a, glycogen and protein phosphatase 1". The Biochemical Journal. 336 (3): 699–704. doi:10.1042/bj3360699. PMC 1219922. PMID 9841883.
- ^ Boraston, A. B.; Healey, M.; Klassen, J.; Ficko-Blean, E.; Lammerts Van Bueren, A.; Law, V. (2005). "A Structural and Functional Analysis of -Glucan Recognition by Family 25 and 26 Carbohydrate-binding Modules Reveals a Conserved Mode of Starch Recognition". Journal of Biological Chemistry. 281 (1): 587–598. doi:10.1074/jbc.M509958200. PMID 16230347.
- ^ Boraston AB, Revett TJ, Boraston CM, Nurizzo D, Davies GJ (June 2003). "Structural and thermodynamic dissection of specific mannan recognition by a carbohydrate binding module, TmCBM27". Structure. 11 (6): 665–75. doi:10.1016/S0969-2126(03)00100-X. PMID 12791255.
- ^ a b Abbot, DW; Eirin-Lopez, JM; Boraston, AB (January 2008). "Insight into ligand diversity and novel biological roles for family 32 carbohydrate-binding modules". Molecular Biology and Evolution. 25 (1): 155–67. doi:10.1093/molbev/msm243. PMID 18032406.
- ^ a b 피코-블란, 엘리자베스; 보라스톤, 알리스데어 "카보하이드레이트 바인딩 모듈 패밀리 32" 웨이백 머신에 2016-08-20 보관,CAZypedia, 2017년 5월 4일.
- ^ Schnellmann, J.; Zeltins, A.; Blaak, H.; Schrempf, H. (1994). "The novel lectin-like protein CHB1 is encoded by a chitin-inducible Streptomyces olivaceoviridis gene and binds specifically to crystalline alpha-chitin of fungi and other organisms". Molecular Microbiology. 13 (5): 807–819. doi:10.1111/j.1365-2958.1994.tb00473.x. PMID 7815940. S2CID 22470447.
- ^ Vaaje-Kolstad, G.; Houston, D. R.; Riemen, A. H.; Eijsink, V. G.; Van Aalten, D. M. (2005). "Crystal Structure and Binding Properties of the Serratia marcescens Chitin-binding Protein CBP21". Journal of Biological Chemistry. 280 (12): 11313–11319. doi:10.1074/jbc.M407175200. PMID 15590674.
- ^ Katsuya, Y.; Mezaki, Y.; Kubota, M.; Matsuura, Y. (1998). "Three-dimensional structure of Pseudomonas isoamylase at 2.2 Å resolution1". Journal of Molecular Biology. 281 (5): 885–897. doi:10.1006/jmbi.1998.1992. PMID 9719642.
- ^ Wiatrowski, H. A.; Van Denderen, B. J.; Berkey, C. D.; Kemp, B. E.; Stapleton, D.; Carlson, M. (2004). "Mutations in the gal83 glycogen-binding domain activate the snf1/gal83 kinase pathway by a glycogen-independent mechanism". Molecular and Cellular Biology. 24 (1): 352–361. doi:10.1128/mcb.24.1.352-361.2004. PMC 303368. PMID 14673168.
- ^ Polekhina, G.; Gupta, A.; Michell, B. J.; Van Denderen, B.; Murthy, S.; Feil, S. C.; Jennings, I. G.; Campbell, D. J.; Witters, L. A.; Parker, M. W.; Kemp, B. E.; Stapleton, D. (2003). "AMPK beta subunit targets metabolic stress sensing to glycogen". Current Biology. 13 (10): 867–871. doi:10.1016/S0960-9822(03)00292-6. PMID 12747837. S2CID 16778615.
- ^ Hudson, E. R.; Pan, D. A.; James, J.; Lucocq, J. M.; Hawley, S. A.; Green, K. A.; Baba, O.; Terashima, T.; Hardie, D. G. (2003). "A novel domain in AMP-activated protein kinase causes glycogen storage bodies similar to those seen in hereditary cardiac arrhythmias". Current Biology. 13 (10): 861–866. doi:10.1016/S0960-9822(03)00249-5. PMID 12747836. S2CID 2295263.
- ^ Urbanowicz BR, Catala C, Irwin D, Wilson DB, Ripoll DR, Rose JK (April 2007). "A tomato endo-beta-1,4-glucanase, SlCel9C1, represents a distinct subclass with a new family of carbohydrate binding modules (CBM49)". J. Biol. Chem. 282 (16): 12066–74. doi:10.1074/jbc.M607925200. PMID 17322304.


