전사 RNA
Transfer RNA| tRNA | |
|---|---|
| 식별자 | |
| 기호. | t |
| Rfam | RF00005 |
| 기타 데이터 | |
| RNA형 | 유전자, tRNA |
| PDB 구조 | PDBe 3icq, 1asy, 1asz, 1il2, 2tra, 3tra, 486d, 1fir, 1yfg, 3eph, 3epj, 3epk, 3epl, 1efw, 1c0a, 2ake, 2azx, 2dr2, 1f7u, 3foz, 2z, 2z, 2z, 2z, 2e, 2zp, 2e, 2e, 02jp, 2e, 2e, 2e, 2e, 2egp, 2egp |
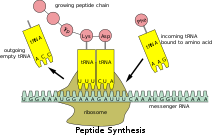
Transfer RNA(용해성[1] RNA의 경우 tRNA로 약칭되며, 이전에는 sRNA로 지칭됨)는 mRNA와 단백질의 아미노산 배열 사이의 물리적 연결 역할을 하는 (진핵생물에서)[2] 전형적으로 길이가 76에서 90 뉴클레오티드로 구성된 어댑터 분자이다.Transfer RNA(tRNA)는 아미노산을 리보솜이라고 불리는 세포의 단백질 합성 기구에 운반함으로써 이것을 한다.메신저 RNA(mRNA) 중 3-뉴클레오티드 코돈을 tRNA의 3-뉴클레오티드 안티코돈에 의해 상보함으로써 mRNA 코드에 기초한 단백질 합성이 이루어진다.이와 같이, tRNA는 유전자 코드에 따른 새로운 단백질의 생물학적 합성인 번역에 필요한 구성요소이다.
개요
mRNA의 특정 뉴클레오티드 배열은 mRNA가 전사되는 유전자의 단백질 생성물에 어떤 아미노산이 포함되는지 규정하는 반면, tRNA의 역할은 유전자 코드로부터의 어떤 배열이 어떤 아미노산에 [3]대응하는지 규정하는 것이다.mRNA는 단백질을 연속된 일련의 코돈으로 부호화하며, 각각의 코돈은 특정 tRNA에 의해 인식된다.tRNA의 한쪽 끝은 안티코돈이라고 불리는 세 개의 핵으로 이루어진 염기서열의 유전자 코드와 일치합니다.안티코돈은 단백질 생합성 중에 mRNA에서 코돈과 세 개의 상보적인 염기쌍을 형성한다.
tRNA의 다른 한쪽 끝에는 항코돈 배열에 대응하는 아미노산에 대한 공유 결합이 있습니다.각각의 tRNA 분자는 오직 하나의 아미노산에만 결합될 수 있기 때문에 각각의 유기체는 많은 종류의 tRNA를 가지고 있다.유전자 코드는 동일한 아미노산을 특정하는 여러 코돈을 포함하고 있기 때문에, 동일한 아미노산을 가지고 있는 다른 항코돈들을 가진 몇 개의 tRNA 분자가 있다.
tRNA 3' 말단에 대한 공유 결합은 아미노아실 tRNA 합성효소라고 불리는 효소에 의해 촉매된다.단백질 합성 중에 아미노산이 부착된 tRNA는 신장인자라고 불리는 단백질에 의해 리보솜에 전달되며, 이는 리보솜과 tRNA의 연관성, 새로운 폴리펩타이드 합성 및 mRNA를 따른 리보솜의 전위(이동)를 돕는다. 리보솜은 성장 중인 폴리펩타이드 사슬을 3' 말단에서 새로 전달된 tRNA의 3' 말단에 부착된 아미노산으로 옮깁니다. 이 반응은 리보솜에 의해 촉매된다.tRNA 분자 내의 다수의 개별 뉴클레오티드는 종종 메틸화 또는 탈아미드에 의해 화학적으로 수정될 수 있다.이러한 특이한 염기는 때때로 리보솜과 tRNA의 상호작용에 영향을 미치기도 하고 염기쌍화 [4]특성을 바꾸기 위해 안티코돈에서 발생하기도 합니다.
구조.
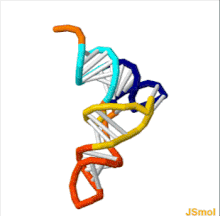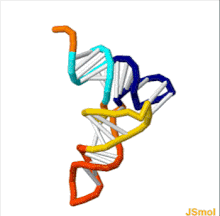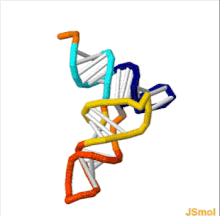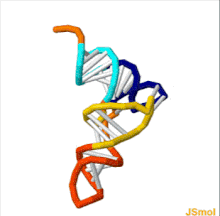
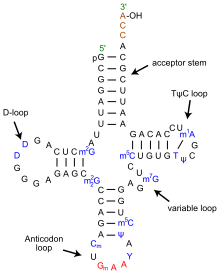
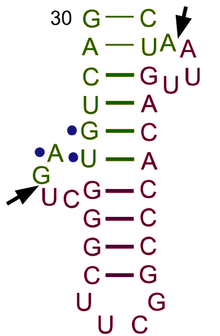
tRNA의 구조는 1차 구조, 2차 구조(일반적으로 클로버 리프 구조) 및 3차[6] 구조(모든 tRNA는 리보솜의 P와 A 부위에 들어갈 수 있는 유사한 L자형 3D 구조를 가진다)로 분해될 수 있다.클로버 리프 구조는 일반적인 RNA 3차 구조 모티브인 나선형의 동축 적재를 통해 3D L자형 구조가 된다.tRNA 분자의 루프 '지름'뿐만 아니라 각 팔의 길이는 [6][7]종마다 다릅니다.tRNA 구조는 다음과 같이 구성됩니다.
- 5' 말단 인산기.
- 수용체 스템은 5' 말단 뉴클레오티드와 3' 말단 뉴클레오티드(아미노산 부착에 사용되는 CCA 3' 말단 그룹을 포함)의 염기쌍에 의해 만들어진 7~9 염기쌍(bp) 스템이다.일반적으로 이러한 3γ 말단 tRNA 유사 구조를 '게놈 태그'라고 한다.수용체 스템에는 Watson-Crick이 아닌 [6][8]염기쌍이 포함될 수 있습니다.
- CCA 꼬리는 tRNA 분자의 3' 말단에 있는 시토신-시토신-아데닌 배열이다.아미노아실 tRNA 합성효소에 의해 tRNA에 적재된 아미노산은 CCA [9]꼬리의 3'-히드록실기에 공유 결합된다.이 배열은 효소에 의한 tRNA의 인식에 중요하며 번역에 중요하다.[10][11]원핵생물에서 CCA 배열은 일부 tRNA 배열에서 전사된다.대부분의 원핵생물 tRNA 및 진핵생물 tRNA에서 CCA 배열은 처리 중에 첨가되므로 tRNA [12]유전자에는 나타나지 않는다.
- D 암은 디히드로우리딘을 포함하는 루프로 끝나는 4-bp ~ 6-bp 줄기입니다.[6]
- 안티코돈 [6]암은 루프에 안티코돈이 포함된 5bp 스템입니다.tRNA 5'에서 3'까지의 프라이머리 구조에는 안티코돈이 포함되어 있습니다만, 5'에서3'까지의 mRNA 판독에는 3'에서 5'까지의 방향성이 필요하기 때문에 역순입니다.
- T 암은 4-bp ~ 5-bp 스템으로, 여기서 δ는 변형된 우리딘인 [6]의사우리딘인 배열 TδC를 포함하는 루프로 끝납니다.
- 특히 메틸화(예: tRNA(과닌-N7--메틸전달효소)에 의해 변형된 염기는 tRNA의 여러 위치에서 발생한다.첫 번째 안티코돈 염기 또는 워블 위치는 때때로 이노신(아데닌에서 유래), 퀘오신(구아닌에서 유래), 우리딘-5-옥시아세트산(우라실로부터 유래), 5-메틸아미노메틸-2-티우리딘(우라실로부터 유래), 또는 리시딘(시토신에서 [13]유래)으로 수정된다.
안티코돈
안티코돈은[14] mRNA 코돈의 3개 염기에 대응하는 3개의 뉴클레오티드의 단위이다.각 tRNA는 아미노산용 하나 이상의 코돈에 대해 3개의 상보적 염기쌍을 형성할 수 있는 독특한 안티코돈 트리플렛 배열을 가진다.일부 안티코돈은 워블 베이스 페어링으로 인해 둘 이상의 코돈과 페어링됩니다.항코돈의 첫 번째 뉴클레오티드는 mRNA에는 없는 이노신이다.이노신은 대응하는 코돈 [4]: 29.3.9 위치에 있는 하나 이상의 염기에 수소가 결합할 수 있다.유전자 코드에서는 단일 아미노산이 4가지 제3위치의 가능성 모두 또는 적어도 피리미딘과 푸린 양쪽에 의해 특정되는 것이 일반적이다.예를 들어 아미노산 글리신은 코돈 배열 GGU, GGC, GGA 및 GGG에 의해 코드화되어 있다.다른 변형된 뉴클레오티드는 미토콘드리아와 [15]같이 유전자 코드에 미묘한 변화를 초래하는 첫 번째 안티코돈 위치(때로는 "흔들림 위치"로 알려져 있음)에서도 나타날 수 있다.tRNA 분자와 아미노산을 특정하는 코돈 사이에 일대일 대응성을 제공하기 위해서는 세포당 61개의 tRNA 타입이 필요하며, 표준 유전자 코드에는 61개의 센스 코돈이 존재하기 때문이다.그러나, 많은 세포들은 61종류의 tRNA를 가지고 있는데, 그 이유는 워블 염기가 반드시 모든 것은 아니지만 특정 아미노산을 지정하는 코돈의 여러 코돈에 결합할 수 있기 때문이다.61개의 모든 감지 [3][16]코돈을 명확하게 번역하려면 적어도 31개의 tRNA가 필요합니다.
아미노아실화
아미노아실화는 아미노아실기를 화합물에 첨가하는 과정이다.아미노산을 tRNA 분자의 CCA 3' 말단에 공유 결합한다.각 tRNA는 아미노아실 tRNA 합성효소에 의해 특정 아미노산과 아미노아실화(또는 대전)된다.아미노산에는 하나 이상의 tRNA와 하나 이상의 항코돈이 있을 수 있음에도 불구하고 일반적으로 각 아미노산에는 하나의 아미노아실 tRNA 합성 효소가 있습니다.합성효소에 의한 적절한 tRNA의 인식은 안티코돈만으로 매개되는 것이 아니며, 수용체 스템이 중요한 [17]역할을 하는 경우가 많다.반응:
특정 유기체는 하나 이상의 아미노인산-tRNA 합성 효소가 결핍될 수 있다.이것은 화학적으로 관련된 아미노산에 의해 tRNA를 충전하게 하고, 효소 또는 효소에 의해 tRNA가 올바르게 충전되도록 변형시킨다.예를 들어 헬리코박터균은 글루타미닐 tRNA 합성효소가 결핍되어 있다.따라서 글루타메이트 tRNA 합성효소는 tRNA-글루타민(tRNA-Gln)에 글루타메이트를 대전시킨다.이어서 아미도전달효소는 글루타메이트의 산측쇄를 아미드로 변환하여 올바르게 대전된 gln-tRNA-Gln을 형성한다.
아미노아실화에 대한 간섭은 몇몇 질병을 치료하기 위한 접근법으로 유용할 수 있다: 암세포는 건강한 세포에 비해 교란된 아미노아실화에 상대적으로 취약할 수 있다.암과 바이러스 생물학과 관련된 단백질 합성은 종종 특정 tRNA 분자에 매우 의존한다.예를 들어, 리신에 의해 충전되는 간암의 경우 tRNA-Lys-CUU는 간암세포의 성장과 전이를 지속하는 반면, 건강한 세포는 세포 [18]생리를 지원하기 위해 이 tRNA에 대한 의존도가 훨씬 낮다.마찬가지로, E형 간염 바이러스는 비감염 [19]세포와 관련된 것과 상당히 다른 tRNA 환경을 필요로 한다.따라서 특정 tRNA종의 아미노아실화 억제는 과잉질환의 합리적인 치료를 위한 유망한 새로운 경로로 간주된다.
리보솜과의 결합
리보솜은 두 개의 리보솜 서브유닛 사이의 공간을 가로지르는 tRNA 분자에 대한 3개의 결합 부위, 즉 A(아미노아실),[21] P(펩티딜), E(출구) 부위를 가지고 있다.또한 리보솜은 mRNA 복호화 중 또는 단백질 합성의 개시 중에 사용되는 tRNA 결합을 위한 두 개의 다른 부위를 가진다.T부위(신장인자 Tu)와 I부위([22][23]개시)이다.관례상 tRNA 결합부위는 처음에 기재된 작은 리보솜 서브유닛 상의 부위와 다음에 기재된 큰 리보솜 서브유닛 상의 부위로 표시된다.예를 들어 A 사이트는 A/A, P 사이트, P 사이트, P/P 및 E 사이트 E/[22]E로 작성되는 경우가 많습니다.A- 및 P- 부위의 L27, L2, L14, L15, L16과 같은 결합 단백질은 A. P. Czernilofsky 등(Proc)의 친화성 라벨링에 의해 결정되었다. Natl. Acad. Sci, USA, 페이지 230-234, 1974).
번역 개시가 완료되면 첫 번째 아미노아실 tRNA는 P/P 부위에 위치하여 아래에 설명된 신장 사이클에 대비합니다.번역 신장 중에 tRNA는 먼저 신장인자 Tu(EF-Tu) 또는 그 진핵생물(eEF-1) 또는 고고학적 대응물과 복합체의 일부로서 리보솜에 결합한다.이 초기 tRNA 결합 부위를 A/T 부위라고 합니다.A/T 부위에서 A 부위의 절반은 mRNA 복호화 부위가 위치한 작은 리보솜 서브유닛에 존재한다.mRNA 디코딩 사이트는 변환 중에 mRNA 코돈이 읽히는 곳입니다.T-사이트의 절반은 EF-Tu 또는 eEF-1이 리보솜과 상호작용하는 큰 리보솜 서브유닛에 주로 존재한다.mRNA 복호화가 완료되면 아미노아실-tRNA는 A/A 부위에 결합되어 다음 펩타이드[24] 결합이 부착된 아미노산에 형성될 준비가 된다.성장 중인 폴리펩타이드를 A/A 부위의 아미노아실-tRNA에 전달하는 펩티딜-tRNA는 P/P 부위에 결합되어 있다.펩타이드 결합이 형성되면 P/P 부위의 tRNA는 아실화되거나 유리 3' 말단을 가지며, A/A 부위의 tRNA는 성장 중인 폴리펩타이드 사슬을 분리한다.다음 신장 사이클을 허용하기 위해 tRNA는 사이클을 완료하기 전에 하이브리드 A/P 및 P/E 결합 부위를 통해 이동하며 P/P 및 E/E 부위에 상주합니다.A/A 및 P/P tRNA가 P/P 및 E/E 사이트로 이동하면 mRNA도 1개의 코돈만큼 이동하며 A/T 사이트는 다음 mRNA 디코딩 라운드를 위해 비워집니다.그런 다음 E/E 사이트에서 결합된 tRNA는 리보솜을 떠납니다.
P/I 부위는 [23]사실 박테리아에서 IF2라고 불리는 개시 인자에 의해 전달되는 아미노아실 tRNA와 결합하는 첫 번째 부위이다.그러나 진핵생물 또는 고고생물 리보솜에서 P/I 부위의 존재는 아직 확인되지 않았다.P-사이트 단백질 L27은 E에 의한 친화성 표기에 의해 결정되었다.콜라츠와 A. P. 체르닐롭스키 (FEBS Let., Vol. 63, 페이지 283–286, 1976).
tRNA유전자
유기체는 게놈에 있는 tRNA 유전자의 수가 다양하다.예를 들어, 유전학 연구에서 흔히 사용되는 모델 유기체인 선충 C. elegans는 핵 게놈에 29,647개의 유전자를 가지고 있으며, 그 중 620개는 tRNA를 [26][27]위해 코드된다.발아하는 효모 사카로미세스 세레비시아에는 게놈에 275개의 tRNA 유전자가 있다.
2013년 1월 추정에 따르면, 총 20,848개의 단백질을 코드하는 유전자를 가진 인간 게놈에는 세포질 tRNA 분자를 코드하는 497개의 핵 유전자와 324개의 tRNA 유래 의사 유전자가 있다.RNA 유전자는 더 이상[29] 기능하지 않는 것으로 생각됩니다(비록 의사 tRNA는 [30]박테리아에서 항생제 내성에 관여하는 것으로 나타났습니다).모든 진핵생물들과 마찬가지로, 인간에게는 22개의 미토콘드리아 tRNA[31][full citation needed] 유전자가 있다.이들 유전자 중 일부의 돌연변이는 MELAS 증후군과 같은 심각한 질병과 연관되어 있다.미토콘드리아 tRNA 유전자와 순서상 매우 유사한 핵염색체 영역도 확인되었다(tRNA-looksike).[32]이러한 tRNA 생김새는 또한 핵 미토콘드리아 DNA(미토콘드리아에서 [32][33]핵으로 전달되는 유전자)의 일부로 간주된다.미토콘드리아 tRNA(tRNA-lookalike)의 다중 핵복사 현상은 인간에서 주머니쥐에[34] 이르는 많은 고등 유기체에서 관찰되었으며, 이는 외모가 기능할 가능성을 시사한다.
세포질 tRNA 유전자는 항코돈 특성에 따라 49족으로 분류할 수 있다.이 유전자들은 22번과 Y번 염색체를 제외한 모든 염색체에서 발견된다.1개의 [29]염색체에서도 6p에서 높은 클러스터링이 관찰된다(140 tRNA 유전자).
HGNC는 Genomic tRNA Database(GtRNAdb) 및 이 분야 전문가와 협력하여 tRNA를 코드하는 인간 유전자의 고유 이름을 승인했다.
진화
tRNA(5' 말단 인산기 및 3' 말단 CCA기로 구성된 T 암 및 수용체 스템)의 위쪽 절반과 아래쪽 절반(D 암 및 안티코돈 암)은 구조 및 기능 면에서 독립된 단위이다.상위 절반은 원래 초기 RNA 세계에서 복제를 위해 tRNA 유사 분자를 표시했을 수 있는 3' 말단 게놈 태그를 포함하여 먼저 진화했을 수 있습니다.아래쪽 절반은 나중에 RNA 세계에서 단백질 합성이 시작되어 리보핵단백질 세계(RNP 세계)로 변하면서 확장으로 진화했을 수 있다.이 제안된 시나리오를 게놈 태그 가설이라고 합니다.실제로 tRNA 및 tRNA 유사 골재는 오늘날에도 복제에 중요한 촉매적 영향(즉, 리보자임)을 가지고 있다.이러한 역할은 RNA 세계의 '[35]분자 화석'으로 여겨질 수 있다.
게놈 tRNA 함량은 생물 영역 간 게놈의 구별되는 특징입니다.고세균은 유전자 복제 수가 균일한 게놈 tRNA 함량 측면에서 가장 단순한 상황을 나타내며 박테리아는 중간 상황을 나타내며 진핵균은 가장 복잡한 [36]상황을 나타낸다.진핵아는 다른 두 왕국보다 tRNA 유전자 함량이 많을 뿐만 아니라 서로 다른 아이소 수용체들 사이에서 유전자 복제 수에서 큰 차이를 보이고 있으며, 이러한 복잡성은 tRNA 유전자의 복제와 안티코돈 특이성의[citation needed] 변화에 기인하는 것으로 보인다.
서로 다른 종에 걸친 tRNA 유전자 복사수의 진화는 특정 tRNA 수정 효소(세균의 우리딘 메틸전달효소, 진핵의 아데노신 탈아미나아제)의 출현과 관련이 있으며, 이는 주어진 tRNA의 [36]해독 능력을 증가시킨다.예를 들어 tRNAALA는 4개의 다른 tRNA isoacceptor(AGC, UGC, GGC 및 CGC)를 부호화합니다.진핵아에서 AGC 아이소 수용체는 나머지 아이소 수용체에 비해 유전자 복사수가 매우 풍부하며, 이는 흔들림 염기의 A-to-I 변형과 상관관계가 있다.이 같은 추세는 대부분의 진핵종 아미노산에서 나타났다.실제로, 이러한 두 가지 tRNA 수정의 효과는 코돈 사용 편견에서도 나타난다.고도로 발현된 유전자는 이러한 변형된 tRNA에 의해 해독되는 코돈을 배타적으로 사용하는 코돈에서 농축된 것으로 보이며, 이는 번역 [36]효율에서 이러한 코돈의 가능한 역할과 결과적으로 이러한 tRNA 수정의 역할을 시사한다.
많은 종들이 진화 과정에서 특정한 tRNA를 잃었다는 것을 주목하는 것이 중요하다.예를 들어, 포유류와 조류 모두 64개의 tRNA 유전자 중 14개가 부족하지만, 다른 생명체들은 이러한 tRNA를 포함하고 있다.[37]정확하게 짝을 이루는 tRNA가 없는 코돈을 번역하기 위해 유기체는 워블링이라고 불리는 전략에 의존하며, 불완전하게 일치하는 tRNA/mRNA 쌍은 여전히 번역을 발생시키지만 이 전략은 번역 오류 [38]성향을 증가시킨다.진화 과정에서 tRNA 유전자가 손실된 이유는 여전히 논의 중에 있지만 바이러스 [39]감염에 대한 저항성의 향상과 관련이 있을 수 있다.뉴클레오티드 세쌍둥이는 아미노산 및 관련 tRNA보다 더 많은 조합을 나타낼 수 있기 때문에, 유전자 코드에는 중복성이 있으며, 여러 개의 다른 3-뉴클레오티드 코돈은 동일한 아미노산을 발현할 수 있다.코돈의 편견이 코돈의 최적화를 필요로 하는 것입니다.또, 타겟 유기체의 tRNA 레퍼토리의 코드 시퀀스를 보면, 발현 [40]시스템에 근거해 코돈을 교환할 수 있습니다.
tRNA유래 단편
tRNA 유래 단편(또는 tRF)은 성숙한 tRNA 또는 전구체 [41][42][43][44]전사체의 분열 후에 나타나는 짧은 분자이다.세포질 tRNA와 미토콘드리아 tRNA는 모두 [45]파편을 만들 수 있다.성숙한 tRNA에서 유래한 것으로 생각되는 tRF에는 비교적 긴 tRNA 반쪽과 짧은 5'-tRF,[41][45][46] 3'-tRF 및 i-tRF를 포함하여 적어도 4가지 구조 유형이 있다.전구체 tRNA는 5' 리더 또는 3' 트레일 시퀀스에서 분자를 생성하기 위해 분해될 수 있다.분해 효소는 앤지게닌, 다이서, RNase Z 및 RNase [41][42]P를 포함한다.특히 Angiogenin의 경우 tRF는 3' 말단에 특징적인 특이한 고리형 인산기를 가지며, 5' [47]말단에 수산기를 가지며, tRF는 RNA 간섭, 특히 복제용 프라이머로 TRNA를 사용하는 레트로바이러스 및 레트로트랜스포존의 억제에 관여하는 것으로 보인다.앤지오제닌에 의해 분해된 절반의 tRNA는 tiRNA로도 알려져 있다.piRNA로 기능하는 파편을 포함한 작은 파편들의 생물 형성은 [48]덜 알려져 있다.
tRF는 성별, 인종 및 질병 [45][49][50]상태 간에 유의한 변화를 보이는 것과 같이 여러 개의 종속성과 역할을 가지고 있다.기능적으로, 그것들은 Ago에 적재되어 RNAi 경로를 [43][46][51]통해 작용하고, 스트레스 과립 [52]형성에 참여하며, RNA 결합[53] 단백질에서 mRNA를 대체하거나 [54]번역을 억제할 수 있다.시스템 또는 생물 수준에서 4가지 유형의 tRF는 다양한 활동 스펙트럼을 가진다.기능적으로, tRF는 바이러스 감염,[55] 암,[46] 세포 증식과 관련이 있으며 후생성 대사의 [56]전세대 조절과도 관련이 있다.
tRF는 사람에게 제한되지 않으며 여러 [46][57][58][59]유기체에 존재하는 것으로 나타났다.
tRF에 대해 더 배우고 싶은 사람들을 위해 두 가지 온라인 도구를 사용할 수 있다. 즉, 미토콘드리아 및 핵 tRNA 단편(MINTbase)[60][61]의 상호작용 탐사를 위한 프레임워크와 Transfer RNA related fragments(tRFdb)[62]의 관계형 데이터베이스이다.MINTbase는 또한 게놈에 의존하지 않는 tRF-license plate(또는 MINTcodes)라고 불리는 tRF의 명명 체계를 제공합니다. 이 체계는 RNA 시퀀스를 더 짧은 문자열로 압축합니다.
엔지니어링된 tRNA
인공억제기 신장기 tRNA는 유전자 코드 배열에 배치된 난센스 코돈에 부자연 아미노산을 함유시키기 위해 사용된다.공학적 개시제 tRNA(metY 유전자에 의해 코드된 CUA 안티코돈을 가진 tRNA)는 호박정지 코돈 UAG에서 번역을 개시하기 위해 사용되어 왔다.이런 유형의 엔지니어링된 tRNA는 보통 UAG 코돈에서 발생하는 변환 정지 신호를 억제하기 때문에 넌센스 서프레서 tRNA라고 불립니다.호박 개시제 tRNA는 강력한 Shine-Dalgarno 배열에 앞서 UAG 코돈에 메티오닌과[63] 글루타민을[64] 삽입합니다.황색 개시제 tRNA의 조사 결과, 유전적으로 기록된 대장균 [63]균주에서 검출 가능한 표적 외 번역 개시 이벤트를 나타내지 않는 일반 AUG 시작 코돈과 직교하는 것으로 나타났다.
tRNA생성
진핵세포에서 tRNA는 RNA 중합효소 III에 의해 [65]핵에서 pre-tRNA로 전사된다.RNA 중합효소 III는 tRNA [2][66][67]유전자 내에서 보존성이 높은 2개의 하류 프로모터 배열, 즉 5µ-ICR, D-컨트롤 영역 또는 A박스 및 3µ-ICR(T-컨트롤 영역 또는 B박스)을 인식한다.첫 번째 프로모터는 성숙한 tRNA의 +8에서 시작되며, 두 번째 프로모터는 첫 번째 프로모터의 다운스트림에 30~60개의 뉴클레오티드에 위치한다.4개 이상의 티미딘이 [2][67]늘어나면 전사가 끝납니다.
전 tRNA는 핵 내부에서 광범위한 수정을 거친다.일부 사전 tRNA는 기능성 tRNA [68]분자를 형성하기 위해 결합되거나 절단되는 인트론을 포함하고 있다; 박테리아에서는 이러한 자가 결합하는 반면, 진핵생물 및 고세균에서는 tRNA 분할 엔도핵산 [69]효소에 의해 제거된다.진핵생물 프리 tRNA는 엔도핵산가수분해효소에 [70]의한 tRNA 인트론의 인식 및 정밀 스플라이싱에 중요한 벌지-나선 벌지(BHB) 구조 모티브를 포함한다.이 모티브의 위치와 구조는 진화적으로 보존됩니다.단, 단세포 조류와 같은 일부 생물은 BHB-모티프뿐만 아니라 스플라이스 인트론 [70]배열의 5µ-ends 및 3µ-ends의 비카논학적 위치를 가진다.5' 배열은 RNase [71]P에 의해 제거되며, 3' 말단은 tRNase Z [72]효소에 의해 제거된다.주목할 만한 예외는 시조 나노아르카이움 균등화합물이며, 시조나노아르카이움 균등화합물은 RNase P 효소를 포함하지 않고 성숙한 tRNA의 5'[73] 말단에서 전사가 시작되도록 배치된 프로모터를 가지고 있다.비템플레이트 3' CCA 꼬리는 뉴클레오티딜 전달 [74]효소에 의해 첨가된다.Los1/Xpo-t에 [75][76]의해 tRNA가 세포질 내로 수출되기 전에 tRNA는 아미노아실화된다.[77]처리 이벤트의 순서가 보존되지 않습니다.예를 들어 효모는 핵이 아닌 미토콘드리아막의 [78]세포질 측에서 스플라이싱을 실시한다.
그럼에도 불구하고, 2021년 3월, 연구자들은 전달 RNA의 예비 형태가 생명체의 초기 발달, 즉 생물의 [79][80]생육에서 복제 분자였을 수 있다는 증거를 보고했다.
역사
tRNA의 존재는 Francis Crick에 의해 "어댑터 가설"로 처음 가설되었는데, 이는 RNA 알파벳을 단백질 알파벳으로 변환하는 것을 매개하는 어댑터 분자가 존재해야 한다는 가정에 바탕을 두고 있다.Paul C Zamecnik과 Mahlon Hoagland는 구조에 대한 중요한 연구가 1960년대 초에 Alex Rich와 Donald Caspar, 두 명의 보스턴 연구원, 프린스턴 대학의 Jacques Presco 그룹과 King's College [82]London에 있는 영국 그룹에 의해 이루어졌다는 것을 발견했습니다.1965년, 로버트 W. 코넬 대학의 Holley는 1차 구조를 보고하고 3개의 2차 [83]구조를 제안했다. tRNA는 로버트 M에 의해 위스콘신주 매디슨에서 처음 결정화 되었다.Bock.[84] 클로버[85] 잎 구조는 그 후 몇 년 동안 여러 다른 연구들에 의해 확인되었고 마침내 1974년 X-ray 결정학 연구를 통해 확인되었다.알렉산더 리치 밑에서 일하는 김성후와 애런 클럭이 이끄는 영국 그룹은 1년 [86][87]만에 같은 결정학 연구 결과를 발표했다.
「 」를 참조해 주세요.
레퍼런스
- ^ Plescia OJ, Palczuk NC, Cora-Figueroa E, Mukherjee A, Braun W (October 1965). "Production of antibodies to soluble RNA (sRNA)". Proceedings of the National Academy of Sciences of the United States of America. 54 (4): 1281–1285. Bibcode:1965PNAS...54.1281P. doi:10.1073/pnas.54.4.1281. PMC 219862. PMID 5219832.
- ^ a b c Sharp SJ, Schaack J, Cooley L, Burke DJ, Söll D (1985). "Structure and transcription of eukaryotic tRNA genes". CRC Critical Reviews in Biochemistry. 19 (2): 107–144. doi:10.3109/10409238509082541. PMID 3905254.
- ^ a b Crick FH (December 1968). "The origin of the genetic code". Journal of Molecular Biology. 38 (3): 367–379. doi:10.1016/0022-2836(68)90392-6. PMID 4887876.
- ^ a b Stryer L, Berg JM, Tymoczko JL (2002). Biochemistry (5th ed.). San Francisco: W. H. Freeman. ISBN 978-0-7167-4955-4.
- ^ "Transfer RNA (tRNA)". Proteopedia.org. Retrieved 7 November 2018.
- ^ a b c d e f Itoh Y, Sekine S, Suetsugu S, Yokoyama S (July 2013). "Tertiary structure of bacterial serenocysteine tRNA". Nucleic Acids Research. 41 (13): 6729–6738. doi:10.1093/nar/gkt321. PMC 3711452. PMID 23649835.
- ^ Goodenbour JM, Pan T (29 October 2006). "Diversity of tRNA genes in eukaryotes". Nucleic Acids Research. 34 (21): 6137–6146. doi:10.1093/nar/gkl725. PMC 1693877. PMID 17088292.
- ^ Jahn M, Rogers MJ, Söll D (July 1991). "Anticodon and acceptor stem nucleotides in tRNA(Gln) are major recognition elements for E. coli glutaminyl-tRNA synthetase". Nature. 352 (6332): 258–260. Bibcode:1991Natur.352..258J. doi:10.1038/352258a0. PMID 1857423. S2CID 4263705.
- ^ Ibba M, Soll D (June 2000). "Aminoacyl-tRNA synthesis". Annual Review of Biochemistry. 69 (1): 617–650. doi:10.1146/annurev.biochem.69.1.617. PMID 10966471.
- ^ Sprinzl M, Cramer F (1979). "The -C-C-A end of tRNA and its role in protein biosynthesis". Progress in Nucleic Acid Research and Molecular Biology. 22: 1–69. doi:10.1016/s0079-6603(08)60798-9. ISBN 978-0-12-540022-0. PMID 392600.
- ^ Green R, Noller HF (1997). "Ribosomes and translation". Annual Review of Biochemistry. 66: 679–716. doi:10.1146/annurev.biochem.66.1.679. PMID 9242921.
- ^ Aebi M, Kirchner G, Chen JY, Vijayraghavan U, Jacobson A, Martin NC, Abelson J, et al. (September 1990). "Isolation of a temperature-sensitive mutant with an altered tRNA nucleotidyltransferase and cloning of the gene encoding tRNA nucleotidyltransferase in the yeast Saccharomyces cerevisiae". The Journal of Biological Chemistry. 265 (27): 16216–16220. doi:10.1016/S0021-9258(17)46210-7. PMID 2204621.
- ^ McCloskey JA, Nishimura S (November 1977). "Modified nucleosides in transfer RNA". Accounts of Chemical Research. 10 (11): 403–410. doi:10.1021/ar50119a004.
- ^ Felsenfeld G, Cantoni GL (May 1964). "Use of thermal denaturation studies to investigate the base sequence of yeast serine sRNA". Proceedings of the National Academy of Sciences of the United States of America. 51 (5): 818–826. Bibcode:1964PNAS...51..818F. doi:10.1073/pnas.51.5.818. PMC 300168. PMID 14172997.
- ^ Suzuki T, Suzuki T (June 2014). "A complete landscape of post-transcriptional modifications in mammalian mitochondrial tRNAs". Nucleic Acids Research. 42 (11): 7346–7357. doi:10.1093/nar/gku390. PMC 4066797. PMID 24831542.
- ^ Lodish H, Berk A, Matsdaira P, 카이저 CA, Krieger M, Scott MP, Zipursky SL, Darnell J. (2004)세포의 분자생물학.WH Freeman :뉴욕.제5판[ISBN missing][page needed]
- ^ Schimmel P, Giegé R, Moras D, Yokoyama S (October 1993). "An operational RNA code for amino acids and possible relationship to genetic code". Proceedings of the National Academy of Sciences of the United States of America. 90 (19): 8763–8768. Bibcode:1993PNAS...90.8763S. doi:10.1073/pnas.90.19.8763. PMC 47440. PMID 7692438.
- ^ Zhang R, Noordam L, Ou X, Ma B, Li Y, Das P, Shi S, Liu J, Wang L, Li P, Verstegen MM, Reddy DS, van der Laan LJ, Peppelenbosch MP, Kwekkeboom J, Smits R, Pan Q (January 2021). "The biological process of lysine-tRNA charging is therapeutically targetable in liver cancer". Liver Int. 41 (1): 206–219. doi:10.1111/liv.14692. PMC 7820958. PMID 33084231.
- ^ Ou X, Ma B, Zhang R, Miao Z, Cheng A, Peppelenbosch MP, Pan Q (June 2020). "A simplified qPCR method revealing tRNAome remodeling upon infection by genotype 3 hepatitis E virus". FEBS Letters. 594 (12): 2005–2015. doi:10.1002/1873-3468.13764. PMID 32133647.
- ^ Dunkle JA, Wang L, Feldman MB, Pulk A, Chen VB, Kapral GJ, Noeske J, Richardson JS, Blanchard SC, Cate JH (May 2011). "Structures of the bacterial ribosome in classical and hybrid states of tRNA binding". Science. 332 (6032): 981–984. Bibcode:2011Sci...332..981D. doi:10.1126/science.1202692. PMC 3176341. PMID 21596992.
- ^ Konevega AL, Soboleva NG, Makhno VI, Semenkov YP, Wintermeyer W, Rodnina MV, Katunin VI (January 2004). "Purine bases at position 37 of tRNA stabilize codon-anticodon interaction in the ribosomal A site by stacking and Mg2+-dependent interactions". RNA. 10 (1): 90–101. doi:10.1261/rna.5142404. PMC 1370521. PMID 14681588.
- ^ a b Agirrezabala X, Frank J (August 2009). "Elongation in translation as a dynamic interaction among the ribosome, tRNA, and elongation factors EF-G and EF-Tu". Quarterly Reviews of Biophysics. 42 (3): 159–200. doi:10.1017/S0033583509990060. PMC 2832932. PMID 20025795.
- ^ a b Allen GS, Zavialov A, Gursky R, Ehrenberg M, Frank J (June 2005). "The cryo-EM structure of a translation initiation complex from Escherichia coli". Cell. 121 (5): 703–712. doi:10.1016/j.cell.2005.03.023. PMID 15935757. S2CID 16146867.
- ^ Tirumalai MR, Rivas M, Tran Q, Fox GE (November 2021). "The Peptidyl Transferase Center: a Window to the Past". Microbiol Mol Biol Rev. 85 (4): e0010421. doi:10.1128/MMBR.00104-21. PMC 8579967. PMID 34756086.
- ^ WormBase 웹사이트, http://www.wormbase.org Archived 2017-04-20 at the Wayback Machine, 릴리즈 WS187, 날짜 2008년 1월 25일
- ^ Spieth J, Lawson D (January 2006). "Overview of gene structure". WormBook: 1–10. doi:10.1895/wormbook.1.65.1. PMC 4781370. PMID 18023127.
- ^ Hartwell LH, Hood L, Goldberg ML, 레이놀즈 AE, Silver LM, Veres RC. (2004)유전학: 유전자부터 게놈까지 제2판.맥그로힐: 뉴욕. 페이지 264
- ^ Ensembl 릴리즈 70 - 2013년 1월 http://www.ensembl.org/Homo_sapiens/Info/StatsTable?db=core 2013-12-15 Wayback Machine 아카이브 완료
- ^ a b Lander ES, Linton LM, Birren B, Nusbaum C, Zody MC, Baldwin J, et al. (International Human Genome Sequencing Consortium) (February 2001). "Initial sequencing and analysis of the human genome" (PDF). Nature. 409 (6822): 860–921. Bibcode:2001Natur.409..860L. doi:10.1038/35057062. PMID 11237011.
- ^ Rogers TE, Ataide SF, Dare K, Katz A, Seveau S, Roy H, Ibba M (2012). "A pseudo-tRNA modulates antibiotic resistance in Bacillus cereus". PLOS ONE. 7 (7): e41248. Bibcode:2012PLoSO...741248R. doi:10.1371/journal.pone.0041248. PMC 3399842. PMID 22815980.
- ^ 약 529페이지.
- ^ a b Telonis AG, Loher P, Kirino Y, Rigoutsos I (2014). "Nuclear and mitochondrial tRNA-lookalikes in the human genome". Frontiers in Genetics. 5: 344. doi:10.3389/fgene.2014.00344. PMC 4189335. PMID 25339973.
- ^ Ramos A, Barbena E, Mateiu L, del Mar González M, Mairal Q, Lima M, Montiel R, Aluja MP, Santos C, et al. (November 2011). "Nuclear insertions of mitochondrial origin: Database updating and usefulness in cancer studies". Mitochondrion. 11 (6): 946–953. doi:10.1016/j.mito.2011.08.009. PMID 21907832.
- ^ Telonis AG, Kirino Y, Rigoutsos I (2015). "Mitochondrial tRNA-lookalikes in nuclear chromosomes: Could they be functional?". RNA Biol. 12 (4): 375–380. doi:10.1080/15476286.2015.1017239. PMC 4615777. PMID 25849196.
- ^ 낸시 메이젤스와 앨런 M.와이너:게놈 태그 가설 – 분자 화석이 tRNA의 진화에 대해 우리에게 알려주는 것: RNA 세계, 제2판에서.1999 콜드 스프링 하버 연구소 프레스 ISBN 978-0-87969-561-3/99, PDF
- ^ a b c Novoa EM, Pavon-Eternod M, Pan T, Ribas de Pouplana L (March 2012). "A role for tRNA modifications in genome structure and codon usage". Cell. 149 (1): 202–213. doi:10.1016/j.cell.2012.01.050. PMID 22464330. S2CID 16487609.
- ^ Ou X, Peng W, Yang Z, Cao J, Wang M, Peppelenbosch MP, Pan Q, Cheng A (November 2020). "Evolutionarily missing and conserved tRNA genes in human and avian". Infect. Genet. Evol. 85: 104460. doi:10.1016/j.meegid.2020.104460. PMID 32679345.
- ^ Ou X, Cao J, Cheng A, Peppelenbosch MP, Pan Q (March 2019). "Errors in translational decoding: tRNA wobbling or misincorporation?". PLOS Genetics. 15 (3): 2979–2986. doi:10.1371/journal.pgen.1008017. PMC 3158919. PMID 21930591.
- ^ Ou X, Wang M, Mao S, Cao J, Cheng A, Zhu D, Chen S, Jia R, Liu M, Yang Q, Wu Y, Zhao X, Zhang S, Liu Y, Yu Y, Zhang L, Chen X, Peppelenbosch MP, Pan Q (July 2018). "Incompatible Translation Drives a Convergent Evolution and Viral Attenuation During the Development of Live Attenuated Vaccine". Front. Cell. Infect. Microbiol. 8: 249. doi:10.3389/fcimb.2018.00249. PMC 6058041. PMID 30073153.
- ^ van Warmerdam, T. "YourBioHelper.com". YourBioHelper.
{{cite web}}: CS1 maint :url-status (링크) - ^ a b c Gebetsberger J, Polacek N (December 2013). "Slicing tRNAs to boost functional ncRNA diversity". RNA Biology. 10 (12): 1798–1806. doi:10.4161/rna.27177. PMC 3917982. PMID 24351723.
- ^ a b Shigematsu M, Honda S, Kirino Y (2014). "Transfer RNA as a source of small functional RNA". Journal of Molecular Biology and Molecular Imaging. 1 (2): 8. PMC 4572697. PMID 26389128.
- ^ a b Sobala A, Hutvagner G (2011). "Transfer RNA-derived fragments: origins, processing, and functions" (PDF). Wiley Interdisciplinary Reviews: RNA. 2 (6): 853–862. doi:10.1002/wrna.96. hdl:10453/18187. PMID 21976287. S2CID 206554146.
- ^ Keam SP, Hutvagner G (November 2015). "tRNA-Derived Fragments (tRFs): Emerging New Roles for an Ancient RNA in the Regulation of Gene Expression". Life. 5 (4): 1638–1651. doi:10.3390/life5041638. PMC 4695841. PMID 26703738.
- ^ a b c Telonis AG, Loher P, Honda S, Jing Y, Palazzo J, Kirino Y, Rigoutsos I (July 2015). "Dissecting tRNA-derived fragment complexities using personalized transcriptomes reveals novel fragment classes and unexpected dependencies". Oncotarget. 6 (28): 24797–822. doi:10.18632/oncotarget.4695. PMC 4694795. PMID 26325506.
- ^ a b c d Kumar P, Anaya J, Mudunuri SB, Dutta A (October 2014). "Meta-analysis of tRNA derived RNA fragments reveals that they are evolutionarily conserved and associate with AGO proteins to recognize specific RNA targets". BMC Biology. 12: 78. doi:10.1186/s12915-014-0078-0. PMC 4203973. PMID 25270025.
- ^ a b Honda S, Loher P, Shigematsu M, Palazzo JP, Suzuki R, Imoto I, Rigoutsos I, Kirino Y (July 2015). "Sex hormone-dependent tRNA halves enhance cell proliferation in breast and prostate cancers". Proceedings of the National Academy of Sciences of the United States of America. 112 (29): E3816–E3825. Bibcode:2015PNAS..112E3816H. doi:10.1073/pnas.1510077112. PMC 4517238. PMID 26124144.
- ^ Schorn, AJ; Martienssen, R (October 2018). "Tie-Break: Host and Retrotransposons Play tRNA". Trends in Cell Biology. 28 (10): 793–806. doi:10.1016/j.tcb.2018.05.006. PMC 6520983. PMID 29934075.
- ^ Telonis AG, Rigoutsos I (March 2018). "Race Disparities in the Contribution of miRNA Isoforms and tRNA-Derived Fragments to Triple-Negative Breast Cancer". Cancer Res. 78 (5): 1140–54. doi:10.1158/0008-5472.CAN-17-1947. PMC 5935570. PMID 29229607.
- ^ Telonis AG, Loher P, Magee R, Pliatsika V, Londin E, Kirino Y, Rigoutsos I (Jun 2019). "tRNA Fragments Show Intertwining with mRNAs of Specific Repeat Content and Have Links to Disparities". Cancer Res. 79 (12): 3034–49. doi:10.1158/0008-5472.CAN-19-0789. PMC 6571059. PMID 30996049.
- ^ Shigematsu M, Kirino Y (2015). "tRNA-Derived Short Non-coding RNA as Interacting Partners of Argonaute Proteins". Gene Regulation and Systems Biology. 9: 27–33. doi:10.4137/GRSB.S29411. PMC 4567038. PMID 26401098.
- ^ Emara MM, Ivanov P, Hickman T, Dawra N, Tisdale S, Kedersha N, Hu GF, Anderson P (April 2010). "Angiogenin-induced tRNA-derived stress-induced RNAs promote stress-induced stress granule assembly". The Journal of Biological Chemistry. 285 (14): 10959–10968. doi:10.1074/jbc.M109.077560. PMC 2856301. PMID 20129916.
- ^ Goodarzi H, Liu X, Nguyen HC, Zhang S, Fish L, Tavazoie SF (May 2015). "Endogenous tRNA-Derived Fragments Suppress Breast Cancer Progression via YBX1 Displacement". Cell. 161 (4): 790–802. doi:10.1016/j.cell.2015.02.053. PMC 4457382. PMID 25957686.
- ^ Ivanov P, Emara MM, Villen J, Gygi SP, Anderson P (August 2011). "Angiogenin-induced tRNA fragments inhibit translation initiation". Molecular Cell. 43 (4): 613–623. doi:10.1016/j.molcel.2011.06.022. PMC 3160621. PMID 21855800.
- ^ Selitsky SR, Baran-Gale J, Honda M, Yamane D, Masaki T, Fannin EE, Guerra B, Shirasaki T, Shimakami T, Kaneko S, Lanford RE, Lemon SM, Sethupathy P (January 2015). "Small tRNA-derived RNAs are increased and more abundant than microRNAs in chronic hepatitis B and C". Scientific Reports. 5: 7675. Bibcode:2015NatSR...5E7675S. doi:10.1038/srep07675. PMC 4286764. PMID 25567797.
- ^ Sharma U, Conine CC, Shea JM, Boskovic A, Derr AG, Bing XY, Belleannee C, Kucukural A, Serra RW, Sun F, Song L, Carone BR, Ricci EP, Li XZ, Fauquier L, Moore MJ, Sullivan R, Mello CC, Garber M, Rando OJ (January 2016). "Biogenesis and function of tRNA fragments during sperm maturation and fertilization in mammals". Science. 351 (6271): 391–396. Bibcode:2016Sci...351..391S. doi:10.1126/science.aad6780. PMC 4888079. PMID 26721685.
- ^ Casas E, Cai G, Neill JD (2015). "Characterization of circulating transfer RNA-derived RNA fragments in cattle". Frontiers in Genetics. 6: 271. doi:10.3389/fgene.2015.00271. PMC 4547532. PMID 26379699.
- ^ Hirose Y, Ikeda KT, Noro E, Hiraoka K, Tomita M, Kanai A (July 2015). "Precise mapping and dynamics of tRNA-derived fragments (tRFs) in the development of Triops cancriformis (tadpole shrimp)". BMC Genetics. 16: 83. doi:10.1186/s12863-015-0245-5. PMC 4501094. PMID 26168920.
- ^ Karaiskos S, Naqvi AS, Swanson KE, Grigoriev A (September 2015). "Age-driven modulation of tRNA-derived fragments in Drosophila and their potential targets". Biology Direct. 10: 51. doi:10.1186/s13062-015-0081-6. PMC 4572633. PMID 26374501.
- ^ Pliatsika V, Loher P, Telonis AG, Rigoutsos I (August 2016). "MINTbase: a framework for the interactive exploration of mitochondrial and nuclear tRNA fragments". Bioinformatics. 32 (16): 2481–2489. doi:10.1093/bioinformatics/btw194. PMC 4978933. PMID 27153631.
- ^ Pliatsika V, Loher P, Magee R, Telonis AG, Londin E, Shigematsu M, Kirino Y, Rigoutsos I (January 2018). "MINTbase v2.0: a comprehensive database for tRNA-derived fragments that includes nuclear and mitochondrial fragments from all The Cancer Genome Atlas projects". Nucleic Acids Research. 46(D1) (D1): D152–D159. doi:10.1093/nar/gkx1075. PMC 5753276. PMID 29186503.
- ^ Kumar P, Mudunuri SB, Anaya J, Dutta A (January 2015). "tRFdb: a database for transfer RNA fragments". Nucleic Acids Research. 43 (Database issue): D141-5. doi:10.1093/nar/gku1138. PMC 4383946. PMID 25392422.
- ^ a b Vincent RM, Wright BW, Jaschke PR (April 2019). "Measuring Amber Initiator tRNA Orthogonality in a Genomically Recoded Organism". ACS Synthetic Biology. 8 (4): 675–685. doi:10.1021/acssynbio.9b00021. PMID 30856316. S2CID 75136654.
- ^ Govindan A, Miryala S, Mondal S, Varshney U (November 2018). "Development of Assay Systems for Amber Codon Decoding at the Steps of Initiation and Elongation in Mycobacteria". Journal of Bacteriology. 200 (22). doi:10.1128/jb.00372-18. PMC 6199473. PMID 30181124.
- ^ White RJ (March 1997). "Regulation of RNA polymerases I and III by the retinoblastoma protein: a mechanism for growth control?". Trends in Biochemical Sciences. 22 (3): 77–80. doi:10.1016/S0968-0004(96)10067-0. PMID 9066256.
- ^ Sharp S, Dingermann T, Söll D (September 1982). "The minimum intragenic sequences required for promotion of eukaryotic tRNA gene transcription". Nucleic Acids Research. 10 (18): 5393–5406. doi:10.1093/nar/10.18.5393. PMC 320884. PMID 6924209.
- ^ a b Dieci G, Fiorino G, Castelnuovo M, Teichmann M, Pagano A (December 2007). "The expanding RNA polymerase III transcriptome". Trends in Genetics. 23 (12): 614–622. doi:10.1016/j.tig.2007.09.001. hdl:11381/1706964. PMID 17977614.
- ^ Tocchini-Valentini GD, Fruscoloni P, Tocchini-Valentini GP (December 2009). "Processing of multiple-intron-containing pretRNA". Proceedings of the National Academy of Sciences of the United States of America. 106 (48): 20246–20251. Bibcode:2009PNAS..10620246T. doi:10.1073/pnas.0911658106. PMC 2787110. PMID 19910528.
- ^ Abelson J, Trotta CR, Li H (May 1998). "tRNA splicing". The Journal of Biological Chemistry. 273 (21): 12685–12688. doi:10.1074/jbc.273.21.12685. PMID 9582290.
- ^ a b Soma A (2014). "Circularly permuted tRNA genes: their expression and implications for their physiological relevance and development". Frontiers in Genetics. 5: 63. doi:10.3389/fgene.2014.00063. PMC 3978253. PMID 24744771.
- ^ Frank DN, Pace NR (1998). "Ribonuclease P: unity and diversity in a tRNA processing ribozyme". Annual Review of Biochemistry. 67 (1): 153–180. doi:10.1146/annurev.biochem.67.1.153. PMID 9759486.
- ^ Ceballos M, Vioque A (2007). "tRNase Z". Protein and Peptide Letters. 14 (2): 137–145. doi:10.2174/092986607779816050. PMID 17305600.
- ^ Randau L, Schröder I, Söll D (May 2008). "Life without RNase P". Nature. 453 (7191): 120–123. Bibcode:2008Natur.453..120R. doi:10.1038/nature06833. PMID 18451863. S2CID 3103527.
- ^ Weiner AM (October 2004). "tRNA maturation: RNA polymerization without a nucleic acid template". Current Biology. 14 (20): R883-5. doi:10.1016/j.cub.2004.09.069. PMID 15498478.
- ^ Kutay U, Lipowsky G, Izaurralde E, Bischoff FR, Schwarzmaier P, Hartmann E, Görlich D (February 1998). "Identification of a tRNA-specific nuclear export receptor". Molecular Cell. 1 (3): 359–369. doi:10.1016/S1097-2765(00)80036-2. PMID 9660920.
- ^ Arts GJ, Fornerod M, Mattaj IW (March 1998). "Identification of a nuclear export receptor for tRNA". Current Biology. 8 (6): 305–314. doi:10.1016/S0960-9822(98)70130-7. PMID 9512417. S2CID 17803674.
- ^ Arts GJ, Kuersten S, Romby P, Ehresmann B, Mattaj IW (December 1998). "The role of exportin-t in selective nuclear export of mature tRNAs". The EMBO Journal. 17 (24): 7430–7441. doi:10.1093/emboj/17.24.7430. PMC 1171087. PMID 9857198.
- ^ Yoshihisa T, Yunoki-Esaki K, Ohshima C, Tanaka N, Endo T (August 2003). "Possibility of cytoplasmic pre-tRNA splicing: the yeast tRNA splicing endonuclease mainly localizes on the mitochondria". Molecular Biology of the Cell. 14 (8): 3266–3279. doi:10.1091/mbc.E02-11-0757. PMC 181566. PMID 12925762.
- ^ Kühnlein, Alexandra; Lanzmich, Simon A.; Brun, Dieter (2 March 2021). "tRNA sequences can assemble into a replicator". eLife. 10. doi:10.7554/eLife.63431. PMC 7924937. PMID 33648631.
- ^ Maximilian, Ludwig (3 April 2021). "Solving the Chicken-and-the-Egg Problem – "A Step Closer to the Reconstruction of the Origin of Life"". SciTechDaily. Retrieved 3 April 2021.
- ^ Kresge, Nicole; Simoni, Robert D.; Hill, Robert L. (October 7, 2005). "The Discovery of tRNA by Paul C. Zamecnik". Journal of Biological Chemistry. 280 (40): e37–e39. doi:10.1016/S0021-9258(20)79029-0 – via www.jbc.org.
- ^ Clark BF (October 2006). "The crystal structure of tRNA" (PDF). Journal of Biosciences. 31 (4): 453–457. doi:10.1007/BF02705184. PMID 17206065. S2CID 19558731.
- ^ Holley RW, Apgar J, Everett GA, Madison JT, Marquisee M, Merrill SH, Penswick JR, Zamir A (March 1965). "Structure of a Ribonucleic Acid". Science. 147 (3664): 1462–1465. Bibcode:1965Sci...147.1462H. doi:10.1126/science.147.3664.1462. PMID 14263761. S2CID 40989800.
- ^ "Obituary". The New York Times. July 4, 1991.
- ^ "The Nobel Prize in Physiology or Medicine 1968: Robert W. Holley – Facts". Nobel Prize Outreach AB. 2022. Retrieved 18 March 2022.
- ^ Ladner JE, Jack A, Robertus JD, Brown RS, Rhodes D, Clark BF, Klug A (November 1975). "Structure of yeast phenylalanine transfer RNA at 2.5 A resolution". Proceedings of the National Academy of Sciences of the United States of America. 72 (11): 4414–4418. Bibcode:1975PNAS...72.4414L. doi:10.1073/pnas.72.11.4414. PMC 388732. PMID 1105583.
- ^ Kim SH, Quigley GJ, Suddath FL, McPherson A, Sneden D, Kim JJ, Weinzierl J, Rich A (January 1973). "Three-dimensional structure of yeast phenylalanine transfer RNA: folding of the polynucleotide chain". Science. 179 (4070): 285–288. Bibcode:1973Sci...179..285K. doi:10.1126/science.179.4070.285. PMID 4566654. S2CID 28916938.