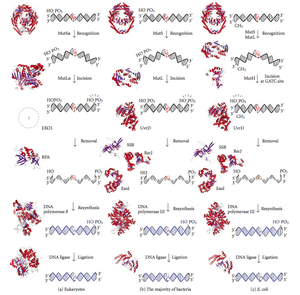
DNA불일치복구
DNA mismatch repairDNA 미스매치 리페어(MMR)는 DNA 복제 및 재조합 중에 발생할 수 있는 염기의 잘못된 삽입, 삭제 및 잘못된 삽입을 인식하고 복구하는 시스템이며, 일부 형태의 DNA [1][2]손상을 복구하는 시스템입니다.
불일치 복구는 가닥에 따라 다릅니다.DNA 합성 중에 새로 합성된 (딸) 가닥은 일반적으로 오류를 포함할 것이다.수리를 시작하기 위해 불일치 수리 기계는 새로 합성된 가닥과 템플릿(부모)을 구분합니다.그램 음성 박테리아에서 일시적인 반메틸화는 가닥을 구별한다(부모는 메틸화 되고 딸은 메틸화되지 않음).그러나 다른 원핵 생물과 진핵 생물에서는 정확한 메커니즘이 명확하지 않다.진핵생물에서 새롭게 합성된 후행선 DNA는 일시적으로 (DNA 연결효소에 의해 봉인되기 전에) 흠집을 포함하고 있으며, 불일치 교정 시스템을 적절한 가닥으로 유도하는 신호를 제공하는 것으로 의심된다.이는 이러한 흠집이 반드시 선두 가닥에 존재해야 한다는 것을 의미하며, 이에 대한 증거가 최근에 발견되었습니다.[3]최근의 연구는[4] 도넛 모양의 단백질의 한쪽 면이 니크의 3'-OH 끝을 향해 나란히 배치되도록 니크가 특정 방향의 복제 슬라이딩 클램프인 증식 세포핵항원(PCNA)의 RFC 의존적 로딩 부위임을 보여주고 있다.그런 다음 로드된 PCNA는 불일치 및 MutSalpha 또는 MutSbeta가 존재하는 경우 MutLalpha 엔도핵산가수분해효소의 액션을 도터 스트랜드로 유도합니다.
DNA의 초나선 구조를 교란시키는 돌연변이 사건은 세포의 유전적 안정성을 해칠 수 있는 가능성을 가지고 있다.손상 검출 및 복구 시스템이 복제 기계 자체만큼이나 복잡하다는 사실은 DNA 충실도에 진화의 중요성을 강조합니다.
일치하지 않는 염기의 예로는 G/T 또는 A/C 쌍이 있습니다(DNA 수리 참조).미스매치는 일반적으로 DNA 복제 중 염기의 호변이성체화에 기인한다.손상은 불일치로 인한 기형을 인식하고 템플릿과 비템플릿 가닥을 결정하며 잘못 삽입된 염기를 제거하고 올바른 뉴클레오티드로 대체함으로써 복구된다.제거 과정은 단순히 일치하지 않는 뉴클레오티드 그 이상을 포함한다.새롭게 합성된 DNA 가닥의 몇 개 또는 수천 개의 염기쌍을 제거할 수 있다.
복구단백질 불일치
| DNA 불일치 복구 단백질, C-말단 도메인 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 hpms2-199gs | |||||||||
| 식별자 | |||||||||
| 기호. | DNA_오류_복구 | ||||||||
| 팜 | PF01119 | ||||||||
| 빠맘 클랜 | CL0329 | ||||||||
| 인터프로 | IPR013507 | ||||||||
| 프로 사이트 | PDOC00057 | ||||||||
| SCOP2 | 1 bkn / SCOPe / SUPFAM | ||||||||
| |||||||||
미스매치 수리는 원핵생물에서 진핵생물까지 고도로 보존된 과정이다.미스매치 복구에 대한 첫 번째 증거는 S. 폐렴(hexA 및 hexB 유전자)에서 얻었다.대장균에 대한 후속 연구는 돌연변이가 불활성화되었을 때, 초변성 변종을 일으키는 많은 유전자를 확인했다.따라서 유전자 생성물은 뮤트 단백질이라고 불리며 불일치 복구 시스템의 주요 활성 성분이다.이들 단백질 중 3가지인 MutS, MutH 및 MutL은 불일치를 검출하고 복구 기계를 유도하는 데 필수적입니다(MutS는 HexA의 상동어이고 MutL은 HexB의 상동어입니다).
MutS는 도터 스트랜드 상의 불일치 염기를 인식하고 돌연변이된 DNA를 결합하는 이합체(MutS2)를 형성합니다.MutH는 딸의 DNA를 따라 반메틸화 부위에 결합하지만 MutS-DNA 복합체를 결합하고 MutS와 MutH 사이의2 매개자 역할을 하는 MutL 이합체(MutL2)에 의해 접촉될 때만 활성화되어 MutS-DNA 복합체를 활성화하는 것으로 그 작용한다.DNA는 루프 아웃되어 불일치에 가장 가까운 d(GATC) 메틸화 부위가 검색됩니다.이 부위는 최대 1kb 떨어져 있을 수 있습니다.MutS-DNA 복합체에 의해 활성화되면 MutH는 반메틸화 부위 근처에 딸 가닥을 긁는다.MutL은 UvrD Helicase(DNA Helicase II)를 모집하여 특정 3'~5' 극성으로 두 가닥을 분리한다.그런 다음 전체 MutSHL 복합체는 DNA를 따라 불일치 방향으로 미끄러져 가면서 제거될 가닥을 해방시킵니다.핵산가수분해효소는 복합체를 추적하여 ss-DNA 꼬리를 소화시킵니다.모집된 핵산가수분해효소(exonuclease)는 MutH가 사슬 – 5' 또는 3' 중 어느 쪽을 절개하느냐에 따라 다르다.MutH에 의해 만들어진 닉이 미스매치의 5' 끝에 있는 경우, RecJ 또는 ExoVII(둘 다 5'~3' 엑소핵산가수분해효소)를 사용한다.단, 닉이 미스매치의 3' 끝에 있는 경우에는 ExoI(3'~5' 효소)를 사용한다.
전체 과정은 불일치 부위를 지나 종료된다. 즉, 사이트 자체와 주변 뉴클레오티드가 모두 완전히 제거된다.엑소핵산가수분해효소에 의해 생성된 단일 가닥 간격은 다른 가닥을 템플릿으로 사용하는 DNA 중합효소 III(단일 가닥 결합 단백질에 의해 지원됨)에 의해 복구될 수 있으며, 최종적으로 DNA 연결효소에 의해 봉인된다.DNA메틸화효소는 딸 가닥을 빠르게 메틸화한다.
MutS 호몰로그
결합 시 MutS2 이합체는 DNA 나선을 구부리고 약 20개의 염기쌍을 차폐합니다.그것은 약한 ATP 효소의 활성을 가지고 있고, ATP의 결합은 분자 표면에 3차 구조를 형성하도록 이끈다.MutS의 결정 구조는 예외적으로 비대칭이며, 액티브한 구성이 다이머인 반면, 2개의 절반 중 1개만이 불일치 사이트와 상호 작용합니다.
진핵생물에서 MutS 호몰로지는 Msh2/Msh6(MutSα)와 Msh2/Msh3(MutSβ)의 두 가지 주요 헤테로디미터를 형성한다.MutSα 경로는 주로 염기 치환 및 소루프 불일치 복구에 관여합니다.MutSβ 경로는 또한 큰 루프(~10개의 뉴클레오티드 루프) 복구와 더불어 작은 루프 복구에도 관여한다.그러나 MutSβ는 염기 치환을 복구하지 않습니다.
MutL 호몰로그
MutL은 또한 약한 ATP 효소 활성을 가지고 있다.MutS 및 MutH와 콤플렉스를 형성하여 DNA에서 MutS 설치 공간을 늘립니다.
그러나 UvrD의 처리성(해리 전 효소가 DNA를 따라 움직일 수 있는 거리)은 40~50bp에 불과하다.MutH에 의해 작성된 닉과 미스매치 사이의 거리는 평균 600bp까지이므로 다른 UvrD가 로드되지 않은 경우 언와인드 섹션은 그 상보적인 스트랜드에 재애널링 할 수 있으므로 프로세스를 다시 시작해야 합니다.단, MutL에 의해 보조될 경우 UvrD 로딩 속도가 크게 향상됩니다.개별 UvrD 분자의 처리성(및 ATP 활용도)은 동일하지만, DNA에 대한 총 효과는 상당히 높아진다. 각 UvrD가 40-50 bp의 DNA를 풀고, 해리를 한 후, 즉시 다른 UvRD로 대체되어 그 과정을 반복하기 때문에 DNA는 재결합할 기회가 없다.이것은 DNA의 큰 부분을 핵산가수분해효소 소화에 노출시켜 잘못된 DNA를 빠르게 절제할 수 있게 한다.
진핵생물에는 MLH1, MLH2, MLH3, PMS1, PMS2로 지정된 5개의 MutL 상동체가 있습니다.그것들은 대장균의 MutL을 모방하는 헤테로디미터를 형성한다.원핵생물 MutL의 인간 호몰로지는 MutLα, MutLβ, MutLγ로 불리는 세 가지 복합체를 형성한다.MutLα 복합체는 MLH1 및 PMS2 서브유닛으로 구성되며, MutLβ 헤테로디머는 MLH1 및 PMS1로 구성되며, MutLδ는 MLH1 및 MLH3으로 구성됩니다.MutLα는 불일치 및 기타 필수 단백질인 MutSα 및 PCNA에 의해 활성화될 때 딸 가닥의 가닥을 파괴하는 엔도핵산가수분해효소 역할을 한다.이러한 가닥의 중단은 불일치 DNA를 제거하는 핵산가수분해효소 활성의 진입점 역할을 합니다. 불일치 복구에서 MutLβ와 MutL†가 수행하는 역할은 잘 알려져 있지 않습니다.
MutH: 대장균과 살모넬라균에 존재하는 핵산내 분해효소
MutH는 매우 약한 핵산가수분해효소이며 MutL(MutS에 결합됨)에 한 번 결합되면 활성화됩니다.그것은 비메틸화 DNA와 반메틸화 DNA의 비메틸화 가닥을 손상시키지만 완전히 메틸화 DNA를 손상시키지는 않는다.실험 결과 어느 가닥도 [citation needed]메틸화되지 않은 경우 미스매치 복구는 무작위인 것으로 나타났습니다.이러한 행동은 MutH가 불일치를 포함하는 가닥을 결정한다는 제안으로 이어졌다.MutH는 진핵생물 상동체가 없다.엔도핵산가수분해효소 기능은 일부 특수화된 5'-3' 엑소핵산가수분해효소 활성을 가진 MutL 호몰로그에 의해 결정된다.진핵생물에서 새로 합성된 딸 가닥의 불일치를 제거하기 위한 가닥 바이어스는 복제 중에 생성된 새로운 가닥의 오카자키 조각의 자유 3' 말단에 의해 제공될 수 있다.
PCNAβ슬라이딩 클램프
PCNA 및 β-슬라이딩 클램프는 각각 MutSα/β 및 MutS와 관련된다.초기 보고서에서는 PCNA-MutSα 복합체가 불일치 [6]인식을 강화할 수 있다고 제시되었지만, 최근[7] PCNA의 유무에 있어 불일치 여부에 대한 MutSα의 친화력에는 뚜렷한 변화가 없는 것으로 입증되었습니다.또한 체외에서 PCNA와 상호작용할 수 없는 MutSα의 돌연변이는 불일치 인식 및 거의 야생형 수준으로 불일치 절제를 수행할 수 있는 능력을 보인다.이러한 돌연변이는 5' 가닥이 끊어지면 나타나는 수리 반응에서 결함이 있어 처음으로 MutSα가 제거 후 단계에서 기능함을 시사합니다.
임상적 의의
불일치 복구에서 상속된 결함
뮤트 단백질의 인간 상동성 돌연변이는 게놈 안정성에 영향을 미치며, 이는 일부 인간 암에 관여하는 마이크로 위성 불안정(MSI)을 초래할 수 있다.구체적으로는 유전성 비폴리포시스 대장암(HNPCC 또는 린치증후군)은 종양억제유전자로 분류되는 MutS 및 MutL 호몰로그 MSH2, MLH1을 각각 코드하는 유전자의 생식계 변이체를 손상시키는 데 기인한다.HNPCC의 한 가지 아형인 뮤어-토레 증후군(MTS)은 피부 종양과 관련이 있다.유전된 MMR 유전자의 두 복사본(알레르)이 해로운 유전자 변형을 수반하는 경우, 이는 매우 드물고 심각한 상태를 초래한다: 불일치 복구 암 증후군(또는 체질 불일치 복구 결핍증, CMMR-D)은 어린 나이에 종양이 여러 번 발생하고 종종 결장과 [8]뇌종양으로 나타난다.
불일치 복구 유전자의 후생성 소음
DNA 수복 결핍을 가진 산발성 암은 DNA 수복 유전자에 돌연변이가 거의 없지만, 대신에 그들은 DNA [9]수복 유전자 발현을 억제하는 프로모터 메틸화 같은 후생유전학적 변화를 갖는 경향이 있다.대장암의 약 13%는 일반적으로 MLH1(9.8%) 또는 때로는 MSH2, MSH6 또는 PMS2(모두 1.5%)[10]의 손실로 인해 DNA 불일치 복구가 부족하다.대부분의 MLH1 결핍 산발성 대장암의 경우, 결핍은 MLH1 프로모터 [10]메틸화 때문이었다.다른 종류의 암은 MLH1 손실의 빈도가 높으며(아래 표 참조), 이는 다시 MLH1 유전자의 프로모터의 메틸화에 의한 결과이다.MMR 결핍의 기초가 되는 다른 후생유전 메커니즘은 마이크로RNA의 과잉 발현을 포함할 수 있다. 예를 들어 miR-155 수준은 대장암에서 [11]MLH1 또는 MSH2의 발현과 반비례한다.
| 암의 종류 | 암의 결핍 빈도 | 인접 필드 결함의 결함 빈도 |
|---|---|---|
| 배 | 32 %[12][13] | 24%-28% |
| 위(공형 종양) | 74 %[14] | 71% |
| 카슈미르 계곡의 위 | 73 %[15] | 20% |
| 식도 | 73 %[16] | 27% |
| 두경부 편평상피암(HNSCC) | 31%~33%[17][18] | 20%-25% |
| 비소세포폐암(NSCLC) | 69 %[19] | 72% |
| 대장균 | 10%[10] |
필드 결함의 MMR 장애
필드 결손(필드 암화)은 후생유전적 또는 유전적 변화에 의해 사전 조정되어 암이 발병하는 경향이 있는 상피 영역입니다.루빈에 의해 지적되었듯이, "...돌연변이형 인간 대장종양에서 발견된 체세포 돌연변이의 80% 이상이 말기 복제 확장이 [20][21]시작되기 전에 발생한다는 증거가 있다."마찬가지로, Vogelstein 외 [22]연구진은 종양에서 확인된 체세포 돌연변이의 절반 이상이 정상세포의 성장 중 종양이 생기기 전 단계(장결함)에서 발생했다고 지적한다.
MLH1 결핍은 종양을 둘러싼 현장 결함(역사적으로 정상 조직)에서 흔했다. 위의 표를 참조하십시오.후생적으로 무음화 또는 돌연변이된 MLH1은 줄기세포에 선택적 이점을 부여하지 않을 가능성이 높지만, 이는 돌연변이율을 증가시키고, 하나 이상의 돌연변이 유전자가 세포에 선택적 이점을 제공할 수 있다.부족한 사람MLH1 유전자는 변이된 줄기세포가 확장된 클론을 생성할 때 선택적으로 거의 중립적인 승객(히치하이커) 유전자로 운반될 수 있다.후생적으로 억제된 MLH1을 가진 클론의 지속적인 존재는 추가적인 돌연변이를 계속 발생시킬 것이며, 그 중 일부는 종양을 발생시킬 수 있다.
사람의 MMR 성분
사람의 경우, 7개의 DNA 불일치 복구(MMR) 단백질(MLH1, MLH3, MSH2, MSH3, MSH6, PMS1 및 PMS2)이 순차적으로 작동하여 DNA [23]불일치 복구를 시작한다.또한 Exo1 의존 MMR 서브패스 및 Exo1 의존 [24]MMR 서브패스도 있습니다.
인간의 불일치 복구(MMR 유전자에 의한 개시 후)에 관여하는 다른 유전자 생성물은 DNA 중합효소 델타, PCNA, RPA, HMGB1, RFC 및 DNA 리가아제 I, 히스톤 및 [25][26]크로마틴 수정 인자를 포함한다.
특정 상황에서 MMR 경로는 오류 발생 가능성이 높은 DNA 중합효소 에타(POLH)를 획득할 수 있다.이것은 체세포의 과변성 동안 B림프구에서 일어나며, 여기서 POLH는 항체 [27]유전자에 유전적 변이를 도입하기 위해 사용된다.그러나, 이러한 오류 발생 가능성이 높은 MMR 경로는 제노톡신에 노출되면 다른 유형의 인간 세포에서 트리거될 수 있으며, 실제로 다양한 인간 암에서 광범위하게 활성화되어 POLH [29]활성의 특징을 지닌 돌연변이를 일으킨다.
MMR 및 돌연변이 빈도
미스매치 및 인델을 인식하고 복구하는 것은 세포에 있어서 중요하다.그렇게 하지 않으면 마이크로 위성 불안정성(MSI)과 자연 돌연변이율(변이자 표현형)이 높아지기 때문이다.다른 암에 비해 MMR 결핍(MSI) 암은 돌연변이의 빈도가 [30]매우 높아 흑색종과 폐암에 가깝고 자외선과 돌연변이 화학물질에 많이 노출되어 발생하는 암이다.
매우 높은 돌연변이 부담 외에도, MMR 결핍은 인간 게놈 전체에 특이한 체세포 돌연변이의 분포를 초래한다: 이것은 MMR이 유전자가 풍부한 조기 복제 유색체 [31]영역을 우선적으로 보호한다는 것을 암시한다.이와는 대조적으로, 유전자 부족, 늦게 복제되는 이색 게놈 영역은 많은 인간 [32]종양에서 높은 돌연변이율을 보인다.
활성 크로마틴의 후생유전학적 마크인 히스톤 수식 H3K36me3는 MSH2-MSH6([33]hMutSα) 복합체를 모집하는 능력을 가지고 있다.일관되게 H3K36me3가 높은 인간 게놈 영역은 MMR [29]활성으로 인한 돌연변이를 적게 축적한다.
종양에서 다중 DNA 복구 경로 손실
MMR의 부족은 종종 다른 DNA 복구 [9]유전자의 손실과 함께 발생한다.예를 들어, MMR 유전자 MLH1과 MLH3뿐만 아니라 다른 11개의 DNA 복구 유전자(MGMT와 많은 NER 경로 유전자 등)는 정상 뇌 [34]조직과 대조적으로 낮은 등급뿐만 아니라 높은 등급의 성상세포종에서도 유의하게 낮은 수준으로 조절되었다.또한 위암 검체 135개에서 MLH1과 MGMT 발현은 밀접한 상관관계가 있었으며, MLH1과 MGMT 손실은 종양 진행 [35]중에 동시에 가속되는 것으로 나타났다.
여러 DNA 복구 유전자의 발현 부족은 종종 [9]암에서 발견되며, 일반적으로 암에서 발견되는 수천 개의 돌연변이에 기여할 수 있다(암에서 돌연변이 빈도 참조).
에이징
중요한 실험적인 지지를 얻지 못한 대중적인 생각은 돌연변이가 DNA 손상과 구별되는 것이 노화의 주요 원인이라는 것이다.mutL 상동 Pms2에 결함이 있는 생쥐는 모든 조직에서 돌연변이 빈도가 약 100배 증가하지만, 더 빨리 [36]늙는 것으로 보이지는 않는다.이 쥐들은 초기 발암과 수컷 불임을 제외하고는 대부분 정상적인 발육과 수명을 보인다.
「 」를 참조해 주세요.
레퍼런스
- ^ Iyer RR, Pluciennik A, Burdett V, Modrich PL (February 2006). "DNA mismatch repair: functions and mechanisms". Chemical Reviews. 106 (2): 302–23. doi:10.1021/cr0404794. PMID 16464007.
- ^ Larrea AA, Lujan SA, Kunkel TA (May 2010). "SnapShot: DNA mismatch repair". Cell. 141 (4): 730–730.e1. doi:10.1016/j.cell.2010.05.002. PMID 20478261. S2CID 26969788.
- ^ Heller RC, Marians KJ (December 2006). "Replisome assembly and the direct restart of stalled replication forks". Nature Reviews. Molecular Cell Biology. 7 (12): 932–43. doi:10.1038/nrm2058. PMID 17139333. S2CID 27666329.
- ^ Pluciennik A, Dzantiev L, Iyer RR, Constantin N, Kadyrov FA, Modrich P (September 2010). "PCNA function in the activation and strand direction of MutLα endonuclease in mismatch repair". Proceedings of the National Academy of Sciences of the United States of America. 107 (37): 16066–71. doi:10.1073/pnas.1010662107. PMC 2941292. PMID 20713735.
- ^ Kadyrov FA, Dzantiev L, Constantin N, Modrich P (July 2006). "Endonucleolytic function of MutLalpha in human mismatch repair". Cell. 126 (2): 297–308. doi:10.1016/j.cell.2006.05.039. PMID 16873062. S2CID 15643051.
- ^ Flores-Rozas H, Clark D, Kolodner RD (November 2000). "Proliferating cell nuclear antigen and Msh2p-Msh6p interact to form an active mispair recognition complex". Nature Genetics. 26 (3): 375–8. doi:10.1038/81708. PMID 11062484. S2CID 20861705.
- ^ Iyer RR, Pohlhaus TJ, Chen S, Hura GL, Dzantiev L, Beese LS, Modrich P (May 2008). "The MutSalpha-proliferating cell nuclear antigen interaction in human DNA mismatch repair". The Journal of Biological Chemistry. 283 (19): 13310–9. doi:10.1074/jbc.M800606200. PMC 2423938. PMID 18326858.
- ^ 온라인 Mendelian In Man (OMIM) : 276300
- ^ a b c Bernstein C, Bernstein H (May 2015). "Epigenetic reduction of DNA repair in progression to gastrointestinal cancer". World Journal of Gastrointestinal Oncology. 7 (5): 30–46. doi:10.4251/wjgo.v7.i5.30. PMC 4434036. PMID 25987950.
- ^ a b c Truninger K, Menigatti M, Luz J, Russell A, Haider R, Gebbers JO, et al. (May 2005). "Immunohistochemical analysis reveals high frequency of PMS2 defects in colorectal cancer". Gastroenterology. 128 (5): 1160–71. doi:10.1053/j.gastro.2005.01.056. PMID 15887099.
- ^ Valeri N, Gasparini P, Fabbri M, Braconi C, Veronese A, Lovat F, et al. (April 2010). "Modulation of mismatch repair and genomic stability by miR-155". Proceedings of the National Academy of Sciences of the United States of America. 107 (15): 6982–7. Bibcode:2010PNAS..107.6982V. doi:10.1073/pnas.1002472107. PMC 2872463. PMID 20351277.
- ^ Kupčinskaitė-Noreikienė R, Skiecevičienė J, Jonaitis L, Ugenskienė R, Kupčinskas J, Markelis R, et al. (2013). "CpG island methylation of the MLH1, MGMT, DAPK, and CASP8 genes in cancerous and adjacent noncancerous stomach tissues". Medicina. 49 (8): 361–6. doi:10.3390/medicina49080056. PMID 24509146.
- ^ Waki T, Tamura G, Tsuchiya T, Sato K, Nishizuka S, Motoyama T (August 2002). "Promoter methylation status of E-cadherin, hMLH1, and p16 genes in nonneoplastic gastric epithelia". The American Journal of Pathology. 161 (2): 399–403. doi:10.1016/S0002-9440(10)64195-8. PMC 1850716. PMID 12163364.
- ^ Endoh Y, Tamura G, Ajioka Y, Watanabe H, Motoyama T (September 2000). "Frequent hypermethylation of the hMLH1 gene promoter in differentiated-type tumors of the stomach with the gastric foveolar phenotype". The American Journal of Pathology. 157 (3): 717–22. doi:10.1016/S0002-9440(10)64584-1. PMC 1949419. PMID 10980110.
- ^ Wani M, Afroze D, Makhdoomi M, Hamid I, Wani B, Bhat G, et al. (2012). "Promoter methylation status of DNA repair gene (hMLH1) in gastric carcinoma patients of the Kashmir valley" (PDF). Asian Pacific Journal of Cancer Prevention. 13 (8): 4177–81. doi:10.7314/apjcp.2012.13.8.4177. PMID 23098428.
- ^ Chang Z, Zhang W, Chang Z, Song M, Qin Y, Chang F, et al. (January 2015). "Expression characteristics of FHIT, p53, BRCA2 and MLH1 in families with a history of oesophageal cancer in a region with a high incidence of oesophageal cancer". Oncology Letters. 9 (1): 430–436. doi:10.3892/ol.2014.2682. PMC 4246613. PMID 25436004.
- ^ Tawfik HM, El-Maqsoud NM, Hak BH, El-Sherbiny YM (2011). "Head and neck squamous cell carcinoma: mismatch repair immunohistochemistry and promoter hypermethylation of hMLH1 gene". American Journal of Otolaryngology. 32 (6): 528–36. doi:10.1016/j.amjoto.2010.11.005. PMID 21353335.
- ^ Zuo C, Zhang H, Spencer HJ, Vural E, Suen JY, Schichman SA, et al. (October 2009). "Increased microsatellite instability and epigenetic inactivation of the hMLH1 gene in head and neck squamous cell carcinoma". Otolaryngology–Head and Neck Surgery. 141 (4): 484–90. doi:10.1016/j.otohns.2009.07.007. PMID 19786217. S2CID 8357370.
- ^ Safar AM, Spencer H, Su X, Coffey M, Cooney CA, Ratnasinghe LD, et al. (June 2005). "Methylation profiling of archived non-small cell lung cancer: a promising prognostic system". Clinical Cancer Research. 11 (12): 4400–5. doi:10.1158/1078-0432.CCR-04-2378. PMID 15958624.
- ^ Rubin H (March 2011). "Fields and field cancerization: the preneoplastic origins of cancer: asymptomatic hyperplastic fields are precursors of neoplasia, and their progression to tumors can be tracked by saturation density in culture". BioEssays. 33 (3): 224–31. doi:10.1002/bies.201000067. PMID 21254148. S2CID 44981539.
- ^ Tsao JL, Yatabe Y, Salovaara R, Järvinen HJ, Mecklin JP, Aaltonen LA, et al. (February 2000). "Genetic reconstruction of individual colorectal tumor histories". Proceedings of the National Academy of Sciences of the United States of America. 97 (3): 1236–41. Bibcode:2000PNAS...97.1236T. doi:10.1073/pnas.97.3.1236. PMC 15581. PMID 10655514.
- ^ Vogelstein B, Papadopoulos N, Velculescu VE, Zhou S, Diaz LA, Kinzler KW (March 2013). "Cancer genome landscapes". Science. 339 (6127): 1546–58. Bibcode:2013Sci...339.1546V. doi:10.1126/science.1235122. PMC 3749880. PMID 23539594.
- ^ Pal T, Permuth-Wey J, Sellers TA (August 2008). "A review of the clinical relevance of mismatch-repair deficiency in ovarian cancer". Cancer. 113 (4): 733–42. doi:10.1002/cncr.23601. PMC 2644411. PMID 18543306.
- ^ Goellner EM, Putnam CD, Kolodner RD (August 2015). "Exonuclease 1-dependent and independent mismatch repair". DNA Repair. 32: 24–32. doi:10.1016/j.dnarep.2015.04.010. PMC 4522362. PMID 25956862.
- ^ Li GM (January 2008). "Mechanisms and functions of DNA mismatch repair". Cell Research. 18 (1): 85–98. doi:10.1038/cr.2007.115. PMID 18157157.
- ^ Li GM (July 2014). "New insights and challenges in mismatch repair: getting over the chromatin hurdle". DNA Repair. 19: 48–54. doi:10.1016/j.dnarep.2014.03.027. PMC 4127414. PMID 24767944.
- ^ Chahwan R, Edelmann W, Scharff MD, Roa S (August 2012). "AIDing antibody diversity by error-prone mismatch repair". Seminars in Immunology. 24 (4): 293–300. doi:10.1016/j.smim.2012.05.005. PMC 3422444. PMID 22703640.
- ^ Hsieh P (September 2012). "DNA mismatch repair: Dr. Jekyll and Mr. Hyde?". Molecular Cell. 47 (5): 665–6. doi:10.1016/j.molcel.2012.08.020. PMC 3457060. PMID 22980456.
- ^ a b Supek F, Lehner B (July 2017). "Clustered Mutation Signatures Reveal that Error-Prone DNA Repair Targets Mutations to Active Genes". Cell. 170 (3): 534–547.e23. doi:10.1016/j.cell.2017.07.003. hdl:10230/35343. PMID 28753428.
- ^ Tuna M, Amos CI (November 2013). "Genomic sequencing in cancer". Cancer Letters. 340 (2): 161–70. doi:10.1016/j.canlet.2012.11.004. PMC 3622788. PMID 23178448.
- ^ Supek F, Lehner B (May 2015). "Differential DNA mismatch repair underlies mutation rate variation across the human genome". Nature. 521 (7550): 81–4. Bibcode:2015Natur.521...81S. doi:10.1038/nature14173. PMC 4425546. PMID 25707793.
- ^ Schuster-Böckler B, Lehner B (August 2012). "Chromatin organization is a major influence on regional mutation rates in human cancer cells". Nature. 488 (7412): 504–7. Bibcode:2012Natur.488..504S. doi:10.1038/nature11273. PMID 22820252. S2CID 205229634.
- ^ Li F, Mao G, Tong D, Huang J, Gu L, Yang W, Li GM (April 2013). "The histone mark H3K36me3 regulates human DNA mismatch repair through its interaction with MutSα". Cell. 153 (3): 590–600. doi:10.1016/j.cell.2013.03.025. PMC 3641580. PMID 23622243.
- ^ Jiang Z, Hu J, Li X, Jiang Y, Zhou W, Lu D (December 2006). "Expression analyses of 27 DNA repair genes in astrocytoma by TaqMan low-density array". Neuroscience Letters. 409 (2): 112–7. doi:10.1016/j.neulet.2006.09.038. PMID 17034947.
- ^ Kitajima Y, Miyazaki K, Matsukura S, Tanaka M, Sekiguchi M (2003). "Loss of expression of DNA repair enzymes MGMT, hMLH1, and hMSH2 during tumor progression in gastric cancer". Gastric Cancer. 6 (2): 86–95. doi:10.1007/s10120-003-0213-z. PMID 12861399.
- ^ Narayanan L, Fritzell JA, Baker SM, Liskay RM, Glazer PM (April 1997). "Elevated levels of mutation in multiple tissues of mice deficient in the DNA mismatch repair gene Pms2". Proceedings of the National Academy of Sciences of the United States of America. 94 (7): 3122–7. doi:10.1073/pnas.94.7.3122. PMC 20332. PMID 9096356.
추가 정보
- Hsieh P, Yamane K (2008). "DNA mismatch repair: molecular mechanism, cancer, and ageing". Mechanisms of Ageing and Development. 129 (7–8): 391–407. doi:10.1016/j.mad.2008.02.012. PMC 2574955. PMID 18406444.
- Iyer RR, Pluciennik A, Burdett V, Modrich PL (February 2006). "DNA mismatch repair: functions and mechanisms". Chemical Reviews. 106 (2): 302–23. doi:10.1021/cr0404794. PMID 16464007.
- Joseph N, Duppatla V, Rao DN (2006). Prokaryotic DNA mismatch repair. Progress in Nucleic Acid Research and Molecular Biology. Vol. 81. pp. 1–49. doi:10.1016/S0079-6603(06)81001-9. ISBN 9780125400817. PMID 16891168.
- Yang W (August 2000). "Structure and function of mismatch repair proteins". Mutation Research. 460 (3–4): 245–56. doi:10.1016/s0921-8777(00)00030-6. PMID 10946232.
- Griffiths JF, Gilbert WM, Lewontin RC, Wessler SR, Suzuki DT, Miller JH (2004). An introduction to genetic analysis (8th ed.). New York, NY: Freeman. ISBN 978-0-7167-4939-4.
- Kunkel TA, Erie DA (2005). "DNA mismatch repair". Annual Review of Biochemistry. 74: 681–710. doi:10.1146/annurev.biochem.74.082803.133243. PMID 15952900.
- Friedberg EC, Walker GC, Siede W, Wood RD, Schultz RA, Ellenberger (2005). DNA repair and mutagenesis (2nd ed.). Washington, D.C.: ASM Press. ISBN 978-1-55581-319-2.
외부 링크
- DNA 복구
- DNA+Mismatch+미국 국립의학도서관의 의학 주제 제목(MeSH) 복구