견고성(진화)
Robustness (evolution)생물학적 시스템의 강건성(생물학적 또는 유전적 강건성이라고도[1] 함)은 동요나 불확실성 조건 하에서 시스템의 특정 특성이나 특성이 지속되는 것이다.[2][3] 개발의 강건성은 운하화라고 알려져 있다.[4][5] 관련된 동요의 종류에 따라, 강건성은 돌연변이, 환경, 재조합 또는 행동 강건성 등으로 분류될 수 있다.[6][7][8] 강건성은 많은 유전적 메커니즘과 분자적 메커니즘의 결합을 통해 달성되며 직접 또는 간접적 선택에 의해 진화할 수 있다. 몇몇 모델 시스템은 강건성과 그것의 진화적 결과를 실험적으로 연구하기 위해 개발되었다.
분류
돌연변이 강건성
돌연변이 강건성( 돌연변이 내성이라고도 함)은 돌연변이에도 불구하고 유기체의 표현형이 일정하게 유지되는 정도를 설명한다.[9] 강건성은 돌연변이를 유도하고 어떤 돌연변이가 같은 표현형, 기능 또는 체력을 유지하는지 측정함으로써 여러 게놈과[10][11] 개별 유전자에[12] 대해 경험적으로 측정할 수 있다. 보다 일반적으로 강건성은 돌연변이의 피트니스 효과 분포에서 중립 대역(즉 돌연변이의 다른 피트니스 빈도)에 해당한다. 지금까지 조사된 단백질은 약 66%의 돌연변이에 대한 내성을 보였다(즉, 돌연변이의 3분의 2가 중성이다).[13]
반대로, 유기체의 측정된 돌연변이 강건성은 매우 다양하다. 예를 들어, C.엘레건에서 포인트 변이의 95%는 검출할 수 있는 효과가[14] 없으며 심지어 대장균에서 단일 유전자 결핍의 90%도 치명적이지 않다.[15] 그러나 바이러스는 돌연변이의 20-40%만 견디기 때문에 돌연변이에 훨씬 더 민감하다.[10]
확률에 대한 강건성
분자 규모의 생물학적 과정은 본질적으로 확률적이다.[16] 그것들은 분자의 물리적 화학적 성질을 감안할 때 발생하는 확률적 사건들의 조합에서 나온다. 예를 들어, 유전자 발현은 본질적으로 시끄럽다. 이는 정확히 동일한 규제 상태의 두 셀이 서로 다른 mRNA 내용을 나타낼 것이라는 것을 의미한다.[17][18] mRNA 함량의[19] 세포군 수준 로그 정규 분포는 중앙 한계 정리 적용에서 유전자 발현 조절의 다단계 특성에 직접 따른다.[20]
환경 강건성
다양한 환경에서 한 조건에 대한 완벽한 적응은 다른 조건에 대한 적응의 희생으로 이루어질 수 있다. 결과적으로, 유기체에 대한 총 선택 압력은 그 환경에서 보낸 시간의 백분율로 가중된 모든 환경에 걸친 평균 선택이다. 따라서 가변적 환경은 표현형이나 피트니스(생물학)의 변화가 거의 없이 유기체가 광범위한 조건에 걸쳐 기능할 수 있는 환경적 강건성을 선택할 수 있다. 어떤 유기체들은 온도, 물의 가용성, 염분 또는 음식물의 가용성의 큰 변화를 견딜 수 있는 적응력을 보여준다. 특히 식물은 환경이 변하면 움직일 수 없기 때문에 환경 건전성을 달성하기 위한 메커니즘의 범위를 보여준다. 마찬가지로, 이것은 단백질에서 광범위한 용매, 이온 농도 또는 온도에 대한 내성으로 볼 수 있다.
유전적, 분자적, 세포적 원인
게놈은 환경 파괴와 불완전한 복제에 의해 변이되지만, 그들은 놀랄만한 내성을 보인다. 이것은 많은 다른 수준에서 모두 강건함에서 온다.
유기체 돌연변이 강건성
게놈의 견고함을 제공하는 많은 메커니즘이 있다. 예를 들어, 유전자 중복은 다중복사 유전자의 어떤 사본에서든 돌연변이의 영향을 감소시킨다.[21] 또한 대사 경로를 통한 유동성은 일반적으로 몇 단계만으로 제한되는데, 이는 많은 효소의 기능 변화가 건강에 거의 영향을 미치지 않는다는 것을 의미한다.[22][23] 마찬가지로 대사 네트워크는 많은 주요 대사물을 생산하기 위한 여러 가지 대체 경로를 가지고 있다.[24]
단백질 돌연변이 강건성
단백질 돌연변이 내성은 유전 코드의 구조와 단백질 구조 건전성의 두 가지 주요 특징의 산물이다.[25][26] 단백질은 많은 염기서열이 매우 유사한 구조 접힘으로 접힐 수 있기 때문에 돌연변이에 내성이 있다.[27] 단백질은 컨포머가 펼치거나 잘못 접힌 상태보다 에너지가 낮기 때문에 제한된 자연 순응의 앙상블을 채택한다(접힘의 ΔG.[28][29] 이것은 협력적 상호작용의 분산된 내부 네트워크(수소성, 극성 및 공밸런트)에 의해 달성된다.[30] 단백질 구조 건전성은 단일 돌연변이가 기능을 손상시킬 정도로 충분히 파괴적이기 때문에 발생한다. 부분적으로 접힌 단백질이 결합하여 크고 반복적이며 불용성인 단백질 섬유질과 덩어리를 형성할 수 있기 때문에 단백질도 집적을[31] 피하기 위해 진화해 왔다.[32] 단백질이 구조에서 집적성 베타시트 모티브의 노출을 줄이기 위해 부정적인 디자인 특징을 보인다는 증거가 있다.[33] 또한 대부분의 포인트 돌연변이가 유사한 아미노산(보수적)으로 이어질 수 있도록 유전자 코드 자체가 최적화될 수 있다는 일부 증거가 있다.[34][35] 이러한 요소들은 함께 많은 수의 중립적 돌연변이와 거의 중립적인 돌연변이를 포함하는 돌연변이의 건강 효과의 분포를 만든다.[12]
유전자 표현 강건성
배아 발달 중 유전자 발현을 시공간적으로 엄격하게 통제해야 완전한 기능 장기가 생긴다. 그러므로 유기체를 개발하는 것은 유전자 발현 확률로 인한 임의의 동요를 다루어야 한다.[36] 다변측정학자의 경우, 강화제 중복성을 통해 유전자 발현의 강건성을 달성할 수 있다. 이는 여러 엔핸서들이 제어하는 유전자의 발현이 동일한 규제 논리를 인코딩할 때 발생한다(즉, 동일한 전사 인자의 집합에 대한 결합 부위를 표시). Drosophila melanogaster에서는 그러한 불필요한 증류제를 종종 그림자 증류제라고 부른다.[37]
더욱이, 발달 상황에는 표현형 결과에 중요한 유전자 발현 타이밍이 있었고, 적절한 유전자 발현을 적시에 보장하기 위한 다양한 메커니즘이 존재한다.[36] 준비된 프로모터들은 RNA 중합효소 II 결합을 보여주는 전사적으로 비활성화된 프로모터들로, 빠른 유도에 대비한다.[38] 또한, 모든 전사 인자가 대상 부지를 압축된 이질 색소 침착으로 묶을 수 있는 것은 아니기 때문에, 선구자 전사인자(Zld 또는 FoxA 등)는 염색체를 열고 유전자 발현을 빠르게 유도할 수 있는 다른 전사 인자의 결합을 허용해야 한다. 개방적인 비활동적인 증류주들은 침착한 증류주라고 불린다.[39]
세포 경쟁은 모자이크 미닛이 야생형 배경에서 세포(리보솜 단백질을 조절하는 것)를 돌연변이시키는 드로소필라에서[40] 처음 설명한 현상이다. 이러한 현상은 초기 생쥐 배아에서도 일어나는데, 이 배아에서는 높은 수준의 myc를 발현하는 세포들이 낮은 수준의 myc 발현을 보이는 이웃을 적극적으로 죽인다. 이것은 균질하게 높은 수준의 마이크를 초래한다.[41][42]
발육성 패터닝 강건성
프랑스 국기 모델에 의해 설명되는 것과 같은 패터닝 메커니즘은 여러 가지 수준(모포겐의 확산의 생산과 확률, 수용기의 생산, 신호 캐스케이드의 확률 등)에서 동요될 수 있다. 그러므로 패터닝은 본질적으로 시끄럽다. 따라서 이 소음과 유전적 섭동에 대한 강건성은 세포가 위치 정보를 정확하게 측정하도록 하기 위해 필요하다. 제브라피쉬 신경관과 안테로-후배성 패터닝에 대한 연구는 잡음 신호는 불완전한 세포 분화를 초래하고 나중에 잘못 배치된 세포의 전분화, 이동 또는 세포 사망에 의해 교정된다는 것을 보여주었다.[43][44][45]
또한 신호 경로의 구조(또는 위상)는 유전적 동요에 대한 강건성에 중요한 역할을 하는 것으로 입증되었다.[46] 자기 강화의 열화는 오랫동안 시스템 생물학에서 강건성의 한 예였다.[47] 마찬가지로, 많은 종에서 도르소벤트럴 패터닝의 강건성은 BMP 신호에 관련된 균형 잡힌 폐쇄-분해 메커니즘에서 나온다.[48][49][50]
진화적 결과
유기체는 끊임없이 유전적, 비유전적 섭동에 노출되기 때문에 표현형의 안정성을 확보하기 위해서는 강건성이 중요하다. 또한 돌연변이-선택 균형 하에서 돌연변이 강건성은 암호화된 유전적 변동을 모집단에 축적할 수 있다. 안정적인 환경에서 표현 중립적인 반면, 이러한 유전적 차이는 환경에 의존하는 방식의 특성 차이(진화 캐패시턴스 참조)로 밝혀질 수 있어 가변 환경에 노출된 모집단의 유전적 표현형식이 더 많이 표현될 수 있다.[51]
건장하다는 것은 진화적으로 안정된 전략으로서 총체적인 체력을 희생시키더라도 선호될 수 있다(가장 평탄한 사람들의 생존이라고도 한다).[52] 피트니스 풍경의 높지만 좁은 산꼭대기는 높은 체력에는 방해가 되지만 대부분의 돌연변이는 엄청난 체력 손실로 이어지기 때문에 낮은 강건성을 가지고 있다. 높은 돌연변이 비율은 더 낮지만 더 넓은 피트니스 피크의 인구를 선호할 수 있다. 기능 감소가 건강에 더 해를 끼치기 때문에 보다 중요한 생물학적 시스템도 건전성에 대한 선택의 폭이 넓어질 수 있다.[53] 변이성 강건성은 이론적인 바이러스성 진폐 형성을 위한 하나의 동인으로 생각된다.
긴급 돌연변이 강건성
자연선택은 건전성을 위해 직간접적으로 선택할 수 있다. 돌연변이 발생률이 높고 인구 규모가 클 경우, 덜 건실한 변종이 생존하는 돌연변이 후손이 적기 때문에 인구는 중립적 네트워크의 밀접하게 연결된 지역으로 이동할 것으로 예측된다.[54] 이러한 방식으로 돌연변이 강건성을 직접적으로 증가시키는 작용을 할 수 있는 선택조건은 제한적이며, 따라서 그러한 선택은 개체 수가 많고 돌연변이 발생률이 높은 소수의 바이러스[55] 및 미생물에만[56] 한정된다고 생각된다. 그러한 긴급한 강건성은 시토크롬 P450s와[57] B-락타마제의 실험적인 진화에서 관찰되었다.[58] 반대로, 돌연변이 강건성은 환경 동요에 대한 강건성을 위한 자연 선택의 부산물로 진화할 수 있다.[59][60][61][62][63]
견고성 및 진화 가능성
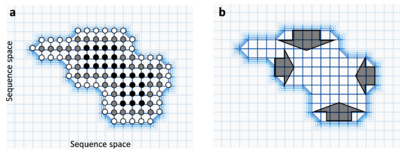
돌연변이 강건성은 단일 유전자형에 대한 뚜렷한 유전적 표현형식의 돌연변이 접근성을 감소시키고 유전적으로 다양한 모집단 내에서 선택적 차이를 감소시키기 때문에 진화가능성에 부정적인 영향을 미치는 것으로 생각되어 왔다.[citation needed] 그러나, 반직관적으로, 돌연변이에 대한 표현형 강건성은 실제로 장기간에 걸쳐 볼 때 유전형 표현형 적응 속도를 증가시킬 수 있다는 가설이 제기되었다.[64][65][66][67]
강건성이 무성의 개체군에서 진화가능성을 촉진하는 한 가지 가설은 피트니스 중립적 유전자형의 연결된 네트워크가 돌연변이 강건성을 야기한다는 것이다. 이는 짧은 시간, 긴 시간, 중성 돌연변이와 유전적 표류로 인해 새로운 유전적 표현형의 접근성을 감소시키는 반면, 개체군이 확산되는 원인이 된다. 유전자형 공간의 [68]더 큰 중립적 네트워크를 통해 이 유전적 다양성은 중성 네트워크의 서로 다른 지점에서 도달할 수 있는 더 많은 수의 뚜렷한 유전적 표현형식에 대한 돌연변이 접근을 모집단에 제공한다.[64][65][67][69][70][71][72] 그러나, 이 메커니즘은 단일 유전적 위치에 의존하는 표현형으로 제한될 수 있다; 다세대 형질의 경우, 무성 생물의 유전적 다양성은 진화 가능성을 크게 증가시키지 않는다.[73]
단백질의 경우, 강건성은 과도한 자유 에너지의 형태로 진화 가능성을 촉진한다.[74] 대부분의 돌연변이는 안정성을 감소시키기 때문에, 과도한 접힘 자유 에너지는 활동에 이롭지만 그렇지 않으면 단백질을 불안정하게 하는 돌연변이를 용인할 수 있다.
성 개체군에서 강건성은 높은 진화 잠재력을 가진 암호화된 유전적 변동의 축적을 이끈다.[75][76]
진화의 가능성은 대부분의 상황에서 높은 강건성과 낮은 강건성 사이에서 전환이 가능한 진화 캐패시턴스와 함께 가역성이 있을 때 높을 수 있다.[77]
방법 및 모델 시스템
건전성을 연구하기 위해 사용되어 온 많은 시스템들이 있다. 실리코 모델에서는 RNA 2차 구조, 단백질 격자 모델 또는 유전자 네트워크를 모델링하는 데 사용되어 왔다. 개별 유전자에 대한 실험 시스템으로는 시토크롬 P450,[57] B-락타마제,[58] RNA 중합효소,[13] LacI가[13] 모두 사용되어 왔다. 전체 유기체 건전성은 RNA 바이러스 적합성,[10] 박테리아 화학작용, 드로필라 피트니스,[15] 세그먼트 극성 네트워크, 신경유전 네트워크 및 뼈 형태유전성 단백질 그라데이션, C.선충 적합성[14] 및 외음 발달, 포유류 순환시계 등에서 조사되었다.[9]
참고 항목
참조
- ^ Kitano, Hiroaki (2004). "Biological robustness". Nature Reviews Genetics. 5 (11): 826–37. doi:10.1038/nrg1471. PMID 15520792. S2CID 7644586.
- ^ Stelling, Jörg; Sauer, Uwe; Szallasi, Zoltan; Doyle, Francis J.; Doyle, John (2004). "Robustness of Cellular Functions". Cell. 118 (6): 675–85. doi:10.1016/j.cell.2004.09.008. PMID 15369668. S2CID 14214978.
- ^ Félix, M-A; Wagner, A (2006). "Robustness and evolution: Concepts, insights and challenges from a developmental model system". Heredity. 100 (2): 132–40. doi:10.1038/sj.hdy.6800915. PMID 17167519.
- ^ Waddington, C. H. (1942). "Canalization of Development and the Inheritance of Acquired Characters". Nature. 150 (3811): 563–5. Bibcode:1942Natur.150..563W. doi:10.1038/150563a0. S2CID 4127926.
- ^ De Visser, JA; Hermisson, J; Wagner, GP; Ancel Meyers, L; Bagheri-Chaichian, H; Blanchard, JL; Chao, L; Cheverud, JM; et al. (2003). "Perspective: Evolution and detection of genetic robustness". Evolution; International Journal of Organic Evolution. 57 (9): 1959–72. doi:10.1111/j.0014-3820.2003.tb00377.x. JSTOR 3448871. PMID 14575319. S2CID 221736785.
- ^ Fernandez-Leon, Jose A. (2011). "Evolving cognitive-behavioural dependencies in situated agents for behavioural robustness". Biosystems. 106 (2–3): 94–110. doi:10.1016/j.biosystems.2011.07.003. PMID 21840371.
- ^ Fernandez-Leon, Jose A. (2011). "Behavioural robustness: A link between distributed mechanisms and coupled transient dynamics". Biosystems. 105 (1): 49–61. doi:10.1016/j.biosystems.2011.03.006. PMID 21466836.
- ^ Fernandez-Leon, Jose A. (2011). "Evolving experience-dependent robust behaviour in embodied agents". Biosystems. 103 (1): 45–56. doi:10.1016/j.biosystems.2010.09.010. PMID 20932875.
- ^ a b Wagner A (2005). Robustness and evolvability in living systems. Princeton Studies in Complexity. Princeton University Press. ISBN 0-691-12240-7.[페이지 필요]
- ^ a b c Sanjuán, R (Jun 27, 2010). "Mutational fitness effects in RNA and single-stranded DNA viruses: common patterns revealed by site-directed mutagenesis studies". Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences. 365 (1548): 1975–82. doi:10.1098/rstb.2010.0063. PMC 2880115. PMID 20478892.
- ^ Eyre-Walker, A; Keightley, PD (Aug 2007). "The distribution of fitness effects of new mutations". Nature Reviews Genetics. 8 (8): 610–8. doi:10.1038/nrg2146. PMID 17637733. S2CID 10868777.
- ^ a b Hietpas, RT; Jensen, JD; Bolon, DN (May 10, 2011). "Experimental illumination of a fitness landscape". Proceedings of the National Academy of Sciences of the United States of America. 108 (19): 7896–901. Bibcode:2011PNAS..108.7896H. doi:10.1073/pnas.1016024108. PMC 3093508. PMID 21464309.
- ^ a b c Guo, HH; Choe, J; Loeb, LA (Jun 22, 2004). "Protein tolerance to random amino acid change". Proceedings of the National Academy of Sciences of the United States of America. 101 (25): 9205–10. Bibcode:2004PNAS..101.9205G. doi:10.1073/pnas.0403255101. PMC 438954. PMID 15197260.
- ^ a b Davies, E. K.; Peters, A. D.; Keightley, P. D. (10 September 1999). "High Frequency of Cryptic Deleterious Mutations in Caenorhabditis elegans". Science. 285 (5434): 1748–1751. doi:10.1126/science.285.5434.1748. PMID 10481013.
- ^ a b Baba, T; Ara, T; Hasegawa, M; Takai, Y; Okumura, Y; Baba, M; Datsenko, KA; Tomita, M; Wanner, BL; Mori, H (2006). "Construction of Escherichia coli K-12 in-frame, single-gene knockout mutants: the Keio collection". Molecular Systems Biology. 2 (1): 2006.0008. doi:10.1038/msb4100050. PMC 1681482. PMID 16738554.
- ^ Bressloff, Paul C. (2014-08-22). Stochastic processes in cell biology. Cham. ISBN 978-3-319-08488-6. OCLC 889941610.
- ^ Elowitz, M. B. (2002-08-16). "Stochastic Gene Expression in a Single Cell". Science. 297 (5584): 1183–1186. Bibcode:2002Sci...297.1183E. doi:10.1126/science.1070919. PMID 12183631. S2CID 10845628.
- ^ Blake, William J.; KÆrn, Mads; Cantor, Charles R.; Collins, J. J. (April 2003). "Noise in eukaryotic gene expression". Nature. 422 (6932): 633–637. Bibcode:2003Natur.422..633B. doi:10.1038/nature01546. PMID 12687005. S2CID 4347106.
- ^ Bengtsson, M.; Ståhlberg, A; Rorsman, P; Kubista, M (16 September 2005). "Gene expression profiling in single cells from the pancreatic islets of Langerhans reveals lognormal distribution of mRNA levels". Genome Research. 15 (10): 1388–1392. doi:10.1101/gr.3820805. PMC 1240081. PMID 16204192.
- ^ Beal, Jacob (1 June 2017). "Biochemical complexity drives log-normal variation in genetic expression". Engineering Biology. 1 (1): 55–60. doi:10.1049/enb.2017.0004. S2CID 31138796.
- ^ Gu, Z; Steinmetz, LM; Gu, X; Scharfe, C; Davis, RW; Li, WH (Jan 2, 2003). "Role of duplicate genes in genetic robustness against null mutations". Nature. 421 (6918): 63–6. Bibcode:2003Natur.421...63G. doi:10.1038/nature01198. PMID 12511954. S2CID 4348693.
- ^ Kauffman, Kenneth J; Prakash, Purusharth; Edwards, Jeremy S (October 2003). "Advances in flux balance analysis". Current Opinion in Biotechnology. 14 (5): 491–496. doi:10.1016/j.copbio.2003.08.001. PMID 14580578.
- ^ Nam, H; Lewis, NE; Lerman, JA; Lee, DH; Chang, RL; Kim, D; Palsson, BO (Aug 31, 2012). "Network context and selection in the evolution to enzyme specificity". Science. 337 (6098): 1101–4. Bibcode:2012Sci...337.1101N. doi:10.1126/science.1216861. PMC 3536066. PMID 22936779.
- ^ Krakauer, DC; Plotkin, JB (Feb 5, 2002). "Redundancy, antiredundancy, and the robustness of genomes". Proceedings of the National Academy of Sciences of the United States of America. 99 (3): 1405–9. Bibcode:2002PNAS...99.1405K. doi:10.1073/pnas.032668599. PMC 122203. PMID 11818563.
- ^ Taverna, DM; Goldstein, RA (Jan 18, 2002). "Why are proteins so robust to site mutations?". Journal of Molecular Biology. 315 (3): 479–84. doi:10.1006/jmbi.2001.5226. PMID 11786027.
- ^ Tokuriki, N; Tawfik, DS (Oct 2009). "Stability effects of mutations and protein evolvability". Current Opinion in Structural Biology. 19 (5): 596–604. doi:10.1016/j.sbi.2009.08.003. PMID 19765975.
- ^ Meyerguz, L; Kleinberg, J; Elber, R (Jul 10, 2007). "The network of sequence flow between protein structures". Proceedings of the National Academy of Sciences of the United States of America. 104 (28): 11627–32. Bibcode:2007PNAS..10411627M. doi:10.1073/pnas.0701393104. PMC 1913895. PMID 17596339.
- ^ Karplus, M (Jun 17, 2011). "Behind the folding funnel diagram". Nature Chemical Biology. 7 (7): 401–4. doi:10.1038/nchembio.565. PMID 21685880.
- ^ Tokuriki, N; Stricher, F; Schymkowitz, J; Serrano, L; Tawfik, DS (Jun 22, 2007). "The stability effects of protein mutations appear to be universally distributed". Journal of Molecular Biology. 369 (5): 1318–32. doi:10.1016/j.jmb.2007.03.069. PMID 17482644.
- ^ Shakhnovich, BE; Deeds, E; Delisi, C; Shakhnovich, E (Mar 2005). "Protein structure and evolutionary history determine sequence space topology". Genome Research. 15 (3): 385–92. arXiv:q-bio/0404040. doi:10.1101/gr.3133605. PMC 551565. PMID 15741509.
- ^ Monsellier, E; Chiti, F (Aug 2007). "Prevention of amyloid-like aggregation as a driving force of protein evolution". EMBO Reports. 8 (8): 737–42. doi:10.1038/sj.embor.7401034. PMC 1978086. PMID 17668004.
- ^ Fink, AL (1998). "Protein aggregation: folding aggregates, inclusion bodies and amyloid". Folding & Design. 3 (1): R9–23. doi:10.1016/s1359-0278(98)00002-9. PMID 9502314.
- ^ Richardson, JS; Richardson, DC (Mar 5, 2002). "Natural beta-sheet proteins use negative design to avoid edge-to-edge aggregation". Proceedings of the National Academy of Sciences of the United States of America. 99 (5): 2754–9. Bibcode:2002PNAS...99.2754R. doi:10.1073/pnas.052706099. PMC 122420. PMID 11880627.
- ^ Müller MM, Allison JR, Hongdilokkul N, Gaillon L, Kast P, van Gunsteren WF, Marlière P, Hilvert D (2013). "Directed evolution of a model primordial enzyme provides insights into the development of the genetic code". PLOS Genetics. 9 (1): e1003187. doi:10.1371/journal.pgen.1003187. PMC 3536711. PMID 23300488.

- ^ Firnberg, E; Ostermeier, M (Aug 2013). "The genetic code constrains yet facilitates Darwinian evolution". Nucleic Acids Research. 41 (15): 7420–8. doi:10.1093/nar/gkt536. PMC 3753648. PMID 23754851.
- ^ a b Lagha, Mounia; Bothma, Jacques P.; Levine, Michael (2012). "Mechanisms of transcriptional precision in animal development". Trends in Genetics. 28 (8): 409–416. doi:10.1016/j.tig.2012.03.006. PMC 4257495. PMID 22513408.
- ^ Perry, Michael W.; Boettiger, Alistair N.; Bothma, Jacques P.; Levine, Michael (2010). "Shadow Enhancers Foster Robustness of Drosophila Gastrulation". Current Biology. 20 (17): 1562–1567. doi:10.1016/j.cub.2010.07.043. PMC 4257487. PMID 20797865.
- ^ Zeitlinger, Julia; Stark, Alexander; Kellis, Manolis; Hong, Joung-Woo; Nechaev, Sergei; Adelman, Karen; Levine, Michael; Young, Richard A (11 November 2007). "RNA polymerase stalling at developmental control genes in the Drosophila melanogaster embryo". Nature Genetics. 39 (12): 1512–1516. doi:10.1038/ng.2007.26. PMC 2824921. PMID 17994019.
- ^ Nien, Chung-Yi; Liang, Hsiao-Lan; Butcher, Stephen; Sun, Yujia; Fu, Shengbo; Gocha, Tenzin; Kirov, Nikolai; Manak, J. Robert; Rushlow, Christine; Barsh, Gregory S. (20 October 2011). "Temporal Coordination of Gene Networks by Zelda in the Early Drosophila Embryo". PLOS Genetics. 7 (10): e1002339. doi:10.1371/journal.pgen.1002339. PMC 3197689. PMID 22028675.
- ^ Morata, Ginés; Ripoll, Pedro (1975). "Minutes: Mutants of Drosophila autonomously affecting cell division rate". Developmental Biology. 42 (2): 211–221. doi:10.1016/0012-1606(75)90330-9. PMID 1116643.
- ^ Clavería, Cristina; Giovinazzo, Giovanna; Sierra, Rocío; Torres, Miguel (10 July 2013). "Myc-driven endogenous cell competition in the early mammalian embryo". Nature. 500 (7460): 39–44. Bibcode:2013Natur.500...39C. doi:10.1038/nature12389. PMID 23842495. S2CID 4414411.
- ^ Sancho, Margarida; Di-Gregorio, Aida; George, Nancy; Pozzi, Sara; Sánchez, Juan Miguel; Pernaute, Barbara; Rodríguez, Tristan A. (2013). "Competitive Interactions Eliminate Unfit Embryonic Stem Cells at the Onset of Differentiation". Developmental Cell. 26 (1): 19–30. doi:10.1016/j.devcel.2013.06.012. PMC 3714589. PMID 23867226.
- ^ Xiong, Fengzhu; Tentner, Andrea R.; Huang, Peng; Gelas, Arnaud; Mosaliganti, Kishore R.; Souhait, Lydie; Rannou, Nicolas; Swinburne, Ian A.; Obholzer, Nikolaus D.; Cowgill, Paul D.; Schier, Alexander F. (2013). "Specified Neural Progenitors Sort to Form Sharp Domains after Noisy Shh Signaling". Cell. 153 (3): 550–561. doi:10.1016/j.cell.2013.03.023. PMC 3674856. PMID 23622240.
- ^ Akieda, Yuki; Ogamino, Shohei; Furuie, Hironobu; Ishitani, Shizuka; Akiyoshi, Ryutaro; Nogami, Jumpei; Masuda, Takamasa; Shimizu, Nobuyuki; Ohkawa, Yasuyuki; Ishitani, Tohru (17 October 2019). "Cell competition corrects noisy Wnt morphogen gradients to achieve robust patterning in the zebrafish embryo". Nature Communications. 10 (1): 4710. Bibcode:2019NatCo..10.4710A. doi:10.1038/s41467-019-12609-4. PMC 6797755. PMID 31624259.
- ^ Kesavan, Gokul; Hans, Stefan; Brand, Michael (2019). "Cell-fate plasticity, adhesion and cell sorting complementarily establish a sharp midbrain-hindbrain boundary". bioRxiv. 147 (11). doi:10.1101/857870. PMID 32439756.
- ^ Eldar, Avigdor; Rosin, Dalia; Shilo, Ben-Zion; Barkai, Naama (2003). "Self-Enhanced Ligand Degradation Underlies Robustness of Morphogen Gradients". Developmental Cell. 5 (4): 635–646. doi:10.1016/S1534-5807(03)00292-2. PMID 14536064.
- ^ Ibañes, Marta; Belmonte, Juan Carlos Izpisúa (25 March 2008). "Theoretical and experimental approaches to understand morphogen gradients". Molecular Systems Biology. 4 (1): 176. doi:10.1038/msb.2008.14. PMC 2290935. PMID 18364710.
- ^ Eldar, Avigdor; Dorfman, Ruslan; Weiss, Daniel; Ashe, Hilary; Shilo, Ben-Zion; Barkai, Naama (September 2002). "Robustness of the BMP morphogen gradient in Drosophila embryonic patterning". Nature. 419 (6904): 304–308. Bibcode:2002Natur.419..304E. doi:10.1038/nature01061. PMID 12239569. S2CID 4397746.
- ^ Genikhovich, Grigory; Fried, Patrick; Prünster, M. Mandela; Schinko, Johannes B.; Gilles, Anna F.; Fredman, David; Meier, Karin; Iber, Dagmar; Technau, Ulrich (2015). "Axis Patterning by BMPs: Cnidarian Network Reveals Evolutionary Constraints". Cell Reports. 10 (10): 1646–1654. doi:10.1016/j.celrep.2015.02.035. PMC 4460265. PMID 25772352.
- ^ Al Asafen, Hadel; Bandodkar, Prasad U.; Carrell-Noel, Sophia; Reeves, Gregory T. (2019-08-19). "Robustness of the Dorsal morphogen gradient with respect to morphogen dosage". doi:10.1101/739292.
{{cite journal}}: Cite 저널은 필요로 한다.journal=(도움말) - ^ Masel J Siegal ML (2009). "Robustness: mechanisms and consequences". Trends in Genetics. 25 (9): 395–403. doi:10.1016/j.tig.2009.07.005. PMC 2770586. PMID 19717203.
- ^ Wilke, CO; Wang, JL; Ofria, C; Lenski, RE; Adami, C (Jul 19, 2001). "Evolution of digital organisms at high mutation rates leads to survival of the flattest". Nature. 412 (6844): 331–3. Bibcode:2001Natur.412..331W. doi:10.1038/35085569. PMID 11460163. S2CID 1482925.
- ^ Van Dijk; Van Mourik, Simon; Van Ham, Roeland C. H. J.; et al. (2012). "Mutational Robustness of Gene Regulatory Networks". PLOS ONE. 7 (1): e30591. Bibcode:2012PLoSO...730591V. doi:10.1371/journal.pone.0030591. PMC 3266278. PMID 22295094.

- ^ van Nimwegen E, Crutchfield JP, Huynen M (1999). "Neutral evolution of mutational robustness". PNAS. 96 (17): 9716–9720. arXiv:adap-org/9903006. Bibcode:1999PNAS...96.9716V. doi:10.1073/pnas.96.17.9716. PMC 22276. PMID 10449760.
- ^ Montville R, Froissart R, Remold SK, Tenaillon O, Turner PE (2005). "Evolution of mutational robustness in an RNA virus". PLOS Biology. 3 (11): 1939–1945. doi:10.1371/journal.pbio.0030381. PMC 1275523. PMID 16248678.

- ^ Masel J, Maughan H; Maughan (2007). "Mutations Leading to Loss of Sporulation Ability in Bacillus subtilis Are Sufficiently Frequent to Favor Genetic Canalization". Genetics. 175 (1): 453–457. doi:10.1534/genetics.106.065201. PMC 1775008. PMID 17110488.
- ^ a b Bloom, JD; Lu, Z; Chen, D; Raval, A; Venturelli, OS; Arnold, FH (Jul 17, 2007). "Evolution favors protein mutational robustness in sufficiently large populations". BMC Biology. 5: 29. arXiv:0704.1885. Bibcode:2007arXiv0704.1885B. doi:10.1186/1741-7007-5-29. PMC 1995189. PMID 17640347.

- ^ a b Bershtein, Shimon; Goldin, Korina; Tawfik, Dan S. (June 2008). "Intense Neutral Drifts Yield Robust and Evolvable Consensus Proteins". Journal of Molecular Biology. 379 (5): 1029–1044. doi:10.1016/j.jmb.2008.04.024. PMID 18495157.
- ^ Meiklejohn CD, Hartl DL (2002). "A single mode of canalization". Trends in Ecology & Evolution. 17 (10): 468–473. doi:10.1016/s0169-5347(02)02596-x.
- ^ Ancel LW, Fontana W (2000). "Plasticity, evolvability, and modularity in RNA". Journal of Experimental Zoology. 288 (3): 242–283. doi:10.1002/1097-010X(20001015)288:3<242::AID-JEZ5>3.0.CO;2-O. PMID 11069142.
- ^ Szöllősi GJ, Derényi I (2009). "Congruent Evolution of Genetic and Environmental Robustness in Micro-RNA". Molecular Biology & Evolution. 26 (4): 867–874. arXiv:0810.2658. doi:10.1093/molbev/msp008. PMID 19168567. S2CID 8935948.
- ^ Wagner GP, Booth G, Bagheri-Chaichian H (1997). "A population genetic theory of canalization". Evolution. 51 (2): 329–347. doi:10.2307/2411105. JSTOR 2411105. PMID 28565347.
- ^ Lehner B (2010). "Genes Confer Similar Robustness to Environmental, Stochastic, and Genetic Perturbations in Yeast". PLOS ONE. 5 (2): 468–473. Bibcode:2010PLoSO...5.9035L. doi:10.1371/journal.pone.0009035. PMC 2815791. PMID 20140261.

- ^ a b Draghi, Jeremy A.; Parsons, Todd L.; Wagner, Günter P.; Plotkin, Joshua B. (2010). "Mutational robustness can facilitate adaptation". Nature. 463 (7279): 353–5. Bibcode:2010Natur.463..353D. doi:10.1038/nature08694. PMC 3071712. PMID 20090752.
- ^ a b Wagner, A. (2008). "Robustness and evolvability: A paradox resolved". Proceedings of the Royal Society B: Biological Sciences. 275 (1630): 91–100. doi:10.1098/rspb.2007.1137. JSTOR 25249473. PMC 2562401. PMID 17971325.
- ^ Masel J, Trotter MV (2010). "Robustness and evolvability". Trends in Genetics. 26 (9): 406–414. doi:10.1016/j.tig.2010.06.002. PMC 3198833. PMID 20598394.
- ^ a b Aldana; Balleza, E; Kauffman, S; Resendiz, O; et al. (2007). "Robustness and evolvability in genetic regulatory networks". Journal of Theoretical Biology. 245 (3): 433–448. doi:10.1016/j.jtbi.2006.10.027. PMID 17188715.
- ^ Ebner, Marc; Shackleton, Mark; Shipman, Rob (2001). "How neutral networks influence evolvability". Complexity. 7 (2): 19–33. Bibcode:2001Cmplx...7b..19E. doi:10.1002/cplx.10021.
- ^ Babajide; Hofacker, I. L.; Sippl, M. J.; Stadler, P. F.; et al. (1997). "Neutral networks in protein space: A computational study based on knowledge-based potentials of mean force". Folding & Design. 2 (5): 261–269. doi:10.1016/s1359-0278(97)00037-0. PMID 9261065.
- ^ van Nimwegen and Crutchfield (2000). "Metastable evolutionary dynamics: Crossing fitness barriers or escaping via neutral paths?". Bulletin of Mathematical Biology. 62 (5): 799–848. arXiv:adap-org/9907002. doi:10.1006/bulm.2000.0180. PMID 11016086. S2CID 17930325.
- ^ Ciliberti; et al. (2007). "Innovation and robustness in complex regulatory gene networks". Proceedings of the National Academy of Sciences, USA. 104 (34): 13591–13596. Bibcode:2007PNAS..10413591C. doi:10.1073/pnas.0705396104. PMC 1959426. PMID 17690244.
- ^ Andreas Wagner (2008). "Neutralism and selectionism: a network-based reconciliation" (PDF). Nature Reviews Genetics. 9 (12): 965–974. doi:10.1038/nrg2473. PMID 18957969. S2CID 10651547.
- ^ Rajon, E.; Masel, J. (18 January 2013). "Compensatory Evolution and the Origins of Innovations". Genetics. 193 (4): 1209–1220. doi:10.1534/genetics.112.148627. PMC 3606098. PMID 23335336.
- ^ Bloom; et al. (2006). "Protein stability promotes evolvability". Proceedings of the National Academy of Sciences. 103 (15): 5869–74. Bibcode:2006PNAS..103.5869B. doi:10.1073/pnas.0510098103. PMC 1458665. PMID 16581913.
- ^ Waddington CH (1957). The strategy of the genes. George Allen & Unwin.
- ^ Masel, J. (30 December 2005). "Cryptic Genetic Variation Is Enriched for Potential Adaptations". Genetics. 172 (3): 1985–1991. doi:10.1534/genetics.105.051649. PMC 1456269. PMID 16387877.
- ^ Masel, J (Sep 30, 2013). "Q&A: Evolutionary capacitance". BMC Biology. 11: 103. doi:10.1186/1741-7007-11-103. PMC 3849687. PMID 24228631.





