진화발달생물학
Evolutionary developmental biology진화발달생물학(information development biology, evo-devo)은 서로 다른 유기체의 발달과정을 비교하여 발달과정이 어떻게 진화했는지를 유추하는 생물학 연구 분야다.
이 분야는 발생학이 미스터리에 직면했던 19세기 초반부터 성장했다. 동물학자들은 분자 수준에서 배아 발달이 어떻게 제어되는지 알지 못했다.찰스 다윈은 유사한 배아를 갖는 것은 공통의 조상을 내포하고 있지만 1970년대까지 거의 진전이 없었다고 지적했다.그리고 나서, 마침내 DNA 재조합 기술은 발생학과 분자 유전학을 결합시켰다.초기 주요 발견은 광범위한 진핵생물의 발달을 조절하는 동종 유전자의 발견이었다.
그 분야는 복수의 핵심 진화 개념으로 구성되어 있다.하나는 곤충, 척추동물, 두족류 연체동물 등의 서로 다른 장기들이 오랫동안 따로 진화했다고 여겨져 온 에보데보 유전자 툴킷의 팩스-6와 같은 유사한 유전자에 의해 조절된다는 발견인 깊은 호몰로지다.이 유전자들은 고대의 것으로서, 식물들 사이에서 매우 보존되어 있다; 그것들은 배아를 형성하고 궁극적으로 유기체의 신체 계획을 형성하는 시공간에서 패턴을 생성한다.또 다른 것은 종들이 효소를 위한 코딩과 같은 구조적인 유전자에 큰 차이가 없다는 것이다; 다른 것은 유전자 발현이 툴킷 유전자에 의해 조절되는 방식이다.이러한 유전자들은 배아의 다른 부분과 다른 발달 단계에서 여러 번 재사용되고, 변하지 않고, 여러 번 다른 조절 유전자를 형성하여, 다른 조절 유전자와 구조 유전자를 정확한 패턴으로 켜고 끄는 복잡한 계단식 대조군을 형성한다.이 다중의 늑막성 재사용을 통해 이러한 유전자들이 보존성이 높은 이유를 설명할 수 있는데, 어떤 변화도 자연 선택이 반대할 수 있는 많은 부정적인 결과를 초래할 것이기 때문이다.
새로운 형태학적 특징과 궁극적으로 새로운 종은 유전자가 새로운 패턴으로 표현될 때 또는 툴킷 유전자가 추가적인 기능을 획득할 때 툴킷의 변화에 의해 생성된다.또 다른 가능성은 후생유전학적 변화가 나중에 유전자 수준에서 통합된다는 네오-라마르크 이론인데, 이것은 다세포 생물 역사 초기에 중요한 것이었을지도 모른다.
역사
재점검
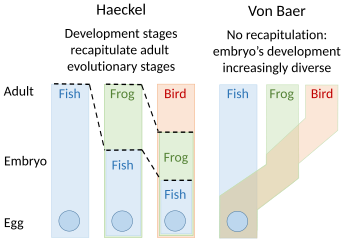
1824–26년 에티엔 세레스가 요한 프리드리히 메켈의 1808개 사상을 반영하여 진화발전에 대한 재현 이론을 제안하였다.그들은 '더 높은' 동물들의 배아가 일련의 단계를 거쳤거나 다시 요약했다고 주장했는데, 각각의 단계는 거대한 존재 사슬 아래쪽에 있는 동물과 닮았다.예를 들어, 인간 배아의 뇌는 처음에는 물고기의 뇌처럼 보였고, 그 다음에는 분명히 인간이 되기 전에 파충류, 새, 포유류의 뇌처럼 보였다.발생학자 칼 에른스트 폰 바어는 1828년 단일한 신체 계획에 근거하여 존재의 위대한 사슬에서와 같이 선형적인 순서가 없고 구조가 분화하는 에피게네시스 과정이 있다고 주장하면서 이에 반대했다.대신에 폰 바어는 네 가지 뚜렷한 동물 신체 계획을 인정했다: 방사, 불가사리, 연체동물, 대합조개, 바닷가재, 그리고 물고기 같은 척추동물.동물학자들은 1866년에 Ernst Haeckel이 그것을 부활시켰지만, 당시 동물학자들은 대체로 재복제를 포기했다.[2][3][4][5][6]
진화 형태학
19세기 초부터 20세기 대부분까지 발생학은 미스터리에 직면했다.동물들은 알로부터 종종 비슷한 단계를 거쳐서 매우 다른 신체 계획을 가진 성인으로 발전하는 것으로 보였지만, 동물학자들은 배아 발달이 어떻게 분자 수준에서 조절되는지 거의 알지 못했고, 따라서 발달 과정이 어떻게 진화했는지에 대해서도 거의 알지 못했다.[7]찰스 다윈은 공유된 배아 구조가 공통의 조상을 내포하고 있다고 주장했다.그 예로 다윈은 1859년 저서 '종의 기원에 관하여'에서 새우와 같은 성충의 유충을 인용했는데, 이 유충의 성충은 다른 절지동물과 전혀 닮지 않았다; 린네우스와 쿠비에르는 그들을 연체동물로 분류했다.[8][9]다윈은 또한 튜니케이트 역시 연체동물이 아니라는 알렉산더 코왈롭스키의 발견에 주목했다. 그러나 유충 단계에서는 척추동물의 동등한 구조물과 동일한 세균 층에서 발달한 노토코드 및 인두막 슬립이 있었고, 따라서 그것들과 함께 화음으로 분류되어야 한다.[8][10]
따라서 19세기 동물학은 태아학을 진화과학으로 전환시켜, 태아의 세균층 사이의 동질성과 연결시켰다.프리츠 뮐러를 포함한 동물학자들은 태아 사이의 유전적 관계를 발견하기 위해 발생학의 사용을 제안했다.뮐러는 갑각류 유충이 나우플리우스 유충을 공유한다는 것을 증명하여 갑각류로 인식되지 않았던 여러 기생 종을 식별하였다.뮐러는 또한 자연 선택이 성인에게 하는 것처럼 유충에게도 작용해야 하며, 자연 선택으로부터 보호받으려면 유충 형태를 필요로 하는 재복제 거짓말을 해야 한다고 인식했다.[8]해켈은 1870년대에 배아 발달의 시기적 변화(히터로시)와 체내 위치의 변화(헤터로토피)가 보조에 비해 후손의 몸의 형태를 변화시킴으로써 진화를 촉진할 것이라고 주장하였다.에스토르의이런 생각들이 옳다고 보여지기까지는 1세기가 걸렸다.[11][12][13]1917년, D'Arcy Thompson은 동물의 모양에 관한 책을 저술했는데, 그는 진화론적 설명보다 기계적인 것을 선호했지만, 간단한 수학으로 위스트포드의 나선형 껍질의 각도와 같은 매개 변수의 작은 변화가 동물의 형태를 얼마나 급진적으로 변화시킬 수 있는지를 보여주었다.[14][15]그러나 다음 세기는 분자적 증거도 없이 진보가 정체되었다.[8]
20세기 초의 현대적 종합
로널드 피셔는 20세기 초의 이른바 현대적 종합에서 자연선택, 유전, 변동을 고집하는 다윈의 진화론과 그레고르 멘델의 유전 법칙을 진화생물학의 일관성 있는 구조로 불러 모았다.생물학자들은 유기체가 그 구성 요소 유전자의 직접적인 반영이라고 추측했다: 단백질을 위해 암호화된 유전자로 유기체의 몸을 만들었다.생화학적 경로(그리고 그들이 추정하는 새로운 종)는 이러한 유전자의 돌연변이를 통해 진화했다.그것은 간단하고 명확하며 거의 포괄적인 그림이었다: 그러나 발생학을 설명하지는 않았다.[8][16]
진화적 발생학자 개빈 드 비어는 1930년 저서 배아와 조상에서 진화적 발달 생물학을 기대했는데,[17] 성인의 청소년 특징 보존과 같은 [18]이질성에 의해 진화가 일어날 수 있다는 것을 보여줌으로써 진화적 발달 생물학을 기대했다.[11]드 비어는 배아가 잘 화석화되지 않기 때문에 화석 기록에 급격한 변화를 일으킬 수 있다고 주장했다.화석 기록의 공백이 다윈의 점진적 진화에 반대하는 주장으로 사용되어 왔기 때문에, 드 비어의 설명은 다윈의 입장을 지지했다.[19]그러나, de Beer에도 불구하고, 인구유전학이 어떻게 형태가 진화했는지에 대한 적절한 설명으로 보이기 때문에, 현대 합성은 유기체의 형태를 설명하기 위한 배아 발달을 대부분 무시했다.[20][21][a]
라크 피연산자
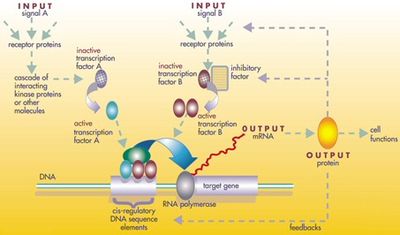
1961년 자크 모노드, 장 피에르 체취스, 프랑수아 제이콥은 대장균 박테리아에서 열상 피연산자를 발견했다.그것은 환경 자극에 의해 "전환"될 때만 제품이 만들어지도록 피드백 제어 루프에 배열된 유전자 군집이었다.이 제품들 중 하나는 설탕, 유당을 갈라놓는 효소였고, 유당 그 자체는 유전자를 바꾼 자극제였다.이는 박테리아처럼 작은 유기체에서도 유전자가 정밀한 통제의 대상이 된다는 것을 처음으로 보여준 계시였다.그 의미는 다른 많은 유전자들도 정교하게 조절된다는 것이었다.[23]
evo-devo의 탄생과 두 번째 합성
1977년, 유전학에서 재조합 DNA 기술이 등장하고, 프랑수아 제이콥의 팅커링에 의한 온토게니와 필로제니의 작품이 등장하면서 진화 및 발달 생물학에 대한 사고의 혁명이 시작되었다.굴드는 해켈의 진화적 발생학 해석을 잠재웠고, 제이콥은 대안 이론을 제시했다.[8]이것은 마침내 발생학뿐만 아니라 분자 유전학, 유전학, 에보-데보를 형성하는 진화 생물학까지 포함하는 두 번째 합성으로 이어졌다.[24][25][26][27]1978년 에드워드 B. 루이스는 드로소필라 초파리에서 배아 발달을 조절하는 유전자를 발견했는데, 모든 곤충이 무척추동물의 주요 식물 중 하나인 절지동물인 것처럼 말이다.[28]빌 맥기니스는 개구리, 새, 포유류와 같은 척추동물의 동물들, 그리고 후에 효모와 같은 곰팡이에서도 발견되었다.[29][30]모든 진핵생물에 걸쳐 발달을 통제하는 유전자들에는 분명히 강한 유사성이 있었다.[31]1980년 크리스티안 누슬린-볼하드와 에릭 위샤우스는 과실파리 배아의 분할 패턴을 만드는 데 도움이 되는 갭 유전자를 묘사했다.[32][33] 그들과 루이스는 1995년 노벨상을 수상하였다.[29][34]
후에 더 구체적인 유사점이 발견되었다: 예를 들어, 원위 없는 유전자는 1989년에 초파리의 맹장이나 팔다리의 발달,[35] 물고기의 지느러미, 닭의 날개, 해양 안네이드 벌레의 파라포디아, 음낭의 앰풀래와 사이펀, 그리고 성게의 관발 발달에 관여한다는 것이 발견되었다.이 유전자는 (약 6억 3천 5백만년 전에 시작된 에디아카란 시대 이전) 쌍방 동물의 마지막 공통 조상으로 거슬러 올라가며 고대의 것이 분명했다.에보데보는 모든 동물의 몸이 발달하는 동안 만들어지는 방법을 밝혀내기 시작했다.[36][37]
차체 구조 제어
딥 호몰로지
대략 다른 동물들의 구면 알은 해파리부터 바닷가재, 나비, 코끼리까지 독특한 형태를 만들어낸다.이러한 유기체들 중 다수는 콜라겐이나 효소와 같은 체형성 단백질에 대해 동일한 구조 유전자를 공유하고 있지만 생물학자들은 각 그룹의 동물들에게 고유한 발달 규칙이 있을 것으로 예상했었다.evo-devo의 놀라운 점은 신체의 형질이 다소 적은 비율의 유전자에 의해 조절되고 있으며, 이러한 규제 유전자는 모든 동물이 공유하는 고대의 것이라는 점이다.기린은 긴 목을 위한 유전자를 가지고 있지 않다. 코끼리가 큰 몸을 위한 유전자를 가지고 있는 것 이상이다.그들의 몸은 다른 특징들의 발달이 더 일찍 또는 나중에 시작되게 하고, 배아의 이 혹은 저 부분에서 발생하게 하고, 더 또는 더 적은 시간 동안 계속되도록 하는 전환 시스템에 의해 패턴화된다.[7]
배아 발달이 어떻게 조절되었는가에 대한 퍼즐은 과일파리 드로소필라 멜라노가스터를 모범생물로 삼아 풀리기 시작했다.태아 발생에 대한 단계별 제어는 배아에서 발현된 유전자에 의해 만들어진 특정 유형의 단백질에 다른 색깔의 형광 염료를 부착함으로써 시각화되었다.[7]녹색 형광 단백질과 같은 염료는 일반적으로 과일파리 단백질에 특화된 항체에 부착되어 살아 있는 배아에서 그 단백질이 언제 어디서 나타났는지 정확한 지표를 형성했다.[38]

1994년 월터 게링은 그러한 기술을 사용하여 초파리의 눈을 형성하는데 필수적인 팩스-6 유전자가 쥐와 인간의 눈 형성 유전자와 정확히 일치한다는 것을 발견했다.같은 유전자는 오징어, 두족류 연체동물과 같은 많은 다른 동물군에서 빠르게 발견되었다.에른스트 메이르를 포함한 생물학자들은 서로 다른 종류의 눈의 해부학적 구조가 매우 다양하기 때문에 적어도 40번은 동물 왕국에서 눈이 발생했다고 믿었다.[7]예를 들어 초파리의 복합눈은 수백 개의 작은 렌즈 구조(옴마티디아)로 이루어져 있는데, 사람의 눈은 시신경이 눈에 들어오는 맹점을 가지고 있고, 신경섬유가 망막 표면 위를 흐르기 때문에 빛은 망막의 검출기 세포에 도달하기 전에 신경섬유층을 통과해야 하기 때문에 구조 I.효과적으로 "하향"; 대조적으로, 두팔로포드 눈은 망막을 가지고 있고, 그 다음 신경 섬유 층을 가지고 있고, 그 다음 눈의 벽은 "올바른 방향"을 가지고 있다.[39]그러나 팍스-6의 증거는 같은 유전자가 이 모든 동물들의 눈의 발달을 통제하여, 모두 공통의 조상으로부터 진화했음을 암시하는 것이었다.[7]고대 유전자들은 수백만 년의 진화를 통해 보존되어 유사한 기능을 위한 서로 다른 구조를 만들어 냈으며, 한때는 순수하게 유사하다고 생각되었던 구조물들 사이의 깊은 호몰로지(homeology)를 보여주었다.[40][41]이 개념은 후에 발생의[42] 진화에까지 확대되었고 진화 생물학에서 동질학의 의미에 대한 급진적인 수정을 초래했다.[40][41][43]
진 툴킷
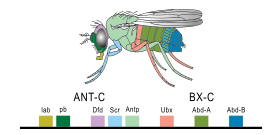
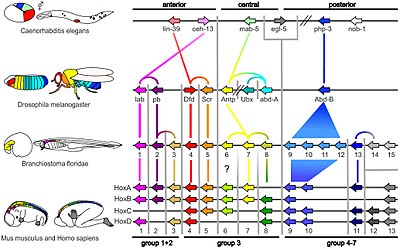
유기체의 게놈에 있는 유전자의 작은 부분은 유기체의 발달을 조절한다.이 유전자들은 발달-유전적 도구 키트라고 불린다.그들은 필라 사이에서 보존성이 매우 뛰어나며, 이것은 그들이 오래되고 널리 분리된 동물군에서 매우 유사하다는 것을 의미한다.툴킷 유전자의 배치 차이는 신체 계획과 신체 부위의 수, 정체성, 패턴에 영향을 미친다.대부분의 툴킷 유전자는 신호 전달 경로의 일부분이다: 그것들은 전사인자, 세포 접착 단백질, 세포 표면 수용체 단백질과 결합하는 신호 리간드를 인코딩하고 배아를 통해 확산되는 형태균을 분비한다.이 모든 것들이 배아에서 미분화 세포의 운명을 규정하는 데 도움이 된다.그들은 함께 배아를 형성하는 시공간에서 패턴을 생성하고, 궁극적으로 유기체의 신체 계획을 형성한다.가장 중요한 도구 키트 유전자 중에는 Hox 유전자가 있다.이러한 전사 인자는 다른 툴킷 유전자에서도 발견되는 호모박스 단백질 결합 DNA 모티브를 포함하고 있으며, 앞뒤 축을 따라 신체의 기본 패턴을 생성한다.[43]홉스 유전자는 뱀의 많은 척추뼈와 같은 반복적인 부분이 발달한 배아나 유충에서 어디서 자랄지 결정한다.[7]이미 언급된 팍스-6는 전형적인 툴킷 유전자다.[44]다른 툴킷 유전자가 식물체 계획 수립에 관여하고 있지만,[45] 식물에서도 홈박스 유전자가 발견되어 모든 진핵생물에 공통적이라는 것을 암시한다.[46][47][48]
배아의 조절 네트워크
규제 도구 키트의 단백질 생산물은 복제와 수정에 의해서가 아니라 플리오트로피의 복잡한 모자이크에 의해 재사용되며, 많은 독립적인 발달 과정에 변하지 않고 적용되어 많은 다른 신체 구조에 패턴을 부여한다.[43]이러한 플리엇틱 툴킷 유전자의 로키는 크고 복잡하며 모듈화된 시스 조절 요소를 가지고 있다.예를 들어 과실파리의 비완성 로도신 유전자는 단지 수백 개의 염기쌍을 가진 시스 조절 원소를 가지고 있는 반면, 플리엇성 무안경 시스 조절 영역은 7000개가 넘는 염기쌍에 6개의 시스 조절 원소를 포함하고 있다.[43]관련된 규제 네트워크는 종종 매우 크다.각 규제 단백질은 "수백 개의 시스 규제 요소"를 제어한다.예를 들어 67개의 과실파리 전사 인자가 각각 평균 124개의 표적 유전자에 의해 제어된다.[43]이 모든 복잡성은 배아의 발달에 관여하는 유전자가 정확한 시간과 정확한 장소에서 켜지거나 꺼지도록 한다.이러한 유전자 중 일부는 구조적인 것으로서 배아의 효소, 조직, 기관을 직접 형성한다.그러나 다른 많은 것들은 그 자체로 규제 유전자들이기 때문에, 스위치를 켜는 것은 종종 발달하는 배아에서 발달 과정을 차례로 켜는 것을 포함하는 정확한 타이밍의 전환이다.[43]
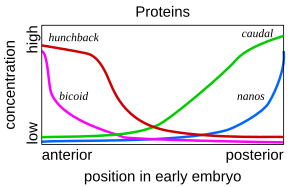
이러한 계단식 규제 네트워크는 과수파리 배아 개발에서 자세히 연구되어 왔다.어린 배아는 럭비공처럼 타원형의 모양을 하고 있다.소수의 유전자는 배아의 긴 축을 따라 농도 구배를 설정하는 메신저 RNA를 생산한다.초기 배아에서, 바이코이드와 꼽추 유전자는 앞쪽 끝 가까이에 높은 농도에 있고, 미래의 머리와 흉부에 패턴을 준다; 코우달과 나노 유전자는 뒤쪽 끝 근처에 높은 농도에 있고, 가장 뒤쪽 복부 부분에 패턴을 준다.이러한 유전자의 효과는 상호 작용한다. 예를 들어, 바이코이드 단백질은 카우달의 메신저 RNA의 번역을 차단하기 때문에 카우달 단백질 농도는 전단에서 낮아진다.Caudal은 나중에 파리의 가장 뒷부분을 만드는 유전자를 사용하지만, 파리가 가장 집중된 뒤쪽 끝부분에서만 유전자를 바꾼다.[49][50]
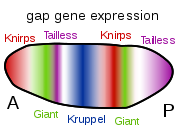
바이코이드, 훈시백, 카우달 단백질은 차례로 거인, 너프, 크뤼펠, 태음인 등 갭 유전자의 전사를 줄무늬 패턴으로 조절해 분할이 되는 1단계 구조를 만든다.[32]이들로부터 나온 단백질은 차례로 쌍규칙 유전자를 조절하는데, 다음 단계에서는 배아의 긴 축을 가로질러 7개의 띠를 설치한다.마지막으로, Engrelated와 같은 세그먼트 극성 유전자는 7개 밴드의 각각을 둘로 분할하여 14개의 미래 세그먼트를 생성했다.[49][50]
이 과정은 툴킷 유전자 배열의 정확한 보존을 설명하며, 이로 인해 서로 다른 동물에서 툴킷 단백질의 깊은 호몰로지 및 기능적 동등성이 초래되었다(예를 들어, 마우스 단백질이 과일파리 발달을 제어하는 경우).전사 인자와 시스 조절 원소 또는 신호 단백질과 수용체들의 상호작용은 다중 사용을 통해 잠기게 되어 거의 모든 돌연변이를 유해하게 만들고 따라서 자연 선택에 의해 제거된다.[43]
신기함의 기원
더욱 놀랍고, 어쩌면, 진화적 발달 생물학에 대한 최근의 연구의 반직관적(신 다윈의 관점에서) 결과들 가운데, 많은 식물들에 걸친 유기체들의 신체 계획과 형태학의 다양성이 유전자의 배열 수준에서 반드시 다양성에 반영되는 것은 아니며, 그 발달된 것을 포함하여, 유전자의 배열의 수준에서도 반드시 다양성에 반영되는 것은 아니라는 것이다.정신 유전자 도구 키트와 다른 유전자들이 발달에 관여했다.사실, 존 게르하트와 마크 키르스치너도 지적했듯이, 명백한 역설은 "우리가 가장 기대했던 다양성을 찾는 곳, 보존, 변화의 부족을 발견한다"는 것이다.[51]그렇다면 서로 다른 혈통들 사이에 관찰된 형태학적 신기함이 유전자 배열의 변화(변이에 의한 등)에서 오는 것이 아니라면, 어디에서 오는 것일까?유전자 조절의 돌연변이 주도의 변화로 인해 새로움이 발생할 수 있다.[43][52][53][54]
툴킷의 변형
도구 키트의 변화는 동물의 형태학적 진화의 큰 부분을 생성했을 수 있다.툴킷은 두 가지 방법으로 진화를 촉진할 수 있다.툴킷 유전자는 BMP 유전자에 의해 다윈의 큰 지느러미 부리가 확대되었을 때,[55] 또는 원위성으로 뱀이 다리를 잃었을 때 다른 파충류들이 사지를 계속 형성하던 곳에서 과소표현되거나 전혀 표현되지 않았을 때, 다른 패턴으로 표현할 수 있다.[56]또는 툴킷 유전자는 척추동물의 하악성,[57][58] 초파리의 다리와 더듬이,[59] 나비 날개의 눈구멍 패턴과 같은 다양한 구조를 제어하는 원위성이 없는 동일한 유전자의 많은 기능에서 볼 수 있듯이 새로운 기능을 획득할 수 있다.[60]도구상자 유전자의 작은 변화가 신체 구조에 중대한 변화를 일으킬 수 있다는 점에서, 그들은 종종 융합적으로 또는 병렬적으로 동일한 기능을 가능하게 했다. 원위-비스는 뮐러 모사인 헬리코니우스 에라토와 헬리코니우스 멜포메네 나비에서 날개 패턴을 생성하는데, 이것은 헬리코니우스 에라토와 헬리코니우스 멜포메네(Melpomene)이다.이른바 촉진 변형에서 이들의 날개 패턴은 서로 다른 진화적 사건에서 발생했지만 동일한 유전자에 의해 제어된다.[61][62]발전적 변화는 명분화에 직접적으로 기여할 수 있다.[63]
후생유전학적 변화의 통합
진화적 혁신은 때때로 유전자 조절 또는 표현형 생성의 후생유전적 변경과 함께 라마르크식 스타일로 시작되며, 이후 유전자 수준의 변화에 의해 통합될 수 있다.후생유전학적 변화에는 가역성 메틸화에 의한 DNA의 수정과 더불어 [64]개발 메커니즘의 내재적 가소성으로 인한 물리적 및 기타 환경적 영향에 의한 미프로그래밍된 유기체의 리마운딩이 포함된다.[65]생물학자 스튜어트 A. 뉴먼과 게르트 B. 뮐러는 다세포 생물의 역사 초기에 유기체가 현대 유기체보다 후생유전적 결정의 이 두 번째 범주에 더 취약해 초기 거시적 진화의 기초를 제공한다고 제안해 왔다.[66]
발달편향
특정 라인의 개발은 주어진 궤적이나 표현형 쪽으로 긍정적으로 편향되거나 [b]또는 특정 유형의 변화를 생산하는 것에서 부정적으로 편향될 수 있다. 즉, 절대적일 수 있거나(변화는 항상 생성되지 않거나 전혀 생성되지 않음) 상대적일 수 있다.진화의 그러한 방향에 대한 증거는 아무리 얻기 어렵고 또한 다양화를 제한하는 개발 제약에서 비롯될 수 있다.[68]예를 들어, 위스트포드에서 달팽이 모양의 껍질은 항상 길이와 직경이 모두 자라는 관으로 만들어진다; 선택으로 인해 이 제약조건들 안에서 평평한 나선형, 카우리와 키 큰 터렛 나선형과 같은 매우 다양한 껍데기 모양을 만들어냈다.지네 중에서 리토비오모르파는 성인으로서 항상 15개의 트렁크 세그먼트를 가지고 있는데, 아마도 홀수 수의 트렁크 세그먼트에 대한 발달 편향의 결과일 것이다.또 다른 지네 순서인 지오필로모르파(Geopipe order, Geophilomorpha)는, 27년과 191년 사이에 종별로 분할의 수가 달라지지만, 그 수가 항상 홀수여서, 이것이 절대적인 제약이 된다; 그 범위에 있는 거의 모든 홀수들은 한 종이나 다른 종에 의해 점유되고 있다.[67][69][70]
생태 진화 발전 생물학
생태 진화 발전 생물학은[c] 발달 생물학과 생태학의 연구를 통합하여 진화 이론과의 관계를 고찰한다.[71]연구자들은 발달 가소성, 후생유전, 유전적 동화, 틈새 건설, 공생과 같은 개념과 메커니즘을 연구한다.[72][73]
참고 항목
- 절지동물 머리 문제
- 세포신호
- 진화발전(저널)
- 인간의 진화적 발달 생물학
- Just So Storys (진화 발전 생물학자들이 보는 바와 같이)
- 식물 진화 발달 생물학
- 재도약 이론
메모들
참조
- ^ Richardson and Keuck (2002), "Haeckel's ABC of evolution and development", Biological Reviews of the Cambridge Philosophical Society, 77 (4): 495–528, CiteSeerX 10.1.1.578.2749, doi:10.1017/s1464793102005948, PMID 12475051, S2CID 23494485
{{citation}}: CS1 maint: 작성자 매개변수(링크) 사용 p.516 - ^ O'Connell, Lindsey (10 July 2013). "The Meckel-Serres Conception of Recapitulation". The Embryo Project Encyclopedia. Retrieved 10 October 2016.
- ^ Desmond, Adrian J. (1989). The politics of evolution: morphology, medicine, and reform in radical London. Chicago: University of Chicago Press. pp. 53–53, 86–88, 337–340, 490–491. ISBN 978-0-226-14374-3.
- ^ 시코드 2003, 페이지 252–253
- ^ Bowler, Peter J. (2003). Evolution: the history of an idea. Berkeley: University of California Press. pp. 120–128, 190–191, 208. ISBN 978-0-520-23693-6.
- ^ 2003년 424페이지 512
- ^ a b c d e f Carroll, Sean B. "The Origins of Form". Natural History. Retrieved 9 October 2016.
Biologists could say, with confidence, that forms change, and that natural selection is an important force for change. Yet they could say nothing about how that change is accomplished. How bodies or body parts change, or how new structures arise, remained complete mysteries.
- ^ a b c d e f Gilbert, Scott F. (2003). "The morphogenesis of evolutionary developmental biology" (PDF). International Journal of Developmental Biology. 47 (7–8): 467–477. PMID 14756322.
- ^ Darwin, Charles (1859). On the Origin of Species. London: John Murray. pp. 439–440. ISBN 978-0-8014-1319-3.
Cirripedes afford a good instance of this: even the illustrious Cuvier did not perceive that a barnacle was, as it certainly is, a crustacean; but a glance at the larva shows this to be the case in an unmistakeable manner.
- ^ Richmond, Marsha (January 2007). "Darwin's Study of the Cirripedia". Darwin Online. Retrieved 9 October 2016.
- ^ a b Hall, B. K. (2003). "Evo-Devo: evolutionary developmental mechanisms". International Journal of Developmental Biology. 47 (7–8): 491–495. PMID 14756324.
- ^ Ridley, Mark (2003). Evolution. Wiley-Blackwell. ISBN 978-1-4051-0345-9.
- ^ Gould 1977, 페이지 221–222.
- ^ Ball, Philip (7 February 2013). "In retrospect: On Growth and Form". Nature. 494 (32–33): 32–33. Bibcode:2013Natur.494...32B. doi:10.1038/494032a. S2CID 205076253.
- ^ Shalizi, Cosma. "Review: The Self-Made Tapestry by Philip Ball". University of Michigan. Retrieved 14 October 2016.
- ^ Bock, Walter J. (July 1981). "Reviewed Work: The Evolutionary Synthesis. Perspectives on the Unification of Biology". The Auk. 98 (3): 644–646. ISSN 0004-8038. JSTOR 4086148.
- ^ Held, Lewis I. (2014). How the Snake Lost its Legs. Curious Tales from the Frontier of Evo-Devo. Cambridge University Press. p. 67. ISBN 978-1-107-62139-8.
- ^ Gould 1977, 페이지 221–222
- ^ Ingo Brigandt (2006). "Homology and heterochrony: the evolutionary embryologist Gavin Rylands de Beer (1899-1972)" (PDF). Journal of Experimental Zoology. 306B (4): 317–328. doi:10.1002/jez.b.21100. PMID 16506229.
- ^ Gilbert, S. F.; Opitz, J. M.; Raff, R. A. (1996). "Resynthesizing evolutionary and developmental biology". Developmental Biology. 173 (2): 357–372. doi:10.1006/dbio.1996.0032. PMID 8605997.
- ^ Adams, M. (1991). Warren, L.; Koprowski, H. (eds.). Through the looking glass: The evolution of Soviet Darwinism. New Perspectives in Evolution. Liss/Wiley. pp. 37–63.
- ^ 1996, 페이지 153
- ^ Monod, Jacques; Changeux, J.P.; Jacob, François (1963). "Allosteric proteins and cellular control systems". Journal of Molecular Biology. 6 (4): 306–329. doi:10.1016/S0022-2836(63)80091-1. PMID 13936070.
- ^ Gilbert, S.F.; Opitz, J.M.; Raff, R.A. (1996). "Resynthesizing Evolutionary and Developmental Biology". Developmental Biology. 173 (2): 357–372. doi:10.1006/dbio.1996.0032. PMID 8605997.
- ^ Müller, G. B. (2007). "Evo–devo: extending the evolutionary synthesis". Nature Reviews Genetics. 8 (12): 943–949. doi:10.1038/nrg2219. PMID 17984972. S2CID 19264907.
- ^ Goodman, C. S.; Coughlin, B. C. (2000). Goodman, C. S.; Coughlin B. S. (eds.). "Special feature: The evolution of evo-devo biology". Proceedings of the National Academy of Sciences. 97 (9): 4424–4456. Bibcode:2000PNAS...97.4424G. doi:10.1073/pnas.97.9.4424. PMC 18255. PMID 10781035.
- ^ Müller GB and Newman SA (Eds.) (2005). "Special issue: Evolutionary Innovation and Morphological Novelty". Journal of Experimental Zoology Part B. 304B (6): 485–631. doi:10.1002/jez.b.21080. PMID 16252267. Archived from the original on 11 December 2012.
- ^ Palmer, R.A. (2004). "Symmetry breaking and the evolution of development". Science. 306 (5697): 828–833. Bibcode:2004Sci...306..828P. CiteSeerX 10.1.1.631.4256. doi:10.1126/science.1103707. PMID 15514148. S2CID 32054147.
- ^ a b Winchester, Guil (2004). "Edward B. Lewis 1918-2004" (PDF). Current Biology (published 21 September 2004). 14 (18): R740–742. doi:10.1016/j.cub.2004.09.007. PMID 15380080. S2CID 32648995.
- ^ Bürglin, Thomas R. "The Homeobox Page". Karolinska Institutet. Retrieved 13 October 2016.
- ^ Holland, P.W. (2013). "Evolution of homeobox genes". Wiley Interdiscip Rev Dev Biol. 2 (1): 31–45. doi:10.1002/wdev.78. PMID 23799629. S2CID 44396110.
Homeobox genes are found in almost all eukaryotes, and have diversified into 11 gene classes and over 100 gene families in animal evolution, and 10 to 14 gene classes in plants.
- ^ a b Nüsslein-Volhard, C.; Wieschaus, E. (October 1980). "Mutations affecting segment number and polarity in Drosophila". Nature. 287 (5785): 795–801. Bibcode:1980Natur.287..795N. doi:10.1038/287795a0. PMID 6776413. S2CID 4337658.
- ^ Arthur, Wallace (14 February 2002). "The emerging conceptual framework of evolutionary developmental biology". Nature. 415 (6873): 757–764. Bibcode:2002Natur.415..757A. doi:10.1038/415757a. PMID 11845200. S2CID 4432164.
- ^ "Eric Wieschaus and Christiane Nüsslein-Volhard: Collaborating to Find Developmental Genes". iBiology. Retrieved 13 October 2016.
- ^ Cohen, S. M.; Jurgens, G. (1989). "Proximal-distal pattern formation in Drosophila: cell autonomous requirement for Distal-less activity in limb development". EMBO J. 8 (7): 2045–2055. doi:10.1002/j.1460-2075.1989.tb03613.x. PMC 401088. PMID 16453891.
- ^ Carroll, Sean B. (2006) [2005]. Endless Forms Most Beautiful: The New Science of Evo Devo and the Making of the Animal Kingdom. Weidenfeld & Nicolson [Norton]. pp. 63–70. ISBN 978-0-297-85094-6.
- ^ Panganiban, G.; Irvine, S. M.; Lowe, C.; Roehl, H.; Corley, L. S.; Sherbon, B.; Grenier, J. K.; Fallon, J. F.; Kimble, J.; Walker, M.; Wray, G. A.; Swalla, B. J.; Martindale, M. Q.; Carroll, S. B. (1997). "The origin and evolution of animal appendages". Proceedings of the National Academy of Sciences of the United States of America. 94 (10): 5162–5166. Bibcode:1997PNAS...94.5162P. doi:10.1073/pnas.94.10.5162. PMC 24649. PMID 9144208.
- ^ "Fluorescent Probes". ThermoFisher Scientific. 2015. Retrieved 12 October 2016.
- ^ Land, M. F.; Fernald, R. D. (1992). "The evolution of eyes". Annual Review of Neuroscience. 15: 1–29. doi:10.1146/annurev.ne.15.030192.000245. PMID 1575438.
- ^ a b Tomarev, Stanislav I.; Callaerts, Patrick; Kos, Lidia; Zinovieva, Rina; Halder, Georg; Gehring, Walter; Piatigorsky, Joram (1997). "Squid Pax-6 and eye development". Proceedings of the National Academy of Sciences. 94 (6): 2421–2426. Bibcode:1997PNAS...94.2421T. doi:10.1073/pnas.94.6.2421. PMC 20103. PMID 9122210.
- ^ a b Pichaud, Franck; Desplan, Claude (August 2002). "Pax genes and eye organogenesis". Current Opinion in Genetics & Development. 12 (4): 430–434. doi:10.1016/S0959-437X(02)00321-0. PMID 12100888.
- ^ Drost, Hajk-Georg; Janitza, Philipp; Grosse, Ivo; Quint, Marcel (2017). "Cross-kingdom comparison of the developmental hourglass". Current Opinion in Genetics & Development. 45: 69–75. doi:10.1016/j.gde.2017.03.003. PMID 28347942.
- ^ a b c d e f g h Carroll, Sean B. (2008). "Evo-Devo and an Expanding Evolutionary Synthesis: A Genetic Theory of Morphological Evolution". Cell. 134 (1): 25–36. doi:10.1016/j.cell.2008.06.030. PMID 18614008. S2CID 2513041.
- ^ Xu, P.X.; Woo, I.; Her, H.; Beier, D. R.; Maas, R. L. (1997). "Mouse Eya homologues of the Drosophila eyes absent gene require Pax6 for expression in lens and nasal placode". Development. 124 (1): 219–231. doi:10.1242/dev.124.1.219. PMID 9006082.
- ^ Quint, Marcel; Drost, Hajk-Georg; Gabel, Alexander; Ullrich, Kristian Karsten; Bönn, Markus; Grosse, Ivo (4 October 2012). "A transcriptomic hourglass in plant embryogenesis". Nature. 490 (7418): 98–101. Bibcode:2012Natur.490...98Q. doi:10.1038/nature11394. ISSN 0028-0836. PMID 22951968. S2CID 4404460.
- ^ Mukherjee, K.; Brocchieri, L.; Bürglin, T.R. (December 2009). "A comprehensive classification and evolutionary analysis of plant homeobox genes". Molecular Biology and Evolution. 26 (12): 2775–94. doi:10.1093/molbev/msp201. PMC 2775110. PMID 19734295.
- ^ Bürglin, T.R. (November 1997). "Analysis of TALE superclass homeobox genes (MEIS, PBC, KNOX, Iroquois, TGIF) reveals a novel domain conserved between plants and animals". Nucleic Acids Research. 25 (21): 4173–80. doi:10.1093/nar/25.21.4173. PMC 147054. PMID 9336443.
{{cite journal}}: CS1 maint: 작성자 매개변수 사용(링크) - ^ Derelle, R.; Lopez, P.; Le Guyader, H.; Manuel, M. (2007). "Homeodomain proteins belong to the ancestral molecular toolkit of eukaryotes". Evolution & Development. 9 (3): 212–9. doi:10.1111/j.1525-142X.2007.00153.x. PMID 17501745. S2CID 9530210.
- ^ a b Russel, Peter (2010). iGenetics: a molecular approach. Pearson Education. pp. 564–571. ISBN 978-0-321-56976-9.
- ^ a b Rivera-Pomar, Rolando; Jackle, Herbert (1996). "From gradients to stripes in Drosophila embryogenesis: Filling in the gaps". Trends in Genetics. 12 (11): 478–483. doi:10.1016/0168-9525(96)10044-5. PMID 8973159.
- ^ Gerhart, John; Kirschner, Marc (1997). Cells, Embryos and Evolution. Blackwell Science. ISBN 978-0-86542-574-3.
- ^ Carroll, Sean B.; Grenier, Jennifer K.; Weatherbee, Scott D. (2005). From DNA to Diversity: Molecular Genetics and the Evolution of Animal Design — Second Edition. Blackwell Publishing. ISBN 978-1-4051-1950-4.
- ^ Carroll, Sean B. (2000). "Endless forms: the evolution of gene regulation and morphological diversity". Cell. 101 (6): 577–80. doi:10.1016/S0092-8674(00)80868-5. PMID 10892643. S2CID 12375030.
- ^ Moczek, Armin P.; et al. (2015). "The Significance and Scope of Evolutionary Developmental Biology: A Vision for the 21st Century" (PDF). Evolution & Development. 17 (3): 198–219. doi:10.1111/ede.12125. hdl:2027.42/111768. PMID 25963198. S2CID 9652129. Archived from the original (PDF) on 17 April 2016. Retrieved 27 November 2015.
- ^ Abzhanov, A.; Protas, M.; Grant, B.R.; Grant, P.R.; Tabin, C.J. (2004). "Bmp4 and Morphological Variation of Beaks in Darwin's Finches". Science. 305 (5689): 1462–1465. Bibcode:2004Sci...305.1462A. doi:10.1126/science.1098095. PMID 15353802. S2CID 17226774.
- ^ Cohn, M.J.; Tickle, C. (1999). "Developmental basis of limblessness and axial patterning in snakes". Nature. 399 (6735): 474–479. Bibcode:1999Natur.399..474C. doi:10.1038/20944. PMID 10365960. S2CID 4309833.
- ^ Beverdam, A.; Merlo, G.R.; Paleari, L.; Mantero, S.; Genova, F.; Barbieri, O.; Janvier, P.; Levi, G. (August 2002). "Jaw Transformation With Gain of Symmetry After DLX5/DLX6 Inactivation: Mirror of the Past?" (PDF). Genesis. 34 (4): 221–227. doi:10.1002/gene.10156. hdl:2318/87307. PMID 12434331. S2CID 19592597.
- ^ Depew, M.J.; Lufkin, T.; Rubenstein, J.L. (October 2002). "Specification of jaw subdivisions by DLX genes". Science. 298 (5592): 381–385. doi:10.1126/science.1075703. PMID 12193642. S2CID 10274300.
- ^ Panganiban, Grace; Rubenstein, John L. R. (2002). "Developmental functions of the Distal-less/Dlx homeobox genes". Development. 129 (19): 4371–4386. doi:10.1242/dev.129.19.4371. PMID 12223397.
- ^ Beldade, P.; Brakefield, P.M.; Long, A.D. (2002). "Contribution of Distal-less to quantitative variation in butterfly eyespots". Nature. 415 (6869): 315–318. doi:10.1038/415315a. PMID 11797007. S2CID 4430563.
- ^ Gerhart, John; Kirschner, Marc (2007). "The theory of facilitated variation". Proceedings of the National Academy of Sciences. 104 (suppl1): 8582–8589. Bibcode:2007PNAS..104.8582G. doi:10.1073/pnas.0701035104. PMC 1876433. PMID 17494755.
- ^ Baxter, S.W.; Papa, R.; Chamberlain, N.; Humphray, S.J.; Joron, M.; Morrison, C.; ffrench-Constant, R.H.; McMillan, W.O.; Jiggins, C.D. (2008). "Convergent Evolution in the Genetic Basis of Mullerian Mimicry in Heliconius Butterflies". Genetics. 180 (3): 1567–1577. doi:10.1534/genetics.107.082982. PMC 2581958. PMID 18791259.
- ^ Pennisi, E. (2002). "Evolutionary Biology:Evo-Devo Enthusiasts Get Down to Details". Science. 298 (5595): 953–955. doi:10.1126/science.298.5595.953. PMID 12411686. S2CID 154023266.
- ^ Jablonka, Eva; Lamb, Marion (1995). Epigenetic Inheritance and Evolution: The Lamarckian Dimension. Oxford, New York: Oxford University Press. ISBN 978-0-19-854063-2.
- ^ West-Eberhard, Mary Jane (2003). Developmental plasticity and evolution. New York: Oxford University Press. ISBN 978-0-19-512235-0.
- ^ Müller, Gerd B.; Newman, Stuart A., eds. (2003). Origination of Organismal Form: Beyond the Gene in Developmental and Evolutionary Biology. MIT Press.
- ^ a b Arthur, W. (July 2001). "Developmental drive: an important determinant of the direction of phenotypic evolution". Evolution & Development. 3 (4): 271–278. doi:10.1046/j.1525-142x.2001.003004271.x. PMID 11478524. S2CID 41698287.
- ^ Drost, Hajk-Georg; Janitza, Philipp; Grosse, Ivo; Quint, Marcel (2017). "Cross-kingdom comparison of the developmental hourglass". Current Opinion in Genetics & Development. 45: 69–75. doi:10.1016/j.gde.2017.03.003. PMID 28347942.
- ^ Arthur, W. (October 2002). "The interaction between developmental bias and natural selection: from centipede segments to a general hypothesis". Heredity. 89 (4): 239–246. doi:10.1038/sj.hdy.6800139. PMID 12242638.
- ^ Chipman, Ariel D.; Arthur, Wallace; Akam, Michael (July 2004). "A Double Segment Periodicity Underlies Segment Generation in Centipede Development". Current Biology. 14 (14): 1250–1255. doi:10.1016/j.cub.2004.07.026. PMID 15268854. S2CID 2371623.
- ^ Abouheif, E.; Favé, M.-J.; Ibarrarán-Viniegra, A. S.; Lesoway, M. P.; Rafiqi, A. M.; Rajakumar, R. (2014). "Eco-Evo-Devo: The Time Has Come". In Landry, C.R.; Aubin-Horth, N. (eds.). Ecological Genomics. Ecological Genomics: Ecology and the Evolution of Genes and Genomes. Advances in Experimental Medicine and Biology. Vol. 781. pp. 107–125. doi:10.1007/978-94-007-7347-9_6. ISBN 978-94-007-7346-2. PMID 24277297.
- ^ Schlichting, C.D. (2009). "An Uneven Guide to Eco-Devo". BioScience. 59 (11): 1000–1001. doi:10.1525/bio.2009.59.11.12. S2CID 116886911.
- ^ Gilbert, S. F.; Bosch, T. C. G.; Ledón-Rettig, C. (2015). "Eco-Evo-Devo: developmental symbiosis and developmental plasticity as evolutionary agents". Nature Reviews Genetics. 16 (10): 611–622. doi:10.1038/nrg3982. PMID 26370902. S2CID 205486234.
원천
- Gould, Stephen Jay (1977). Ontogeny and Phylogeny. Cambridge, MA: Belknap Press of Harvard University Press. ISBN 978-0-674-63940-9. LCCN 76045765. OCLC 2508336.
- Secord, James A. (2003). Victorian sensation: the extraordinary publication, reception, and secret authorship of Vestiges of the natural history of creation. Chicago: University of Chicago Press. ISBN 978-0-226-74410-0.
- Smocovitis, Vassiliki Betty (1996). Unifying Biology: The Evolutionary Synthesis and Evolutionary Biology. Journal of the History of Biology. Vol. 25. Princeton, NJ: Princeton University Press. pp. 1–65. doi:10.1007/BF01947504. ISBN 978-0-691-03343-3. LCCN 96005605. OCLC 34411399. PMID 11623198. S2CID 189833728.