히가시모나스아루기노사
Pseudomonas aeruginosa| 히가시모나스아루기노사 | |
|---|---|
 | |
| 혈액 한천에 사는 P. Aeruginosa 군락 | |
| 과학적 분류 | |
| 도메인: | 박테리아 |
| 문: | 슈도모나도타속 |
| 클래스: | 감마프로테오박테리아 |
| 주문: | 슈도모나데스속 |
| 패밀리: | 슈도모나과 |
| 속: | 슈도모나스속 |
| 종류: | 아오시노사 |
| 이항명 | |
| 히가시모나스아루기노사 (슈뢰터 1872) 미굴라 1900 | |
| 동의어 | |
| |
Pseudomonas aeruginosa는 [1][2]인간을 포함한 식물과 동물에게 질병을 일으킬 수 있는 캡슐화된 그램 음성, 호기성 혐기성 막대 모양의 일반적인 박테리아이다.의학적으로 매우 중요한 종인 P. aeruginosa는 다제내성 병원체로, 그 보편성, 본질적으로 진보된 항생제 내성 메커니즘, 그리고 인공호흡기 관련 폐렴과 다양한 패혈증 신드롬과 같은 병원 후천성 감염과 같은 심각한 질병과의 연관성으로 알려져 있다.
기존의 질병이나 상태, 특히 낭포성 섬유증과 외상성 화상 중에 심각한 감염이 종종 발생하는 한, 이 유기체는 기회주의자로 간주됩니다.그것은 일반적으로 면역 결핍에 영향을 미치지만 또한 온수욕조 모낭염에서와 같이 면역 결핍제를 감염시킬 수 있다.P. Aeruginosa 감염은 항생제에 대한 내성이 있기 때문에 치료가 어려울 수 있습니다.더 발전된 항생제 요법이 필요할 때 역효과가 발생할 수 있다.
구연산염, 카탈라아제, 산화효소 양성입니다.그것은 토양, 물, 피부 식물, 그리고 전 세계의 대부분의 인공 환경에서 발견됩니다.그것은 정상적인 대기뿐만 아니라 저산소 대기에서도 번성하여 많은 자연환경과 인공환경에서 서식해왔다.그것은 음식을 위해 광범위한 유기 물질을 사용합니다; 동물에서, 그것의 다용도는 유기체가 손상된 조직이나 면역력이 떨어진 사람들을 감염시킬 수 있게 합니다.이러한 감염의 증상은 전신성 염증과 패혈증이다.폐, 요로, 신장과 같은 중요한 신체 장기에 이러한 정착이 일어나면,[3] 그 결과는 치명적일 수 있다.이 박테리아는 습한 표면에서 번식하기 때문에 카테터를 포함한 의료 장비에서도 발견되며 병원과 클리닉에서 교차 감염을 일으킨다.그것은 또한 탄화수소를 분해할 수 있고 [4]기름 유출로 인한 타르볼과 기름을 분해하는 데 사용되어 왔다.P. Aeruginosa는 다른 주요 병원성 박테리아 종(예: 그램 양성 황색포도상구균과 스트렙토코커스 화원균)에 비해 매우 독성이 있지는 않지만, P. Aeruginosa는 광범위한 군집화가 가능하며 지속적인 생체막으로 [5]집적될 수 있다.
명명법
Pseudomonas라는 단어는 그리스어 pseud s(그리스어: οδ false, false)와 (라틴어: monas, 그리스어: μον,, 단일 단위)에서 "거짓 단위"를 의미합니다.줄기 단어 mon은 미생물학 역사에서 초기에 세균, 예를 들어 Monera [6]왕국을 지칭하기 위해 사용되었습니다.
아루기노사(aeruginosa)라는 이름은 라틴어로 버디그리스(verdigris)라는 뜻이며, 실험실 문화의 청록색을 가리킨다.이 청록색 색소는 피오시아닌(파란색)과 피오베르딘(녹색)의 두 가지 대사물의 조합으로,[6] 문화의 청록색 특성을 부여한다.1956년의 또 다른 주장은 아루기노사가 그리스어 접두사 ae-에서 유래한 것일 수 있으며, 접미사 ruginosa는 주름이나 [7]울퉁불퉁함을 의미한다.
'피오시아닌'과 '피오베르딘'이라는 이름은 그리스어에서 유래했으며, [8]'푸스', '푸른', '푸른'을 뜻하는 시아닌, '녹색'[citation needed]을 뜻하는 '베르딘'이 있다.따라서, "피시아니 박테리아"라는 용어는 특히 P. Aeruginosa 감염의 특징인 "푸른 고름"을 가리킨다.피오브딘은 피오브시아닌이 없는 형광-노란색이다.[citation needed]
생물학
게놈
Pseudomonas aeruginosa의 게놈은 5,500에서 6,000개의 열린 판독 프레임을 운반하는 비교적 큰 원형 염색체(5.5-6.8Mb)와 때로는 [9]변종에 따라 다양한 크기의 플라스미드로 구성됩니다.각기 다른 P. Aeruginosa 균주의 389개의 게놈을 비교한 결과, 단지 17.5%만이 공유되는 것으로 나타났다.게놈의 이 부분은 P. Aeruginosa 핵심 [10]게놈입니다.
| 변형률: | VRFPA04 | C3719 | PAO1 | PA14 | PACS2 |
|---|---|---|---|---|---|
| 염색체 크기(bp) | 6,818,030 | 6,222,097 | 6,264,404 | 6,537,648 | 6,492,423 |
| ORF | 5,939 | 5,578 | 5,571 | 5,905 | 5,676 |
비교 게놈 연구(2020년)는 Pseudomonas속의 완전한 게놈 494개를 분석했으며, 그 중 189개는 P. Aeruginosa [11]균주였다.연구 결과 단백질 수치와 GC 함량은 각각 [11]5500~7352(평균: 6192)와 65.6~66.9%(평균: 66.1%)로 나타났다.이 비교 분석을 통해 단백질의 30% 이상을 차지하는 1811개의 아루기노사 코어 단백질을 추가로 확인했다.후자의 분석에서 Aeruginosa 핵심 단백질의 높은 비율은 부분적으로 완전한 게놈의 사용에 기인할 수 있다.P. aeruginosa는 계통학적으로나 ANIm 값 면에서 매우 잘 정의된 단통류 종이지만 단백질 함량 면에서 놀라울 정도로 다양하여 여러 [11][12][13][14]분석에 따르면 매우 역동적인 보조 단백질체를 드러낸다.평균적으로 산업용 균주가 가장 큰 게놈을 가지고 있고, 그 다음으로 환경적 균주, 그리고 임상적 [11][15]분리주들이 있습니다.동일한 비교 연구(494개의 Pseudomonas 균주, 189개는 P. aeruginosa)에서 1811개의 P. aeruginosa 핵심 단백질 중 41개가 이 종에만 존재하고 다른 어떤 종에도 존재하지 않으며, (41개 중) 26개가 가설로 주석되어 있음을 확인했다.또, 적어도 188/189 P. Aeruginosa 균주에 19개의 직립단백질군이 존재하며,[citation needed] 이 균주의 다른 모든 균주에는 존재하지 않는다.
인구 구조
P. aeruginosa의 개체군은 세 개의 주요 계통으로 분류될 수 있으며, 모델 변종 PAO1, PA14, 그리고 더 다양한 PA7에 [16]의해 유전적으로 특징지어진다.
반면 P.aeruginosa은 일반적으로 기회주의적인 병원 균으로 생각해 본 몇가지 광범위한 복제 낭포성 섬유증 환자들 특히에서 UK,[17]DK2 Denmark,[18]과 AUST-02에 호주에서 또한 이전에 AES-로 알려진에서 주로 발견되는 리버풀 전염병 변형(LES)을 포함할 필요가 더 특수 병원 균, 추정되고 있습니다.2와 P2말의 [20][21]생식기관을 감염시키는 클론도 자주 볼 수 있다.[19]
대사
P. Aeruginosa는 부분적 또는 전체적 산소 고갈 조건에서 증식하는 데 잘 적응하기 때문에 통성 혐기성 동물이다.이 유기체는 질산염이나 아질산염을 말단 전자수용체로 하여 혐기성 성장을 달성할 수 있다.산소, 질산염, 아질산염이 없을 때 기질수준의 [22]인산화로 아르기닌과 피루브산을 발효시킬 수 있다.미세 혐기성 또는 혐기성 환경에 대한 적응은 예를 들어, 무코이드 박테리아 세포를 둘러싼 두꺼운 폐 점액층과 세균적으로 생성된 알긴산염의 두꺼운 층이 산소의 확산을 제한할 수 있는 낭포성 섬유증 및 일차 섬모성 이상에서 폐 감염되는 동안 P. aeruginosa의 특정 라이프스타일에 필수적이다.박테리아가 면역체계를 압도하는 바이오필름을 형성할 때까지 인체 내에서 무증상이 나타날 수 있다.이 바이오필름들은 낭포성 섬유증과 원발성 섬모성 운동장애를 가진 사람들의 폐에서 발견되어 [23][24][25][26][27][28]치명적일 수 있다.
세포협력
철분은 성장을 위한 영양 공급원으로서 철분에 의존합니다.하지만, 철은 환경에서 흔히 볼 수 없기 때문에 쉽게 접근할 수 없습니다.철은 대개 용해되지 않는 [29]철 형태로 발견됩니다.또한 철분 농도가 지나치게 높으면 독성이 있을 수 있습니다.이를 극복하고 철분 섭취를 조절하기 위해 철분을 [30]결합하고 운반하는 분비 분자인 시데로포어를 사용한다.그러나 이러한 철-시데로포 복합체는 특이하지 않다.시데로포어를 만든 박테리아가 반드시 철분 섭취의 직접적인 혜택을 받는 것은 아니다.오히려, 세포 집단의 모든 구성원들은 철-사이더로포 복합체에 동등하게 접근할 가능성이 있다.이러한 시데로포어를 효율적으로 생산할 수 있는 세포 집단의 구성원은 일반적으로 협력자라고 불리며, 시데로포어를 거의 또는 전혀 생산하지 않는 구성원은 종종 부정행위자라고 불립니다.연구에 따르면 협동조합과 부정행위자가 함께 성장하면 협동조합의 체력은 떨어지는 반면 부정행위자는 [31]체력이 증가하는 것으로 나타났습니다.철제한이 [32]증가함에 따라 피트니스 변화의 크기가 증가합니다.피트니스가 증가하면 부정행위자는 협력자를 능가할 수 있습니다. 이는 충분한 사이드로포어 생산 부족으로 인해 그룹의 피트니스가 전반적으로 감소합니다.이러한 관찰은 협력자와 부정행위자의 혼합이 P. aeruginosa의 [31]독성을 감소시킬 수 있다는 것을 암시한다.
효소
LigDs는 DNA 연결효소의 하위 패밀리를 형성합니다.이들은 모두 LigDom/리가수분해효소 도메인을 가지고 있지만, 많은 세균 LigDs는 별도의 중합효소 도메인/PolDoms 및 뉴클레아제 도메인/뉴클라아제 도메인/뉴클라아제 도메인/뉴클라아제 도메인/뉴클라아제 도메인/뉴클라아제 도메인/뉴클라아제 도메인/뉴클라아P. aeruginosa의 경우 뉴클레아제 도메인은 N-말단이고, 중합효소 도메인은 [33]단일 중심 리가아제 도메인의 확장인 C-말단이다.
병인 발생
면역력이 약해진 개인의 기회주의적 병원체인 P. aeruginosa는 일반적으로 기도, 요로, 화상 및 상처를 감염시키고 다른 혈액 [34]감염을 일으킨다.
| 감염 | 상세 및 공통 연관성 | 고위험군 |
|---|---|---|
| 폐렴 | 비만성 기관지 폐렴 | 낭포성 섬유증, 비CF 기관지 확장증 환자 |
| 패혈성 쇼크 | 보라색-검은색 피부병변과 관련된 신경절외반증 | 호중구증 환자 |
| 요로 감염 | 요로 카테터 삽입 | |
| 위장 감염 | 괴사성 장염 | 미숙아 및 호중구 감소성 암 환자 |
| 피부 및 연조직 감염 | 출혈과 괴사 | 화상 또는 상처 감염이 있는 사람 |
화상 손상과 외이도(외이도염) 감염의 가장 일반적인 원인이며 의료기기(예: 카테터)의 가장 빈번한 콜로나이저이다.의사도모나스는 오염되어 세척이 제대로 되지 않은 장비나 의료 [35]종사자의 손에 의해 전파될 수 있다.의사도모나스는 드문 경우지만, 여러 [37]연구에서 격리된 가장 일반적인 물질 중 하나인 인공호흡기 관련 폐렴뿐만 아니라 지역사회에 의해 획득되는 폐렴을 [36]일으킬 수 있다.피오시아닌은 박테리아의 독성 인자로 산화 스트레스에 의해 C. 엘레건에서 사망을 일으키는 것으로 알려져 있다.그러나 살리실산은 피시아닌 [38]생성을 억제할 수 있다.병원 감염자 10명 중 1명은 Pseudomonas에 의한 것입니다.낭포성 섬유증 환자도 [39]돌연변이의 결과로 세포막을 통과하는 염화물 이온 이동의 기능 상실로 인해 폐의 P. Aeruginosa 감염에 걸리기 쉽다.P. Aeruginosa는 또한 수질에 대한 적절하고 주기적인 주의 부족으로 인해 발생하는 "뜨거운 욕조 발진"(피부염)의 흔한 원인일 수 있다.이러한 박테리아는 뜨거운 욕조나 수영장 같은 습한 환경에서 잘 자라기 때문에 피부 발진이나 수영하는 사람의 [35]귀를 유발할 수 있다.슈도모나스는 또한 요골 각막절개 수술 환자들에게 수술 후 감염의 흔한 원인이다.그 유기체는 또한 피부병변 ectyma gangrenosum과 관련이 있다.P. Aeruginosa는 종종 발의 천자 상처를 수반하는 골수염과 관련이 있으며, 이는 테니스화에서 발견되는 폼 패딩을 통해 P. Aeruginosa를 직접 접종함으로써 발생하며, 당뇨병 환자들에게 더 높은 위험이 있다.
189개의 완전한 P. Aeruginosa 게놈을 포함한 494개의 유사모나스 게놈의 비교 게놈 분석 결과, 대부분의 P. Aeruginosa 균주가 공유하는 여러 단백질을 확인했지만 다른 분석된 유사모나스 [11]게놈에서는 관찰되지 않았다.CntL, CntM, PlcB, Acp1, MucE, SrfA, Tse1, Tsi2, Tse3 및 EsrC와 같은 이러한 Aeruginosa 특이 핵심 단백질은 이 종의 병원성에 [11]중요한 역할을 하는 것으로 알려져 있다.
독소
P. Aeruginosa는 디프테리아 독소와 마찬가지로 숙주 세포에서 ADP-리보실화를 통해 진핵신장인자 2를 비활성화하기 위해 독성인자 엑소톡신 A를 사용한다.신장인자 2가 없으면 진핵세포는 단백질을 합성하고 괴사시킬 수 없다.세포내 내용물의 방출은 면역적합환자의 면역학적 반응을 유도한다.또한 진핵세포의 혈장막을 분해하여 용융을 일으키는 엑조엔자임인 엑소유(Exoenzyme)를 사용한다.철분 취득 시데로포인 피오베르딘도 미토콘드리아에서 철분을 제거해 이 세포에 [40][41]손상을 입히는 독소 역할을 한다는 인식이 확산되고 있다.
페나진
페나진은 녹농균에 의해 생성되는 산화환원 활성 색소이다.이 색소는 쿼럼 감지, 독성 및 철분 [42]획득에 관여합니다.P. aeruginosa는 모두 생합성 경로를 통해 생성되는 여러 색소를 생산한다. 페나진-1-카르복사미드(PCA), 1-히드록시페나진, 5-메틸페나진-1-카르복실산 베타인, 피오시아닌 및 아에루기노신 A.페나진 생합성에는 phzA1B1C1D1E1F1G1과 phzA2B2C2D2F2G2의 [43][44][45]2개의 오퍼론이 관여한다.이들 오퍼론에 의해 코드된 효소는 맥락산을 PCA로 변환한다.그리고 phzH, phzM, phzS의 세 가지 핵심 유전자의 산물은 PCA를 위에 언급된 다른 페나진으로 변환시킨다.페나진 생합성은 잘 연구되고 있지만 갈색 페나진 피오멜라닌의 최종 구조에 대해서는 의문이 남는다.
피오시아닌 생합성이 억제되면 [45]체외에서 P. Aeruginosa 병원성 저하가 관찰된다.이는 피오시아닌이 체내 P. Aeruginosa의 초기 콜로니화에 가장 큰 영향을 미친다는 것을 시사한다.
트리거
낮은 인산염 수치로, P. aeruginosa는 양성 공생에서 활성화되어 장내 치사 독소를 발현하고 숙주를 심각하게 손상시키거나 죽이는 것으로 밝혀졌으며, 이는 [46]항생제 대신 과잉 인산염을 공급함으로써 완화될 수 있다.
식물 및 무척추동물
고등 식물에서, 예를 들어 아라비도시스 [47]탈리아나 락투카 사티바(Lactuca sativa)[48][49]와 같이, P. aeruginosa는 부드러운 부패를 유도한다.선충류인 케노하브디티스 엘레강스,[50][51] 초파리 드로소필라,[52] 나방 갤러리아멜로넬라 [53]등 무척추동물에게도 병원성이 있다.독성 인자의 연관성은 식물과 동물 [48][54]감염에 있어서 동일하다.곤충과 식물 모두에서 P. Aeruginosa의 독성은 높은 쿼럼 감지(QS)[55]에 의존합니다.그 QS는 차례로 아실-호모세린-락톤 합성효소, lasI 및 [56]rhlI와 같은 유전자에 크게 의존한다.
쿼럼 감지
P. Aeruginosa는 영양소나 군락을 위해 다른 종들과 경쟁하기 위해 유전자 발현을 조정하는 능력을 가진 기회주의적 병원체이다.유전자 발현 조절은 외부 환경으로 방출되는 자가 인듀서라고 불리는 작은 분자의 생산을 통해 세포-세포 통신이나 쿼럼 감지(QS)를 통해 일어날 수 있다.이러한 신호는 특정 모집단 세포 밀도와 상관된 특정 농도에 도달하면 각각의 조절기를 활성화하여 유전자 발현과 조정 행동을 변화시킨다.P. aeruginosa는 5개의 상호 연결된 QS 시스템(las, rhl, pqs, iqs 및 pch)을 사용하여 각각 고유한 신호 [57]분자를 생성합니다.las와 rhl 시스템은 수많은 QS 제어 유전자의 활성화에 책임이 있으며, pqs 시스템은 퀴놀론 시그널링에 관여하며 iqs 시스템은 세포간 [58]통신에 중요한 역할을 한다.P. aeruginosa의 QS는 계층적으로 구성된다.라스 레귤레이터는 rhl과 같은 다수의 다른 레귤레이터의 전사를 활성화함으로써 QS 조절 시스템을 개시하기 때문에 시그널링 계층의 상위는 라스 시스템입니다.따라서, 라스 시스템은 라스로부터 [59]rhl 레귤론까지의 계층형 QS 캐스케이드를 정의합니다.이들 분자의 검출은 낭포성 섬유증 [60]환자의 폐에서 생물막으로 성장한다는 것을 의미한다.그러나 QS, 특히 라스 시스템이 P. aeruginosa의 병원성에 미치는 영향은 명확하지 않다.연구에 따르면 lasR 결핍 돌연변이는 낭포성 섬유증[61] 환자에서 더 심각한 결과와 관련이 있으며 QS 활동이 손상되었음에도 불구하고 만성 감염 낭포성 섬유증[62] 환자의 최대 63%에서 발견된다.
QS는 색소 피오시아닌을 포함한 많은 독성 인자의 발현을 계층적으로 제어하는 것으로 알려져 있다.그러나, 라스 시스템이 유전자 발현 조절을 시작하지만, 그것의 부재가 독성 인자의 상실로 이어지지는 않는다.최근에는 rhl 시스템이 엘라스트 분해 및 포도상구균 분해 활동을 담당하는 단백질 분해 효소와 같은 라스 특이 인자를 부분적으로 제어하지만 지연되는 방식으로 제어한다는 것이 입증되었다.라스는 QS 제어 [58]유전자의 직간접 조절 장치입니다박테리아가 주변 변화에 빠르게 적응할 수 있도록 하는 또 다른 형태의 유전자 조절은 환경 신호를 통해서이다.최근의 연구는 혐기성 생물이 QS의 주요 조절 회로에 큰 영향을 미칠 수 있다는 것을 발견했다.QS와 혐기성 생물 사이의 이러한 중요한 연관성은 이 [63]유기체의 독성 인자의 생산에 중요한 영향을 미친다.마늘은 대마늘의 [64]쿼럼 감지를 실험적으로 차단한다.
생체막 형성 및 순환 di-GMP
대부분의 Gram 음성세균과 마찬가지로 P. aeruginosa 바이오막 형성은 단일 분자, 즉 순환 di-GMP에 의해 조절된다. 낮은 순환 di-GMP 농도에서는 P. aeruginosa는 자유수영 모드를 갖는다.그러나 주기적인 di-GMP 수치가 증가하면, P. aeruginosa는 표면에 sessile 군집을 형성하기 시작한다.주기적 di-GMP의 세포 내 농도는 P. aeruginosa가 표면에 닿으면 수 초 이내에 증가한다(예: 암석, 플라스틱, 숙주 조직 [65]등).이렇게 하면 점착필리의 생산이 활성화되고, 점착필리는 "앵커"로 작용하여 표면에 점착된 점착필리를 안정화를 도모할 수 있습니다.나중에 박테리아는 강한 접착성 매트릭스를 생성함으로써 돌이킬 수 없을 정도로 부착되기 시작합니다.동시에, 고리형 di-GMP는 편모기계의 합성을 억제하고, 편모기계의 유영을 방지한다.억제하면 생체막의 접착력이 떨어지고 치료도 쉬워집니다.P. Aeruginosa의 생체막 매트릭스는 핵산, 아미노산, 탄수화물 및 다양한 이온으로 구성되어 있습니다.그것은 면역 체계와 일부 독성 화합물에 의한 공격으로부터 P. aeruginosa를 기계적, 화학적으로 보호합니다.P. Aeruginosa 바이오필름의 매트릭스는 PSL과 PEL이라는 두 가지 유형의 당(또는 "엑소폴리사카리드")으로 구성되어 있다.
- 다당류 합성궤적(PSL)과 순환 di-GMP는 양의 피드백 루프를 형성한다.PSL은 주기적인 di-GMP 생성을 촉진하고, 높은 주기적인 di-GMP는 오퍼론을 켜고 오퍼론의 액티비티를 증가시킵니다.이 15-유전자 오퍼론은 세포 통신에 필요한 세포-세포 및 세포-표면 상호작용을 담당합니다.또한 세포외 고분자 물질 [66][better source needed]매트릭스의 시퀘스터화에도 관여한다.
- PEL은 P. Aeruginosa 바이오 필름 [67]매트릭스에서 세포외 DNA를 가교하는 양이온성 엑소다당류이다.
특정 신호 또는 응력에 따라 P. Aeruginosa는 바이오 필름 프로그램을 되돌리고 분리합니다.최근 연구에 따르면 P. Aeruginosa 바이오필름의 분산세포는 순환성 di-GMP 수치가 낮으며 플랑크톤 및 바이오필름 [68][69]세포와는 생리가 다르다.이렇게 분산된 세포는 대식세포와 C.엘레건에는 매우 독성이 있지만 플랑크톤 [68]세포에 비해 철분 스트레스에 매우 민감하다.
생체막 및 처리 저항성
P. Aeruginosa의 생체 필름은 만성 기회주의 감염을 일으킬 수 있으며, 이는 산업 사회의 의료, 특히 면역 결핍 환자와 노인에게 심각한 문제이다.그들은 종종 전통적인 항생제 치료법으로는 효과적으로 치료될 수 없다.바이오필름은 이러한 박테리아를 환경적 요인으로부터 보호하는 것으로 보인다.P. Aeruginosa는 병원내 감염을 일으킬 수 있으며 항생제 내성 박테리아 연구의 모범 유기체로 간주됩니다.연구자들은 플랑크톤 성장에서 생물막 표현형으로 전환하게 하는 분자 메커니즘과 P. aeruginosa와 같은 치료 저항성 박테리아에서 QS의 역할에 대해 더 많이 배우는 것이 중요하다고 생각한다.이는 만성 감염 환자의 더 나은 임상 관리에 기여해야 하며, [63]신약 개발로 이어져야 한다.
최근 과학자들은 토브라마이신과 같은 항생제에 대한 P. Aeruginosa 내성의 가능한 유전적 근거를 조사하고 있다.이 종에서 내성의 중요한 유전적 결정인자로 식별되는 한 가지 흔적은 ndvB이며, 이는 항생제와 상호작용하여 그것들을 주증막으로 격리시키는 원인이 될 수 있는 주증막성 글루칸을 암호화한다.이러한 결과는 세균 항생제 내성 이면에 단순히 항생제에 [70]대한 확산 장벽 역할을 하는 바이오필름이 아니라 유전적 근거가 존재함을 시사한다.
진단.
감염의 성질에 따라 적절한 검체를 채취하여 세균학 실험실로 보내 식별한다.대부분의 세균학적 시료와 마찬가지로 그램염색을 실시하여 그램음성봉 및/또는 백혈구를 나타낼 수 있다.P. Aeruginosa는 세균학적 매체에서 특징적인 "가죽 같은" 또는 "신선한 토르티야" 냄새를 가진 군락을 생산합니다.혼합 배양에서, 그것은 산화효소에 양성 반응을 보이는 MacConkey 한천(유당을 발효시키지 않기 때문에)에서 투명한 군체로 분리될 수 있습니다.확인 테스트에는 세트리미드 한천에서 청록색 색소 피오시아닌의 생성과 42°C에서의 성장이 포함됩니다.TSI 경사도는 분변 [citation needed]시료에서 비발효성 의사모나스 종과 장내 병원체를 구별하기 위해 종종 사용된다.
P. aeruginosa가 정상적으로 멸균된 장소(피, 뼈, 깊은 채집)에서 분리되면 일반적으로 위험한 것으로 간주되며, 거의 항상 [71][72]치료가 필요하다.그러나 비살균 부위(입가래, 가래 등)에서 자주 분리되며, 이러한 상황에서는 감염이 아닌 식민지화를 나타낼 수 있다.따라서 비살균 검체로부터 P. aeruginosa의 분리는 신중하게 해석해야 하며, 치료를 시작하기 전에 미생물학자 또는 전염병 의사/약국의 조언을 구해야 한다.치료가 [citation needed]필요 없는 경우가 많습니다.
분류
Pseudomonas aeruginosa의 형태학적, 생리학적, 생화학적 특성은 아래 표에 나와 있다.
| 테스트 타입 | 시험 | 특성. |
| 콜로니 문자 | 크기 | 큰. |
| 유형 | 매끄러운 | |
| 색. | ||
| 모양. | 평평한 | |
| 형태학적 문자 | 모양. | 로드 |
| 생리학적 특징 | 운동성 | + |
| 6.5%의 NaCl 성장률 | - | |
| 생화학적 특성 | 그램 염색 | - |
| 산화효소 | + | |
| 카탈라아제 | + | |
| 산화 발효제 | ||
| 운동성 | + | |
| 메틸레드 | - | |
| 보제스 프로스카우어 | - | |
| 인돌 | - | |
| HS2 프로덕션 | - | |
| 우레아제 | - | |
| 질산 환원효소 | + | |
| β-갈락토시다아제 | ||
| 페닐알라닌탈아미나아제 | - | |
| 디엔에스 | - | |
| 리파아제 | + | |
| 리신탈카르복실화효소 | - | |
| 안료 | + (연두색 색소 침착) | |
| 용혈 | 베타/변수 | |
| 가수 분해 | 젤라틴 | + |
| 카세인 | ||
| 사용률 | 글리세롤 | + |
| 갈락토스 | - | |
| D-포도당 | + | |
| D-과당 | + | |
| D만노스 | - | |
| 만니톨 | + | |
| 구연산염 | + | |
| 말토스 | - | |
| 수크로스 | - | |
| 유당 | - |
참고: + = 양, - = 음
P. aeruginosa는 단극 운동성을 [73]가진 그램 음성, 호기성(때로는 통칭 혐기성)의 막대 모양의 박테리아이다.그것은 인간과 [74]식물 모두의 기회주의적 병원체로 확인되었다.P. aeruginosa는 Pseudomonas의 [75]표준종이다.
P. aeruginosa의 식별은 개별 격리체가 종종 운동성이 부족하다는 사실 때문에 복잡할 수 있다.군체의 형태학 자체도 여러 가지 변종을 보여준다.크게는 평탄한 모서리와 높은 중앙을 가진 크고 매끄러운 두 가지 타입과 작고 거칠고 [76]볼록한 타입이다.세 번째 타입인 뮤코이드도 발견될 수 있다.큰 군락은 일반적으로 클리니얼 환경에서 발견되고 작은 군락은 [76]자연에서 발견됩니다.그러나 세 번째는 생물학적 환경에 존재하며 호흡기와 [76]요로에서 발견되었습니다.또한, 유전자 lasR의 돌연변이는 콜로니 형태를 크게 변화시켜 전형적으로 젤라틴 또는 용혈의 [citation needed]가수분해 실패로 이어진다.
특정 조건에서 P. aeruginosa는 피오시아닌(파란색), 피오베르딘(노란색 및 형광색), 피오루빈(빨간색) 및 피오멜라닌(갈색)을 포함한 다양한 색소를 분비할 수 있습니다.이것들은 [77]유기체의 신원을 확인하는 데 사용될 수 있다.
P. aeruginosa의 임상적 확인에는 42°C에서 성장하는 능력뿐만 아니라 피오시아닌과 플루오레세인의 생산 확인도 포함될 수 있다.P. Aeruginosa는 디젤 및 제트 연료에서 성장할 수 있으며, 탄화수소를 사용하는 미생물로 알려져 미생물 [78]부식을 일으킵니다.그것은 때때로 그들의 [citation needed]외모 때문에 부적절하게 "조류"라고 불리는 어둡고 젤리 같은 매트를 만듭니다.
치료
많은 P. Aeruginosa 분리제는 광범위한 항생제에 내성이 있으며 치료 실패 후 추가적인 내성을 보일 수 있습니다.일반적으로 항생제를 경험적으로 선택하는 것이 아니라 실험실 민감도에 따라 치료를 유도할 수 있어야 한다.만약 항생제가 경험적으로 시작된다면, 배양물을 얻기 위해 모든 노력을 해야 하고, 배양 결과가 나올 때 항생제의 선택을 재검토해야 한다.
많은 일반적인 일선 항생제에 대한 내성이 널리 퍼졌기 때문에, 카르바페넴, 폴리믹신, 그리고 최근에는 티게사이클린 등이 선택된 약물로 간주되었다. 그러나, 이러한 약물에 대한 내성도 보고되었다.그럼에도 불구하고, 그것들은 아직 저항이 보고되지 않은 지역에서 여전히 사용되고 있다.설박탐과 같은 β-락타마아제 억제제의 사용은 일정 수준의 내성이 존재하는 경우에도 항균 작용을 강화하기 위해 항생제와 함께 권고되었다.엄격한 항균 감수성 테스트 후 조합 요법은 다제내성 P. Aeruginosa 치료에 있어 최선의 조치인 것으로 밝혀졌다.P. aeruginosa에 대해 활성으로 보고된 일부 차세대 항생제에는 도리페넴, 세프토비폴린, 세프타롤린이 있다.그러나 표준화를 위해서는 더 많은 임상시험이 필요하다.그러므로, P. Aeruginosa에 대한 새로운 항생제와 약물의 발견을 위한 연구가 매우 필요하다.P. aeruginosa에 대해 활성이 있을 수 있는 항생제에는 다음이 포함된다.
- 아미노글리코시드(겐타마이신, 아미카신, 토브라마이신, 단 카나마이신은 제외)
- 퀴놀론(히드로플록사신, 레보플록사신, 단 묵시플록사신은 제외)
- 세팔로스포린(세페타지타임, 세페라존, 세페로폼, 세페토비폴롤, 그러나 세페로시임, 세페토락시임 또는 세페트리아손 제외)
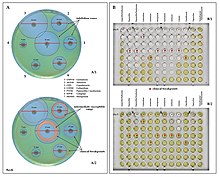 P. Aeruginosa 항생제 감수성 테스트의 예.디스크 확산 테스트(A) 및 MIC 테스트(B).P. Aeruginosa는 암피실린/술박탐, 티게사이클린 및 트리메토프림/술파메톡사졸(임그에는 중단점이 없음)에 내성이 있다.B)
P. Aeruginosa 항생제 감수성 테스트의 예.디스크 확산 테스트(A) 및 MIC 테스트(B).P. Aeruginosa는 암피실린/술박탐, 티게사이클린 및 트리메토프림/술파메톡사졸(임그에는 중단점이 없음)에 내성이 있다.B) - 항이피소도모날 페니실린: 카르복시 페니실린(카르베니실린 및 티카실린)과 우레이도피니실린(메즐로실린, 아즐로실린, 피페라실린)입니다.P. Aeruginosa는 본질적으로 다른 모든 페니실린에 내성이 있다.
- 카르바페넴(meropenem, imipenem, doripenem, 단, Eertapenem은 제외)
- 폴리믹신(폴리믹신B 및 콜리스틴)[79]
- 모노박탐스(아즈트레오남)
플루오로퀴놀론은 P. Aeruginosa에 널리 효과가 있는 몇 안 되는 항생제 클래스 중 하나이기 때문에 일부 병원에서는 내성 균주의 발생을 피하기 위해 사용이 엄격하게 제한된다.감염이 표면적이고 제한적인 드문 경우(예: 귀 감염 또는 손톱 감염)에는 국소 겐타마이신 또는 콜리스틴을 사용할 수 있습니다.
의사도모나스 창상 감염의 경우 0.5%에서 5%의 농도의 아세트산이 창상 세균을 제거하는 데 효과적인 정균제가 될 수 있다.보통 아세트산을 적신 멸균 거즈를 일반 식염수로 세척한 후 상처 부위에 올려놓습니다.드레싱은 하루에 한 번 할 것이다.의사도모나스는 보통 10~14일 치료 [80]후 90%가 제거된다.
항생제 내성
P. aeruginosa의 가장 우려되는 특징 중 하나는 낮은 항생제 감수성이며, 이는 염색체 부호화 항생제 내성 유전자(예: mexAB, mexXY 등)와 함께 다약물 유출 펌프의 일치된 작용과 세균 세포 [81]외피의 낮은 투과성 때문이다.이러한 내인성 외에도, P. aeruginosa는 염색체 부호화 유전자의 돌연변이 또는 항생제 내성 결정 인자의 수평 유전자 전달에 의해 후천성 내성을 쉽게 발달시킨다.P. Aeruginosa 분리주들에 의한 다제내성의 개발은 다른 돌연변이의 획득 및/또는 항생제 내성 유전자의 수평적 이전을 포함한 몇 가지 다른 유전적 사건을 필요로 한다.과변성은 만성 감염을 일으키는 P. aeruginosa 균주의 돌연변이 주도 항생제 내성 선택을 선호하며, 인테그론에서 여러 다른 항생제 내성 유전자의 클러스터링은 항생제 내성 결정 인자의 공동 획득을 선호한다.일부 최근의 연구는 생물막 형성이나 소결장 변종 출현과 관련된 표현형 내성이 항생제 [63]치료에 대한 P. Aeruginosa 집단의 반응에 중요할 수 있다는 것을 보여주었다.
항생제 내성의 기초가 되는 메커니즘에는 항생제 분해 또는 항생제 불활성화 효소, 항생제 퇴출을 위한 외막 단백질 및 항생제 표적을 바꾸기 위한 돌연변이의 생산이 포함되어 있는 것으로 밝혀졌다.PER-1, PER-2, VEB-1, AmpC 세팔로스포리나아제 등의 확장 스펙트럼β-락타마아제, 세린옥사실리나제 등의 카르바페네마아제, 메탈로-b-락타마아제, 옥사형 카르바페나아제, 아미노글리제 등의 항생제 분해 효소의 존재는 기타 효소 중 존재해 왔다.P. Aeruginosa는 또한 항생제 작용의 대상을 수정할 수 있다. 예를 들어, 아미노글리코시드의 결합과 DNA의 변형을 방지하기 위한 16S rRNA의 메틸화 또는 퀴놀론의 작용으로부터 보호하기 위한 토포이소머라아제이다.P. Aeruginosa는 또한 다수의 항생제 등급에 대해 내성을 부여하는 다제 배수 펌프 시스템을 보유하고 있는 것으로 보고되었으며, MexAB-OprM(Resistance-Nodulation-Division, RND) 패밀리가 가장[82] 중요한 것으로 간주된다.항생제 내성과 관련이 있는 것으로 밝혀진 중요한 요인은 내성 균주의 독성 능력의 감소이다.이러한 발견은 리팜피신 내성 및 콜리스틴 내성 균주의 경우 보고되었으며, 이 경우 감염 능력, 쿼럼 감지 및 운동성의 감소가 확인되었다.[83]
DNA 자이라아제 돌연변이는 일반적으로 P. Aeruginosa의 항생제 내성과 관련이 있다.이 돌연변이는 다른 돌연변이와 결합하면 생존에 지장을 주지 않고 높은 저항을 일으킨다.또한 사이클릭 di-GMP 시그널링에 관여하는 유전자는 내성에 기여할 수 있다.낭포성 섬유증 환자의 폐를 모방하도록 설계된 체외 환경에서 성장하면,[84] 이 유전자들은 반복적으로 돌연변이를 일으킨다.
두 개의 작은 RNA: Sr0161과 ErsA는 카르바페넴 항생제를 주배경으로 흡수하는 주요 포린 OprD를 코드하는 mRNA와 상호작용하는 것으로 나타났다.sRNA는 5'에 결합합니다.oprD의 UTR은 meropenem에 대한 세균 내성을 증가시킨다.또 다른 sRNA: Sr006은 지질 A의 탈아실화를 담당하는 효소인 PagL의 발현을 (전사 후) 적극적으로 조절하는 것이 제안되었다.이것은 지질 [85]A의 소염 특성을 감소시킨다.또한 PagL 발현에 대한 살모넬라[86] Sr006의 연구와 마찬가지로 폴리믹신 B의 [85]내성을 돕는 것이 제안되었다.
예방
프로바이오틱스 예방은 ICU [87]환경에서 Pseudomonas 감염의 정착과 지연 시작을 예방할 수 있습니다.Pseudomonas에 대한 면역 친화증이 [88]연구되고 있다.물웅덩이, 뜨거운 욕조 및 기타 체수조를 피하고 정기적으로 습기와 접촉하는 장비(예: 콘택트렌즈 장비 및 용액)를 정기적으로 소독 및/또는 교체하며 손을 자주 씻는 것(다른 많은 병원체로부터도 보호됨)을 통해 P. Aeruginosa에 걸릴 위험을 줄일 수 있습니다.그러나,[89] 환경에서의 P. Aeruginosa가 얼마나 흔한지를 감안할 때, 최상의 위생 관행이라 할지라도 P. Aeruginosa로부터 개인을 완전히 보호할 수는 없습니다.
실험 요법
P. Aeruginosa에 대한 파지 요법은 항생제와 결합할 수 있는 가능한 효과적인 치료법으로 조사되었으며, 금기사항이 없으며 부작용이 최소화되었습니다.페이지는 섭취, 용도 [90]등에 적합한 멸균 액체로 생산됩니다.P. Aeruginosa에 의한 귀 감염에 대한 파지 요법은 2009년 [91]8월 임상 이비인후과 저널에 보고되었다.
조사.
2013년 Joang Xavier는 먹이를 얻기 위해 떼를 지어 다녀야 하는 반복적인 조건에 노출되었을 때, 기준 유기체는 단일 [92]편모를 발달시키는 반면 기준 유기체는 기준 유기체보다 25% 빠른 속도로 "고온"할 수 있는 능력을 발전시킨 실험에 대해 설명했다.이 결과는 반복 가능성이 [93]높다는 점에서 실험 진화 분야에서 두드러졌다.
P. Aeruginosa는 도시 고체 [94]폐기물의 생물 조정 및 폴리에틸렌 처리에 사용하기 위해 연구되어 왔다.
분배
해충 위험 분석
2019년 현재[update] 동아프리카공동체는 방역우려종으로 간주하고 있다.케냐의 나머지 지역에 P. aeruginosa의 Phaseolus vultis-pathogenic 변종 존재.1989년 케냐에서 [95]Kaaya & Darji가 처음 검출된 이후 EAC에 의한 해충 위험 분석은 이 박테리아의 CABI의 농작물 보호 요약 목록에 기초했다.
「 」를 참조해 주세요.
- 세균수 분석
- 오염 관리
- 병원내 감염
- NrsZ소형 RNA
- 아스폰A 안티센스 RNA
- 열충격 유전자 발현 억제(ROSE) 요소
- Pseudomon-1 RNA 모티브(ErsA sRNA)
- PrrF RNA
- Pseudomonas sRNA P16 (RgsA sRNA)
레퍼런스
- ^ "UK Standards for Microbiology Investigations: Identification of Pseudomonas species and other Non- Glucose Fermenters" (PDF). Public Health England. 13 April 2015. Retrieved 4 May 2022.
- ^ Diggle S, Whiteley, M (2020). "Microbe Profile: Pseudomonas aeruginosa:opportunistic pathogen and lab rat". Microbiology. 166 (1): 30–33. doi:10.1099/mic.0.000860. PMC 7273324. PMID 31597590.
- ^ Balcht A, Smith R (1994). Pseudomonas aeruginosa: Infections and Treatment. Informa Health Care. pp. 83–84. ISBN 978-0-8247-9210-7.
- ^ Itah A, Essien J (2005). "Growth Profile and Hydrocarbonoclastic Potential of Microorganisms Isolated from Tarballs in the Bight of Bonny, Nigeria". World Journal of Microbiology and Biotechnology. 21 (6–7): 1317–22. doi:10.1007/s11274-004-6694-z. S2CID 84888286.
- ^ Høiby N, Ciofu O, Bjarnsholt T (November 2010). "Pseudomonas aeruginosa biofilms in cystic fibrosis". Future Microbiology. 5 (11): 1663–74. doi:10.2217/fmb.10.125. PMID 21133688.
- ^ a b Henry, R. (August 2012). "Etymologia: Pseudomonas". Emerging Infectious Diseases. 18 (8): 1241. doi:10.3201/eid1808.ET1808. PMC 3423701.
- ^ Brown RW (1956). Composition of Scientific Words. Smithsonian Institutional Press. ISBN 978-0-87474-286-2.
- ^ Tzouchas A (2014). WestBow Press. Greek Words. p. 550. ISBN 978-1490726106.
- ^ Klockgether J, Cramer N, Wiehlmann L, et al. (2011). "Pseudomonas aeruginosa Genomic Structure and Diversity". Frontiers in Microbiology. 2: 150. doi:10.3389/fmicb.2011.00150. PMC 3139241. PMID 21808635.
- ^ De Smet J, Hendrix H, Blasdel BG, et al. (September 2017). "Pseudomonas predators: understanding and exploiting phage-host interactions". Nature Reviews. Microbiology. 15 (9): 517–530. doi:10.1038/nrmicro.2017.61. PMID 28649138. S2CID 826136.
- ^ a b c d e f Nikolaidis M, Mossialos D, Oliver SG, Amoutzias GD (2020-07-24). "Comparative Analysis of the Core Proteomes among the Pseudomonas Major Evolutionary Groups Reveals Species-Specific Adaptations for Pseudomonas aeruginosa and Pseudomonas chlororaphis". Diversity. 12 (8): 289. doi:10.3390/d12080289. ISSN 1424-2818.
- ^ Ozer EA, Allen JP, Hauser AR (August 2014). "Characterization of the core and accessory genomes of Pseudomonas aeruginosa using bioinformatic tools Spine and AGEnt". BMC Genomics. 15 (1): 737. doi:10.1186/1471-2164-15-737. PMC 4155085. PMID 25168460.
- ^ Subedi D, Vijay AK, Kohli GS, et al. (October 2018). "Comparative genomics of clinical strains of Pseudomonas aeruginosa strains isolated from different geographic sites". Scientific Reports. 8 (1): 15668. Bibcode:2018NatSR...815668S. doi:10.1038/s41598-018-34020-7. PMC 6199293. PMID 30353070.
- ^ Freschi L, Vincent AT, Jeukens J, et al. (January 2019). Martin B (ed.). "The Pseudomonas aeruginosa Pan-Genome Provides New Insights on Its Population Structure, Horizontal Gene Transfer, and Pathogenicity". Genome Biology and Evolution. 11 (1): 109–120. doi:10.1093/gbe/evy259. PMC 6328365. PMID 30496396.
- ^ Weiser R, Green AE, Bull MJ, et al. (July 2019). "Not all Pseudomonas aeruginosa are equal: strains from industrial sources possess uniquely large multireplicon genomes". Microbial Genomics. 5 (7). doi:10.1099/mgen.0.000276. PMC 6700666. PMID 31170060.
- ^ Roy PH, Tetu SG, Larouche A, et al. (January 2010). "Complete genome sequence of the multiresistant taxonomic outlier Pseudomonas aeruginosa PA7". PLOS ONE. 5 (1): e8842. Bibcode:2010PLoSO...5.8842R. doi:10.1371/journal.pone.0008842. PMC 2809737. PMID 20107499.
- ^ Winstanley C, Langille MG, Fothergill JL, et al. (January 2009). "Newly introduced genomic prophage islands are critical determinants of in vivo competitiveness in the Liverpool Epidemic Strain of Pseudomonas aeruginosa". Genome Research. 19 (1): 12–23. doi:10.1101/gr.086082.108. PMC 2612960. PMID 19047519.
- ^ Marvig RL, Johansen HK, Molin S, Jelsbak L (2013). "Genome analysis of a transmissible lineage of pseudomonas aeruginosa reveals pathoadaptive mutations and distinct evolutionary paths of hypermutators". PLOS Genetics. 9 (9): e1003741. doi:10.1371/journal.pgen.1003741. PMC 3764201. PMID 24039595.
- ^ Wee BA, Tai AS, Sherrard LJ, et al. (August 2018). "Whole genome sequencing reveals the emergence of a Pseudomonas aeruginosa shared strain sub-lineage among patients treated within a single cystic fibrosis centre". BMC Genomics. 19 (1): 644. doi:10.1186/s12864-018-5018-x. PMC 6117919. PMID 30165811.
- ^ Kidd TJ, Ritchie SR, Ramsay KA, et al. (6 September 2012). "Pseudomonas aeruginosa exhibits frequent recombination, but only a limited association between genotype and ecological setting". PLOS ONE. 7 (9): e44199. Bibcode:2012PLoSO...744199K. doi:10.1371/journal.pone.0044199. PMC 3435406. PMID 22970178.
- ^ Kidd TJ, Gibson JS, Moss S, et al. (May 2011). "Clonal complex Pseudomonas aeruginosa in horses". Veterinary Microbiology. 149 (3–4): 508–12. doi:10.1016/j.vetmic.2010.11.030. PMID 21183294.
- ^ Schobert M, Jahn D (December 2010). "Anaerobic physiology of Pseudomonas aeruginosa in the cystic fibrosis lung". International Journal of Medical Microbiology. 300 (8): 549–56. doi:10.1016/j.ijmm.2010.08.007. PMID 20951638.
- ^ Gerard, Funke, Case (2016). Microbiology: An Introduction (12th ed.). Pearson Education. p. 54. ISBN 978-0-321-92915-0.
- ^ Hassett DJ (December 1996). "Anaerobic production of alginate by Pseudomonas aeruginosa: alginate restricts diffusion of oxygen". Journal of Bacteriology. 178 (24): 7322–5. doi:10.1128/jb.178.24.7322-7325.1996. PMC 178651. PMID 8955420.
- ^ Worlitzsch D, Tarran R, Ulrich M, et al. (February 2002). "Effects of reduced mucus oxygen concentration in airway Pseudomonas infections of cystic fibrosis patients". The Journal of Clinical Investigation. 109 (3): 317–25. doi:10.1172/JCI13870. PMC 150856. PMID 11827991.
- ^ Cooper M, Tavankar GR, Williams HD (May 2003). "Regulation of expression of the cyanide-insensitive terminal oxidase in Pseudomonas aeruginosa". Microbiology. 149 (Pt 5): 1275–1284. doi:10.1099/mic.0.26017-0. PMID 12724389.
- ^ Williams HD, Zlosnik JE, Ryall B (2007). Oxygen, cyanide and energy generation in the cystic fibrosis pathogen Pseudomonas aeruginosa. Advances in Microbial Physiology. Vol. 52. pp. 1–71. doi:10.1016/S0065-2911(06)52001-6. ISBN 9780120277520. PMID 17027370.
- ^ Leach R, Moore K, Bell D (2016). Oxford Desk Reference: Acute Medicine. Oxford University Press. p. 244. ISBN 9780191007149.
- ^ Buckling A, Harrison F, Vos M, et al. (November 2007). "Siderophore-mediated cooperation and virulence in Pseudomonas aeruginosa". FEMS Microbiology Ecology. 62 (2): 135–41. doi:10.1111/j.1574-6941.2007.00388.x. PMID 17919300.
- ^ Nguyen AT, Jones JW, Ruge MA, et al. (July 2015). "Iron Depletion Enhances Production of Antimicrobials by Pseudomonas aeruginosa". Journal of Bacteriology. 197 (14): 2265–75. doi:10.1128/JB.00072-15. PMC 4524187. PMID 25917911.
- ^ a b Harrison F, Browning LE, Vos M, Buckling A (July 2006). "Cooperation and virulence in acute Pseudomonas aeruginosa infections". BMC Biology. 4: 21. doi:10.1186/1741-7007-4-21. PMC 1526758. PMID 16827933.
- ^ Griffin AS, West SA, Buckling A (August 2004). "Cooperation and competition in pathogenic bacteria". Nature. 430 (7003): 1024–7. Bibcode:2004Natur.430.1024G. doi:10.1038/nature02744. hdl:1842/698. PMID 15329720. S2CID 4429250.
- ^ Pitcher RS, Brissett NC, Doherty AJ (2007). "Nonhomologous end-joining in bacteria: a microbial perspective". Annual Review of Microbiology. Annual Reviews. 61 (1): 259–282. doi:10.1146/annurev.micro.61.080706.093354. PMID 17506672.
- ^ 토다의 세균학 온라인 교과서.Textbookofbacteriology.net (2004-06-04)2011년 10월 9일에 취득.
- ^ a b "Pseudomonas aeruginosa in Healthcare Settings". Healthcare-associated Infections (HAI): Diseases and Organisms. Centers for Disease Control and Prevention. 7 May 2014.
- ^ Fine MJ, Smith MA, Carson CA, et al. (January 1996). "Prognosis and outcomes of patients with community-acquired pneumonia. A meta-analysis". JAMA. 275 (2): 134–41. doi:10.1001/jama.275.2.134. PMID 8531309.
- ^ Diekema DJ, Pfaller MA, Jones RN, et al. (September 1999). "Survey of bloodstream infections due to gram-negative bacilli: frequency of occurrence and antimicrobial susceptibility of isolates collected in the United States, Canada, and Latin America for the SENTRY Antimicrobial Surveillance Program, 1997". Clinical Infectious Diseases. 29 (3): 595–607. doi:10.1086/598640. PMID 10530454.
- ^ Prithiviraj B, Bais HP, Weir T, et al. (September 2005). "Down regulation of virulence factors of Pseudomonas aeruginosa by salicylic acid attenuates its virulence on Arabidopsis thaliana and Caenorhabditis elegans". Infection and Immunity. 73 (9): 5319–28. doi:10.1128/IAI.73.9.5319-5328.2005. PMC 1231131. PMID 16113247.
- ^ Johnson PA (March 2019). "Novel understandings of host cell mechanisms involved in chronic lung infection: Pseudomonas aeruginosa in the cystic fibrotic lung". Journal of Infection and Public Health. 12 (2): 242–246. doi:10.1016/j.jiph.2018.10.014. PMID 30459101.
- ^ Kirienko NV, Ausubel FM, Ruvkun G (February 2015). "Mitophagy confers resistance to siderophore-mediated killing by Pseudomonas aeruginosa". Proceedings of the National Academy of Sciences of the United States of America. 112 (6): 1821–6. Bibcode:2015PNAS..112.1821K. doi:10.1073/pnas.1424954112. PMC 4330731. PMID 25624506.
- ^ Kirienko NV, Kirienko DR, Larkins-Ford J, et al. (April 2013). "Pseudomonas aeruginosa disrupts Caenorhabditis elegans iron homeostasis, causing a hypoxic response and death". Cell Host & Microbe. 13 (4): 406–16. doi:10.1016/j.chom.2013.03.003. PMC 3641844. PMID 23601103.
- ^ Dietrich LE, Price-Whelan A, Petersen A, et al. (September 2006). "The phenazine pyocyanin is a terminal signalling factor in the quorum sensing network of Pseudomonas aeruginosa" (PDF). Molecular Microbiology. 61 (5): 1308–21. doi:10.1111/j.1365-2958.2006.05306.x. PMID 16879411. S2CID 4985392.
- ^ Abu EA, Su S, Sallans L, et al. (August 2013). "Cyclic voltammetric, fluorescence and biological analysis of purified aeruginosin A, a secreted red pigment of Pseudomonas aeruginosa PAO1". Microbiology. 159 (Pt 8): 1736–1747. doi:10.1099/mic.0.065235-0. PMID 23782801.
- ^ Mavrodi DV, Bonsall RF, Delaney SM, et al. (November 2001). "Functional analysis of genes for biosynthesis of pyocyanin and phenazine-1-carboxamide from Pseudomonas aeruginosa PAO1". Journal of Bacteriology. 183 (21): 6454–65. doi:10.1128/JB.183.21.6454-6465.2001. PMC 100142. PMID 11591691.
- ^ a b Ho Sui SJ, Lo R, Fernandes AR, et al. (September 2012). "Raloxifene attenuates Pseudomonas aeruginosa pyocyanin production and virulence". International Journal of Antimicrobial Agents. 40 (3): 246–51. doi:10.1016/j.ijantimicag.2012.05.009. PMC 5511546. PMID 22819149.
- ^ "Research could lead to new non-antibiotic drugs to counter hospital infections" (Press release). University of Chicago Medical Center. 2009-04-14. Retrieved 26 June 2022.
- ^ Walker TS, Bais HP, Déziel E, et al. (January 2004). "Pseudomonas aeruginosa-plant root interactions. Pathogenicity, biofilm formation, and root exudation". Plant Physiology. 134 (1): 320–31. doi:10.1104/pp.103.027888. PMC 316311. PMID 14701912.
- ^ a b Rahme LG, Stevens EJ, Wolfort SF, et al. (June 1995). "Common virulence factors for bacterial pathogenicity in plants and animals". Science. 268 (5219): 1899–902. Bibcode:1995Sci...268.1899R. doi:10.1126/science.7604262. PMID 7604262.
- ^ Rahme LG, Tan MW, Le L, et al. (November 1997). "Use of model plant hosts to identify Pseudomonas aeruginosa virulence factors". Proceedings of the National Academy of Sciences of the United States of America. 94 (24): 13245–50. Bibcode:1997PNAS...9413245R. doi:10.1073/pnas.94.24.13245. PMC 24294. PMID 9371831.
- ^ Mahajan-Miklos S, Tan MW, Rahme LG, Ausubel FM (January 1999). "Molecular mechanisms of bacterial virulence elucidated using a Pseudomonas aeruginosa-Caenorhabditis elegans pathogenesis model". Cell. 96 (1): 47–56. doi:10.1016/S0092-8674(00)80958-7. PMID 9989496. S2CID 11207155.
- ^ Martínez C, Pons E, Prats G, León J (January 2004). "Salicylic acid regulates flowering time and links defence responses and reproductive development". The Plant Journal. 37 (2): 209–17. doi:10.1046/j.1365-313X.2003.01954.x. PMID 14690505.
- ^ D'Argenio DA, Gallagher LA, Berg CA, Manoil C (February 2001). "Drosophila as a model host for Pseudomonas aeruginosa infection". Journal of Bacteriology. 183 (4): 1466–71. doi:10.1128/JB.183.4.1466-1471.2001. PMC 95024. PMID 11157963.
- ^ Miyata S, Casey M, Frank DW, et al. (May 2003). "Use of the Galleria mellonella caterpillar as a model host to study the role of the type III secretion system in Pseudomonas aeruginosa pathogenesis". Infection and Immunity. 71 (5): 2404–13. doi:10.1128/IAI.71.5.2404-2413.2003. PMC 153283. PMID 12704110.
- ^ Rahme LG, Ausubel FM, Cao H, et al. (August 2000). "Plants and animals share functionally common bacterial virulence factors". Proceedings of the National Academy of Sciences of the United States of America. 97 (16): 8815–21. Bibcode:2000PNAS...97.8815R. doi:10.1073/pnas.97.16.8815. PMC 34017. PMID 10922040.
- ^ Rumbaugh KP, Griswold JA, Iglewski BH, Hamood AN (Nov 1999). "Contribution of quorum sensing to the virulence of Pseudomonas aeruginosa in burn wound infections". Infect Immun. 67 (11): 5854–62. doi:10.1128/IAI.67.11.5854-5862.1999. PMC 96966. PMID 10531240.
- ^ Azimi S, Klementiev AD, Whiteley M, Diggle SP (September 2020). "Bacterial Quorum Sensing During Infection". Annual Review of Microbiology. Annual Reviews. 74 (1): 201–219. doi:10.1146/annurev-micro-032020-093845. PMID 32660382. S2CID 220518911.
- ^ Allesen-Holm M, Barken KB, Yang L, et al. (February 2006). "A characterization of DNA release in Pseudomonas aeruginosa cultures and biofilms". Molecular Microbiology. 59 (4): 1114–28. doi:10.1111/j.1365-2958.2005.05008.x. PMID 16430688. S2CID 11915780.
- ^ a b Dekimpe V, Déziel E (March 2009). "Revisiting the quorum-sensing hierarchy in Pseudomonas aeruginosa: the transcriptional regulator RhlR regulates LasR-specific factors". Microbiology. 155 (Pt 3): 712–723. doi:10.1099/mic.0.022764-0. PMID 19246742.
- ^ Lee J, Zhang L (January 2015). "The hierarchy quorum sensing network in Pseudomonas aeruginosa". Protein & Cell. 6 (1): 26–41. doi:10.1007/s13238-014-0100-x. PMC 4286720. PMID 25249263.
- ^ Winstanley C, Fothergill JL (January 2009). "The role of quorum sensing in chronic cystic fibrosis Pseudomonas aeruginosa infections". FEMS Microbiology Letters. 290 (1): 1–9. doi:10.1111/j.1574-6968.2008.01394.x. PMID 19016870.
- ^ Hoffman LR, Kulasekara HD, Emerson J, et al. (January 2009). "Pseudomonas aeruginosa lasR mutants are associated with cystic fibrosis lung disease progression". Journal of Cystic Fibrosis. 8 (1): 66–70. doi:10.1016/j.jcf.2008.09.006. PMC 2631641. PMID 18974024.
- ^ Feltner JB, Wolter DJ, Pope CE, et al. (October 2016). "LasR Variant Cystic Fibrosis Isolates Reveal an Adaptable Quorum-Sensing Hierarchy in Pseudomonas aeruginosa". mBio. 7 (5): e01513–16, /mbio/7/5/e01513–16.atom. doi:10.1128/mBio.01513-16. PMC 5050340. PMID 27703072.
- ^ a b c Cornelis P (2008). Pseudomonas: Genomics and Molecular Biology (1st ed.). Caister Academic Press. ISBN 978-1-904455-19-6.
- ^ Bjarnsholt T, Jensen PØ, Rasmussen TB, et al. (December 2005). "Garlic blocks quorum sensing and promotes rapid clearing of pulmonary Pseudomonas aeruginosa infections". Microbiology. 151 (Pt 12): 3873–3880. doi:10.1099/mic.0.27955-0. PMID 16339933.
- ^ Laventie BJ, Sangermani M, Estermann F, et al. (January 2019). "A Surface-Induced Asymmetric Program Promotes Tissue Colonization by Pseudomonas aeruginosa". Cell Host & Microbe. 25 (1): 140–152.e6. doi:10.1016/j.chom.2018.11.008. PMID 30581112.
- ^ 콜빈 외, 2013년
- ^ Jennings LK, Storek KM, Ledvina HE, et al. (September 2015). "Pel is a cationic exopolysaccharide that cross-links extracellular DNA in the Pseudomonas aeruginosa biofilm matrix". Proceedings of the National Academy of Sciences of the United States of America. 112 (36): 11353–8. Bibcode:2015PNAS..11211353J. doi:10.1073/pnas.1503058112. PMC 4568648. PMID 26311845.
- ^ a b Chua SL, Liu Y, Yam JK, et al. (July 2014). "Dispersed cells represent a distinct stage in the transition from bacterial biofilm to planktonic lifestyles". Nature Communications. 5: 4462. Bibcode:2014NatCo...5.4462C. doi:10.1038/ncomms5462. PMID 25042103.
- ^ Chua SL, Hultqvist LD, Yuan M, et al. (August 2015). "In vitro and in vivo generation and characterization of Pseudomonas aeruginosa biofilm-dispersed cells via c-di-GMP manipulation". Nature Protocols. 10 (8): 1165–80. doi:10.1038/nprot.2015.067. hdl:10356/84100. PMID 26158442. S2CID 20235088.
- ^ Mah TF, Pitts B, Pellock B, et al. (November 2003). "A genetic basis for Pseudomonas aeruginosa biofilm antibiotic resistance". Nature. 426 (6964): 306–10. Bibcode:2003Natur.426..306M. doi:10.1038/nature02122. PMID 14628055. S2CID 4412747.
- ^ Wheeler, Tyler. "What Is a Pseudomonas Infection?". MedicineNet. Retrieved 8 December 2020.
- ^ "Pseudomonas aeruginosa in Healthcare Settings". Center for Disease Control and Prevention. U.S. Department of Health & Human Services. 6 November 2019. Retrieved 8 December 2020.
- ^ Ryan KJ, Ray CG, eds. (2004). Sherris Medical Microbiology (4th ed.). McGraw Hill. ISBN 978-0-8385-8529-0.
- ^ Iglewski BH (1996). "Pseudomonas". In Baron S, et al. (eds.). Baron's Medical Microbiology (4th ed.). University of Texas Medical Branch. ISBN 978-0-9631172-1-2.
- ^ Anzai Y, Kim H, Park JY, et al. (July 2000). "Phylogenetic affiliation of the pseudomonads based on 16S rRNA sequence". International Journal of Systematic and Evolutionary Microbiology. 50 Pt 4 (4): 1563–1589. doi:10.1099/00207713-50-4-1563. PMID 10939664.
- ^ a b c Boone DR, Castenholz RW, Garrity GM (2005). Bergey's manual of systematic bacteriology (2nd ed.). New York: Springer. pp. 323–442. ISBN 0-387-98771-1. OCLC 45951601.
- ^ King EO, Ward MK, Raney DE (August 1954). "Two simple media for the demonstration of pyocyanin and fluorescin". The Journal of Laboratory and Clinical Medicine. 44 (2): 301–7. PMID 13184240.
- ^ Striebich RC, Smart CE, Gunasekera TS, et al. (September 2014). "Characterization of the F-76 diesel and Jet-A aviation fuel hydrocarbon degradation profiles of Pseudomonas aeruginosa and Marinobacter hydrocarbonoclasticus". International Biodeterioration & Biodegradation. 93: 33–43. doi:10.1016/j.ibiod.2014.04.024.
- ^ Hachem RY, Chemaly RF, Ahmar CA, et al. (June 2007). "Colistin is effective in treatment of infections caused by multidrug-resistant Pseudomonas aeruginosa in cancer patients". Antimicrobial Agents and Chemotherapy. 51 (6): 1905–11. doi:10.1128/AAC.01015-06. PMC 1891378. PMID 17387153.
- ^ Nagoba BS, Selkar SP, Wadher BJ, Gandhi RC (December 2013). "Acetic acid treatment of pseudomonal wound infections--a review". Journal of Infection and Public Health. 6 (6): 410–5. doi:10.1016/j.jiph.2013.05.005. PMID 23999348.
- ^ Poole K (January 2004). "Efflux-mediated multiresistance in Gram-negative bacteria". Clinical Microbiology and Infection. 10 (1): 12–26. doi:10.1111/j.1469-0691.2004.00763.x. PMID 14706082.
- ^ Rampioni G, Pillai CR, Longo F, et al. (September 2017). "Effect of efflux pump inhibition on Pseudomonas aeruginosa transcriptome and virulence". Scientific Reports. 7 (1): 11392. Bibcode:2017NatSR...711392R. doi:10.1038/s41598-017-11892-9. PMC 5596013. PMID 28900249.
- ^ Aghapour Z, Gholizadeh P, Ganbarov K, et al. (2019). "Molecular mechanisms related to colistin resistance in Enterobacteriaceae". Infection and Drug Resistance. 12: 965–975. doi:10.2147/IDR.S199844. PMC 6519339. PMID 31190901.
- ^ Wong A, Rodrigue N, Kassen R (September 2012). "Genomics of adaptation during experimental evolution of the opportunistic pathogen Pseudomonas aeruginosa". PLOS Genetics. 8 (9): e1002928. doi:10.1371/journal.pgen.1002928. PMC 3441735. PMID 23028345.
- ^ a b Zhang YF, Han K, Chandler CE, et al. (December 2017). "Probing the sRNA regulatory landscape of P. aeruginosa: post-transcriptional control of determinants of pathogenicity and antibiotic susceptibility". Molecular Microbiology. 106 (6): 919–937. doi:10.1111/mmi.13857. PMC 5738928. PMID 28976035.
- ^ Kawasaki K, China K, Nishijima M (July 2007). "Release of the lipopolysaccharide deacylase PagL from latency compensates for a lack of lipopolysaccharide aminoarabinose modification-dependent resistance to the antimicrobial peptide polymyxin B in Salmonella enterica". Journal of Bacteriology. 189 (13): 4911–9. doi:10.1128/JB.00451-07. PMC 1913436. PMID 17483225.
- ^ Forestier C, Guelon D, Cluytens V, et al. (2008). "Oral probiotic and prevention of Pseudomonas aeruginosa infections: a randomized, double-blind, placebo-controlled pilot study in intensive care unit patients". Critical Care. 12 (3): R69. doi:10.1186/cc6907. PMC 2481460. PMID 18489775.
- ^ Döring G, Pier GB (February 2008). "Vaccines and immunotherapy against Pseudomonas aeruginosa". Vaccine. 26 (8): 1011–24. doi:10.1016/j.vaccine.2007.12.007. PMID 18242792.
- ^ "Pseudomonas Aeruginosa Fact Sheet" (PDF). Children's Hospital of Illinois. Archived from the original (PDF) on 2016-05-09. Retrieved 2014-11-15.
- ^ Sulakvelidze A, Alavidze Z, Morris JG (March 2001). "Bacteriophage therapy". Antimicrobial Agents and Chemotherapy. 45 (3): 649–59. doi:10.1128/AAC.45.3.649-659.2001. PMC 90351. PMID 11181338.
- ^ Wright A, Hawkins CH, Anggård EE, Harper DR (August 2009). "A controlled clinical trial of a therapeutic bacteriophage preparation in chronic otitis due to antibiotic-resistant Pseudomonas aeruginosa; a preliminary report of efficacy". Clinical Otolaryngology. 34 (4): 349–57. doi:10.1111/j.1749-4486.2009.01973.x. PMID 19673983. S2CID 379471.
- ^ van Ditmarsch D, Boyle KE, Sakhtah H, et al. (August 2013). "Convergent evolution of hyperswarming leads to impaired biofilm formation in pathogenic bacteria". Cell Reports. 4 (4): 697–708. doi:10.1016/j.celrep.2013.07.026. PMC 3770465. PMID 23954787.
- ^ Zimmer C. "Watching Bacteria Evolve, With Predictable Results". Retrieved 2 February 2016.
- ^ Pathak, Vinay Mohan (23 March 2017). "Review on the current status of polymer degradation: a microbial approach". Bioresources and Bioprocessing. 4: 15. doi:10.1186/s40643-017-0145-9. ISSN 2197-4365.
- ^ East African Community (2019-11-29). Pest Risk Analysis (PRA) for Grain and Seed of Beans, Phaseolus vulgaris L. within East African Countries (Kenya, Burundi, Rwanda, Tanzania and Uganda): A Qualitative, Pathway-Initiated Risk Analysis) (Report). hdl:11671/24138.
- Breidenstein EB, de la Fuente-Núñez C, Hancock RE (August 2011). "Pseudomonas aeruginosa: all roads lead to resistance". Trends in Microbiology. 19 (8): 419–26. doi:10.1016/j.tim.2011.04.005. PMID 21664819.
외부 링크
- BacDive의 Pseudomonas aeruginosa 모식주 - 세균다양성 메타다타베이스
- 요한나 M.Swere 등(2019):박테리오파지는 항바이러스 면역성을 유발하고 세균 감염의 정화를 방지한다, Science, 2019년 3월 29일: Vol. 363, 6434, eaat9691. doi:10.1126/science.aat9691. Pf-phages, Inoviridae.다음 항목도 참조하십시오.
- UM 연구원들은 박테리아 바이러스에 대한 새로운 발견을 발표했다.점등: Eurek Alert!2019년 4월 1일.출처:몬태나 대학교