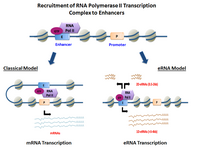
인핸서 RNA
Enhancer RNA인핸서 RNA(eRNA)는 인핸서 영역의 DNA 배열에서 전사된 비교적 긴 비코드 RNA 분자(50-2000 뉴클레오티드)의 클래스를 나타냅니다.이들은 RNA-seq와 [1][2]ChIP-seq와 같은 게놈 전체 기술을 사용하여 2010년에 처음 검출되었다. eRNA는 크기, 폴리아데닐화 상태 및 전사 방향성 [3]측면에서 주로 다른 1D eRNA와 2D eRNA의 두 가지 주요 클래스로 나눌 수 있다.주어진 eRNA의 발현은 표적 [4]유전자의 해당 강화제의 활성과 관련이 있다.증가하는 증거는 eRNA가 cis 및 트랜스(trans)에서 전사 조절에 적극적으로 역할을 한다는 것을 시사하며, 그 작용 메커니즘이 불분명하지만, 몇 가지 모델이 [3]제안되었다.
검출
RNA 중합효소 II(RNA pol II) 결합 및 비코드 RNA [1][2]전사의 공통 영역으로 증강제를 식별한 게놈 전체 연구에서 외부 유전자 전사의 부위로 증강제가 처음 발견되었다.RNA pol II-enhancer 상호작용과 RNA 전사 형성의 수준은 이러한 초기 연구에서 매우 가변적인 것으로 밝혀졌다.명시적 크로마틴 시그니처 피크를 사용하여 상당 부분(~70%)의 외인성 RNA Pol II 전사 시작 부위가 쥐 대식세포에서 [5]인핸서 부위와 겹치는 것으로 확인되었다.마우스 게놈의 12,000개의 신경 증강제 중, 거의 25%의 부위가 RNA Pol II와 결합하고 [6]전사물을 생성하는 것으로 밝혀졌다.병렬 연구에서 전사를 [2]유도하기 위해 염증 매개체 리포다당류에 자극된 쥐 대식세포에서 4,588개의 고신뢰 외부 RNA Pol II 결합 부위가 확인되었다.이러한 eRNA는 메신저 RNA(mRNA)와 달리 폴리아데닐화에 의한 수정이 부족했고, 일반적으로 짧고 부호화되지 않았으며, 양방향으로 전사되었다.이후 연구에서 단방향 전사를 통해 생성된 다른 유형의 eRNA의 전사가 더 길고 폴리 A [7]꼬리를 포함하는 것으로 밝혀졌다.또한, eRNA 수준은 인근 유전자의 mRNA 수준과 상관되어 이러한 비부호화 인핸서 RNA [1]분자의 잠재적 조절 및 기능적 역할을 시사했다.
생물 생성
요약
eRNA는 유전자 외 인핸서 [8]영역의 상류 및 하류 DNA 배열에서 전사된다.지금까지 여러 모델 인핸서가 RNA Pol II 및 일반 전사 인자를 직접 모집하고 유전자 프로모터에서 전사 시작 부위 전에 사전 개시 복합체(PIC)를 형성하는 능력을 입증했다.특정 세포 유형에서, 활성화된 증강제는 RNA Pol II를 모집하는 능력과 또한 그들의 국소 [2][1]서열의 능동적인 전사를 위한 템플릿을 제공하는 능력을 입증했다.
전사의 방향성에 따라 인핸서 영역은 2종류의 비코드 트랜스크립트, 1D-eRNA와 2D-eRNA를 생성합니다.사전 개시 복합체 및 인핸서에 모집된 특정 전사 인자의 성질에 따라 생성되는 eRNA의 유형이 제어될 수 있습니다.전사 후에도 대부분의 eRNA는 핵에 [9]남습니다.일반적으로, eRNA는 매우 불안정하고 핵 엑소좀에 의해 활발하게 분해된다.모든 인핸서가 전사되는 것은 아니며, 모든 세포 [5]유형에서 수십만 개의 크기로 전사된 인핸서보다 비전사된 인핸서가 훨씬 많습니다.
1D eRNA
대부분의 경우 인핸서 영역의 단방향 전사는 긴(4kb 이상) 폴리아데닐화 eRNA를 생성한다.폴리A+ eRNA를 생성하는 [7]인핸서는 2D-eRNA보다 염색질 시그니처의 H3K4me1/me3 비율이 낮다.PolyA+ eRNA는 유전자 내 인핸서에서의 전사 개시에 의해 생성되는 긴 다극성 폴리 트랜스크립트(meRNA)와는 다릅니다.대체적인 첫 번째 엑손(exon)을 제외하고 숙주 유전자의 구조를 정확하게 반영하는 이 긴 비부호화 RNA들은 낮은 [10]부호화 잠재력을 보인다.그 결과 폴리A+1D-eRNA는 진정한 증강제 템플릿 RNA와 다전자 RNA의 혼합기를 나타낼 수 있다.
2D eRNA
인핸서 사이트에서의 쌍방향 전사는 비교적 짧은(0.5~2kb) 및 비폴리아데닐화 eRNA를 생성한다.폴리A-eRNA를 생성하는 인핸서는 1D-eRNA보다 H3K4me1/me3 비율이 높은 크로마틴 시그니처를 가진다.일반적으로 증강제 전사 및 양방향 eRNA의 생산은 유전자 [11]전사에 대한 증강제 활성의 강한 상관관계를 보여준다.
eRNA 발현 빈도 및 타이밍
Arner 외 [12]연구진은 서로 다른 조건과 자극 타이밍에 따라 33개의 서로 다른 세포 유형에서 65,423개의 전사된 증강제(eRNA 생성)를 확인했다.증강제의 전사는 일반적으로 전사인자의 전사에 선행했고, 차례로 유전자의 메신저 RNA(mRNA) 전사에 선행했다.
카룰로 외 [13]연구진은 (1차 뉴런 배양으로부터) 하나의 특정 세포 유형인 뉴런을 조사했다.그들은 eRNA를 생성하는 28,492개의 추정 증강제를 전시했다.이러한 eRNA는 종종 인핸서 DNA의 양쪽 가닥에서 반대 방향으로 전사되었다.카룰로 [13]등표적 유전자의 mRNA와 비교하여 특정 증강 eRNA의 타이밍을 조사하기 위해 배양된 뉴런을 사용했다.배양된 뉴런은 활성화되고 RNA는 활성화 후 0분, 3.75, 5, 7.5, 15, 30, 60분에 해당 뉴런에서 분리되었다.이러한 실험 조건에서, 그들은 즉시 초기 유전자(IEG) FOS의 5개 강화제 중 2개, 즉 FOS 강화제 1과 FOS 강화제 3이 활성화되어 그들의 eRNAs(eRNA1과 eRNA3)의 전사가 시작되었음을 발견했다.FOS eRNA1과 eRNA3는 7.5분 이내에 유의하게 상향 조절된 반면, FOS mRNA는 자극 후 15분 후에 상향 조절되었다.IEGs FOSb와 NR4A1에서도 유사한 패턴이 발생했으며, 이는 많은 IEG에서 신경 활성화에 대한 반응으로 eRNA 유도가 mRNA 유도에 선행함을 나타낸다.
일부 인핸서는 eRNA를 전사하지 않고 표적 유전자에서 표적 프로모터를 활성화할 수 있지만, 대부분의 활성 인핸서는 표적 [14]프로모터의 활성화 중에 eRNA를 전사합니다.
2013년부터 2021년까지 발견된 eRNA의 기능
아래에 설명된 eRNA의 기능은 다양한 생물학적 시스템에서 보고되었으며, 종종 소수의 특정 증강제-대상 유전자 쌍으로 입증되었다.여기서 설명하는 eRNA의 기능이 대부분의 eRNA에 어느 정도까지 일반화 될 수 있는지는 명확하지 않다.
루프 형성 중인 eRNA

그림에 표시된 염색체 루프는 표적 유전자의 프로모터에 인핸서를 가져오는 것으로 인핸서가 활성화된 후 인핸서로부터 전사된 eRNA에 의해 지시되고 형성될 수 있다.
Atranscribed으로서 RNA(eRNA)Mediator의 단지로 상호 작용하는 프로틴(그림 보), 특히 Mediator 벅차12(MED12), 다정한 사이가 되는 강화제의 표적 유전자의 5유전자 라이(알에 의해 연구되어의 경우 프로모터로 강화제를 일으키는 염색체 고리 형성에 필수적일 것이라는.[15][16][17]Hou와 [18]Kraus는 유사한 결과를 보고하는 다른 두 연구를 설명한다.Arnold 등은 [19]eRNA가 인핸서-프로모터 루프를 형성하는 데 적극적인 다른 5가지 사례를 검토한다.
eRNA는 전사에 영향을 미치기 위해 단백질과 상호작용한다
잘 연구된 eRNA 중 하나는 전립선특이항원([20]PSA) 유전자의 프로모터와 상호작용하는 인핸서의 eRNA이다.PSA eRNA는 안드로겐 수용체에 의해 강하게 상향 조절된다.높은 PSA eRNA는 도미노 효과를 일으킨다.PSA eRNA는 RNA 중합효소 II(RNAP II)를 인산화할 수 있는 양성 전사 신장 인자 P-TEFb 단백질 복합체에 결합 및 활성화하여 mRNA 생성에 있어 그 활성을 개시한다. P-TEFb는 또한 음의 신장 인자 NELF(MNA 개시 후 60 뉴클레오티드 내에서 RNA II를 일시 정지)를 인산화할 수 있다.인산화 NELF는 RNA II에서 방출되어 RNA II가 생산적인 mRNA 진행을 갖도록 한다(그림 참조).따라서 상향조절된 PSA eRNA는 586개의 안드로겐 수용체 응답 유전자의 발현을 증가시킨다.PSA eRNA의 녹다운 또는 PSA eRNA에서 일련의 뉴클레오티드를 삭제하면 이러한 유전자에서 인산화(활성) RNAP II의 존재가 감소하여 전사가 감소한다.
음의 신장인자 NELF 단백질은 또한 일부 eRNA와의 직접적인 상호작용에 의해 RNA II와의 상호작용에서 방출될 수 있다.샤우코비치 외 연구진은 [21]두 개의 즉시 초기 유전자(IEG)의 eRNA가 NELF 단백질과 직접 상호작용하여 RNAP II에서 NELF를 방출함으로써 이 두 개의 유전자가 발현될 수 있음을 보여주었다.
또한, eRNA는 30개의 다른 [19][17][18]단백질과 상호작용하는 것으로 보인다.
2013년까지 제안된 기능 메커니즘
모든 증강제가 동시에 전사되는 것은 아니며 eRNA 전사가 증강제 특이적 활동과 상관된다는 개념은 개별 eRNA가 구별되고 관련성이 있는 생물학적 [3]기능을 가지고 있다는 생각을 뒷받침한다.그러나 eRNA의 기능적 중요성에 대해서는 아직 합의가 이루어지지 않았다.또한 eRNA는 엑소좀과 난센스 매개 붕괴를 통해 쉽게 분해될 수 있으며, 이는 중요한 전사 [22]조절제로서의 잠재력을 제한한다.지금까지 eRNA 기능의 네 가지 주요 모델이 [3]제안되었으며, 각각 다른 실험 증거 라인에 의해 뒷받침되었다.
문자 변환 노이즈
여러 연구에서 RNA Pol II가 매우 많은 수의 외부 유전자 영역에서 발견될 수 있다는 것이 밝혀졌기 때문에, eRNA는 단순히 무작위 "누출" 전사의 산물을 나타내며 기능적 의미를 [5]가지지 않을 수 있습니다.따라서 RNA Pol II의 비특이적 활성은 염색질이 이미 개방적이고 전사적으로 적합한 상태에 있는 부위에서 외부 유전자 전사 노이즈를 허용한다.개방된 부위도 조직 특이적이기 때문에 조직 특이적 eRNA[23] 발현도 설명할 수 있습니다.
전사의존효과
RNA Pol II 매개 유전자 전사는 에크로마틴 형성을 촉진하는 히스톤 아세틸전달효소 및 기타 히스톤 수식제들의 신병을 통해 크로마틴 상태의 국소 개방을 유도한다.이러한 효소의 존재는 또한 보통 먼 곳에 존재하지만 [24]DNA의 루프를 통해 유전자를 목표로 삼기 위해 모집될 수 있는 인핸서 영역에서 염색질의 개방을 유도할 수 있다고 제안되었다.따라서 이 모델에서 eRNA는 RNA Pol II 전사에 반응하여 발현되므로 생물학적 기능이 없다.
cis에서의 기능 활동
이전의 두 모델은 eRNA가 기능적으로 관련이 없음을 암시했지만, 이 메커니즘은 eRNA가 cis 활성을 보이는 기능 분자임을 명시한다.이 모델에서 eRNA는 자체 합성 부위에서 국소적으로 조절 단백질을 모집할 수 있다.이 가설을 뒷받침하는 것으로, 사이클린 D1 유전자의 상류에 있는 인핸서로부터 유래한 전사물은 히스톤아세틸전달효소의 신병을 위한 어댑터 역할을 하는 것으로 생각된다.이러한 eRNA의 고갈은 Cyclin D1 전사 [9]소음으로 이어졌다.
트랜스 기능 활동
마지막 모델은 먼 염색체 위치에서 eRNA에 의한 전사 조절을 포함한다.단백질 복합체의 차이적 모집을 통해, eRNA는 특정 위치의 전사 능력에 영향을 미칠 수 있다.Evf-2는 Dlx2의 발현을 유도하여 Dlx5 및 Dlx6 인핸서의 [25]활성을 높일 수 있는 트랜스 레귤레이터 eRNA의 좋은 예입니다.트랜스액션 eRNA는 cis에서도 동작할 수 있으며, 그 반대도 마찬가지입니다.
실험 검출
eRNA의 검출은 상당히 최근(2010년)이며, RNA 배열 분석(RNA-seq) 및 염색질 면역침강 배열 분석(ChIP-seq)[1]과 같은 게놈 전반의 조사 기법을 사용하여 가능해졌다.RNA-seq는 생체정보 [26]분석을 통해 검출된 전사물을 대응하는 인핸서 배열에 일치시킴으로써 eRNA의 직접적인 식별을 가능하게 한다.ChIP-seq는 인핸서 전사를 평가하는 덜 직접적인 방법을 나타내지만 활성 인핸서와 [27]관련된 특정 크로마틴 마크가 있기 때문에 중요한 정보를 제공할 수도 있습니다.일부 데이터는 여전히 논란의 여지가 있지만, 문헌에서는 H2AZ, H3K27ac 및 H3K4me3 [27][28][29]대비 H3K4me1의 높은 비율의 활성 증강제에서 히스톤 번역 후 수정의 최선의 조합이 이루어졌다는 데 의견이 일치한다.ChIP 실험은 또한 RNA Pol II를 인식하는 항체로 수행될 수 있으며, 이는 활성 전사 [5]부위에서 발견될 수 있다.eRNA의 실험적인 검출은 엑소좀 분해와 난센스 매개 [22]붕괴에 의해 부여되는 낮은 내인성 안정성으로 인해 복잡하다.높은 처리량 시퀀싱 기술을 사용한 런온 검사는 eRNA를 검출하는 또 다른 방법입니다.Precision Run-On Sequencing(PRO-seq)은 활발하게 전사되고 있으며 최대 1개의 [30]염기쌍의 분해능을 가진 RNA를 검출할 수 있습니다.그럼에도 불구하고 eRNA가 활성 증강제에서 발현되는 경향이 있다는 사실은 활성 증강제와 비활성 증강제를 구별하는 데 유용한 도구가 될 수 있다.
개발 및 질병에 미치는 영향
eRNA가 인핸서 활성화 및 유전자 전사의 효율성에 다운스트림 효과를 일으킨다는 증거는 eRNA의 기능적 능력과 잠재적 중요성을 시사한다.전사인자 p53은 p53 의존적인 방식으로 [31]인핸서 영역을 결합하고 eRNA를 생성하는 것으로 입증되었다.암에서 p53은 [32]종양의 50%에서 유전자의 돌연변이가 나타나면서 종양 억제에 중추적인 역할을 한다.이러한 p53 결합 인핸서 영역(p53BERs)은 세포 증식과 생존에 관여하는 여러 국소 및 원위 유전자 표적과 상호작용하는 것으로 나타났다.또한 p53 활성화에 의해 생성된 eRNA는BERs는 종양 억제 및 암에서 eRNA의 중요한 조절 역할을 나타내는 p53 표적 유전자의 효율적인 전사에 필요한 것으로 나타났다.
인핸서의 변이는 인간의 질병과 관련이 있지만 인핸서 활동을 조작하는 치료적 접근은 현재 가능하지 않다.eRNA의 출현으로 RNAi와 같은 강력한 치료 도구는 유전자 발현 장애를 목표로 하는 유망한 경로를 제공할 수 있다.
레퍼런스
- ^ a b c d e Kim TK, Hemberg M, Gray JM, Costa AM, Bear DM, Wu J, et al. (May 2010). "Widespread transcription at neuronal activity-regulated enhancers". Nature. 465 (7295): 182–7. Bibcode:2010Natur.465..182K. doi:10.1038/nature09033. PMC 3020079. PMID 20393465.
- ^ a b c d De Santa, Francesca; Barozzi, Iros; Mietton, Flore; Ghisletti, Serena; Polletti, Sara; Tusi, Betsabeh Khoramian; Muller, Heiko; Ragoussis, Jiannis; Wei, Chia-Lin; Natoli, Gioacchino (2010-05-11). "A large fraction of extragenic RNA pol II transcription sites overlap enhancers". PLOS Biology. 8 (5): e1000384. doi:10.1371/journal.pbio.1000384. ISSN 1545-7885. PMC 2867938. PMID 20485488.
- ^ a b c d Natoli G, Andrau JC (2012). "Noncoding transcription at enhancers: general principles and functional models". Annual Review of Genetics. 46: 1–19. doi:10.1146/annurev-genet-110711-155459. PMID 22905871.
- ^ Burren OS, Rubio García A, Javierre BM, Rainbow DB, Cairns J, Cooper NJ, et al. (September 2017). "Chromosome contacts in activated T cells identify autoimmune disease candidate genes". Genome Biology. 18 (1): 165. doi:10.1186/s13059-017-1285-0. PMC 5584004. PMID 28870212.
- ^ a b c d De Santa F, Barozzi I, Mietton F, Ghisletti S, Polletti S, Tusi BK, Muller H, Ragoussis J, Wei CL, Natoli G (May 2010). Mattick JS (ed.). "A large fraction of extragenic RNA pol II transcription sites overlap enhancers". PLOS Biology. 8 (5): e1000384. doi:10.1371/journal.pbio.1000384. PMC 2867938. PMID 20485488.
- ^ Heintzman ND, Stuart RK, Hon G, Fu Y, Ching CW, Hawkins RD, Barrera LO, Van Calcar S, Qu C, Ching KA, Wang W, Weng Z, Green RD, Crawford GE, Ren B (March 2007). "Distinct and predictive chromatin signatures of transcriptional promoters and enhancers in the human genome". Nature Genetics. 39 (3): 311–8. doi:10.1038/ng1966. PMID 17277777. S2CID 1595885.
- ^ a b Koch F, Fenouil R, Gut M, Cauchy P, Albert TK, Zacarias-Cabeza J, Spicuglia S, de la Chapelle AL, Heidemann M, Hintermair C, Eick D, Gut I, Ferrier P, Andrau JC (July 2011). "Transcription initiation platforms and GTF recruitment at tissue-specific enhancers and promoters". Nature Structural & Molecular Biology. 18 (8): 956–63. doi:10.1038/nsmb.2085. PMID 21765417. S2CID 12778976.
- ^ Fedoseeva DM, Kretova OV, Tchurikov NA (2012). "Molecular analysis of enhancer RNAs and chromatin modifications in the region of their synthesis in Drosophila cells possessing genetic constructs". Doklady. Biochemistry and Biophysics. 442: 7–11. doi:10.1134/S1607672912010012. PMID 22419084. S2CID 14304312.
- ^ a b Wang X, Arai S, Song X, Reichart D, Du K, Pascual G, Tempst P, Rosenfeld MG, Glass CK, Kurokawa R (July 2008). "Induced ncRNAs allosterically modify RNA-binding proteins in cis to inhibit transcription". Nature. 454 (7200): 126–30. Bibcode:2008Natur.454..126W. doi:10.1038/nature06992. PMC 2823488. PMID 18509338.
- ^ Kowalczyk MS, Hughes JR, Garrick D, Lynch MD, Sharpe JA, Sloane-Stanley JA, McGowan SJ, De Gobbi M, Hosseini M, Vernimmen D, Brown JM, Gray NE, Collavin L, Gibbons RJ, Flint J, Taylor S, Buckle VJ, Milne TA, Wood WG, Higgs DR (February 2012). "Intragenic enhancers act as alternative promoters". Molecular Cell. 45 (4): 447–58. doi:10.1016/j.molcel.2011.12.021. PMID 22264824.
- ^ Wang D, Garcia-Bassets I, Benner C, Li W, Su X, Zhou Y, Qiu J, Liu W, Kaikkonen MU, Ohgi KA, Glass CK, Rosenfeld MG, Fu XD (May 2011). "Reprogramming transcription by distinct classes of enhancers functionally defined by eRNA". Nature. 474 (7351): 390–4. doi:10.1038/nature10006. PMC 3117022. PMID 21572438.
- ^ Arner E, Daub CO, Vitting-Seerup K, Andersson R, Lilje B, Drabløs F, Lennartsson A, Rönnerblad M, Hrydziuszko O, Vitezic M, Freeman TC, Alhendi AM, Arner P, Axton R, Baillie JK, Beckhouse A, Bodega B, Briggs J, Brombacher F, Davis M, Detmar M, Ehrlund A, Endoh M, Eslami A, Fagiolini M, Fairbairn L, Faulkner GJ, Ferrai C, Fisher ME, Forrester L, Goldowitz D, Guler R, Ha T, Hara M, Herlyn M, Ikawa T, Kai C, Kawamoto H, Khachigian LM, Klinken SP, Kojima S, Koseki H, Klein S, Mejhert N, Miyaguchi K, Mizuno Y, Morimoto M, Morris KJ, Mummery C, Nakachi Y, Ogishima S, Okada-Hatakeyama M, Okazaki Y, Orlando V, Ovchinnikov D, Passier R, Patrikakis M, Pombo A, Qin XY, Roy S, Sato H, Savvi S, Saxena A, Schwegmann A, Sugiyama D, Swoboda R, Tanaka H, Tomoiu A, Winteringham LN, Wolvetang E, Yanagi-Mizuochi C, Yoneda M, Zabierowski S, Zhang P, Abugessaisa I, Bertin N, Diehl AD, Fukuda S, Furuno M, Harshbarger J, Hasegawa A, Hori F, Ishikawa-Kato S, Ishizu Y, Itoh M, Kawashima T, Kojima M, Kondo N, Lizio M, Meehan TF, Mungall CJ, Murata M, Nishiyori-Sueki H, Sahin S, Nagao-Sato S, Severin J, de Hoon MJ, Kawai J, Kasukawa T, Lassmann T, Suzuki H, Kawaji H, Summers KM, Wells C, Hume DA, Forrest AR, Sandelin A, Carninci P, Hayashizaki Y (February 2015). "Transcribed enhancers lead waves of coordinated transcription in transitioning mammalian cells". Science. 347 (6225): 1010–4. Bibcode:2015Sci...347.1010A. doi:10.1126/science.1259418. PMC 4681433. PMID 25678556.
- ^ a b Carullo NV, Phillips Iii RA, Simon RC, Soto SA, Hinds JE, Salisbury AJ, Revanna JS, Bunner KD, Ianov L, Sultan FA, Savell KE, Gersbach CA, Day JJ (September 2020). "Enhancer RNAs predict enhancer-gene regulatory links and are critical for enhancer function in neuronal systems". Nucleic Acids Res. 48 (17): 9550–9570. doi:10.1093/nar/gkaa671. PMC 7515708. PMID 32810208.
- ^ Mikhaylichenko O, Bondarenko V, Harnett D, Schor IE, Males M, Viales RR, Furlong EE (January 2018). "The degree of enhancer or promoter activity is reflected by the levels and directionality of eRNA transcription". Genes Dev. 32 (1): 42–57. doi:10.1101/gad.308619.117. PMC 5828394. PMID 29378788.
- ^ Lai F, Orom UA, Cesaroni M, Beringer M, Taatjes DJ, Blobel GA, Shiekhattar R (February 2013). "Activating RNAs associate with Mediator to enhance chromatin architecture and transcription". Nature. 494 (7438): 497–501. Bibcode:2013Natur.494..497L. doi:10.1038/nature11884. PMC 4109059. PMID 23417068.
- ^ Allen BL, Taatjes DJ (March 2015). "The Mediator complex: a central integrator of transcription". Nat Rev Mol Cell Biol. 16 (3): 155–66. doi:10.1038/nrm3951. PMC 4963239. PMID 25693131.
- ^ a b Sartorelli V, Lauberth SM (June 2020). "Enhancer RNAs are an important regulatory layer of the epigenome". Nat Struct Mol Biol. 27 (6): 521–528. doi:10.1038/s41594-020-0446-0. PMC 7343394. PMID 32514177.
- ^ a b Hou TY, Kraus WL (February 2021). "Spirits in the Material World: Enhancer RNAs in Transcriptional Regulation". Trends Biochem Sci. 46 (2): 138–153. doi:10.1016/j.tibs.2020.08.007. PMC 7855021. PMID 32888773.
- ^ a b Arnold PR, Wells AD, Li XC (2019). "Diversity and Emerging Roles of Enhancer RNA in Regulation of Gene Expression and Cell Fate". Front Cell Dev Biol. 7: 377. doi:10.3389/fcell.2019.00377. PMC 6971116. PMID 31993419.
- ^ Zhao Y, Wang L, Ren S, Wang L, Blackburn PR, McNulty MS, Gao X, Qiao M, Vessella RL, Kohli M, Zhang J, Karnes RJ, Tindall DJ, Kim Y, MacLeod R, Ekker SC, Kang T, Sun Y, Huang H (April 2016). "Activation of P-TEFb by Androgen Receptor-Regulated Enhancer RNAs in Castration-Resistant Prostate Cancer". Cell Rep. 15 (3): 599–610. doi:10.1016/j.celrep.2016.03.038. PMC 5395199. PMID 27068475.
- ^ Schaukowitch K, Joo JY, Liu X, Watts JK, Martinez C, Kim TK (October 2014). "Enhancer RNA facilitates NELF release from immediate early genes". Mol Cell. 56 (1): 29–42. doi:10.1016/j.molcel.2014.08.023. PMC 4186258. PMID 25263592.
- ^ a b Wyers F, Rougemaille M, Badis G, Rousselle JC, Dufour ME, Boulay J, Régnault B, Devaux F, Namane A, Séraphin B, Libri D, Jacquier A (June 2005). "Cryptic pol II transcripts are degraded by a nuclear quality control pathway involving a new poly(A) polymerase". Cell. 121 (5): 725–37. doi:10.1016/j.cell.2005.04.030. PMID 15935759.
- ^ Ren B (May 2010). "Transcription: Enhancers make non-coding RNA". Nature. 465 (7295): 173–4. Bibcode:2010Natur.465..173R. doi:10.1038/465173a. PMID 20463730.
- ^ Obrdlik A, Kukalev A, Louvet E, Farrants AK, Caputo L, Percipalle P (October 2008). "The histone acetyltransferase PCAF associates with actin and hnRNP U for RNA polymerase II transcription". Molecular and Cellular Biology. 28 (20): 6342–57. doi:10.1128/MCB.00766-08. PMC 2577438. PMID 18710935.
- ^ Feng J, Bi C, Clark BS, Mady R, Shah P, Kohtz JD (June 2006). "The Evf-2 noncoding RNA is transcribed from the Dlx-5/6 ultraconserved region and functions as a Dlx-2 transcriptional coactivator". Genes & Development. 20 (11): 1470–84. doi:10.1101/gad.1416106. PMC 1475760. PMID 16705037.
- ^ Wang Z, Gerstein M, Snyder M (January 2009). "RNA-Seq: a revolutionary tool for transcriptomics". Nature Reviews. Genetics. 10 (1): 57–63. doi:10.1038/nrg2484. PMC 2949280. PMID 19015660.
- ^ a b Barski A, Cuddapah S, Cui K, Roh TY, Schones DE, Wang Z, Wei G, Chepelev I, Zhao K (May 2007). "High-resolution profiling of histone methylations in the human genome". Cell. 129 (4): 823–37. doi:10.1016/j.cell.2007.05.009. PMID 17512414.
- ^ Melgar MF, Collins FS, Sethupathy P (November 2011). "Discovery of active enhancers through bidirectional expression of short transcripts". Genome Biology. 12 (11): R113. doi:10.1186/gb-2011-12-11-r113. PMC 3334599. PMID 22082242.
- ^ Creyghton MP, Cheng AW, Welstead GG, Kooistra T, Carey BW, Steine EJ, Hanna J, Lodato MA, Frampton GM, Sharp PA, Boyer LA, Young RA, Jaenisch R (December 2010). "Histone H3K27ac separates active from poised enhancers and predicts developmental state". Proceedings of the National Academy of Sciences of the United States of America. 107 (50): 21931–6. doi:10.1073/pnas.1016071107. PMC 3003124. PMID 21106759.
- ^ Mahat DB, Kwak H, Booth GT, Jonkers IH, Danko CG, Patel RK, Waters CT, Munson K, Core LJ, Lis JT (August 2016). "Base-pair-resolution genome-wide mapping of active RNA polymerases using precision nuclear run-on (PRO-seq)". Nature Protocols. 11 (8): 1455–76. doi:10.1038/nprot.2016.086. PMC 5502525. PMID 27442863.
- ^ Melo CA, Drost J, Wijchers PJ, van de Werken H, de Wit E, Oude Vrielink JA, Elkon R, Melo SA, Léveillé N, Kalluri R, de Laat W, Agami R (February 2013). "eRNAs are required for p53-dependent enhancer activity and gene transcription". Molecular Cell. 49 (3): 524–35. doi:10.1016/j.molcel.2012.11.021. PMID 23273978.
- ^ Vousden KH, Lu X (August 2002). "Live or let die: the cell's response to p53". Nature Reviews. Cancer. 2 (8): 594–604. doi:10.1038/nrc864. PMID 12154352. S2CID 6412605.