마이크로RNA
microRNA마이크로RNA(약칭 miRNA)는 식물, 동물 및 일부 바이러스에서 발견되는 작은 단일사슬 비코드 RNA 분자(약 22개의 뉴클레오티드를 포함한다)로, RNA 사일런트화 및 유전자 [1][2]발현의 전사 후 조절에서 기능한다.miRNA는 mRNA [3]분자 내에서 상보적인 염기쌍을 통해 기능한다.그 결과, 이들 mRNA 분자는 (1) mRNA 가닥의 2분할, (2) 폴리(A) 꼬리의 단축에 의한 mRNA의 불안정화, (3) 리보솜에 [3][4]의한 단백질로의 mRNA의 저효율화 중 하나 이상의 프로세스에 의해 무음화된다.
miRNA는 RNA 간섭 경로의 작은 간섭 RNA(siRNA)와 유사하지만, miRNA는 짧은 머리핀을 형성하기 위해 뒤로 접히는 RNA 전사 영역에서 파생되는 반면, siRNA는 이중 가닥 [5]RNA의 긴 영역에서 파생됩니다.인간 게놈은 1900개 이상의 miRNA를 [6]부호화할 수 있지만, 더 최근의 분석에서는 그 [7]수가 2,300개에 가깝다고 한다.그러나 수동 경화 miRNA 유전자 데이터베이스 MirGeneDB에서는 [8]인간 마이크로RNA가 약 500개만 진정한 miRNA를 나타낸다.
miRNA는 많은 포유동물 세포[9][10] 유형 및 세포 외 순환 miRNA로 [11]풍부합니다.순환하는 miRNA는 혈액과 뇌척수액을 포함한 체액에 방출되어 많은 질병에서 [11][12]바이오마커로 이용될 가능성이 있다.MiRNA는 인간과 다른 [13][14]포유류의 유전자의 약 60%를 목표로 하는 것으로 보인다.많은 miRNA는 진화적으로 보존되는데, 이것은 그들이 중요한 생물학적 [15][1]기능을 가지고 있다는 것을 의미한다.예를 들어, 90개 과의 miRNA는 적어도 포유류와 어류의 공통 조상 때부터 보존되어 왔고, 이러한 보존된 miRNA의 대부분은 중요한 기능을 가지고 있는데, 이는 한 과의 한 명 이상의 유전자가 [1]쥐에서 녹아웃된 연구에서 알 수 있다.
역사
최초의 miRNA는 1990년대 [16]초에 발견되었다.그러나 miRNAs 생물학적 조정자들 뚜렷한 클래스로 2000년대 초반까지 인식되지 않았다.[17][18][19][20][21]miRNA 연구 miRNAs의 다른 형태를 다양한 종류의 세포와 tissues[10][22], 식물과 동물 개발과 많은 다른 생물학적 과정에서 miRNAs에 다양한 역할로 표현된다고 밝혔다.[23][24][25][26][27][28][29]이상 miRNA 발현은 질병 상태와 관련이 있다.MiRNA 기반의 치료법이 [30][31][32][33]연구되고 있다.
첫 번째 miRNA는 1993년 이 씨와 파인바움 씨를 포함한 암브로스가 이끄는 그룹에 의해 발견되었다.그러나 동작 방식에 대한 추가적인 통찰은 Wightman과 [16][34]Ha를 포함한 Ruvkun의 팀이 동시에 발표한 작업을 필요로 했습니다.이들 그룹은 린-14 유전자를 억제함으로써 엘레강스의 애벌레 발생 시기를 조절하는 것으로 알려진 린-4 유전자에 대한 연속 논문을 발표했다.Lee 등이 lin-4 miRNA를 분리했을 때, 그들은 단백질을 코드하는 mRNA를 생성하는 대신 짧은 비코드 RNA를 생성했으며, 그 중 하나는 lin-14 MRNA의 [16]3' UTR의 여러 배열에 부분적으로 상보적인 배열을 포함하는 ~22-뉴클레오티드 RNA였다.이러한 상호보완성은 LIN-14 mRNA가 LIN-14 단백질로 변환되는 것을 억제하기 위해 제안되었다.그 당시, lin-4 작은 RNA는 선충 특이성으로 생각되었다.
2000년, 두 번째 작은 RNA가 특징지어졌습니다: let-7 RNA는 C. elegans의 [17]후발달 전이를 촉진하기 위해 lin-41을 억제합니다.let-7 RNA는 많은 종에서 보존되는 것으로 밝혀졌고, [18]let-7 RNA와 추가적인 "작은 시간 RNA"가 인간을 포함한 다양한 동물들의 발달 시기를 조절할 수 있다는 제안을 이끌어냈다.
1년 후, line-4와 let-7 RNA는 C. 엘레건, 드로소필라, 인간 [19][20][21]세포에 존재하는 많은 종류의 작은 RNA의 일부인 것으로 밝혀졌다.이 클래스의 많은 RNA는 발현 패턴이 발달 시기를 조절하는 역할과 일치하지 않는다는 점을 제외하고는 lin-4 및 let-7 RNA와 유사했다.이는 대부분이 다른 유형의 규제 경로에서 기능할 수 있음을 시사했다.이 시점에서, 연구자들은 "마이크로 RNA"[19][20][21]라는 용어를 이러한 종류의 작은 조절 RNA를 지칭하기 시작했다.
miRNA의 규제완화와 관련된 첫 번째 인간질환은 만성 림프구성 백혈병이었다.이 장애에서, miRNA는 종양 억제제와 종양 발생원 [35]둘 다로 작용하는 두 가지 역할을 한다.
명명법
표준 명명법 체계에서는 발표 [36][37]전에 실험적으로 확인된 miRNA에 이름이 할당된다.접두사 "miR" 뒤에 대시와 숫자가 오고, 대시는 종종 명명 순서를 나타냅니다.예를 들어 miR-124는 miR-456보다 먼저 이름이 붙여져 검출되었을 가능성이 있습니다.대문자 "miR-"는 miRNA의 성숙한 형태를 나타내며, 자본화되지 않은 "mir-"는 miRNA [38]및 pri-miRNA를 나타냅니다.또한 유전자를 코드하는 miRNA는 유기체 유전자 명명 규칙에 따라 동일한 세 글자의 접두사를 사용하여 명명됩니다.예를 들어, 일부 유기체의 공식 miRNA 유전자 이름은 "C. elegans와 Drosophila의 mir-1, Rattus norveicus의 Mir-1, 인간의 MIR-25이다.
하나 또는 두 개의 뉴클레오티드를 제외하고 거의 동일한 배열을 가진 miRNA에는 추가 소문자가 주석을 달았다.예를 들어 miR-124a는 miR-124b와 밀접하게 관련되어 있습니다.예를 들어 다음과 같습니다.
- hsa-miR-181a: aacauucaACgcugucggugAgu
- hsa-miR-181b: aacauucaUUgcugucggugGgu
100% 동일한 성숙한 miRNA를 유도하지만 게놈의 다른 위치에 위치한 프리미RNA, pri-miRNA 및 유전자는 추가 대시 번호 접미사로 표시된다.예를 들어 pre-miRNAs hsa-mir-194-1 및 hsa-mir-194-2는 동일한 성숙한 miRNA(hsa-miR-194)를 발생시키지만 다른 게놈 영역에 위치한 유전자에서 유래한다.
원산지는 3글자 접두사로 지정되며, 예를 들어 hsa-miR-124는 인간(호모 사피엔스) miRNA, oar-miR-124는 양(Ovis aries) miRNA이다.다른 일반적인 접두사에는 바이러스의 "v"와 Drosophila miRNA의 "d"가 있습니다.
두 개의 성숙한 마이크로RNA가 동일한 사전 miRNA의 반대쪽 팔에서 유래하여 대략 비슷한 양으로 발견될 경우, 이들은 -3p 또는 -5p 접미사로 표시된다(과거에는 "s"(의식)와 "as"(반감)로 구분되기도 했다).그러나 머리핀의 한쪽 팔에서 발견되는 성숙한 마이크로RNA는 다른 쪽 [5]팔에서 발견되는 것보다 훨씬 더 풍부합니다. 이 경우, 이름 뒤에 나오는 별표는 머리핀의 반대쪽 팔에서 낮은 수준에서 발견되는 성숙한 종을 나타냅니다.예를 들어 miR-124와 miR-124*는 miRNA 전 헤어핀을 공유하지만 miR-124는 세포 내에서 훨씬 더 많이 발견됩니다.
대상
식물의 miRNA는 보통 mRNA 표적과 거의 완벽한 쌍을 이루는데, 이것은 표적 [23][39]전사의 분열을 통해 유전자 억제를 유도한다.이와는 대조적으로, 동물 miRNA는 [13][40][41]miRNA의 5' 말단에서 6~8개의 뉴클레오티드(종자 영역)를 사용하여 표적 mRNA를 인식할 수 있으며, 이는 표적 mRNA의 [3]분열을 유도하기에 충분한 쌍이 아니다.조합 조절은 [3][42]동물에서 miRNA 조절의 특징이다.특정 miRNA는 수백 개의 다른 mRNA 표적을 가질 수 있으며, 특정 표적은 여러 miRNA에 [14][43]의해 조절될 수 있다.
일반적인 miRNA에 의한 억제 대상이 되는 고유한 메신저 RNA의 평균 개수의 [44]추정치는 추정 방법에 따라 다르지만, 여러 접근법에 따르면 포유류의 miRNA는 많은 고유한 대상을 가질 수 있다.예를 들어 척추동물에서 고도로 보존된 miRNA의 분석은 각각 평균 약 400개의 보존된 [14]표적을 가지고 있음을 보여준다.마찬가지로, 실험은 단일 miRNA 종이 수백 개의 독특한 메신저 RNA의 안정성을 [45]감소시킬 수 있다는 것을 보여준다.다른 실험들은 단일 miRNA 종이 수백 개의 단백질 생성을 억제할 수 있지만, 이러한 억제 효과는 종종 비교적 경미하다는 것을 보여준다.[46][47]miRNA의 규제완화와 관련이 있는 것으로 밝혀진 첫 번째 인간질환은 만성 림프구성 백혈병이었다.다른 B세포 악성종양이 뒤따랐다.
생물 생성
miRNA 유전자의 40%가 다른 [48]유전자의 인트론 또는 엑손에 있을 수 있다.이것들은, 배타적이지는 않지만, 보통 의미 [49][50]방향에서 발견되고, 따라서 보통 그들의 숙주 [48][51][52]유전자와 함께 조절된다.
DNA 템플릿은 성숙한 miRNA 생산에 대한 최종 단어는 아닙니다. 인간 miRNA의 6%는 DNA에 의해 암호화된 것과 다른 RNA 서열의 부위별 수정인 RNA 편집(IsomiRs)을 보여줍니다.이로 인해 miRNA 작용의 다양성과 범위가 게놈에서만 나타나는 것보다 더 넓어집니다.
문자 변환
miRNA 유전자는 보통 RNA 중합효소 II(Pol II)[53][54]에 의해 전사된다.중합효소는 종종 DNA 배열 근처에서 발견되는 프로모터에 결합하여 무엇이 전 miRNA의 머리핀 고리가 될 것인지를 코드화한다.그 결과 생성된 전사물은 5' 말단에서 특수하게 변형된 뉴클레오티드로 덮이고, 다중 아데노신(폴리(A)[53][49] 꼬리)으로 처리된 폴리아데닐화 및 스플라이싱된다.동물 miRNA는 처음에 pri-miRNA라고 불리는 수백 개의 nucleotide-length miRNA [53][49]전구체의 일부를 형성하는 약 80개의 nucleotide RNA 스템 루프의 일부로서 전사된다.3' UTR에서 스템루프 전구체가 발견되면 전사체는 pri-miRNA [49]및 mRNA로 기능할 수 있다.RNA 중합효소 III(Pol III)는 일부 miRNA, 특히 업스트림 Alu 배열을 가진 miRNA, 전달 RNA(tRNAs) 및 포유류의 광역산재 반복 프로모터([55]MWIR) 단위로 전사한다.
핵처리
단일 pri-miRNA는 1~6개의 miRNA 전구체를 포함할 수 있다.이 헤어핀 루프 구조는 각각 약 70개의 뉴클레오티드로 구성되어 있습니다.각 머리핀은 효율적인 처리에 필요한 배열로 양옆에 배치되어 있다.
pri-miRNA에서 머리핀의 이중가닥 RNA(dsRNA) 구조는 디조지 증후군과의 연관성으로 이름 붙여진 무척추동물에서 디조지 증후군 임계 영역 8(DGCR8 또는 "파샤")로 알려진 핵단백질에 의해 인식된다.DGCR8은 마이크로프로세서 [56][57]복합체를 형성하기 위해 RNA를 절단하는 단백질인 Drosha 효소와 결합합니다.이 복합체에서 DGCR8은 Drosha의 촉매 RNase III 도메인을 오리엔테이션하여 머리핀 베이스로부터 약 11개의 뉴클레오티드를 절단함으로써 머리핀을 pri-miRNA로부터 해방시킨다(하나의 나선형 dsRNA는 [58][59]줄기로 변화).결과물은 3' 말단에 2-뉴클레오티드 돌출부가 있으며, 3' 하이드록실기와 5' 인산기를 가지고 있다.이것은 종종 pre-miRNA(precursor-miRNA)라고 불린다.효율적인 처리에 중요한 사전 miRNA의 하류 배열 모티브가 확인되었다.[60][61][62]
마이크로프로세서 콤플렉스를 우회하여 인트론에서 직접 스플라이스된 프리미리온(pre-miRNA)을 "미트론"이라고 합니다.원래는 드로소필라와 엘레강스에만 존재한다고 생각되었던 미르톤은 이제 [63]포유동물에서 발견되었다.
핵 RNA [64][65][66]편집을 통해 사전 miRNA의 16%가 변경될 수 있습니다.가장 일반적으로, RNA에 작용하는 아데노신 탈아미나아제(ADARs)로 알려진 효소는 아데노신을 이노신(A에서 I) 전환으로 촉매한다.RNA 편집은 핵처리(예를 들어 리보핵산가수분해효소 Tudor-SN에 의한 분해로 이어지는 pri-miR-142)를 중지하고 세포질 miRNA 처리 및 표적 특이성을 포함한 하류 프로세스(예를 들어 [64]중추신경계에서의 miR-376의 종자 영역 변경)를 변경할 수 있다.
핵수출
핵세포질 셔터 Exportin-5를 수반하는 공정에서 miRNA 전 헤어핀을 핵에서 내보낸다.카리오페린 계열의 이 단백질은 miRNA 전 머리핀의 3' 말단에 RNAase III 효소 Drosha가 남긴 2-뉴클레오티드 돌출부를 인식한다.세포질로의 [67]exportin-5 매개 전달은 Ran 단백질에 결합된 구아노신 삼인산(GTP)을 사용하여 에너지 의존적이다.
세포질 처리
이 세포질에서 프리미RNA 헤어핀은 RNase III 효소 Dicer에 [68]의해 절단된다.이 엔도리보핵산가수분해효소는 머리핀의[69] 5' 및 3' 말단과 상호작용하여 3' 및 5' 팔과 결합하는 루프를 절단하여 길이가 [68]약 22 뉴클레오티드인 불완전한 miRNA:miRNA* 이중화를 생성한다.전체적인 머리핀 길이와 루프 크기는 다이서 가공의 효율에 영향을 미칩니다.miRNA:miRNA*[68][70] 쌍의 불완전한 특성도 분할에 영향을 미칩니다.G가 풍부한 프리미RNA 중 일부는 표준 스템 루프 구조의 대안으로 G-쿼드플렉스 구조를 잠재적으로 채택할 수 있다.예를 들어 인간 프리미RNA92b는 세포질 중 [71]다이서 매개분할에 내성이 있는 G-쿼드플렉스 구조를 채용하고 있다.듀플렉스의 어느 한 가닥이 기능성 miRNA로 작용할 수 있지만, 보통 miRNA와 그 mRNA 타겟이 상호작용하는 RNA 유도 사일링 복합체(RISC)에 1 가닥만 포함된다.
대부분의 miRNA가 세포 내에 위치하는 반면, 일반적으로 순환하는 miRNA 또는 세포외 miRNA로 알려진 일부 miRNA는 다양한 생물학적 액체와 세포외 배양 [72][73]배지를 포함한 세포외 환경에서도 발견되었다.
식물의 생물 생성
식물의 miRNA 생물 형성은 주로 핵 처리와 수출 단계에서 동물 생물 형성과 다르다.두 개의 다른 효소에 의해 분해되는 대신, 식물의 miRNA의 분할은 Dicer-like1 (DL1)이라고 불리는 Dicer 호몰로그에 의해 수행됩니다.DL1은 식물 세포의 핵에서만 발현되며, 이는 두 반응이 모두 핵 안에서 일어난다는 것을 나타낸다.식물의 miRNA:miRNA* 듀플렉스가 핵 밖으로 운반되기 전에, 그 3' 돌출부는 Hua-Enhancer1(HEN1)이라는 RNA 메틸전달효소 단백질에 의해 메틸화된다.그런 다음 이중성은 Exportin 5 호몰로그(Homolog)라고 불리는 단백질에 의해 핵에서 세포질로 운반되며, 여기서 분해되고 성숙한 miRNA가 RISC에 [74]통합됩니다.
RNA유기사일런트복합체
성숙한 miRNA는 Dicer와 많은 관련 [75]단백질을 포함하는 활성 RNA 유도 사일런싱 복합체(RISC)의 일부이다.RISC는 마이크로RNA 리보핵단백질복합체(miRNP)[76]로도 알려져 있으며, miRNA가 통합된 RISC는 때때로 "miRISC"라고 불립니다.
pre-miRNA의 다이서 처리는 듀플렉스의 풀림과 결합되어 있다고 생각됩니다.일반적으로 miRISC에는 열역학적 불안정성과 다른 [77][78][79]한 가닥에 비해 5' 끝의 약한 염기쌍을 기준으로 한 가닥만 통합된다.스템 루프의 위치도 스트랜드 [80]선택에 영향을 줄 수 있습니다.정상 상태에서 레벨이 낮기 때문에 승객용 스트랜드라고 불리는 다른 스트랜드는 별표(*)로 표시되며 일반적으로 열화됩니다.어떤 경우, 이중성의 두 가닥은 생존 가능하며 다른 mRNA [81]모집단을 대상으로 하는 기능성 miRNA가 된다.
Argonaute(Ago) 단백질 패밀리의 구성원은 RISC 기능의 중심입니다.아르고네이트는 miRNA 유도 사일런싱에 필요하며, 성숙한 miRNA의 단일 가닥 3' 말단을 결합할 수 있는 PAZ 도메인과 구조적으로 리보핵산가수분해효소-H와 유사하며 가이드 가닥의 5' 말단과 상호작용하는 기능인 PIWI 도메인 두 개의 보존 RNA 결합 도메인을 포함한다.그들은 성숙한 miRNA를 결합하고 표적 mRNA와의 상호작용을 위해 방향을 잡는다.예를 들어 인간 아고네이트는 표적 전사를 직접 절단한다.아르고네이트는 번역 [82]억제를 달성하기 위해 추가 단백질을 모집할 수도 있다.인간 게놈은 배열 유사성에 의해 두 개의 패밀리로 나뉘어진 8개의 아르고네이트 단백질을 암호화한다: AGO (모든 포유동물 세포에 [76][82]4개의 구성원을 가지고 있고 인간의 경우 E1F2C/hAgo라고 불린다)와 PIWI (배아줄기와 조혈모세포에서 발견됨).
추가 리스크 요소 TRBP[인체 면역 결핍 바이러스(HIV)전사 촉진하는. 반응 RNA(TAR)결합 단백질],[83]PACT(그interferon-induced의 단백질 단백질 활성기 인산화 효소), SMN 단지, 곧 사라진 X정신 지체 단백질(FMRP), 튜더 포도상 구균 nuclease-domain-containing 단백질(Tudor-SN), 추정 상의 DNAhel을 포함한다.icas단백질 TNRC6B를 [67][84][85]포함한 RNA 인식 모티브.
소음 모드 및 규제 루프
유전자 사일런싱은 mRNA 분해 또는 mRNA 번역을 방해함으로써 발생할 수 있다.예를 들어 miR16은 3'에서 발견된 AU가 풍부한 원소를 보완하는 배열을 포함한다.TNF 알파 [86]또는 GM-CSF와 같은 많은 불안정한 mRNA의 UTR.miRNA와 표적 mRNA 배열 사이에 완전한 상보성이 주어졌을 때, Ago2는 mRNA를 분해하여 직접 mRNA 분해로 이어질 수 있다는 것이 입증되었다.상호보완성이 없는 경우 변환을 [45]방지함으로써 소음화를 달성할 수 있습니다.miRNA와 대상 mRNA의 관계는 대상 mRNA의 단순한 음성 조절에 기초할 수 있지만, 일반적인 시나리오는 "일관성 피드포워드 루프", "상호 네거티브 피드백 루프" 및 "양성 피드백/피드포워드 루프"를 사용하는 것으로 보인다.일부 miRNA는 전사, 번역 및 단백질 안정성의 확률적 사건으로 인해 발생하는 무작위 유전자 발현 변화의 완충제 역할을 한다.이러한 조절은 일반적으로 mRNA 전사로부터 출력되는 단백질의 음의 피드백 루프 또는 일관되지 않은 피드 포워드 루프에 의해 달성된다.
이직률.
miRNA 발현 프로파일의 급격한 변화를 위해서는 성숙한 miRNA의 교체가 필요하다.세포질에서 miRNA가 성숙하는 동안, Argonaute 단백질에 의한 흡수는 가이드 가닥을 안정시키는 반면, 반대편(* 또는 "승객") 가닥은 우선적으로 파괴된다."사용 또는 손실" 전략이라고 불리는 전략에서, 아르고네이트는 타겟이 적거나 전혀 없는 miRNA보다 타겟이 많은 miRNA를 우선적으로 보유할 수 있으며, 이는 비타겟팅 [87]분자의 분해를 초래할 수 있다.
케노하브도염 엘레강스에서 성숙한 miRNA의 붕괴는 Rat1p로도 [88]알려진 5'-3' 엑시보핵산가수분해효소 XRN2에 의해 매개된다.식물에서 SDN(Small RNA decreading nuclease) 패밀리 구성원은 miRNA를 반대(3'to-5') 방향으로 분해한다.유사한 효소가 동물의 게놈에 암호화되어 있지만, 그 역할은 [87]아직 밝혀지지 않았다.
몇 가지 miRNA 수정은 miRNA 안정성에 영향을 미칩니다.모델 생물 Arabidopsis thaliana(Thale cress)의 연구에서 알 수 있듯이 성숙한 식물 miRNA는 3' 말단에 메틸기를 첨가함으로써 안정화되는 것으로 보인다.2'-O 결합 메틸기는 miRNA 분해와 관련될 수 있는 변형인 우리딜전달효소(Uridyl transferase) 효소에 의한 우라실(U) 잔기의 첨가를 차단한다.그러나 요실화는 또한 일부 miRNA를 보호할 수 있다. 이러한 변경의 결과는 완전히 파악되지 않았다.일부 동물의 miRNA의 요릴화가 보고되었다.miRNA의 3' 말단에 아데닌(A) 잔류물을 첨가함으로써 식물 및 동물 miRNA를 모두 변경할 수 있다.C형 간염에서 중요한 간농축 miRNA인 포유동물 miR-122의 말단에 첨가된 추가 A는 분자를 안정시키고 아데닌 잔기로 끝나는 식물 miRNA는 붕괴 [87]속도가 느리다.
셀룰러 기능

miRNA의 기능은 유전자 조절에 있는 것으로 보인다.이를 위해 miRNA는 하나 이상의 메신저 RNA(mRNA)의 일부에 상보적이다.동물 miRNA는 일반적으로 3' UTR의 부위와 보완적인 반면, 식물 miRNA는 일반적으로 mRNA의 [90]코딩 영역을 보완한다.표적 RNA와의 완전 또는 거의 완벽한 염기쌍은 [91]RNA의 분열을 촉진한다.이것이 식물의 miRNA의 [92]주요 모드입니다.동물에게는 짝짓기가 불완전하다.
부분보완 마이크로RNA가 대상을 인식하려면 miRNA의 뉴클레오티드 2-7('씨 영역')[13][40]이 완벽하게 [93]상호보완적이어야 한다.동물 miRNA는 표적 mRNA의 단백질 번역을 억제한다(이는 존재하지만 [92]식물에서는 덜 흔하다).부분적으로 상보적인 마이크로RNA는 데데닐화를 가속화하여 mRNA를 더 [95]빨리 분해할 수 있습니다.miRNA 표적 mRNA의 열화는 잘 문서화되어 있지만 mRNA 열화, 번역 억제 또는 둘의 조합에 의해 번역 억제가 달성되는지 여부는 뜨거운 논쟁이다.제브라피쉬의 miR-430과 드로소필라 배양세포의 bantam-miRNA 및 miR-9에 대한 최근 연구는 번역 억제가 mRNA [96][97]데데데닐화와는 무관하게 번역 개시의 중단에 의해 발생한다는 것을 보여준다.
또한 miRNA는 때때로 표적 [98][99]유전자의 발현에 영향을 미치는 프로모터 부위의 히스톤 수정과 DNA 메틸화를 일으킨다.
miRNA 작용의 9가지 메커니즘이 설명되고 통합된 수학적 [89]모델로 조립된다.
- Cap-40S 개시 억제
- 60S 리보솜 단위 결합 억제
- 신장 억제
- 리보솜 드롭오프(사전 종료);
- 동시 번역 초기 단백질 분해
- P-body에서의 격리
- mRNA 붕괴(파괴);
- mRNA 분할
- 마이크로RNA 매개 크로마틴 재구성을 통한 전사 억제 후 유전자 사일런싱.
정상 반응 속도에 대한 실험 데이터를 사용하여 이러한 메커니즘을 식별하는 것은 종종 불가능하다.그럼에도 불구하고, 그들은 역학적으로 구별되고 다른 운동학적 [89]신호를 가지고 있다.
식물의 마이크로RNA와 달리, 동물의 마이크로RNA는 다양한 [40]유전자를 대상으로 한다.그러나 유전자 발현과 같은 모든 세포에 공통적인 기능에 관여하는 유전자는 상대적으로 마이크로RNA 표적 부위가 적고 마이크로RNA [100]표적 부위가 선택되지 않는 것으로 보인다.
dsRNA는 또한 "작은 RNA 유도 유전자 활성화" 또는 RNAa라고 불리는 메커니즘인 유전자 발현을 활성화할 수 있습니다. dsRNA는 유전자 촉진제를 대상으로 하는 강력한 전사 활성화를 유도할 수 있습니다.이는 소형 활성화 RNA(saRNA)[101]라고 불리는 합성 dsRNA를 사용하는 인체 세포에서 입증되었지만 내인성 마이크로RNA에서도 [102]입증되었다.
마이크로RNA와 유전자 상보적 염기서열 간의 상호작용은 심지어 염기서열 호몰로지를 공유하는 유사 유전자(공통 조상 유전자로부터의 분리를 나타내는 유사한 구조를 가진 유전자) 사이의 발현 수준을 조절하는 통신의 백채널로 생각된다."경쟁하는 내인성 RNA(ceRNA)"라는 이름에서, 이러한 마이크로 RNA는 유전자와 의사 유전자에 있는 "마이크로 RNA 응답 요소"와 결합하고 비코드 [103]DNA의 지속성에 대한 또 다른 설명을 제공할 수 있다.
일부 연구에 따르면 엑소좀의 mRNA 화물은 주입에 역할을 할 수 있으며, 영양아세포와 자궁내막 사이의 유착을 맹렬히 확산시키거나 접착/침입에 [104]관여하는 유전자의 발현을 하향 조절 또는 상향 조절함으로써 유착을 지원할 수 있다.
또한 miR-183/96/182로서의 miRNA는 일주기 [105]리듬에 중요한 역할을 하는 것으로 보인다.
진화
miRNA는 식물과 동물 모두에서 잘 보존되어 있고,[106][107][108][109][110] 유전자 조절의 필수적이고 진화적으로 오래된 구성요소라고 생각됩니다.마이크로RNA 경로의 핵심 구성요소는 식물과 동물 사이에 보존되는 반면, 두 왕국의 miRNA 레퍼토리는 서로 다른 주요 [111][112]작용 모드로 독립적으로 나타난 것으로 보인다.
마이크로RNA는 겉으로 보기에는 낮은 [113]진화율 때문에 유용한 계통 발생학적 지표이다. 마이크로RNA의 기원은 바이러스와 [114]같은 외부 유전 물질에 대한 방어책으로 사용되었던 이전의 RNAi 기계에서 개발된 조절 메커니즘이다.그들의 기원은 형태학적 혁신의 개발을 허락했을지도 모르고, 유전자 발현을 보다 구체적이고 '미세 조정 가능'하게 함으로써, 복잡한[115] 장기의 발생을 허용했고,[110] 아마도 궁극적으로, 복잡한 생명체의 발생을 허락했을지도 모른다.형태학적 혁신의 빠른 폭발은 일반적으로 높은 마이크로RNA [113][115]축적 속도와 관련이 있다.
새로운 마이크로 RNA는 여러 가지 방법으로 생성됩니다.b.microRNAs 머리 핀의 DNA(i.e. 인트론 또는intergene 지역)의"non-coding"부분에 무작위로 형성,라 중복이나 기존 microRNAs의 변형에 의해에서 가져온 것일 수 있습니다.[116]microRNAs 또한 protein-coding 시퀀스의foldback 머리 핀 구조가 탄생할 수 있는 역 duplications에서 형성될 수 있다.[117]진화(i.e. 뉴클레오티드 대체)의 최근 유래된 microRNAs의 비율은 다른 곳에non-coding DNA에 있는 중립 추세에서 진화는 것을 암시하고 있지만, 나이 든 microRNAs 변화의 훨씬 더 낮은 수준(100만년당은 한 대체)[110]이 한번 microRNA functio을 얻음을 시사하는 것이 비유할 수 있다.n, 나는정제 [116]선택을 거칩니다.miRNA 유전자 내의 개별 영역은 서로 다른 진화 압력에 직면하며, 여기서 처리와 기능에 필수적인 영역은 더 높은 수준의 [118]보존을 가진다.이 시점에서, 새로운 마이크로 RNA는 [116]종종 상실되지만, 동물의 [110]게놈에서 마이크로 RNA는 거의 손실되지 않는다.아라비도시스 탈리아나에서 miRNA 유전자의 순유속은 백만 [119]년당 1.2에서 3.3 사이의 유전자로 예측되어 왔다.이것은 그것들을 귀중한 계통 발생학적 지표로 만들고, 절지동물의 [120]관계와 같은 뛰어난 계통 발생학적 문제에 대한 가능한 해결책으로 간주되고 있다.한편, 복수의 경우 마이크로RNA는 계통발생과 상관관계가 낮으며, 이들의 계통발생학적 일치성은 대부분 마이크로RNA의 [121]제한된 표본추출을 반영할 수 있다.
마이크로 RNA는 갈색[122] 조류에서 동물에 이르는 대부분의 진핵 생물들의 게놈에서 특징지어진다.그러나 이러한 마이크로 RNA가 어떻게 기능하고 처리되는 방법의 차이는 마이크로 RNA가 식물과 [123]동물에서 독립적으로 발생했음을 시사한다.
동물에 초점을 맞춘 므네미옵시스 레이디의[124] 게놈은 표준 마이크로 RNA 생물 형성에 중요한 핵단백질 드로샤와 파샤뿐만 아니라 인식 가능한 마이크로 RNA가 부족한 것으로 보인다.지금까지 드로샤가 사라진 것으로 보고된 유일한 동물이다.마이크로RNA는 플라코조아문(placozoa)[125]의 유일한 멤버인 트리코플락스 아드헤렌스를 제외하고 지금까지 조사된 모든 비구식동물에서 유전자 발현 조절에 중요한 역할을 한다.
2010년 [126]3월까지 모든 종에서 5000개 이상의 miRNA가 확인되었다.광범위하게 유사한 기능의 짧은 RNA 배열(50 – 수백 개의 염기쌍)이 박테리아에서 발생하는 반면, 박테리아는 진정한 마이크로 RNA가 부족하다.[127]
실험적인 검출과 조작
연구자들이 생리학적, 병리학적 과정에서 miRNA 발현에 초점을 맞춘 반면, 마이크로RNA 격리와 관련된 다양한 기술적 변수들이 나타났다.저장된 miRNA 샘플의 안정성에 [73]의문이 제기되었습니다. 마이크로RNA는 mRNA보다 훨씬 쉽게 분해됩니다. 일부는 길이 때문이기도 하지만, 또한 보편적으로 존재하는 RNase 때문이기도 합니다.따라서 얼음 위에서 샘플을 냉각하고 RNase가 없는 [128]장비를 사용해야 합니다.
마이크로RNA 발현은 수식 RT-PCR의 2단계 중합효소 연쇄반응 프로세스에서 정량화할 수 있다.이 메서드의 변경의 상대 위치나 절대 수량화를 떨치다그래서 miRNAs의 상대적 수준 다른 샘플에서 결정될 수 있[129]miRNAs 또한 마이크로 어레이, 미끄럼틀이나 칩들에 수백 혹은miRNA 목표물 수천개의 탐침과, hybridized 수 있다.[130]microRNAs고 실험해 염기 서열 분석 방법에 의해(mi 발견될 수 있다.croRNA 시퀀싱).[131]miRNA의 활성은 잠금 핵산(LNA) 올리고, 모르포리노 올리고[132][133] 또는 2'-O-메틸 RNA [134]올리고를 사용하여 실험적으로 억제할 수 있다.특정 miRNA는 상보적인 항고미르에 의해 무음화할 수 있으며, 마이크로RNA 성숙은 스테릭 블록올리고에 [135][136]의해 몇 가지 점에서 억제할 수 있다.mRNA 전사체의 miRNA 표적부위도 입체차단올리고에 [137]의해 차단될 수 있다.miRNA의[138] "위치" 검출을[139] 위해 LNA 또는 Morpholino 프로브를 사용할 수 있습니다.LNA의 잠금 배치는 교배 특성을 향상시키고 민감도와 선택성을 증가시켜 짧은 miRNA [140]검출에 이상적입니다.
miRNA의 높은 throughput 정량화는 방법론적 문제를 동반하는 큰 분산(mRNA에 비해)에 오류가 발생하기 쉽다. 따라서 mRNA 발현은 종종 miRNA 수준의 영향을 확인하기 위해 분석된다(예[141]: 의 경우).데이터베이스는 기본 [142][143]시퀀스를 기반으로 miRNA 대상을 예측하는 mRNA와 miRNA 데이터를 쌍으로 구성하는 데 사용할 수 있다.이는 일반적으로 관심 있는 miRNA가 검출된 후에 수행되지만(예: 높은 발현 수준 때문에), mRNA와 miRNA 발현 정보를 통합하는 분석 도구에 대한 아이디어가 [144][145]제안되었다.
질병
miRNA가 진핵세포의 정상적인 기능에 관여하는 것처럼 miRNA의 조절 장애도 질병과 관련이 있다.수동으로 큐레이션되고 공개적으로 이용 가능한 데이터베이스 miR2Disease는 miRNA 규제 이상과 인간 [146]질병 사이의 알려진 관계를 문서화한다.
유전병
miR-96의 종자 영역의 돌연변이는 유전성 진행성 난청을 [147]일으킨다.
miR-184의 종자 영역의 돌연변이는 전극성 [148]백내장과 함께 유전성 각화과를 일으킨다.
miR-17~92 클러스터를 삭제하면 골격 [149]및 성장 결함이 발생합니다.
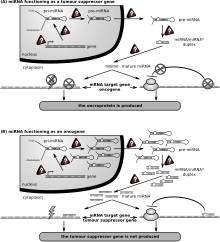
암
miRNA 규제완화와 관련이 있는 것으로 알려진 첫 번째 인간질환은 만성 림프구성 백혈병이었다.다른 많은 miRNA도 암과 관련이 있으며, 따라서 "온코미르"로 불리기도 한다.악성 B세포에서 miRNA는 B세포수용체(BCR) 시그널링, B세포이동/접착, 면역니치에서의 세포간 상호작용 및 면역글로불린의 생성 및 클래스 스위칭과 같은 B세포 발달에 필수적인 경로에 관여한다.MiRNA는 B세포의 성숙, 사전, 한계대, 모낭, B1, 혈장 및 기억 B세포의 생성에 영향을 미친다.
암에서 miRNA의 또 다른 역할은 암의 발현 수준을 예후에 사용하는 것이다.NSCLC 검체에서는 miR-324a 레벨이 낮으면 [150]생존율이 낮다는 징후가 될 수 있습니다.높은 miR-185 또는 낮은 miR-133b 수치는 [151]대장암에서 전이 및 생존율 저하와 상관관계가 있을 수 있다.
또한 특정 miRNA는 대장암의 특정 조직학적 하위 유형과 관련될 수 있다.예를 들어 miR-205와 miR-373의 발현 수준은 점액성 대장암과 점액성 대장염 관련 대장암에서 증가하지만 점액성 [152]성분이 결여된 산발성 대장선암에서는 증가하지 않는 것으로 나타났다.시험관내 연구에 따르면 miR-205와 miR-373은 기능적으로 장상피세포에서 [152]점액 관련 종양 진행의 다른 특징을 유도할 수 있다.
간세포암세포 증식은 종양억제유전자 [153]MAP2K3와의 miR-21 상호작용에서 발생할 수 있다.암에 대한 최적의 치료에는 위험 계층화 치료의 환자를 정확하게 식별하는 것이 포함된다.초기 치료에 빠른 반응을 보이는 사람들은 정확한 질병 대응 측정의 가치를 보여주는 잘린 치료 요법의 혜택을 받을 수 있다.무세포 순환 miRNA(cimiRNA)는 혈액에서 매우 안정적이며, 암에서 과도하게 발현되며, 진단 실험실 내에서 정량화할 수 있다.고전적인 호지킨 림프종에서 혈장 miR-21, miR-494 및 miR-1973은 유망한 질병 반응 바이오마커이다.[154]순환하는 miRNA는 임상 의사 결정을 돕고 컴퓨터 단층 촬영과 결합된 양전자 방출 단층 촬영의 해석을 도울 수 있는 잠재력을 가지고 있습니다.그들은 질병 반응을 평가하고 재발을 발견하기 위해 각 상담에서 수행될 수 있다.
마이크로RNA는 다양한 [155]암을 치료하기 위한 도구 또는 표적으로 사용될 가능성이 있다.특정 마이크로RNA인 miR-506은 여러 연구에서 종양 길항제 역할을 하는 것으로 밝혀졌다.상당수의 자궁경부암 검체가 miR-506의 하향 조절 발현을 보이는 것으로 밝혀졌다.또한 miR-506은 직접 표적 고슴도치 경로 전사인자 Gli3를 [156][157]통해 자궁경부암 세포의 아포토시스를 촉진한다.
DNA수복과 암
암은 DNA 손상이나 DNA [158]복제의 수정되지 않은 오류로 인한 돌연변이의 축적에 의해 발생한다.DNA 수복의 결함은 돌연변이의 축적을 유발하여 [159]암을 유발할 수 있다.DNA 수복에 관여하는 몇몇 유전자들은 마이크로RNA에 [160]의해 조절된다.
DNA 복구 유전자의 생식계 돌연변이는 대장암 [161]사례의 2 - 5%만 유발한다.그러나, DNA 복구 결핍을 야기하는 미세 RNA의 변화된 발현은 암과 자주 관련되며 중요한 인과적 요인이 될 수 있다.DNA 불일치 복구 단백질 MLH1의 발현을 감소시킨 68개의 산발성 대장암 중 대부분은 MLH1 [162]유전자의 CpG 섬의 후생적 메틸화에 의해 결핍된 것으로 밝혀졌다.그러나 산발성 대장암에서 MLH1 결핍의 최대 15%는 MLH1 [163]발현을 억제하는 마이크로RNA miR-155의 과잉 발현으로 나타났다.
교아세포종의 29~[164][165]66%에서 MGMT 유전자의 후생적 메틸화에 의해 DNA 복구가 부족하여 MGMT의 단백질 발현을 감소시킨다. 그러나 교아세포종의 28%에서는 MGMT 단백질은 결핍되지만 MGMT 프로모터는 [164]메틸화되지 않는다.메틸화 MGMT 촉진제가 없는 교아세포종에서 마이크로RNA miR-181d의 수준은 MGMT의 단백질 발현과 역상관되며, miR-181d의 직접 타깃은 MGMT mRNA 3'UTR(MGMT MRNA의 [164]3대 미번역 영역)이다.따라서 교아종의 28%에서 miR-181d의 발현 증가와 DNA 복구 효소 MGMT의 발현 감소가 원인 요인이 될 수 있다.
HMGA 단백질(HMGA1a, HMGA1b 및 HMGA2)은 암에 관여하며, 이러한 단백질의 발현은 마이크로RNA에 의해 조절된다.HMGA 발현은 분화된 성인 조직에서는 거의 검출되지 않지만, 많은 암에서 증가한다.HMGA 단백질은 모듈러 배열 구성에 의해 특징지어지는 최대 100개의 아미노산 잔류물의 폴리펩타이드이다.이 단백질들은 AT 후크라고 불리는 3개의 양전하를 띤 영역을 가지고 있으며, 이는 특정 DNA 영역에서 AT가 풍부한 DNA의 작은 홈과 결합한다.갑상선, 전립선, 자궁경부, 대장암, 췌장암, 난소암을 포함한 인간 종양은 HMGA1a와 HMGA1b 단백질의 [166]강한 증가를 보인다.림프세포를 대상으로 한 HMGA1을 가진 트랜스제닉 마우스는 공격성 림프종을 발생시켜 높은 HMGA1 발현이 암과 관련되며 HMGA1이 [167]종양유전자로 작용할 수 있음을 보여준다.HMGA2 단백질은 특히 ERCC1의 프로모터를 표적으로 하여 이 DNA 수복 [168]유전자의 발현을 감소시킨다.ERCC1 단백질 발현은 평가된 대장암 47개 중 100%에서 결핍되었다(단, HGMA2가 관여한 정도는 알려지지 [169]않았다.
단일 뉴클레오티드 다형성(SNP)은 miRNA의 결합을 3'로 바꿀 수 있다.예를 들어 CDON 종양 억제 유전자의 [170]hsa-mir181a 및 hsa-mir181b의 경우 UTRs.
심장병
심장에서 miRNA 기능의 전체적인 역할은 쥐 심장의 miRNA 성숙을 조건부로 억제함으로써 다루어졌습니다.이것은 miRNA가 [171][172]그 발달 과정에서 필수적인 역할을 한다는 것을 밝혀냈다. miRNA 발현 프로파일링 연구는 질병이 있는 사람의 심장에서 특정 miRNA의 발현 수준이 변화한다는 것을 보여주며 심근병증에 [173][174][175]관여하는 것을 지적했다.또한 특정 miRNA에 대한 동물 연구는 심장발달 중 및 심장발달에 중요한 핵심인자의 조절, 비대 성장 반응 및 심장전도도를 [172][176][177][178][179][180]포함한 병리학적 조건 하에서 miRNA에 대해 구별되는 역할을 확인했다.심혈관 질환에서 miRNA의 또 다른 역할은 진단, 예후 또는 위험 [181]계층화를 위해 발현 수준을 사용하는 것입니다. 동물 모델에서의 miRNA는 또한 콜레스테롤 대사 및 조절과 관련이 있습니다.
miRNA-712
Murine microRNA-712는 지질 [182]유지 및 염증과 관련된 동맥벽의 심혈관 질환인 아테롬성 경화증의 잠재적 바이오마커(즉, 예측 변수)이다.비층혈류도 내피세포의 기계 세너가 교란된 흐름의 전단력에 반응하기 때문에 아테롬성 동맥경화증의 발생과 관련이 있다.[183]매트릭스 금속단백질가수분해효소(MMP)를 포함한 다수의 친아테로겐 유전자는 [183]d-흐름에 의해 상향조절되어 친염증 및 친혈관신호를 매개한다.이러한 발견들은 d-flow의 효과를 모방하기 위해 생쥐의 결합 경동맥에서 관찰되었다.24시간 이내에 기존의 미성숙한 miR-712가 성숙한 miR-712를 형성하여 miR-712가 흐름에 [183]민감함을 나타냅니다.이러한 결과와 일치하여 miR-712는 [183]대동맥궁의 큰 곡률에서 자연적으로 발생하는 d-흐름에 노출된 내피세포에서도 상향 조절된다.
기원.
miR-712의 프리mRNA 배열은 내부 전사 스페이서 영역 2(ITS2)[183]의 뮤린 리보솜 RN45s 유전자로부터 생성된다.XRN1은 RN45s [183]처리 중에 ITS2 영역을 분해하는 엑소뉴클레아제이다.따라서 d-flow 조건 하에서 XRN1이 감소하면 miR-712가 [183]축적됩니다.
메커니즘
MiR-712는 금속단백질가수분해효소3(TIMP3)[183]의 조직억제제를 표적으로 한다.TIMP는 보통 세포외 매트릭스(ECM)를 분해하는 매트릭스 메탈로프로테아제(MMP)의 활성을 조절한다.동맥 ECM은 주로 콜라겐과 엘라스틴 섬유로 구성되어 [184]동맥의 구조적 지지 및 반동 특성을 제공합니다.이러한 섬유는 혈관 염증과 투과성의 조절에 중요한 역할을 하며, 아테롬성 [185]경화증의 발달에 중요합니다.TIMP3는 내피 셀로 표현되며 ECM에 연결된 유일한 TIMP입니다.[184]TIMP3 발현 감소는 d-flow 존재 시 ECM 열화를 증가시킵니다.이러한 발견과 일관되게, 프리 miR712의 억제는 난류에 [183]노출되어도 세포에서 TIMP3의 발현을 증가시킨다.
TIMP3는 또한 [183]난류 중에 TNFα(소염 억제제)의 발현을 감소시킵니다.난류에서 TNFα의 활성은 혈액에서 TNFα 변환 효소(TACE)의 발현으로 측정되었다.miR-712가 억제되거나 TIMP3가 [183]과압축된 경우 TNFα가 감소하여 miR-712 및 TIMP3가 난류 조건에서 TACE 활성을 조절하는 것으로 나타났습니다.
안티미R-712는 d-flow에 의한 miR-712 발현을 효과적으로 억제하고 TIMP3 [183]발현을 증가시킨다.또한 항미R-712는 혈관 과투과성을 억제하여 아테롬성 동맥경화 병변 발생 및 면역세포 [183]침투를 현저하게 감소시킨다.
인간 상동 마이크로RNA-205
miR-712의 인간 상동 유전자는 [183]생쥐와 유사한 miRNA를 유지하는 RN45s 상동 유전자에서 발견되었다.사람의 MiR-205는 쥐의 miR-712와 유사한 서열을 공유하며 대부분의 척추 동물에 [183]보존된다.또한 MiR-205와 miR-712는 TIMP3를 [183]포함한 셀시그널링 타깃의 50% 이상을 공유합니다
시험했을 때, d-flow는 생쥐 내피 세포에서와 마찬가지로 인간에서 XRN1의 발현을 감소시켰고,[183] 이는 인간에서 XRN1의 잠재적으로 일반적인 역할을 나타냈다.
신장병
쥐 모델에서 FoxD1 유래 신장 전구 세포에서 Dicer의 표적 결실은 네프론 전구 세포 확장, 레닌 세포 감소, 평활근 동맥, 진행성 중정맥 [186]손실 및 사구체 동맥류를 포함한 복합 신장 표현형을 초래했다.FoxD1-Dicer 녹아웃 마우스 모델의 높은 throughput 전체 전사체 프로파일링 결과, 박스, Trp53inp1, Jun, Cdkn1, Mmp2, 건조 단백질 수준을 포함한 p53 이펙터 유전자의 증가와 함께 친아포토시스 유전자, Bcl2L11(Bim) 및 p53 경로의 이소성 상승 조절 불량이 밝혀졌다.al miRNA는 p53-이펙터 유전자를 직접적으로 억제한다.FoxD1 유래 세포의 miRNA 프로파일링은 형광활성화 세포 분류에 이은 혈통 추적 접근법을 사용하여 혈관 발달에 중요한 miRNA의 전사 풍경을 포괄적으로 정의했을 뿐만 아니라 부재 시 신장 표현형을 변조할 가능성이 있는 핵심 miRNA를 식별했다.이러한 miRNA에는 Bcl2L11(Bim) 및 miRs1515b, 18a, 19b, 24, 30c, 92a, 92a, 106a, 106a, 106a, 130a, 152, 181a, 214, 222, 302a, 370 및 381이 포함되어 있습니다.프로파일링 결과와 일관되게, FoxD1 유도 전구 계통의 세포 유도체에서 이소성 아포토시스가 관찰되었으며 세포 항상성에서 [186]신장 간질 miRNA의 중요성을 재차 강조했다.
신경계
miRNA는 신경계의 [187]발달과 기능을 조절하는 것으로 보인다.신경 miRNA는 수지상 형성(miR-132, miR-134 및 miR-124 포함), 시냅스[188] 형성 및 시냅스 성숙(miR-134 및 miR-138이 [189]관여하는 것으로 생각됨)을 포함한 시냅스 발달의 다양한 단계에 관여한다.Dicer의 실험적인 소음에 의한 생쥐의 miRNA 형성의 제거는 선조체[190] 뉴런에서 무음화 시 신경 크기 감소 및 운동 이상, 전뇌 [191]뉴런에서 무음화 시 신경 변성과 같은 병리학적 결과를 가져왔다.일부 연구는 정신분열증,[192] 조울증, 주요 우울증, 불안 [193][194][195]장애뿐만 아니라 알츠하이머병에서도 miRNA 발현 변화를 발견한다.
스트로크
질병통제예방센터에 따르면 뇌졸중은 미국에서 사망과 장기 장애의 주요 원인 중 하나이다.87%는 허혈성 뇌졸중으로 산소가 풍부한 혈액을 운반하는 뇌동맥이 막혀 발생한다.혈류의 방해의 뇌와 이산화 탄소와 같은 폐기물 제거가 산소와 포도당 같은 필요한 영양소를 공급 받지 못할 수 있다는 것을 의미한다.[196][197]miRNAs한 염증 혈관 신생은,apoptotic 경로 같은 대뇌 허혈의 발병에 유전자를 표적으로 하여 번역 후의 유전자 것이었다에서 역할을 한다.[198]
알코올 중독
유전자 발현에서 miRNA의 중요한 역할은 중독, 특히 알코올 [199]중독에 중요하다.뇌 기능에 있어 계속적인 변화의 만성 알코올 남용 결과 부분의 유전자 발현을 고치면서 양당을 중재했다.많은 하류의 유전자[199]miRNA 글로벌 규제 중요한 재편이나 시냅스 연결이나 장기적으로 신경 적응 알코올 소비 withdrawal 그리고/까지의 행동의 변화 의혹과 관련해 존경하고 있다.또는 의존.[200]최대 35개의 다른 miRNA가 알코올성 사후 뇌에서 변화된 것으로 밝혀졌으며, 이들 모두는 세포 주기, 아포토시스, 세포 접착, 신경계 발달 및 세포 [199]신호 전달을 포함하는 유전자를 대상으로 한다.알코올 의존 생쥐의 내측 전전두피질에서 miRNA 수치가 변화하여 번역 불균형을 조정하고 복잡한 인지 행동과 의사결정이 발생할 [201]가능성이 있는 뇌 영역 내에서 차등적으로 발현되는 단백질의 생성에 miRNA의 역할을 시사했다.
miRNA는 만성 알코올 사용에 따라 상향 조절되거나 하향 조절될 수 있습니다. miR-206 발현이 알코올 의존 쥐의 전전두피질에서 증가하여 전사인자 뇌유래 신경영양인자(BDNF)를 대상으로 하여 궁극적으로 그 발현을 감소시킵니다.BDNF 새로운 뉴런과 시냅스의 형성과 성숙에, 시냅스growth/synaptic 가소성에 술 침해국 범주에 가능한 함축을 제안하는 중요한 역할을 한다.[202]miR-155,alcohol-induced neuroinflammation 반응 통제하는 중요한, microglia일 뿐 선동하기 위한 생화학제의 역할을 제안하는 upregulated 것이 발견되었다.알코올 병태 [203]생리학miR-382의 하향조절은 근저 전뇌의 구조인 핵 어컴벤스에서 발견되었다. miR-382는 그러한 힘의 동기부여 습관을 조절하는 보상의 감정을 중요하게 한다. miR-382는 도파민 수용체 D1(DRD1)의 타겟이며, 그 과잉발현은 DRD1과 델타 fOSB의 상향조절을 초래하여 전사 인자를 활성화시킨다.궁극적으로 중독성 [204]행동을 초래하는 핵의 전사 사건의 오류.또는 miR-382를 과도하게 발현하면 음주가 약화되고 알코올 중독의 랫드 모델에서 DRD1 및 델타 fosB 상향 조절이 억제되어 치료에 [204]miRNA 표적 의약품을 사용할 수 있는 가능성을 보여주었다.
비만
miRNA는 지방세포로 [205]분화하는 줄기세포 전구체의 조절에 중요한 역할을 한다.지방 형성에 만능 줄기세포가 어떤 역할을 하는지 결정하기 위한 연구는 불멸화된 인간 골수 유래 간질 세포주 hMSC-Tert20에서 [206]조사되었다.miR-155, miR-221 및 miR-222의 발현 감소는 불멸화 및 1차 hMSC의 지방 발생 프로그래밍 중에 발견되었으며, 이는 이들이 분화의 음성 조절 인자로 작용함을 시사한다.반대로 miRNA 155, 221, 222의 이소성 발현은 지방 형성을 유의하게 억제하고 마스터 조절제 PPAR and 및 CCAAT/enhancer-binding protein alpha(CEBPA)[207]의 유도를 억제했다.이것은 가능한 유전자 비만 치료법을 위한 길을 열어준다.
인슐린 저항성, 비만, 당뇨병을 조절하는 또 다른 종류의 miRNA는 let-7 계열이다.Let-7은 [208]노화 과정에서 인체 조직에 축적된다.let-7이 가속화된 노화를 모방하기 위해 외부적으로 과도하게 압박을 받았을 때, 생쥐는 인슐린 저항성을 갖게 되었고, 따라서 고지방 식단에 의해 유발되는 비만과 [209]당뇨병에 더 잘 걸리게 되었다.let-7 특이적 항고마이르 주입에 의해 let-7이 억제되었을 때, 생쥐는 인슐린에 더 민감해지고 고지방 식단에 의해 유발되는 비만과 당뇨병에 현저하게 저항하게 된다.let-7 억제는 비만과 당뇨병을 예방할 수 있을 뿐만 아니라,[210] 이 상태를 되돌리고 치료할 수도 있다.이러한 실험 결과는 let-7 억제가 비만과 제2형 당뇨병에 대한 새로운 치료법을 나타낼 수 있다는 것을 암시한다.
지혈
miRNA는 또한 지혈 혈액 응고 [211]시스템을 포함한 복잡한 효소 캐스케이드의 조절에 중요한 역할을 한다.기능성 miRNA 타겟팅에 대한 대규모 연구는 최근 지혈 [212][213]시스템에서 이론적 치료 대상을 밝혀냈다.
비부호화 RNA
1999년 인간 게놈 프로젝트가 첫 번째 염색체를 지도화했을 때, 게놈에는 10만 개 이상의 단백질을 코드하는 유전자가 포함될 것으로 예측되었다.하지만,[214] 겨우 약 20,000명만이 결국 확인되었다.그 이후로, 생물 정보학 접근법의 출현 유전체 tiling 연구 전체 길이 cDNAlibraries[216]와 실험적 validation[217](miRNA의 탄생 안티 센스 올리고 핵산염 antagomirs라고 불리는 파생된 포함)의 transcriptome,[215]체계적인 염기 서열 결정 법을 살펴보고 함께 많은 대화 내용을 비단 백질다고 밝혔다 연합.-coding RNA, 나는여러 snoRNA 및 miRNA를 [218]제외합니다.
바이러스
바이러스 마이크로RNA는 바이러스에 이로운 바이러스 및/또는 숙주 유전자의 유전자 발현 조절에 중요한 역할을 한다.따라서 miRNA는 숙주-바이러스 상호작용과 바이러스 [219][220]질환의 병인에 중요한 역할을 한다.인간 헤르페스 바이러스-6 DNA에 의한 전사 활성제의 발현은 바이러스 miRNA에 [221]의해 조절되는 것으로 생각된다.
대상 예측
miRNA는 단백질 코드화 유전자의 표적 메신저 RNA(mRNA) 전사에 결합하여 이들의 번역을 부정적으로 제어하거나 mRNA 분해를 일으킬 수 있다.miRNA 대상을 [222]정확하게 식별하는 것이 중요합니다.18 in silico 알고리즘의 예측 성능을 비교할 [223]수 있습니다.기능성 miRNA 표적에 대한 대규모 연구는 표적 예측 [212]알고리즘에 의해 많은 기능성 miRNA가 누락될 수 있음을 시사한다.
「 」를 참조해 주세요.
- 항miRNA올리고뉴클레오티드
- 유전자 발현
- miRNA 유전자 예측 도구 목록
- miRNA 표적 예측 도구 목록
- 마이크로DNA
- MiRNEST
- MIR222
- miR-324-5p
- RNA 간섭
- 작은 간섭 RNA
- 소핵소 RNA유래 마이크로RNA
레퍼런스
- ^ a b c Bartel DP (March 2018). "Metazoan MicroRNAs". Cell. 173 (1): 20–51. doi:10.1016/j.cell.2018.03.006. PMC 6091663. PMID 29570994.
- ^ Qureshi A, Thakur N, Monga I, Thakur A, Kumar M (1 January 2014). "VIRmiRNA: a comprehensive resource for experimentally validated viral miRNAs and their targets". Database. 2014: bau103. doi:10.1093/database/bau103. PMC 4224276. PMID 25380780.
- ^ a b c d Bartel DP (January 2009). "MicroRNAs: target recognition and regulatory functions". Cell. 136 (2): 215–33. doi:10.1016/j.cell.2009.01.002. PMC 3794896. PMID 19167326.
- ^ Fabian MR, Sonenberg N, Filipowicz W (2010). "Regulation of mRNA translation and stability by microRNAs". Annual Review of Biochemistry. 79: 351–79. doi:10.1146/annurev-biochem-060308-103103. PMID 20533884.
- ^ a b Bartel DP (January 2004). "MicroRNAs: genomics, biogenesis, mechanism, and function". Cell. 116 (2): 281–297. doi:10.1016/S0092-8674(04)00045-5. PMID 14744438.
- ^ 맨체스터 대학의 miRBase에서 Homo sapiens miRNA가 검출되었습니다.
- ^ Alles J, Fehlmann T, Fischer U, Backes C, Galata V, Minet M, et al. (April 2019). "An estimate of the total number of true human miRNAs". Nucleic Acids Research. 47 (7): 3353–3364. doi:10.1093/nar/gkz097. PMC 6468295. PMID 30820533.
- ^ Fromm B, Domanska D, Høye E, Ovchinnikov V, Kang W, Aparicio-Puerta E, et al. (January 2020). "MirGeneDB 2.0: the metazoan microRNA complement". Nucleic Acids Research. 48 (D1): D132–D141. doi:10.1093/nar/gkz885. PMC 6943042. PMID 31598695.
- ^ Lim LP, Lau NC, Weinstein EG, Abdelhakim A, Yekta S, Rhoades MW, Burge CB, Bartel DP (April 2003). "The microRNAs of Caenorhabditis elegans". Genes & Development. 17 (8): 991–1008. doi:10.1101/gad.1074403. PMC 196042. PMID 12672692.
- ^ a b Lagos-Quintana M, Rauhut R, Yalcin A, Meyer J, Lendeckel W, Tuschl T (April 2002). "Identification of tissue-specific microRNAs from mouse". Current Biology. 12 (9): 735–9. doi:10.1016/S0960-9822(02)00809-6. PMID 12007417.
- ^ a b Kumar S, Reddy PH (September 2016). "Are circulating microRNAs peripheral biomarkers for Alzheimer's disease?". Biochim Biophys Acta. 1862 (9): 1617–27. doi:10.1016/j.bbadis.2016.06.001. PMC 5343750. PMID 27264337.
- ^ van den Berg MM, Krauskopf J, Ramaekers JG, et al. (February 2020). "Circulating microRNAs as potential biomarkers for psychiatric and neurodegenerative disorders". Prog Neurobiol. 185: 101732. doi:10.1016/j.pneurobio.2019.101732. PMID 31816349.
- ^ a b c Lewis BP, Burge CB, Bartel DP (January 2005). "Conserved seed pairing, often flanked by adenosines, indicates that thousands of human genes are microRNA targets". Cell. 120 (1): 15–20. doi:10.1016/j.cell.2004.12.035. PMID 15652477.
- ^ a b c Friedman RC, Farh KK, Burge CB, Bartel DP (January 2009). "Most mammalian mRNAs are conserved targets of microRNAs". Genome Research. 19 (1): 92–105. doi:10.1101/gr.082701.108. PMC 2612969. PMID 18955434.
- ^ Fromm B, Billipp T, Peck LE, Johansen M, Tarver JE, King BL, et al. (2015). "A Uniform System for the Annotation of Vertebrate microRNA Genes and the Evolution of the Human microRNAome". Annual Review of Genetics. 49: 213–42. doi:10.1146/annurev-genet-120213-092023. PMC 4743252. PMID 26473382.
- ^ a b c Lee RC, Feinbaum RL, Ambros V (December 1993). "The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14". Cell. 75 (5): 843–54. doi:10.1016/0092-8674(93)90529-Y. PMID 8252621.
- ^ a b Reinhart BJ, Slack FJ, Basson M, Pasquinelli AE, Bettinger JC, Rougvie AE, Horvitz HR, Ruvkun G (February 2000). "The 21-nucleotide let-7 RNA regulates developmental timing in Caenorhabditis elegans". Nature. 403 (6772): 901–6. Bibcode:2000Natur.403..901R. doi:10.1038/35002607. PMID 10706289. S2CID 4384503.
- ^ a b Pasquinelli AE, Reinhart BJ, Slack F, Martindale MQ, Kuroda MI, Maller B, Hayward DC, Ball EE, Degnan B, Müller P, Spring J, Srinivasan A, Fishman M, Finnerty J, Corbo J, Levine M, Leahy P, Davidson E, Ruvkun G (November 2000). "Conservation of the sequence and temporal expression of let-7 heterochronic regulatory RNA". Nature. 408 (6808): 86–9. Bibcode:2000Natur.408...86P. doi:10.1038/35040556. PMID 11081512. S2CID 4401732.
- ^ a b c Lagos-Quintana M, Rauhut R, Lendeckel W, Tuschl T (October 2001). "Identification of novel genes coding for small expressed RNAs". Science. 294 (5543): 853–8. Bibcode:2001Sci...294..853L. doi:10.1126/science.1064921. hdl:11858/00-001M-0000-0012-F65F-2. PMID 11679670. S2CID 18101169.
- ^ a b c Lau NC, Lim LP, Weinstein EG, Bartel DP (October 2001). "An abundant class of tiny RNAs with probable regulatory roles in Caenorhabditis elegans". Science. 294 (5543): 858–62. Bibcode:2001Sci...294..858L. doi:10.1126/science.1065062. PMID 11679671. S2CID 43262684.
- ^ a b c Lee RC, Ambros V (October 2001). "An extensive class of small RNAs in Caenorhabditis elegans". Science. 294 (5543): 862–4. Bibcode:2001Sci...294..862L. doi:10.1126/science.1065329. PMID 11679672. S2CID 33480585.
- ^ Wienholds E, Kloosterman WP, Miska E, Alvarez-Saavedra E, Berezikov E, de Bruijn E, Horvitz HR, Kauppinen S, Plasterk RH (July 2005). "MicroRNA expression in zebrafish embryonic development". Science. 309 (5732): 310–1. Bibcode:2005Sci...309..310W. doi:10.1126/science.1114519. PMID 15919954. S2CID 38939571.
- ^ a b Jones-Rhoades MW, Bartel DP, Bartel B (2006). "MicroRNAS and their regulatory roles in plants". Annual Review of Plant Biology. 57: 19–53. doi:10.1146/annurev.arplant.57.032905.105218. PMID 16669754.
- ^ Brennecke J, Hipfner DR, Stark A, Russell RB, Cohen SM (April 2003). "bantam encodes a developmentally regulated microRNA that controls cell proliferation and regulates the proapoptotic gene hid in Drosophila". Cell. 113 (1): 25–36. doi:10.1016/S0092-8674(03)00231-9. PMID 12679032.
- ^ Cuellar TL, McManus MT (December 2005). "MicroRNAs and endocrine biology". The Journal of Endocrinology. 187 (3): 327–32. doi:10.1677/joe.1.06426. PMID 16423811.
- ^ Poy MN, Eliasson L, Krutzfeldt J, Kuwajima S, Ma X, Macdonald PE, Pfeffer S, Tuschl T, Rajewsky N, Rorsman P, Stoffel M (November 2004). "A pancreatic islet-specific microRNA regulates insulin secretion". Nature. 432 (7014): 226–30. Bibcode:2004Natur.432..226P. doi:10.1038/nature03076. PMID 15538371. S2CID 4415988.
- ^ Chen CZ, Li L, Lodish HF, Bartel DP (January 2004). "MicroRNAs modulate hematopoietic lineage differentiation". Science. 303 (5654): 83–6. Bibcode:2004Sci...303...83C. doi:10.1126/science.1091903. hdl:1721.1/7483. PMID 14657504. S2CID 7044929.
- ^ Wilfred BR, Wang WX, Nelson PT (July 2007). "Energizing miRNA research: a review of the role of miRNAs in lipid metabolism, with a prediction that miR-103/107 regulates human metabolic pathways". Molecular Genetics and Metabolism. 91 (3): 209–17. doi:10.1016/j.ymgme.2007.03.011. PMC 1978064. PMID 17521938.
- ^ Harfe BD, McManus MT, Mansfield JH, Hornstein E, Tabin CJ (August 2005). "The RNaseIII enzyme Dicer is required for morphogenesis but not patterning of the vertebrate limb". Proceedings of the National Academy of Sciences of the United States of America. 102 (31): 10898–903. Bibcode:2005PNAS..10210898H. doi:10.1073/pnas.0504834102. PMC 1182454. PMID 16040801.
- ^ Trang P, Weidhaas JB, Slack FJ (December 2008). "MicroRNAs as potential cancer therapeutics". Oncogene. 27 Suppl 2: S52–7. doi:10.1038/onc.2009.353. PMID 19956180.
- ^ Li C, Feng Y, Coukos G, Zhang L (December 2009). "Therapeutic microRNA strategies in human cancer". The AAPS Journal. 11 (4): 747–57. doi:10.1208/s12248-009-9145-9. PMC 2782079. PMID 19876744.
- ^ Fasanaro P, Greco S, Ivan M, Capogrossi MC, Martelli F (January 2010). "microRNA: emerging therapeutic targets in acute ischemic diseases". Pharmacology & Therapeutics. 125 (1): 92–104. doi:10.1016/j.pharmthera.2009.10.003. PMID 19896977.
- ^ Hydbring P, Badalian-Very G (August 2013). "Clinical applications of microRNAs". F1000Research. 2: 136. doi:10.12688/f1000research.2-136.v2. PMC 3917658. PMID 24627783.
- ^ Wightman B, Ha I, Ruvkun G (December 1993). "Posttranscriptional regulation of the heterochronic gene lin-14 by lin-4 mediates temporal pattern formation in C. elegans". Cell. 75 (5): 855–62. doi:10.1016/0092-8674(93)90530-4. PMID 8252622.
- ^ Giza DE, Calin GA (2015). "microRNA and Chronic Lymphocytic Leukemia". Advances in Experimental Medicine and Biology. 889: 23–40. doi:10.1007/978-3-319-23730-5_2. ISBN 978-3-319-23729-9. PMID 26658994.
- ^ Ambros V, Bartel B, Bartel DP, Burge CB, Carrington JC, Chen X, Dreyfuss G, Eddy SR, Griffiths-Jones S, Marshall M, Matzke M, Ruvkun G, Tuschl T (March 2003). "A uniform system for microRNA annotation". RNA. 9 (3): 277–9. doi:10.1261/rna.2183803. PMC 1370393. PMID 12592000.
- ^ Griffiths-Jones S, Grocock RJ, van Dongen S, Bateman A, Enright AJ (January 2006). "miRBase: microRNA sequences, targets and gene nomenclature". Nucleic Acids Research. 34 (Database issue): D140–4. doi:10.1093/nar/gkj112. PMC 1347474. PMID 16381832.
- ^ Wright MW, Bruford EA (January 2011). "Naming 'junk': human non-protein coding RNA (ncRNA) gene nomenclature". Human Genomics. 5 (2): 90–8. doi:10.1186/1479-7364-5-2-90. PMC 3051107. PMID 21296742.
- ^ Hunt M, Banerjee S, Surana P, Liu M, Fuerst G, Mathioni S, Meyers BC, Nettleton D, Wise RP (2019). "Small RNA discovery in the interaction between barley and the powdery mildew pathogen". BMC Genomics. 20 (1): 19–53. doi:10.1186/s12864-019-5947-z. PMC 6657096. PMID 31345162.
- ^ a b c Lewis BP, Shih IH, Jones-Rhoades MW, Bartel DP, Burge CB (December 2003). "Prediction of mammalian microRNA targets". Cell. 115 (7): 787–98. doi:10.1016/S0092-8674(03)01018-3. PMID 14697198.
- ^ Ellwanger DC, Büttner FA, Mewes HW, Stümpflen V (May 2011). "The sufficient minimal set of miRNA seed types". Bioinformatics. 27 (10): 1346–50. doi:10.1093/bioinformatics/btr149. PMC 3087955. PMID 21441577.
- ^ Rajewsky N (June 2006). "microRNA target predictions in animals". Nature Genetics. 38 Suppl (6s): S8–13. doi:10.1038/ng1798. PMID 16736023. S2CID 23496396.
- ^ Krek A, Grün D, Poy MN, Wolf R, Rosenberg L, Epstein EJ, MacMenamin P, da Piedade I, Gunsalus KC, Stoffel M, Rajewsky N (May 2005). "Combinatorial microRNA target predictions". Nature Genetics. 37 (5): 495–500. doi:10.1038/ng1536. PMID 15806104. S2CID 22672750.
- ^ Thomson DW, Bracken CP, Goodall GJ (September 2011). "Experimental strategies for microRNA target identification". Nucleic Acids Research. 39 (16): 6845–53. doi:10.1093/nar/gkr330. PMC 3167600. PMID 21652644.
- ^ a b Lim LP, Lau NC, Garrett-Engele P, Grimson A, Schelter JM, Castle J, Bartel DP, Linsley PS, Johnson JM (February 2005). "Microarray analysis shows that some microRNAs downregulate large numbers of target mRNAs". Nature. 433 (7027): 769–73. Bibcode:2005Natur.433..769L. doi:10.1038/nature03315. PMID 15685193. S2CID 4430576.
- ^ Selbach M, Schwanhäusser B, Thierfelder N, Fang Z, Khanin R, Rajewsky N (September 2008). "Widespread changes in protein synthesis induced by microRNAs". Nature. 455 (7209): 58–63. Bibcode:2008Natur.455...58S. doi:10.1038/nature07228. PMID 18668040. S2CID 4429008.
- ^ Baek D, Villén J, Shin C, Camargo FD, Gygi SP, Bartel DP (September 2008). "The impact of microRNAs on protein output". Nature. 455 (7209): 64–71. Bibcode:2008Natur.455...64B. doi:10.1038/nature07242. PMC 2745094. PMID 18668037.
- ^ a b Rodriguez A, Griffiths-Jones S, Ashurst JL, Bradley A (October 2004). "Identification of mammalian microRNA host genes and transcription units". Genome Research. 14 (10A): 1902–10. doi:10.1101/gr.2722704. PMC 524413. PMID 15364901.
- ^ a b c d Cai X, Hagedorn CH, Cullen BR (December 2004). "Human microRNAs are processed from capped, polyadenylated transcripts that can also function as mRNAs". RNA. 10 (12): 1957–66. doi:10.1261/rna.7135204. PMC 1370684. PMID 15525708.
- ^ Weber MJ (January 2005). "New human and mouse microRNA genes found by homology search". The FEBS Journal. 272 (1): 59–73. doi:10.1111/j.1432-1033.2004.04389.x. PMID 15634332. S2CID 32923462.
- ^ Kim YK, Kim VN (February 2007). "Processing of intronic microRNAs". The EMBO Journal. 26 (3): 775–83. doi:10.1038/sj.emboj.7601512. PMC 1794378. PMID 17255951.
- ^ Baskerville S, Bartel DP (March 2005). "Microarray profiling of microRNAs reveals frequent coexpression with neighboring miRNAs and host genes". RNA. 11 (3): 241–7. doi:10.1261/rna.7240905. PMC 1370713. PMID 15701730.
- ^ a b c Lee Y, Kim M, Han J, Yeom KH, Lee S, Baek SH, Kim VN (October 2004). "MicroRNA genes are transcribed by RNA polymerase II". The EMBO Journal. 23 (20): 4051–60. doi:10.1038/sj.emboj.7600385. PMC 524334. PMID 15372072.
- ^ Zhou X, Ruan J, Wang G, Zhang W (March 2007). "Characterization and identification of microRNA core promoters in four model species". PLOS Computational Biology. 3 (3): e37. Bibcode:2007PLSCB...3...37Z. doi:10.1371/journal.pcbi.0030037. PMC 1817659. PMID 17352530.
- ^ Faller M, Guo F (November 2008). "MicroRNA biogenesis: there's more than one way to skin a cat". Biochimica et Biophysica Acta (BBA) - Gene Regulatory Mechanisms. 1779 (11): 663–7. doi:10.1016/j.bbagrm.2008.08.005. PMC 2633599. PMID 18778799.
- ^ Lee Y, Ahn C, Han J, Choi H, Kim J, Yim J, Lee J, Provost P, Rådmark O, Kim S, Kim VN (September 2003). "The nuclear RNase III Drosha initiates microRNA processing". Nature. 425 (6956): 415–9. Bibcode:2003Natur.425..415L. doi:10.1038/nature01957. PMID 14508493. S2CID 4421030.
- ^ Gregory RI, Chendrimada TP, Shiekhattar R (2006). "MicroRNA biogenesis: isolation and characterization of the microprocessor complex". MicroRNA Protocols. Methods in Molecular Biology. Vol. 342. pp. 33–47. doi:10.1385/1-59745-123-1:33. ISBN 978-1-59745-123-9. PMID 16957365.
- ^ Han J, Lee Y, Yeom KH, Kim YK, Jin H, Kim VN (December 2004). "The Drosha-DGCR8 complex in primary microRNA processing". Genes & Development. 18 (24): 3016–27. doi:10.1101/gad.1262504. PMC 535913. PMID 15574589.
- ^ Han J, Lee Y, Yeom KH, Nam JW, Heo I, Rhee JK, Sohn SY, Cho Y, Zhang BT, Kim VN (June 2006). "Molecular basis for the recognition of primary microRNAs by the Drosha-DGCR8 complex". Cell. 125 (5): 887–901. doi:10.1016/j.cell.2006.03.043. PMID 16751099.
- ^ Conrad T, Marsico A, Gehre M, Orom UA (October 2014). "Microprocessor activity controls differential miRNA biogenesis in Vivo". Cell Reports. 9 (2): 542–54. doi:10.1016/j.celrep.2014.09.007. PMID 25310978.
- ^ Auyeung VC, Ulitsky I, McGeary SE, Bartel DP (February 2013). "Beyond secondary structure: primary-sequence determinants license pri-miRNA hairpins for processing". Cell. 152 (4): 844–58. doi:10.1016/j.cell.2013.01.031. PMC 3707628. PMID 23415231.
- ^ Ali PS, Ghoshdastider U, Hoffmann J, Brutschy B, Filipek S (November 2012). "Recognition of the let-7g miRNA precursor by human Lin28B". FEBS Letters. 586 (22): 3986–90. doi:10.1016/j.febslet.2012.09.034. PMID 23063642. S2CID 28899778.
- ^ Berezikov E, Chung WJ, Willis J, Cuppen E, Lai EC (October 2007). "Mammalian mirtron genes". Molecular Cell. 28 (2): 328–36. doi:10.1016/j.molcel.2007.09.028. PMC 2763384. PMID 17964270.
- ^ a b Kawahara Y, Megraw M, Kreider E, Iizasa H, Valente L, Hatzigeorgiou AG, Nishikura K (September 2008). "Frequency and fate of microRNA editing in human brain". Nucleic Acids Research. 36 (16): 5270–80. doi:10.1093/nar/gkn479. PMC 2532740. PMID 18684997.
- ^ Winter J, Jung S, Keller S, Gregory RI, Diederichs S (March 2009). "Many roads to maturity: microRNA biogenesis pathways and their regulation". Nature Cell Biology. 11 (3): 228–34. doi:10.1038/ncb0309-228. PMID 19255566. S2CID 205286318.
- ^ Ohman M (October 2007). "A-to-I editing challenger or ally to the microRNA process". Biochimie. 89 (10): 1171–6. doi:10.1016/j.biochi.2007.06.002. PMID 17628290.
- ^ a b Murchison EP, Hannon GJ (June 2004). "miRNAs on the move: miRNA biogenesis and the RNAi machinery". Current Opinion in Cell Biology. 16 (3): 223–9. doi:10.1016/j.ceb.2004.04.003. PMID 15145345.

- ^ a b c Lund E, Dahlberg JE (2006). "Substrate selectivity of exportin 5 and Dicer in the biogenesis of microRNAs". Cold Spring Harbor Symposia on Quantitative Biology. 71: 59–66. doi:10.1101/sqb.2006.71.050. PMID 17381281.
- ^ Park JE, Heo I, Tian Y, Simanshu DK, Chang H, Jee D, Patel DJ, Kim VN (July 2011). "Dicer recognizes the 5' end of RNA for efficient and accurate processing". Nature. 475 (7355): 201–5. doi:10.1038/nature10198. PMC 4693635. PMID 21753850.
- ^ Ji X (2008). "The mechanism of RNase III action: how dicer dices". RNA Interference. Current Topics in Microbiology and Immunology. Vol. 320. pp. 99–116. doi:10.1007/978-3-540-75157-1_5. ISBN 978-3-540-75156-4. PMID 18268841.
- ^ Mirihana Arachchilage G, Dassanayake AC, Basu S (February 2015). "A potassium ion-dependent RNA structural switch regulates human pre-miRNA 92b maturation". Chemistry & Biology. 22 (2): 262–72. doi:10.1016/j.chembiol.2014.12.013. PMID 25641166.
- ^ Sohel MH (2016). "Extracellular/Circulating MicroRNAs: Release Mechanisms, Functions and Challenges". Achievements in the Life Sciences. 10 (2): 175–186. doi:10.1016/j.als.2016.11.007.
- ^ a b Boeckel JN, Reis SM, Leistner D, Thomé CE, Zeiher AM, Fichtlscherer S, Keller T (April 2014). "From heart to toe: heart's contribution on peripheral microRNA levels". International Journal of Cardiology. 172 (3): 616–7. doi:10.1016/j.ijcard.2014.01.082. PMID 24508494.
- ^ Lelandais-Brière C, Sorin C, Declerck M, Benslimane A, Crespi M, Hartmann C (March 2010). "Small RNA diversity in plants and its impact in development". Current Genomics. 11 (1): 14–23. doi:10.2174/138920210790217918. PMC 2851111. PMID 20808519.
- ^ Rana TM (January 2007). "Illuminating the silence: understanding the structure and function of small RNAs". Nature Reviews Molecular Cell Biology. 8 (1): 23–36. doi:10.1038/nrm2085. PMID 17183358. S2CID 8966239.
- ^ a b Schwarz DS, Zamore PD (May 2002). "Why do miRNAs live in the miRNP?". Genes & Development. 16 (9): 1025–31. doi:10.1101/gad.992502. PMID 12000786.
- ^ Krol J, Sobczak K, Wilczynska U, Drath M, Jasinska A, Kaczynska D, Krzyzosiak WJ (October 2004). "Structural features of microRNA (miRNA) precursors and their relevance to miRNA biogenesis and small interfering RNA/short hairpin RNA design". The Journal of Biological Chemistry. 279 (40): 42230–9. doi:10.1074/jbc.M404931200. PMID 15292246.
- ^ Khvorova A, Reynolds A, Jayasena SD (October 2003). "Functional siRNAs and miRNAs exhibit strand bias". Cell. 115 (2): 209–16. doi:10.1016/S0092-8674(03)00801-8. PMID 14567918.
- ^ Schwarz DS, Hutvágner G, Du T, Xu Z, Aronin N, Zamore PD (October 2003). "Asymmetry in the assembly of the RNAi enzyme complex". Cell. 115 (2): 199–208. doi:10.1016/S0092-8674(03)00759-1. PMID 14567917.
- ^ Lin SL, Chang D, Ying SY (August 2005). "Asymmetry of intronic pre-miRNA structures in functional RISC assembly". Gene. 356: 32–8. doi:10.1016/j.gene.2005.04.036. PMC 1788082. PMID 16005165.
- ^ Okamura K, Chung WJ, Lai EC (September 2008). "The long and short of inverted repeat genes in animals: microRNAs, mirtrons and hairpin RNAs". Cell Cycle. 7 (18): 2840–5. doi:10.4161/cc.7.18.6734. PMC 2697033. PMID 18769156.
- ^ a b Pratt AJ, MacRae IJ (July 2009). "The RNA-induced silencing complex: a versatile gene-silencing machine". The Journal of Biological Chemistry. 284 (27): 17897–901. doi:10.1074/jbc.R900012200. PMC 2709356. PMID 19342379.
- ^ MacRae IJ, Ma E, Zhou M, Robinson CV, Doudna JA (January 2008). "In vitro reconstitution of the human RISC-loading complex". Proceedings of the National Academy of Sciences of the United States of America. 105 (2): 512–7. Bibcode:2008PNAS..105..512M. doi:10.1073/pnas.0710869105. PMC 2206567. PMID 18178619.
- ^ Mourelatos Z, Dostie J, Paushkin S, Sharma A, Charroux B, Abel L, Rappsilber J, Mann M, Dreyfuss G (March 2002). "miRNPs: a novel class of ribonucleoproteins containing numerous microRNAs". Genes & Development. 16 (6): 720–8. doi:10.1101/gad.974702. PMC 155365. PMID 11914277.
- ^ Meister G, Landthaler M, Peters L, Chen PY, Urlaub H, Lührmann R, Tuschl T (December 2005). "Identification of novel argonaute-associated proteins". Current Biology. 15 (23): 2149–55. doi:10.1016/j.cub.2005.10.048. PMID 16289642.
- ^ Jing Q, Huang S, Guth S, Zarubin T, Motoyama A, Chen J, Di Padova F, Lin SC, Gram H, Han J (March 2005). "Involvement of microRNA in AU-rich element-mediated mRNA instability". Cell. 120 (5): 623–34. doi:10.1016/j.cell.2004.12.038. PMID 15766526.
- ^ a b c Kai ZS, Pasquinelli AE (January 2010). "MicroRNA assassins: factors that regulate the disappearance of miRNAs". Nature Structural & Molecular Biology. 17 (1): 5–10. doi:10.1038/nsmb.1762. PMC 6417416. PMID 20051982.
- ^ Chatterjee S, Grosshans H (September 2009). "Active turnover modulates mature microRNA activity in Caenorhabditis elegans". Nature. 461 (7263): 546–9. Bibcode:2009Natur.461..546C. doi:10.1038/nature08349. PMID 19734881. S2CID 4414841.
- ^ a b c Morozova N, Zinovyev A, Nonne N, Pritchard LL, Gorban AN, Harel-Bellan A (September 2012). "Kinetic signatures of microRNA modes of action". RNA. 18 (9): 1635–55. doi:10.1261/rna.032284.112. PMC 3425779. PMID 22850425.
- ^ Wang XJ, Reyes JL, Chua NH, Gaasterland T (2004). "Prediction and identification of Arabidopsis thaliana microRNAs and their mRNA targets". Genome Biology. 5 (9): R65. doi:10.1186/gb-2004-5-9-r65. PMC 522872. PMID 15345049.
- ^ Kawasaki H, Taira K (2004). "MicroRNA-196 inhibits HOXB8 expression in myeloid differentiation of HL60 cells". Nucleic Acids Symposium Series. 48 (1): 211–2. doi:10.1093/nass/48.1.211. PMID 17150553.
- ^ a b Moxon S, Jing R, Szittya G, Schwach F, Rusholme Pilcher RL, Moulton V, Dalmay T (October 2008). "Deep sequencing of tomato short RNAs identifies microRNAs targeting genes involved in fruit ripening". Genome Research. 18 (10): 1602–9. doi:10.1101/gr.080127.108. PMC 2556272. PMID 18653800.
- ^ Mazière P, Enright AJ (June 2007). "Prediction of microRNA targets". Drug Discovery Today. 12 (11–12): 452–8. doi:10.1016/j.drudis.2007.04.002. PMID 17532529.
- ^ Williams AE (February 2008). "Functional aspects of animal microRNAs". Cellular and Molecular Life Sciences. 65 (4): 545–62. doi:10.1007/s00018-007-7355-9. PMID 17965831. S2CID 5708394.
- ^ Eulalio A, Huntzinger E, Nishihara T, Rehwinkel J, Fauser M, Izaurralde E (January 2009). "Deadenylation is a widespread effect of miRNA regulation". RNA. 15 (1): 21–32. doi:10.1261/rna.1399509. PMC 2612776. PMID 19029310.
- ^ Bazzini AA, Lee MT, Giraldez AJ (April 2012). "Ribosome profiling shows that miR-430 reduces translation before causing mRNA decay in zebrafish". Science. 336 (6078): 233–7. Bibcode:2012Sci...336..233B. doi:10.1126/science.1215704. PMC 3547538. PMID 22422859.
- ^ Djuranovic S, Nahvi A, Green R (April 2012). "miRNA-mediated gene silencing by translational repression followed by mRNA deadenylation and decay". Science. 336 (6078): 237–40. Bibcode:2012Sci...336..237D. doi:10.1126/science.1215691. PMC 3971879. PMID 22499947.
- ^ Tan Y, Zhang B, Wu T, Skogerbø G, Zhu X, Guo X, He S, Chen R (February 2009). "Transcriptional inhibiton of Hoxd4 expression by miRNA-10a in human breast cancer cells". BMC Molecular Biology. 10 (1): 12. doi:10.1186/1471-2199-10-12. PMC 2680403. PMID 19232136.
- ^ Hawkins PG, Morris KV (March 2008). "RNA and transcriptional modulation of gene expression". Cell Cycle. 7 (5): 602–7. doi:10.4161/cc.7.5.5522. PMC 2877389. PMID 18256543.
- ^ Stark A, Brennecke J, Bushati N, Russell RB, Cohen SM (December 2005). "Animal MicroRNAs confer robustness to gene expression and have a significant impact on 3'UTR evolution". Cell. 123 (6): 1133–46. doi:10.1016/j.cell.2005.11.023. PMID 16337999.
- ^ Li LC (2008). "Small RNA-Mediated Gene Activation". In Morris KV (ed.). RNA and the Regulation of Gene Expression: A Hidden Layer of Complexity. Horizon Scientific Press. ISBN 978-1-904455-25-7.
- ^ Place RF, Li LC, Pookot D, Noonan EJ, Dahiya R (February 2008). "MicroRNA-373 induces expression of genes with complementary promoter sequences". Proceedings of the National Academy of Sciences of the United States of America. 105 (5): 1608–13. Bibcode:2008PNAS..105.1608P. doi:10.1073/pnas.0707594105. PMC 2234192. PMID 18227514.
- ^ Salmena L, Poliseno L, Tay Y, Kats L, Pandolfi PP (August 2011). "A ceRNA hypothesis: the Rosetta Stone of a hidden RNA language?". Cell. 146 (3): 353–8. doi:10.1016/j.cell.2011.07.014. PMC 3235919. PMID 21802130.
- ^ Cuman C, Van Sinderen M, Gantier MP, Rainczuk K, Sorby K, Rombauts L, et al. (October 2015). "Human Blastocyst Secreted microRNA Regulate Endometrial Epithelial Cell Adhesion". EBioMedicine. 2 (10): 1528–1535. doi:10.1016/j.ebiom.2015.09.003. PMC 4634783. PMID 26629549.
- ^ Zhou L, Miller C, Miraglia LJ, Romero A, Mure LS, Panda S, Kay SA (January 2021). "A genome-wide microRNA screen identifies the microRNA-183/96/182 cluster as a modulator of circadian rhythms". Proceedings of the National Academy of Sciences of the United States of America. 118 (1): e2020454118. doi:10.1073/pnas.2020454118. PMC 7817116. PMID 33443164. S2CID 230713808.
- "MicroRNAs Play Key Role in Regulation of Circadian Rhythms". Science News. 6 January 2021.
- ^ Axtell MJ, Bartel DP (June 2005). "Antiquity of microRNAs and their targets in land plants". The Plant Cell. 17 (6): 1658–73. doi:10.1105/tpc.105.032185. PMC 1143068. PMID 15849273.
- ^ Tanzer A, Stadler PF (May 2004). "Molecular evolution of a microRNA cluster". Journal of Molecular Biology. 339 (2): 327–35. CiteSeerX 10.1.1.194.1598. doi:10.1016/j.jmb.2004.03.065. PMID 15136036.
- ^ Chen K, Rajewsky N (February 2007). "The evolution of gene regulation by transcription factors and microRNAs". Nature Reviews Genetics. 8 (2): 93–103. doi:10.1038/nrg1990. PMID 17230196. S2CID 174231.
- ^ Lee CT, Risom T, Strauss WM (April 2007). "Evolutionary conservation of microRNA regulatory circuits: an examination of microRNA gene complexity and conserved microRNA-target interactions through metazoan phylogeny". DNA and Cell Biology. 26 (4): 209–18. doi:10.1089/dna.2006.0545. PMID 17465887.
- ^ a b c d Peterson KJ, Dietrich MR, McPeek MA (July 2009). "MicroRNAs and metazoan macroevolution: insights into canalization, complexity, and the Cambrian explosion". BioEssays. 31 (7): 736–47. doi:10.1002/bies.200900033. PMID 19472371. S2CID 15364875.
- ^ Shabalina SA, Koonin EV (October 2008). "Origins and evolution of eukaryotic RNA interference". Trends in Ecology & Evolution. 23 (10): 578–87. doi:10.1016/j.tree.2008.06.005. PMC 2695246. PMID 18715673.
- ^ Axtell MJ, Westholm JO, Lai EC (2011). "Vive la différence: biogenesis and evolution of microRNAs in plants and animals". Genome Biology. 12 (4): 221. doi:10.1186/gb-2011-12-4-221. PMC 3218855. PMID 21554756.
- ^ a b Wheeler BM, Heimberg AM, Moy VN, Sperling EA, Holstein TW, Heber S, Peterson KJ (2009). "The deep evolution of metazoan microRNAs". Evolution & Development. 11 (1): 50–68. doi:10.1111/j.1525-142X.2008.00302.x. PMID 19196333. S2CID 14924603.
- ^ Pashkovskiy PP, Ryazansky SS (June 2013). "Biogenesis, evolution, and functions of plant microRNAs". Biochemistry. Biokhimiia. 78 (6): 627–37. doi:10.1134/S0006297913060084. PMID 23980889. S2CID 12025420.
- ^ a b Heimberg AM, Sempere LF, Moy VN, Donoghue PC, Peterson KJ (February 2008). "MicroRNAs and the advent of vertebrate morphological complexity". Proceedings of the National Academy of Sciences of the United States of America. 105 (8): 2946–50. Bibcode:2008PNAS..105.2946H. doi:10.1073/pnas.0712259105. PMC 2268565. PMID 18287013.
- ^ a b c Nozawa M, Miura S, Nei M (July 2010). "Origins and evolution of microRNA genes in Drosophila species". Genome Biology and Evolution. 2: 180–9. doi:10.1093/gbe/evq009. PMC 2942034. PMID 20624724.
- ^ Allen E, Xie Z, Gustafson AM, Sung GH, Spatafora JW, Carrington JC (December 2004). "Evolution of microRNA genes by inverted duplication of target gene sequences in Arabidopsis thaliana". Nature Genetics. 36 (12): 1282–90. doi:10.1038/ng1478. PMID 15565108. S2CID 11997028.
- ^ Warthmann N, Das S, Lanz C, Weigel D (May 2008). "Comparative analysis of the MIR319a microRNA locus in Arabidopsis and related Brassicaceae". Molecular Biology and Evolution. 25 (5): 892–902. doi:10.1093/molbev/msn029. PMID 18296705.
- ^ Fahlgren N, Jogdeo S, Kasschau KD, Sullivan CM, Chapman EJ, Laubinger S, Smith LM, Dasenko M, Givan SA, Weigel D, Carrington JC (April 2010). "MicroRNA gene evolution in Arabidopsis lyrata and Arabidopsis thaliana". The Plant Cell. 22 (4): 1074–89. doi:10.1105/tpc.110.073999. PMC 2879733. PMID 20407027.
- ^ Caravas J, Friedrich M (June 2010). "Of mites and millipedes: recent progress in resolving the base of the arthropod tree". BioEssays. 32 (6): 488–95. doi:10.1002/bies.201000005. PMID 20486135. S2CID 20548122.
- ^ Kenny NJ, Namigai EK, Marlétaz F, Hui JH, Shimeld SM (December 2015). "Draft genome assemblies and predicted microRNA complements of the intertidal lophotrochozoans Patella vulgata (Mollusca, Patellogastropoda) and Spirobranchus (Pomatoceros) lamarcki (Annelida, Serpulida)". Marine Genomics. 24 (2): 139–46. doi:10.1016/j.margen.2015.07.004. PMID 26319627.
- ^ Cock JM, Sterck L, Rouzé P, Scornet D, Allen AE, Amoutzias G, et al. (June 2010). "The Ectocarpus genome and the independent evolution of multicellularity in brown algae". Nature. 465 (7298): 617–21. Bibcode:2010Natur.465..617C. doi:10.1038/nature09016. PMID 20520714.
- ^ Cuperus JT, Fahlgren N, Carrington JC (February 2011). "Evolution and functional diversification of MIRNA genes". The Plant Cell. 23 (2): 431–42. doi:10.1105/tpc.110.082784. PMC 3077775. PMID 21317375.
- ^ Ryan JF, Pang K, Schnitzler CE, Nguyen AD, Moreland RT, Simmons DK, Koch BJ, Francis WR, Havlak P, Smith SA, Putnam NH, Haddock SH, Dunn CW, Wolfsberg TG, Mullikin JC, Martindale MQ, Baxevanis AD (December 2013). "The genome of the ctenophore Mnemiopsis leidyi and its implications for cell type evolution". Science. 342 (6164): 1242592. doi:10.1126/science.1242592. PMC 3920664. PMID 24337300.
- ^ Maxwell EK, Ryan JF, Schnitzler CE, Browne WE, Baxevanis AD (December 2012). "MicroRNAs and essential components of the microRNA processing machinery are not encoded in the genome of the ctenophore Mnemiopsis leidyi". BMC Genomics. 13 (1): 714. doi:10.1186/1471-2164-13-714. PMC 3563456. PMID 23256903.
- ^ Dimond PF (15 March 2010). "miRNAs' Therapeutic Potential". Genetic Engineering & Biotechnology News. 30 (6): 1. Archived from the original on 19 July 2010. Retrieved 10 July 2010.
- ^ Tjaden B, Goodwin SS, Opdyke JA, Guillier M, Fu DX, Gottesman S, Storz G (2006). "Target prediction for small, noncoding RNAs in bacteria". Nucleic Acids Research. 34 (9): 2791–802. doi:10.1093/nar/gkl356. PMC 1464411. PMID 16717284.
- ^ Liu CG, Calin GA, Volinia S, Croce CM (2008). "MicroRNA expression profiling using microarrays". Nature Protocols. 3 (4): 563–78. doi:10.1038/nprot.2008.14. PMID 18388938. S2CID 2441105.
- ^ Chen C, Ridzon DA, Broomer AJ, Zhou Z, Lee DH, Nguyen JT, Barbisin M, Xu NL, Mahuvakar VR, Andersen MR, Lao KQ, Livak KJ, Guegler KJ (November 2005). "Real-time quantification of microRNAs by stem-loop RT-PCR". Nucleic Acids Research. 33 (20): e179. doi:10.1093/nar/gni178. PMC 1292995. PMID 16314309.
- ^ Shingara J, Keiger K, Shelton J, Laosinchai-Wolf W, Powers P, Conrad R, Brown D, Labourier E (September 2005). "An optimized isolation and labeling platform for accurate microRNA expression profiling". RNA. 11 (9): 1461–70. doi:10.1261/rna.2610405. PMC 1370829. PMID 16043497.
- ^ Buermans HP, Ariyurek Y, van Ommen G, den Dunnen JT, 't Hoen PA (December 2010). "New methods for next generation sequencing based microRNA expression profiling". BMC Genomics. 11: 716. doi:10.1186/1471-2164-11-716. PMC 3022920. PMID 21171994.
- ^ Kloosterman WP, Wienholds E, Ketting RF, Plasterk RH (2004). "Substrate requirements for let-7 function in the developing zebrafish embryo". Nucleic Acids Research. 32 (21): 6284–91. doi:10.1093/nar/gkh968. PMC 535676. PMID 15585662.
- ^ Flynt AS, Li N, Thatcher EJ, Solnica-Krezel L, Patton JG (February 2007). "Zebrafish miR-214 modulates Hedgehog signaling to specify muscle cell fate". Nature Genetics. 39 (2): 259–63. doi:10.1038/ng1953. PMC 3982799. PMID 17220889.
- ^ Meister G, Landthaler M, Dorsett Y, Tuschl T (March 2004). "Sequence-specific inhibition of microRNA- and siRNA-induced RNA silencing". RNA. 10 (3): 544–50. doi:10.1261/rna.5235104. PMC 1370948. PMID 14970398.
- ^ Kloosterman WP, Lagendijk AK, Ketting RF, Moulton JD, Plasterk RH (August 2007). "Targeted inhibition of miRNA maturation with morpholinos reveals a role for miR-375 in pancreatic islet development". PLOS Biology. 5 (8): e203. doi:10.1371/journal.pbio.0050203. PMC 1925136. PMID 17676975.
- ^ Gebert LF, Rebhan MA, Crivelli SE, Denzler R, Stoffel M, Hall J (January 2014). "Miravirsen (SPC3649) can inhibit the biogenesis of miR-122". Nucleic Acids Research. 42 (1): 609–21. doi:10.1093/nar/gkt852. PMC 3874169. PMID 24068553.
- ^ Choi WY, Giraldez AJ, Schier AF (October 2007). "Target protectors reveal dampening and balancing of Nodal agonist and antagonist by miR-430". Science. 318 (5848): 271–4. Bibcode:2007Sci...318..271C. doi:10.1126/science.1147535. PMID 17761850. S2CID 30461594.
- ^ You Y, Moreira BG, Behlke MA, Owczarzy R (May 2006). "Design of LNA probes that improve mismatch discrimination". Nucleic Acids Research. 34 (8): e60. doi:10.1093/nar/gkl175. PMC 1456327. PMID 16670427.
- ^ Lagendijk AK, Moulton JD, Bakkers J (June 2012). "Revealing details: whole mount microRNA in situ hybridization protocol for zebrafish embryos and adult tissues". Biology Open. 1 (6): 566–9. doi:10.1242/bio.2012810. PMC 3509442. PMID 23213449.
- ^ Kaur H, Arora A, Wengel J, Maiti S (June 2006). "Thermodynamic, counterion, and hydration effects for the incorporation of locked nucleic acid nucleotides into DNA duplexes". Biochemistry. 45 (23): 7347–55. doi:10.1021/bi060307w. PMID 16752924.
- ^ Nielsen JA, Lau P, Maric D, Barker JL, Hudson LD (August 2009). "Integrating microRNA and mRNA expression profiles of neuronal progenitors to identify regulatory networks underlying the onset of cortical neurogenesis". BMC Neuroscience. 10: 98. doi:10.1186/1471-2202-10-98. PMC 2736963. PMID 19689821.
- ^ Grimson A, Farh KK, Johnston WK, Garrett-Engele P, Lim LP, Bartel DP (July 2007). "MicroRNA targeting specificity in mammals: determinants beyond seed pairing". Molecular Cell. 27 (1): 91–105. doi:10.1016/j.molcel.2007.06.017. PMC 3800283. PMID 17612493.
- ^ Griffiths-Jones S, Saini HK, van Dongen S, Enright AJ (January 2008). "miRBase: tools for microRNA genomics". Nucleic Acids Research. 36 (Database issue): D154–8. doi:10.1093/nar/gkm952. PMC 2238936. PMID 17991681.
- ^ Nam S, Li M, Choi K, Balch C, Kim S, Nephew KP (July 2009). "MicroRNA and mRNA integrated analysis (MMIA): a web tool for examining biological functions of microRNA expression". Nucleic Acids Research. 37 (Web Server issue): W356–62. doi:10.1093/nar/gkp294. PMC 2703907. PMID 19420067.
- ^ Artmann S, Jung K, Bleckmann A, Beissbarth T (2012). Provero P (ed.). "Detection of simultaneous group effects in microRNA expression and related target gene sets". PLOS ONE. 7 (6): e38365. Bibcode:2012PLoSO...738365A. doi:10.1371/journal.pone.0038365. PMC 3378551. PMID 22723856.
- ^ Jiang Q, Wang Y, Hao Y, Juan L, Teng M, Zhang X, Li M, Wang G, Liu Y (January 2009). "miR2Disease: a manually curated database for microRNA deregulation in human disease". Nucleic Acids Research. 37. 37 (Database issue): D98–104. doi:10.1093/nar/gkn714. PMC 2686559. PMID 18927107.
- ^ Mencía A, Modamio-Høybjør S, Redshaw N, Morín M, Mayo-Merino F, Olavarrieta L, Aguirre LA, del Castillo I, Steel KP, Dalmay T, Moreno F, Moreno-Pelayo MA (May 2009). "Mutations in the seed region of human miR-96 are responsible for nonsyndromic progressive hearing loss". Nature Genetics. 41 (5): 609–13. doi:10.1038/ng.355. PMID 19363479. S2CID 11113852.
- ^ Hughes AE, Bradley DT, Campbell M, Lechner J, Dash DP, Simpson DA, Willoughby CE (November 2011). "Mutation altering the miR-184 seed region causes familial keratoconus with cataract". American Journal of Human Genetics. 89 (5): 628–33. doi:10.1016/j.ajhg.2011.09.014. PMC 3213395. PMID 21996275.
- ^ de Pontual L, Yao E, Callier P, Faivre L, Drouin V, Cariou S, Van Haeringen A, Geneviève D, Goldenberg A, Oufadem M, Manouvrier S, Munnich A, Vidigal JA, Vekemans M, Lyonnet S, Henrion-Caude A, Ventura A, Amiel J (September 2011). "Germline deletion of the miR-17∼92 cluster causes skeletal and growth defects in humans". Nature Genetics. 43 (10): 1026–30. doi:10.1038/ng.915. PMC 3184212. PMID 21892160.
- ^ Võsa U, Vooder T, Kolde R, Fischer K, Välk K, Tõnisson N, Roosipuu R, Vilo J, Metspalu A, Annilo T (October 2011). "Identification of miR-374a as a prognostic marker for survival in patients with early-stage nonsmall cell lung cancer". Genes, Chromosomes & Cancer. 50 (10): 812–22. doi:10.1002/gcc.20902. PMID 21748820. S2CID 9746594.
- ^ Akçakaya P, Ekelund S, Kolosenko I, Caramuta S, Ozata DM, Xie H, Lindforss U, Olivecrona H, Lui WO (August 2011). "miR-185 and miR-133b deregulation is associated with overall survival and metastasis in colorectal cancer". International Journal of Oncology. 39 (2): 311–8. doi:10.3892/ijo.2011.1043. PMID 21573504.
- ^ a b Eyking A, Reis H, Frank M, Gerken G, Schmid KW, Cario E (2016). "MiR-205 and MiR-373 Are Associated with Aggressive Human Mucinous Colorectal Cancer". PLOS ONE. 11 (6): e0156871. Bibcode:2016PLoSO..1156871E. doi:10.1371/journal.pone.0156871. PMC 4894642. PMID 27271572.
- ^ MicroRNA-21은 마이트젠 활성화 단백질 키나아제3의 억제를 통해 간세포암 HepG2 세포 증식을 촉진한다.광셴 쉬 외, 2013년
- ^ Jones K, Nourse JP, Keane C, Bhatnagar A, Gandhi MK (January 2014). "Plasma microRNA are disease response biomarkers in classical Hodgkin lymphoma". Clinical Cancer Research. 20 (1): 253–64. doi:10.1158/1078-0432.CCR-13-1024. PMID 24222179.
- ^ Hosseinahli N, Aghapour M, Duijf PH, Baradaran B (August 2018). "Treating cancer with microRNA replacement therapy: A literature review". Journal of Cellular Physiology. 233 (8): 5574–5588. doi:10.1002/jcp.26514. PMID 29521426. S2CID 3766576.
- ^ Liu G, Sun Y, Ji P, Li X, Cogdell D, Yang D, Parker Kerrigan BC, Shmulevich I, Chen K, Sood AK, Xue F, Zhang W (July 2014). "MiR-506 suppresses proliferation and induces senescence by directly targeting the CDK4/6-FOXM1 axis in ovarian cancer". The Journal of Pathology. 233 (3): 308–18. doi:10.1002/path.4348. PMC 4144705. PMID 24604117.
- ^ Wen SY, Lin Y, Yu YQ, Cao SJ, Zhang R, Yang XM, Li J, Zhang YL, Wang YH, Ma MZ, Sun WW, Lou XL, Wang JH, Teng YC, Zhang ZG (February 2015). "miR-506 acts as a tumor suppressor by directly targeting the hedgehog pathway transcription factor Gli3 in human cervical cancer". Oncogene. 34 (6): 717–25. doi:10.1038/onc.2014.9. PMID 24608427. S2CID 20603801.
- ^ Loeb KR, Loeb LA (March 2000). "Significance of multiple mutations in cancer". Carcinogenesis. 21 (3): 379–385. doi:10.1093/carcin/21.3.379. PMID 10688858.
- ^ Lodish H, Berk A, Kaiser CA, Krieger M, Bretscher A, Ploegh H, Amon A, Martin KC (2016). Molecular Cell Biology (8th ed.). New York: W. H. Freeman and Company. p. 203. ISBN 978-1-4641-8339-3.
- ^ Hu H, Gatti RA (June 2011). "MicroRNAs: new players in the DNA damage response". Journal of Molecular Cell Biology. 3 (3): 151–158. doi:10.1093/jmcb/mjq042. PMC 3104011. PMID 21183529.
- ^ Jasperson KW, Tuohy TM, Neklason DW, Burt RW (June 2010). "Hereditary and familial colon cancer". Gastroenterology. 138 (6): 2044–58. doi:10.1053/j.gastro.2010.01.054. PMC 3057468. PMID 20420945.
- ^ Truninger K, Menigatti M, Luz J, Russell A, Haider R, Gebbers JO, Bannwart F, Yurtsever H, Neuweiler J, Riehle HM, Cattaruzza MS, Heinimann K, Schär P, Jiricny J, Marra G (May 2005). "Immunohistochemical analysis reveals high frequency of PMS2 defects in colorectal cancer". Gastroenterology. 128 (5): 1160–71. doi:10.1053/j.gastro.2005.01.056. PMID 15887099.
- ^ Valeri N, Gasparini P, Fabbri M, Braconi C, Veronese A, Lovat F, Adair B, Vannini I, Fanini F, Bottoni A, Costinean S, Sandhu SK, Nuovo GJ, Alder H, Gafa R, Calore F, Ferracin M, Lanza G, Volinia S, Negrini M, McIlhatton MA, Amadori D, Fishel R, Croce CM (April 2010). "Modulation of mismatch repair and genomic stability by miR-155". Proceedings of the National Academy of Sciences of the United States of America. 107 (15): 6982–7. Bibcode:2010PNAS..107.6982V. doi:10.1073/pnas.1002472107. PMC 2872463. PMID 20351277.
- ^ a b c Zhang W, Zhang J, Hoadley K, Kushwaha D, Ramakrishnan V, Li S, Kang C, You Y, Jiang C, Song SW, Jiang T, Chen CC (June 2012). "miR-181d: a predictive glioblastoma biomarker that downregulates MGMT expression". Neuro-Oncology. 14 (6): 712–9. doi:10.1093/neuonc/nos089. PMC 3367855. PMID 22570426.
- ^ Spiegl-Kreinecker S, Pirker C, Filipits M, Lötsch D, Buchroithner J, Pichler J, Silye R, Weis S, Micksche M, Fischer J, Berger W (January 2010). "O6-Methylguanine DNA methyltransferase protein expression in tumor cells predicts outcome of temozolomide therapy in glioblastoma patients". Neuro-Oncology. 12 (1): 28–36. doi:10.1093/neuonc/nop003. PMC 2940563. PMID 20150365.
- ^ Sgarra R, Rustighi A, Tessari MA, Di Bernardo J, Altamura S, Fusco A, Manfioletti G, Giancotti V (September 2004). "Nuclear phosphoproteins HMGA and their relationship with chromatin structure and cancer". FEBS Letters. 574 (1–3): 1–8. doi:10.1016/j.febslet.2004.08.013. PMID 15358530. S2CID 28903539.
- ^ Xu Y, Sumter TF, Bhattacharya R, Tesfaye A, Fuchs EJ, Wood LJ, Huso DL, Resar LM (May 2004). "The HMG-I oncogene causes highly penetrant, aggressive lymphoid malignancy in transgenic mice and is overexpressed in human leukemia". Cancer Research. 64 (10): 3371–5. doi:10.1158/0008-5472.CAN-04-0044. PMID 15150086.
- ^ Borrmann L, Schwanbeck R, Heyduk T, Seebeck B, Rogalla P, Bullerdiek J, Wisniewski JR (December 2003). "High mobility group A2 protein and its derivatives bind a specific region of the promoter of DNA repair gene ERCC1 and modulate its activity". Nucleic Acids Research. 31 (23): 6841–51. doi:10.1093/nar/gkg884. PMC 290254. PMID 14627817.
- ^ Facista A, Nguyen H, Lewis C, Prasad AR, Ramsey L, Zaitlin B, Nfonsam V, Krouse RS, Bernstein H, Payne CM, Stern S, Oatman N, Banerjee B, Bernstein C (April 2012). "Deficient expression of DNA repair enzymes in early progression to sporadic colon cancer". Genome Integrity. 3 (1): 3. doi:10.1186/2041-9414-3-3. PMC 3351028. PMID 22494821.
- ^ Gibert B, Delloye-Bourgeois C, Gattolliat CH, Meurette O, Le Guernevel S, Fombonne J, et al. (November 2014). "Regulation by miR181 family of the dependence receptor CDON tumor suppressive activity in neuroblastoma". Journal of the National Cancer Institute. 106 (11). doi:10.1093/jnci/dju318. PMID 25313246.
- ^ Chen JF, Murchison EP, Tang R, Callis TE, Tatsuguchi M, Deng Z, Rojas M, Hammond SM, Schneider MD, Selzman CH, Meissner G, Patterson C, Hannon GJ, Wang DZ (February 2008). "Targeted deletion of Dicer in the heart leads to dilated cardiomyopathy and heart failure". Proceedings of the National Academy of Sciences of the United States of America. 105 (6): 2111–6. Bibcode:2008PNAS..105.2111C. doi:10.1073/pnas.0710228105. PMC 2542870. PMID 18256189.
- ^ a b Zhao Y, Ransom JF, Li A, Vedantham V, von Drehle M, Muth AN, Tsuchihashi T, McManus MT, Schwartz RJ, Srivastava D (April 2007). "Dysregulation of cardiogenesis, cardiac conduction, and cell cycle in mice lacking miRNA-1-2". Cell. 129 (2): 303–17. doi:10.1016/j.cell.2007.03.030. PMID 17397913.
- ^ Thum T, Galuppo P, Wolf C, Fiedler J, Kneitz S, van Laake LW, Doevendans PA, Mummery CL, Borlak J, Haverich A, Gross C, Engelhardt S, Ertl G, Bauersachs J (July 2007). "MicroRNAs in the human heart: a clue to fetal gene reprogramming in heart failure". Circulation. 116 (3): 258–67. doi:10.1161/CIRCULATIONAHA.107.687947. PMID 17606841.
- ^ van Rooij E, Sutherland LB, Liu N, Williams AH, McAnally J, Gerard RD, Richardson JA, Olson EN (November 2006). "A signature pattern of stress-responsive microRNAs that can evoke cardiac hypertrophy and heart failure". Proceedings of the National Academy of Sciences of the United States of America. 103 (48): 18255–60. Bibcode:2006PNAS..10318255V. doi:10.1073/pnas.0608791103. PMC 1838739. PMID 17108080.
- ^ Tatsuguchi M, Seok HY, Callis TE, Thomson JM, Chen JF, Newman M, Rojas M, Hammond SM, Wang DZ (June 2007). "Expression of microRNAs is dynamically regulated during cardiomyocyte hypertrophy". Journal of Molecular and Cellular Cardiology. 42 (6): 1137–41. doi:10.1016/j.yjmcc.2007.04.004. PMC 1934409. PMID 17498736.
- ^ Zhao Y, Samal E, Srivastava D (July 2005). "Serum response factor regulates a muscle-specific microRNA that targets Hand2 during cardiogenesis". Nature. 436 (7048): 214–20. Bibcode:2005Natur.436..214Z. doi:10.1038/nature03817. PMID 15951802. S2CID 4340449.
- ^ Xiao J, Luo X, Lin H, Zhang Y, Lu Y, Wang N, Zhang Y, Yang B, Wang Z (April 2007). "MicroRNA miR-133 represses HERG K+ channel expression contributing to QT prolongation in diabetic hearts". The Journal of Biological Chemistry. 282 (17): 12363–7. doi:10.1074/jbc.C700015200. PMID 17344217.
- ^ Yang B, Lin H, Xiao J, Lu Y, Luo X, Li B, Zhang Y, Xu C, Bai Y, Wang H, Chen G, Wang Z (April 2007). "The muscle-specific microRNA miR-1 regulates cardiac arrhythmogenic potential by targeting GJA1 and KCNJ2". Nature Medicine. 13 (4): 486–91. doi:10.1038/nm1569. PMID 17401374. S2CID 1935811.
- ^ Carè A, Catalucci D, Felicetti F, Bonci D, Addario A, Gallo P, Bang ML, Segnalini P, Gu Y, Dalton ND, Elia L, Latronico MV, Høydal M, Autore C, Russo MA, Dorn GW, Ellingsen O, Ruiz-Lozano P, Peterson KL, Croce CM, Peschle C, Condorelli G (May 2007). "MicroRNA-133 controls cardiac hypertrophy". Nature Medicine. 13 (5): 613–8. doi:10.1038/nm1582. PMID 17468766. S2CID 10097893.
- ^ van Rooij E, Sutherland LB, Qi X, Richardson JA, Hill J, Olson EN (April 2007). "Control of stress-dependent cardiac growth and gene expression by a microRNA". Science. 316 (5824): 575–9. Bibcode:2007Sci...316..575V. doi:10.1126/science.1139089. PMID 17379774. S2CID 1927839.
- ^ Keller T, Boeckel JN, Groß S, Klotsche J, Palapies L, Leistner D, et al. (July 2017). "Improved risk stratification in prevention by use of a panel of selected circulating microRNAs". Scientific Reports. 7 (1): 4511. Bibcode:2017NatSR...7.4511K. doi:10.1038/s41598-017-04040-w. PMC 5495799. PMID 28674420.
- ^ Insull W (January 2009). "The pathology of atherosclerosis: plaque development and plaque responses to medical treatment". The American Journal of Medicine. 122 (1 Suppl): S3–S14. doi:10.1016/j.amjmed.2008.10.013. PMID 19110086.
- ^ a b c d e f g h i j k l m n o p q Son DJ, Kumar S, Takabe W, Kim CW, Ni CW, Alberts-Grill N, Jang IH, Kim S, Kim W, Won Kang S, Baker AH, Woong Seo J, Ferrara KW, Jo H (2013). "The atypical mechanosensitive microRNA-712 derived from pre-ribosomal RNA induces endothelial inflammation and atherosclerosis". Nature Communications. 4: 3000. Bibcode:2013NatCo...4.3000S. doi:10.1038/ncomms4000. PMC 3923891. PMID 24346612.
- ^ a b Basu R, Fan D, Kandalam V, Lee J, Das SK, Wang X, Baldwin TA, Oudit GY, Kassiri Z (December 2012). "Loss of Timp3 gene leads to abdominal aortic aneurysm formation in response to angiotensin II". The Journal of Biological Chemistry. 287 (53): 44083–96. doi:10.1074/jbc.M112.425652. PMC 3531724. PMID 23144462.
- ^ Libby P (2002). "Inflammation in atherosclerosis". Nature. 420 (6917): 868–74. Bibcode:2002Natur.420..868L. doi:10.1038/nature01323. PMID 12490960. S2CID 407449.
- ^ a b Phua YL, Chu JY, Marrone AK, Bodnar AJ, Sims-Lucas S, Ho J (October 2015). "Renal stromal miRNAs are required for normal nephrogenesis and glomerular mesangial survival". Physiological Reports. 3 (10): e12537. doi:10.14814/phy2.12537. PMC 4632944. PMID 26438731.
- ^ Maes OC, Chertkow HM, Wang E, Schipper HM (May 2009). "MicroRNA: Implications for Alzheimer Disease and other Human CNS Disorders". Current Genomics. 10 (3): 154–68. doi:10.2174/138920209788185252. PMC 2705849. PMID 19881909.
- ^ Amin ND, Bai G, Klug JR, Bonanomi D, Pankratz MT, Gifford WD, Hinckley CA, Sternfeld MJ, Driscoll SP, Dominguez B, Lee KF, Jin X, Pfaff SL (December 2015). "Loss of motoneuron-specific microRNA-218 causes systemic neuromuscular failure". Science. 350 (6267): 1525–9. Bibcode:2015Sci...350.1525A. doi:10.1126/science.aad2509. PMC 4913787. PMID 26680198.
- ^ Schratt G (December 2009). "microRNAs at the synapse". Nature Reviews. Neuroscience. 10 (12): 842–9. doi:10.1038/nrn2763. PMID 19888283. S2CID 3507952.
- ^ Cuellar TL, Davis TH, Nelson PT, Loeb GB, Harfe BD, Ullian E, McManus MT (April 2008). "Dicer loss in striatal neurons produces behavioral and neuroanatomical phenotypes in the absence of neurodegeneration". Proceedings of the National Academy of Sciences. 105 (14): 5614–19. Bibcode:2008PNAS..105.5614C. doi:10.1073/pnas.0801689105. PMC 2291142. PMID 18385371.
- ^ Hébert SS, Papadopoulou AS, Smith P, Galas M, Planel E, Silahtaroglu AN, Sergeant N, Buée L, De Strooper B (October 2010). "Genetic ablation of Dicer in adult forebrain neurons results in abnormal tau hyperphosphorylation and neurodegeneration". Human Molecular Genetics. 19 (20): 3959–69. doi:10.1093/hmg/ddq311. PMID 20660113.
- ^ Hosseinian S, Arefian A, Rakhsh-Khorshid H (2020). "A meta-analysis of gene expression data highlights synaptic dysfunction in the hippocampus of brains with Alzheimer's disease". Scientific Reports. 10 (1): 8384. Bibcode:2020NatSR..10.8384H. doi:10.1038/s41598-020-64452-z. PMC 7239885. PMID 32433480.
- ^ Hommers LG, Domschke K, Deckert J (January 2015). "Heterogeneity and individuality: microRNAs in mental disorders". Journal of Neural Transmission. 122 (1): 79–97. doi:10.1007/s00702-014-1338-4. PMID 25395183. S2CID 25088900.
- ^ Feng J, Sun G, Yan J, Noltner K, Li W, Buzin CH, Longmate J, Heston LL, Rossi J, Sommer SS (July 2009). Reif A (ed.). "Evidence for X-chromosomal schizophrenia associated with microRNA alterations". PLOS ONE. 4 (7): e6121. Bibcode:2009PLoSO...4.6121F. doi:10.1371/journal.pone.0006121. PMC 2699475. PMID 19568434.
- ^ Beveridge NJ, Gardiner E, Carroll AP, Tooney PA, Cairns MJ (December 2010). "Schizophrenia is associated with an increase in cortical microRNA biogenesis". Molecular Psychiatry. 15 (12): 1176–89. doi:10.1038/mp.2009.84. PMC 2990188. PMID 19721432.
- ^ "Stroke Facts cdc.gov". www.cdc.gov. 15 March 2019. Retrieved 5 December 2019.
- ^ Rink C, Khanna S (May 2011). "MicroRNA in ischemic stroke etiology and pathology". Physiological Genomics. 43 (10): 521–528. doi:10.1152/physiolgenomics.00158.2010. PMC 3110894. PMID 20841499.
- ^ Ouyang YB, Stary CM, Yang GY, Giffard R (January 2013). "microRNAs: innovative targets for cerebral ischemia and stroke". Current Drug Targets. 14 (1): 90–101. doi:10.2174/138945013804806424. PMC 3673881. PMID 23170800.
- ^ a b c Lewohl JM, Nunez YO, Dodd PR, Tiwari GR, Harris RA, Mayfield RD (November 2011). "Up-regulation of microRNAs in brain of human alcoholics". Alcoholism, Clinical and Experimental Research. 35 (11): 1928–37. doi:10.1111/j.1530-0277.2011.01544.x. PMC 3170679. PMID 21651580.
- ^ Tapocik JD, Solomon M, Flanigan M, Meinhardt M, Barbier E, Schank JR, Schwandt M, Sommer WH, Heilig M (June 2013). "Coordinated dysregulation of mRNAs and microRNAs in the rat medial prefrontal cortex following a history of alcohol dependence". The Pharmacogenomics Journal. 13 (3): 286–96. doi:10.1038/tpj.2012.17. PMC 3546132. PMID 22614244.
- ^ Gorini G, Nunez YO, Mayfield RD (2013). "Integration of miRNA and protein profiling reveals coordinated neuroadaptations in the alcohol-dependent mouse brain". PLOS ONE. 8 (12): e82565. Bibcode:2013PLoSO...882565G. doi:10.1371/journal.pone.0082565. PMC 3865091. PMID 24358208.
- ^ Tapocik JD, Barbier E, Flanigan M, Solomon M, Pincus A, Pilling A, Sun H, Schank JR, King C, Heilig M (March 2014). "microRNA-206 in rat medial prefrontal cortex regulates BDNF expression and alcohol drinking". The Journal of Neuroscience. 34 (13): 4581–8. doi:10.1523/JNEUROSCI.0445-14.2014. PMC 3965783. PMID 24672003.
- ^ Lippai D, Bala S, Csak T, Kurt-Jones EA, Szabo G (2013). "Chronic alcohol-induced microRNA-155 contributes to neuroinflammation in a TLR4-dependent manner in mice". PLOS ONE. 8 (8): e70945. Bibcode:2013PLoSO...870945L. doi:10.1371/journal.pone.0070945. PMC 3739772. PMID 23951048.
- ^ a b Li J, Li J, Liu X, Qin S, Guan Y, Liu Y, Cheng Y, Chen X, Li W, Wang S, Xiong M, Kuzhikandathil EV, Ye JH, Zhang C (September 2013). "MicroRNA expression profile and functional analysis reveal that miR-382 is a critical novel gene of alcohol addiction". EMBO Molecular Medicine. 5 (9): 1402–14. doi:10.1002/emmm.201201900. PMC 3799494. PMID 23873704.
- ^ Romao JM, Jin W, Dodson MV, Hausman GJ, Moore SS, Guan LL (September 2011). "MicroRNA regulation in mammalian adipogenesis". Experimental Biology and Medicine. 236 (9): 997–1004. doi:10.1258/ebm.2011.011101. PMID 21844119. S2CID 30646787.
- ^ Skårn M, Namløs HM, Noordhuis P, Wang MY, Meza-Zepeda LA, Myklebost O (April 2012). "Adipocyte differentiation of human bone marrow-derived stromal cells is modulated by microRNA-155, microRNA-221, and microRNA-222". Stem Cells and Development. 21 (6): 873–83. doi:10.1089/scd.2010.0503. hdl:10852/40423. PMID 21756067.
- ^ Zuo Y, Qiang L, Farmer SR (March 2006). "Activation of CCAAT/enhancer-binding protein (C/EBP) alpha expression by C/EBP beta during adipogenesis requires a peroxisome proliferator-activated receptor-gamma-associated repression of HDAC1 at the C/ebp alpha gene promoter". The Journal of Biological Chemistry. 281 (12): 7960–7. doi:10.1074/jbc.M510682200. PMID 16431920.
- ^ Jun-Hao ET, Gupta RR, Shyh-Chang N (March 2016). "Lin28 and let-7 in the Metabolic Physiology of Aging". Trends in Endocrinology and Metabolism. 27 (3): 132–141. doi:10.1016/j.tem.2015.12.006. PMID 26811207. S2CID 3614126.
- ^ Zhu H, Shyh-Chang N, Segrè AV, Shinoda G, Shah SP, Einhorn WS, Takeuchi A, Engreitz JM, Hagan JP, Kharas MG, Urbach A, Thornton JE, Triboulet R, Gregory RI, Altshuler D, Daley GQ (September 2011). "The Lin28/let-7 axis regulates glucose metabolism". Cell. 147 (1): 81–94. doi:10.1016/j.cell.2011.08.033. PMC 3353524. PMID 21962509.
- ^ Frost RJ, Olson EN (December 2011). "Control of glucose homeostasis and insulin sensitivity by the Let-7 family of microRNAs". Proceedings of the National Academy of Sciences of the United States of America. 108 (52): 21075–80. Bibcode:2011PNAS..10821075F. doi:10.1073/pnas.1118922109. PMC 3248488. PMID 22160727.
- ^ Teruel-Montoya R, Rosendaal FR, Martínez C (February 2015). "MicroRNAs in hemostasis". J Thromb Haemost. 13 (2): 170–181. doi:10.1111/jth.12788. PMID 25400249.
- ^ a b Nourse J, Braun J, Lackner K, Hüttelmaier S, Danckwardt S (September 2018). "Large-scale identification of functional microRNA targeting reveals cooperative regulation of the hemostatic system". J Thromb Haemost. 16 (11): 2233–2245. doi:10.1111/jth.14290. PMID 30207063.
- ^ Nourse J, Danckwardt S (September 2020). "A novel rationale for targeting FXI: Insights from the hemostatic microRNA targetome for emerging anticoagulant strategies". Pharmacol Ther. 218: 107676. doi:10.1016/j.pharmthera.2020.107676. PMID 32898547.
- ^ Pheasant M, Mattick JS (September 2007). "Raising the estimate of functional human sequences". Genome Research. 17 (9): 1245–53. doi:10.1101/gr.6406307. PMID 17690206.
- ^ Bertone P, Stolc V, Royce TE, Rozowsky JS, Urban AE, Zhu X, Rinn JL, Tongprasit W, Samanta M, Weissman S, Gerstein M, Snyder M (December 2004). "Global identification of human transcribed sequences with genome tiling arrays". Science. 306 (5705): 2242–6. Bibcode:2004Sci...306.2242B. doi:10.1126/science.1103388. PMID 15539566. S2CID 396518.
- ^ Ota T, Suzuki Y, Nishikawa T, Otsuki T, Sugiyama T, Irie R, et al. (January 2004). "Complete sequencing and characterization of 21,243 full-length human cDNAs". Nature Genetics. 36 (1): 40–5. doi:10.1038/ng1285. PMID 14702039.
- ^ Kuhn DE, Martin MM, Feldman DS, Terry AV, Nuovo GJ, Elton TS (January 2008). "Experimental validation of miRNA targets". Methods. 44 (1): 47–54. doi:10.1016/j.ymeth.2007.09.005. PMC 2237914. PMID 18158132.
- ^ Hüttenhofer A, Schattner P, Polacek N (May 2005). "Non-coding RNAs: hope or hype?". Trends in Genetics. 21 (5): 289–97. doi:10.1016/j.tig.2005.03.007. PMID 15851066.
- ^ Qureshi A, Thakur N, Monga I, Thakur A, Kumar M (1 January 2014). "VIRmiRNA: a comprehensive resource for experimentally validated viral miRNAs and their targets". Database. 2014: bau103. doi:10.1093/database/bau103. PMC 4224276. PMID 25380780.
- ^ Kumar M. "VIRmiRNA". Resource for experimental viral miRNA and their targets. Bioinformatics center, CSIR-IMTECH.
- ^ Tuddenham L, Jung JS, Chane-Woon-Ming B, Dölken L, Pfeffer S (February 2012). "Small RNA deep sequencing identifies microRNAs and other small noncoding RNAs from human herpesvirus 6B". Journal of Virology. 86 (3): 1638–49. doi:10.1128/JVI.05911-11. PMC 3264354. PMID 22114334.
- ^ Zheng H, Fu R, Wang JT, Liu Q, Chen H, Jiang SW (April 2013). "Advances in the techniques for the prediction of microRNA targets". International Journal of Molecular Sciences. 14 (4): 8179–87. doi:10.3390/ijms14048179. PMC 3645737. PMID 23591837.
- ^ Agarwal V, Bell GW, Nam JW, Bartel DP (August 2015). "Predicting effective microRNA target sites in mammalian mRNAs". eLife. 4: e05005. doi:10.7554/eLife.05005. PMC 4532895. PMID 26267216.
추가 정보
- miRNA 정의 및 분류:
- 소형 RNA의 과학 리뷰:
- 최초로 발견된 miRNA인 lin-4의 검출:
외부 링크
- miRBase 데이터베이스
- miRTARBase, 실험적으로 검증된 마이크로RNA 타깃 상호작용 데이터베이스.
- semirna, 식물 게놈에서 마이크로RNA를 검색하기 위한 웹 애플리케이션입니다.
- ONCO.IO: 암의 마이크로RNA 및 전사 인자 분석을 위한 통합 자원.
- MirOB: MicroRNA는 데이터베이스 및 데이터 분석 및 데이터비즈 도구를 대상으로 합니다.
- ChIPBase 데이터베이스:ChIP-seq 데이터로부터의 마이크로RNA 전사에 관여하거나 영향을 준 전사인자를 디코딩하기 위한 오픈액세스 데이터베이스
- 마이크로RNA 생물 생성 과정을 애니메이션화한 영상입니다.
- 내인성 성숙 마이크로RNA 기능의 상향 조절 또는 억제를 가능하게 하는 miRNA 변조 시약